LSm
LSm이 글은 대부분의 독자들이 이해하기에는 너무 기술적인 것일 수도 있다. 정보를 할 수 하십시오.(2013년 8월)(이 및 |
| LSM 도메인 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 p. heavssi의 sm 관련 단백질의 결정 구조 생물학적 단위는 헵타머다. | |||||||||
| 식별자 | |||||||||
| 기호 | LSM | ||||||||
| Pfam | PF01423 | ||||||||
| 인터프로 | IPR001163 | ||||||||
| SCOP2 | 1d3b / SCOPe / SUPFAM | ||||||||
| CDD | cd00600 | ||||||||
| |||||||||
분자생물학에서 LSM 단백질은 거의 모든 세포 유기체에서 발견되는 RNA 결합 단백질이다.LSm은 'Sm처럼'의 수축인데, LSm 단백질 계열의 첫 번째 구성원이 sm 단백질이었기 때문이다.LSm 단백질은 특징적인 3차원 구조와 그 조립에 의해 6개 또는 7개의 개별 LSm 단백질 분자의 고리로 정의되며, mRNA 처리와 조절에서 많은 다양한 역할을 한다.null
sm 단백질은 쇠약해지는 자가면역질환인 전신 루푸스 에리테마토스(SLE)의 형태를 가진 환자에게서 이른바 항Sm 항체가 표적이 된 항원으로 처음 발견됐다.이들은 SLE를 앓은 환자 스테파니 스미스를 기리기 위해 sm 단백질로 명명됐다.[1]이후 매우 유사한 구조를 가진 다른 단백질들이 발견되었고 LSm 단백질이라고 명명되었다.LSm 단백질 계열의 새로운 구성원이 계속 확인되고 보고되고 있다.null
구조가 비슷한 단백질은 단백질 계열, 슈퍼 패밀리와 접히는 계층으로 분류된다.LSm 단백질 구조는 작은 베타 시트를 짧은 배럴로 접은 예다.개별 LSm 단백질은 6, 7개의 멤버 도넛 링(더 적절하게 torus라고 불림)으로 조립되는데, 보통 작은 RNA 분자에 결합해 리보핵단백질 복합체를 형성한다.LSm torus는 RNA 분자가 적절한 3차원 구조를 가정하고 유지하도록 돕는다.이 리보뉴클레오프로틴 복합체는 어떤 LSm 단백질과 RNA 분자가 관여하는가에 따라 분해, 편집, 스플라이싱, 조절 등 다양한 RNA 처리를 용이하게 한다.null
LSm 계열의 대체 용어는 LSm 폴드와 SM 유사 폴드이며, LSM, LSM과 같은 대체 대문자화 스타일이 일반적이며 동등하게 허용된다.null
역사
스미스 항원 발견
최초의 LSM 단백질이 발견되었다는 이야기는 1959년 전신 루푸스 에리테마토스(SLE) 진단을 받은 젊은 여성 스테파니 스미스가 결국 22세의 나이로 1969년 질병의 합병증에 굴복하면서 시작된다.[1]이 기간 동안 그녀는 뉴욕 록펠러 대학 병원에서 박사의 보살핌을 받으며 치료를 받았다.헨리 쿤켈과 엥 탄 박사.SLE 환자는 자가면역질환을 앓고 있는 환자로서 세포핵에서 항원에 대한 항체를 생성하는데, 가장 빈번하게 자신의 DNA에 대한 항체를 생성한다.하지만, 쿤켈 박사와 닥터.탄은 1966년 스미스 씨가 핵 단백질 세트에 항체를 생산한 것을 발견했는데, 그들은 이를 '스미스 항원'(Smith antig)이라고 이름 붙였다.[2]SLE 환자의 약 30%는 이중으로 좌초된 DNA와는 반대로 이러한 단백질에 대한 항체를 생성한다.이 발견으로 SLE에 대한 진단 테스트가 개선되었지만, 이 항원의 성격과 기능은 알 수 없었다.null
SM 단백질, snRNP, 스플라이소솜 및 메신저 RNA 스플라이싱
연구는 1970년대와 1980년대 초반에 계속되었다.스미스 항원은 리보핵산(RNA) 분자와 여러 단백질의 복합체인 것으로 밝혀졌다.우리딘이 풍부한 소형 핵 RNA(snRNA) 분자 세트는 이 복합체의 일부였으며, U1, U2, U4, U5, U6라는 이름을 붙였다.이들 snRNA(U1, U2, U4, U5) 중 4개(U1, U2, U5)는 크기가 줄어드는 순서로 smB, smD, smE, smF, smG라고 이름 붙여진 여러 개의 작은 단백질과 밀접하게 결합되어 있는 것으로 밝혀졌다.smB는 대체적으로 분열된 변종인 smB'를 가지고 있으며, 매우 유사한 단백질인 smN은 특정 (대부분 신경) 조직에서 smB'/B를 대체한다.smD는 나중에 smD1, smD2, smD3라는 이름의 세 개의 단백질이 혼합된 것으로 밝혀졌다.이 9가지 단백질(SmB, smB'), smN, smD1, smD2, smD3, smE, smF, smG)은 sm핵단백질, 또는 단순히 sm 단백질로 알려지게 되었다.snRNA는 sm핵심 단백질과 다른 단백질들로 복잡하게 이루어져 작은 핵 리보핵단백질 또는 snRNP라고 불리는 세포 핵에 입자를 형성한다.1980년대 중반까지, 이러한 snRNPs는 인트론이라고 불리는 mRNA 이전의 부분을 배설하고 코딩 부분(exons)을 함께 스플라이소솜이라고 불리는 큰 (4.8 MD 분자량) 콤플렉스를 형성하는데 도움을 준다는 것이 명백해졌다.[3]몇 번 더 수정한 후에 분열된 프리 mRNA는 메신저 RNA(mRNA)가 되고, 그 다음 핵에서 수출되어 리보솜에 의해 단백질로 변환된다.null
sm 단백질과 유사한 단백질 발견
snRNA U6(U1, U2, U4, U5)는 U6 snRNP가 스플라이소솜의 중심 성분임에도 불구하고 sm 단백질과 연관되지 않는다.1999년에 특별히 U6에 결합하는 단백질 이성질체가 발견되었고, sm 단백질에 명백히 동질성이 있는 7개의 단백질로 구성되었다.이러한 단백질은 LSm(Sm과 유사) 단백질(LSm1, LSm2, LSm3, LSm4, LSm5, LSm6, LSm7)로 표시되었으며, 유사한 LSm8 단백질이 나중에 식별되었다.대장균 박테리아에서 유전자 hfq에 의해 인코딩된 sm 유사 단백질 HF-I는 1968년 RNA 박테리오파지 Qβ 복제를 위한 필수 숙주 요인으로 설명되었다.사카로마이오스 세레비시아아(Baker's Eastiae)의 게놈은 1990년대 중반에 서열화되어 이러한 인간 단백질의 동질성을 식별하는 풍부한 자원을 제공하였다.그 후, eukaryotes 게놈의 염기서열이 더 많아짐에 따라, 일반적으로 eukaryotes는 7Sm과 8 LSm 단백질의 동일한 집합에 동음이의어를 공유한다는 것이 명백해졌다.[4]곧이어 이 진핵생물 LSm 단백질에 동질인 단백질이 고고아(Sm1과 sm2)와 박테리아(Hfq와 YlxS 호몰로그램)에서 발견됐다.[5]고고학적 LSm 단백질은 박테리아 LSm 단백질보다 eukaryote LSm 단백질과 더 유사하다.지금까지 설명한 LSm 단백질은 인간 SmG의 경우 76개의 아미노산(8.7kD 분자량)에서 인간 SmB의 경우 231개의 아미노산(29kD 분자량)에 이르기까지 다소 작은 단백질이었다.그러나 최근에는 다른 단백질 구조 영역(LSm10, LSm11, LSm12, LSm13, LSm14, LSm15, LSm16, 아탁신-2 등) 외에 LSm 구조 영역을 포함하는 더 큰 단백질이 발견되었다.null
LSm 폴드 발견
1995년경, 다양한 LSm 호몰로겐들 간의 비교에서 각 LSm 호몰로겐에서 매우 유사한 길이 32개(아미노산 14개)의 두 시퀀스 모티브가 확인되었고, 가변 길이의 비보존 영역으로 분리되었다.이는 이 두 시퀀스 모티브(Sm1과 sm2)의 중요성을 나타냈으며, 모든 LSm 단백질 유전자가 단일 조상의 유전자에서 진화했음을 시사했다.[6]1999년에는 재조합형 SM 단백질의 결정체가 준비되어 X선 결정술과 3차원의 원자 구조를 결정할 수 있었다.[7]이것은 LSm 단백질이 짧은 알파 나선과 5줄 접힌 베타 시트의 유사한 3차원 접힘과 LSm 접힘이라는 이름을 공유한다는 것을 보여주었다.다른 조사 결과 LSm 단백질은 6~7개의 LSm 단백질의 토러스(도넛 모양의 고리)로 조립되며 RNA는 각 LSm 단백질에 1개의 뉴클레오티드가 결합돼 토러스 내부에 결합하는 것으로 나타났다.null
구조
우리딘 인산염은 고고학적 Sm1에서 β2b/β3a 루프와 β4b/β5 루프 사이에 결합된다.우라실은 히스티딘과 아르기닌 잔여물 사이에 쌓이고 아스파라긴 잔여물에 대한 수소 결합과 아스파라긴 잔여물과 리보스 사이의 수소 결합에 의해 안정화된다.LSm 단백질은 베타 시트(이차 구조)가 특징이며, LSm 접이식(제3차 구조)으로 접이식(제3차 구조), 중합성(제6 또는 제7의 멤버 토루스(제2차 구조), RNA 올리고뉴클레오티드에 결합한다.[8]현대 패러다임은 단백질 구조에 기초하여 단백질을 분류하고 현재 활동 중인 분야로, SCOP(단백질 구조분류), Cath(클래스, 아키텍처, 위상, 호몰로컬 슈퍼패밀리), FSSP/DALI(구조적으로 유사한 단백질의 가족)의 3대 접근법이 있다.null
이차적
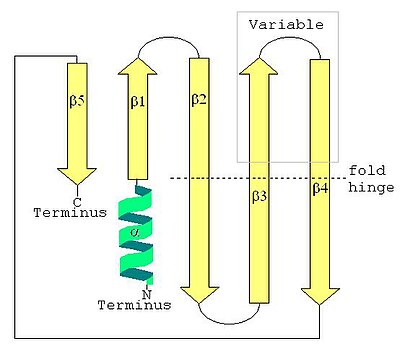
LSm 단백질의 2차 구조는 작은 5-스트랜드 반병렬 베타 시트로서, N-단자 끝에서 C-단자 끝까지의 가닥은 β1, β2, β3, β4, β5로 식별된다.올 베타 단백질의 SCOP 등급과 주로 베타의 CAT 등급은 주로 베타 시트인 단백질 구조로 정의되며, 따라서 LSm을 포함한다.SM1 시퀀스 모티브는 β1, β2, β3 Strand에 해당하며, SM2 시퀀스 모티브는 β4 및 β5 Strand에 해당한다.처음 네 개의 베타 가닥은 서로 인접해 있지만 β5는 β1에 인접해 있어 전체 구조가 짧은 배럴로 바뀐다.이 구조 위상은 51234로 설명된다.대부분의 LSm 단백질에도 짧은 (2~4회전) N단자 알파나선이 존재한다.β3와 β4 가닥은 일부 LSm 단백질에서 짧고 가변 길이의 비정형 코일에 의해 분리된다.β2, β3 및 β4 가닥은 중간점에서 약 120° 정도로 강하게 휘어져 있다 이들 가닥의 굴곡은 흔히 글리신이며, 베타 배럴 내부의 사이드 체인은 흔히 소수성 잔류물 발레린, 류신, 이솔레우신, 메티오닌이다.null
제3차
SCOP는 LSm 구조를 중간 그룹화 없이 149개의 베타 단백질 접힘 중 하나인 SM형 접힘으로 분류한다.LSm 베타 시트는 날카롭게 구부러져 있으며, CAT에서는 롤 아키텍처(CAT에서는 20개의 서로 다른 베타 단백질 아키텍처 중 하나)로 설명된다.베타 가닥 중 하나(LSM의 β5)가 롤의 열린 가장자리를 가로지르며 작은 SH3형 배럴 위상(CAT의 33개 베타 롤 위상 중 하나)을 형성한다(CAT의 33개 베타 롤 위상 중 하나). Cath에는 SH3형 배럴 위상(CAT 시스템의 RNA 바인딩 단백질)을 가진 23개의 호몰로 구성된 슈퍼 패밀리가 나열된다.SCOP는 Fold to Superfamily, Family, Domain에 이어 구조 분류를 계속하는 반면, Cath는 Sequence Family로 계속 진행되지만, 이러한 분할은 "진화 및 계통 발생" 섹션에 더 적절하게 설명되어 있다.null
LSm 폴드의 SH3형 배럴 3차 구조는 강하게 굽힘(약 120°)에 의해 형성된다.배럴 구조가 β5 스트랜드에 의해 닫힌 상태에서 β2, β3, β4 스트랜드.3차 구조를 강조하면서 각 구부러진 베타 가닥은 두 개의 짧은 베타 가닥으로 설명할 수 있다.LSm 접히는 8스트랜드 반병렬 베타 샌드위치로 볼 수 있는데, 한 평면에 5개의 가닥이 있고, 두 개의 베타 샌드위치의 절반 사이 피치 각도가 약 45°인 평행 평면에 3개의 가닥이 있다.짧은 (2~4회전) N단자 알파나선은 베타 샌드위치의 한쪽 가장자리에서 발생한다.This alpha helix and the beta strands can be labeled (from the N-terminus to the C-terminus) α, β1, β2a, β2b, β3a, β3b, β4a, β4b, β5 where the a and b refer to either the two halves of a bent strand in the five-strand description, or to the individual strands in the eight-strand description.각 스트랜드(8줄 설명)는 5개의 아미노산 잔류물로 형성된다.가닥과 알파나선 사이의 굴곡과 루프를 포함하면 약 60개의 아미노산 잔류물이 LSm 접힘에 기여하지만, 이는 띠간 루프, 알파나선, 심지어 β3b와 β4a 가닥의 길이 등의 변화로 인해 호몰로사이에 변화한다.null
쿼터너리
LSm 단백질은 일반적으로 2나노미터 구멍이 있는 직경 약 7나노미터의 LSm 링, 6, 7개의 멤버 토러스, 즉 직경 7나노미터로 조립된다.조상 조건은 동일한 LSm 하위 단위의 호모헥사머 또는 호모헵타머이다.eukaryotes의 LSm 단백질은 sm 단백질과 같은 7개의 다른 LSm 하위 단위의 헤테로헥타머를 형성한다.LSm 단백질 사이의 결합은 LSm 접힘에 대한 8-strand 설명으로 가장 잘 이해된다.The five-strand half of the beta sandwich of one subunit aligns with the three-strand half of the beta sandwich of the adjacent subunit, forming a twisted 8-strand beta sheet Aβ4a/Aβ3b/Aβ2a/Aβ1/Aβ5/Bβ4b/Bβ3a/Bβ2b, where the A and B refer to the two different subunits.두 LSm 단백질 서브유닛의 Aβ5와 Bβ4b 베타 가닥 사이에 수소 본딩 이외에도 접촉 영역 내부의 소수성 아미노산 사이드 체인 간에는 활력적으로 유리한 접점이 있으며, 접촉 영역 주변 주변의 친수성 아미노산 사이드 체인 간에는 정력적으로 유리한 접점이 있다.null
RNA 올리고뉴클레오티드 결합
LSm 링은 RNA 올리고뉴클레오티드로 리보뉴클레오프로테인 콤플렉스를 형성하며, 매우 안정적인 콤플렉스(Sm class snRNP 등)부터 과도 콤플렉스까지 결합 강도가 다양하다.RNA 올리고뉴클레오티드는 일반적으로 LSm 소단위당 1개의 뉴클레오티드인 LSm torus의 구멍(루멘) 내부에 결합하지만, 링의 상단(α 나선측)에는 추가적인 뉴클레오티드 결합 부위가 보고되었다.이 결합의 정확한 화학적 성질은 다양하지만 공통적인 모티브에는 두 아미노산의 평면측 체인 사이에 이질적 염기(종종 요라실), 이질적 염기 및/또는 리보스에 대한 수소 결합, 인산염 그룹에 대한 염기 교량 쌓기가 포함된다.null
기능들
다양한 종류의 LSm 링은 RNA 올리고뉴클레오티드의 비계 또는 샤페로네 역할을 하여 RNA가 적절한 3차원 구조를 가정하고 유지할 수 있도록 돕는다.어떤 경우에는, 이것은 올리고뉴클레오티드 RNA가 리보핵으로서 촉매적으로 기능할 수 있게 한다.다른 경우에는 RNA, 즉 리보핵단백질 복합체의 조립, 저장 및 세포내 이동을 용이하게 한다.[9]null
sm링
sm 링은 모든 eukaryotes의 핵(증식 인간 세포당 약 2.5 x 10복사6)에서 발견되며, 가장 잘 이해되는 기능을 가지고 있다.sm 반지는 이단 헵타머다.The Sm-class snRNA molecule (in the 5' to 3' direction) enters the lumen (doughnut hole) at the SmE subunit and proceeds sequentially in a clockwise fashion (looking from the α helix side) inside the lumen (doughnut hole) to the SmG, SmD3, SmB, SmD1, SmD2 subunits, exiting at the SmF subunit.[10](SmB는 신경 조직에서 스플라이스 변종 SmB'로 대체될 수 있다.)sm 링은 U1, U2, U4 및 U5 snRNA에 영구적으로 바인딩되며, 이는 주요 스니소솜을 구성하는 5개의 snRNP 중 4개를 형성한다.sm 링은 또한 작은 이음매를 구성하는 5개의 snRNP(U5 snRNP 포함) 중 4개를 형성하는 U11, U12, U4atac snRNA에 영구적으로 결합된다.이 두 스플라이소솜은 모두 전 mRNA에서 온 메신저 RNA 성숙기에 있는 중심 RNA 처리 복합체다.sm 단백질은 텔로머레이즈의 리보핵단백질 성분에도 포함된 것으로 보고되었다.[11]null
Lsm2-8 링
두 개의 Lsm2-8 snRNP(U6 및 U6atac)는 주 및 부 스플라이소솜에 주요 촉매 기능을 가지고 있다.이러한 snRNP는 sm 링을 포함하지 않고 헤테로 heethopticanic Lsm2-8 링을 사용한다.LSm 링은 sm 링보다 20배가량 풍부하지 않다.The order of these seven LSm proteins in this ring is not known, but based on amino acid sequence homology with the Sm proteins, it is speculated that the snRNA (in the 5' to 3' direction) may bind first to LSm5, and precedes sequentially clockwise to the LSm7, LSm4, LSm8, LSm2, LSm3, and exiting at the LSm6 subunit.Experiments with Saccharomyces cerevisiae (budding yeast) mutations suggest that the Lsm2-8 ring assists the reassociation of the U4 and U6 snRNPs into the U4/U6 di-snRNP.[12] (After completion of exon deletion and intron splicing, these two snRNPs must reassociate for the spliceosome to initiate another exon/intron splicing cycle.이 역할에서 Lsm2-8 링은 RNA 비계 대신 RNA 샤페론 역할을 한다.)Lsm2-8 링은 또한 U8 소핵 RNA(snoRNA)로 snRNP를 형성하는데, 이는 뉴클레오스에 국부화된다.이 리보핵단백질 복합체는 리보솜 RNA를 처리하고 RNA를 성숙한 형태로 전달하는데 필요하다.[13]Lsm2-8 링은 사전 P RNA를 RNase P RNA로 가공하는 역할을 하는 것으로 보고되고 있다.Sm 링과 달리 Lsm2-8 링은 snRNA와 snoRNA에 영구적으로 결합되지 않는다.
Sm10/Sm11 링
LSm10이 SmD1을 대체하고 LSm11이 SmD2를 대체하는 두 번째 유형의 Sm 링이 존재한다. LSm11은 C-단자 도메인이 LSm 도메인인 두 개의 도메인 단백질이다.이 헤테로헵타머 링은 U7 snRNP에서 U7 snRNA와 결합한다.U7 snRNP는 핵에 있는 히스톤 mRNA의 3' UTR 스템 루프 처리를 중재한다.[14]sm 링처럼 특수 SMN 콤플렉스에 의해 U7 snRNA에 세포질 속에 조립된다.null
Lsm1-7 링
LSM 링의 두 번째 유형은 LSM1-7 링으로 LSm1이 LSm8을 대체한다는 점을 제외하면 LSM2-8 링과 구조가 같다.Lsm2-8 링과는 대조적으로 Lsm1-7 링은 리보뉴클레오프로틴 복합체에서 메신저 RNA를 분해하는 데 도움을 주는 세포질에서 국소화된다.이 과정은 단백질에 대한 mRNA의 리보솜 번역이 세포에 의한 메신저 RNA로의 DNA 전사 변화에 빠르게 반응하도록 메신저 RNA의 이직률을 조절한다.null
제민6호와 제민7호
SMN 복합체("snRNPs의 생물 발생"에서 설명함)는 SMN 단백질과 제민2-8로 구성되어 있다.이 중 두 가지인 제민6호와 제민7호는 LSm 구조를 갖고 있으며, 이단계를 형성하고 있는 것으로 밝혀졌다.SM급 snRNA에 SM 링의 형성을 돕기 위해 SMN 콤플렉스에 샤퍼론 기능이 있을 수 있다.[15] PRMT5 콤플렉스는 PRMT5, pICln, WD45(Mep50)로 구성된다. pICln은 SMN 콤플렉스에 SM 오픈 링 형성을 돕는다.SMN 콤플렉스는 SMN 콤플렉스에서 SM 링이 공개 순응 상태에 있는 snRNP 조립을 보조하며, 이 sm 링은 SMN 콤플렉스에 의해 snRNA에 로딩된다.[16]null
LSm12-16 및 기타 다중 도메인 LSm 단백질
LSm12-16 단백질은 매우 최근에 설명되었다.이들은 N단자 LSm 도메인과 C단자 메틸전달효소 도메인을 가진 2개 영역 단백질이다.[17]이러한 단백질의 기능에 대해서는 거의 알려져 있지 않지만, 아마도 RNA와 상호작용하는 LSm-도메인 링의 일원일 것이다.LSm12가 mRNA 분해에 관여하고 LSm13-16이 유사분열 조절에 역할을 할 수 있다는 일부 증거가 있다.뜻밖에도 LSm12는 최근 두 번째 뉴클레오티드2+ 메신저인 NADP(Nicotinic acid [18]adenine dinucleotide phosphate)의 중간 결합단백질 역할을 하며 칼슘 신호에 관여했다.이는 NADP가 AD 도메인이 아닌 LSm 도메인에 바인딩함으로써 발생했다.[18]신경퇴행성 질환인 스피노세레벨라 아탁시아 타입 2와 연관된 알 수 없는 기능의 큰 단백질인 아탁신-2도 N단자 LSm 도메인을 가지고 있다.null
고고학적 sm 고리
두 개의 LSm 단백질은 생명의 두 번째 영역인 고세아에서 발견된다.이들은 같은 고고 단백질인 smAP1과 smAP2로 확인되기도 한다sm2 단백질(sm1과 sm2 시퀀스 모티브와 혼동해서는 안 된다)이며, 이러한 이유로 sm과 sm1과.[19]sm1과 sm2는 일반적으로 호모헵타메르 반지를 형성하지만 호모헥사메르 반지는 관찰되어 왔다.sm1 링은 RNA가 없을 때 형성된다는 점에서 eukaryote Lsm 링과 유사한 반면, sm2 링은 형성을 위해 우리딘이 풍부한 RNA가 필요하다는 점에서 eukaryote sm 링과 유사하다.그들은 RNase P RNA와 연관되어 전달 RNA 처리의 역할을 제안하는 것으로 보고되었지만, 이 과정에서 (그리고 리보솜 RNA와 같은 다른 RNA 처리) 고고학에서의 그들의 기능은 대부분 알려져 있지 않다.고고학의 두 가지 주요 가지 중 하나인, 세레나카에오테는 세 번째로 알려진 고고 LSm 단백질을 가지고 있다.이것은 호모헵타머 링을 형성하는 N단자 LSm 도메인을 가진 2개 영역 단백질이다.이 LSm 단백질의 기능에 대해 알려진 것은 없지만, 아마도 이 단백질은 이 유기체에서 RNA와 상호작용하고 아마도 처리를 도울 것이다.null
박테리아 LSM 링
몇몇 LSm 단백질이 생명의 세 번째 영역인 박테리아에서 보고되었다.Hfq 단백질은 호모헥사메르 고리를 형성하며, 박테리오파지 Qβ에 의한 감염에 필요한 것으로서 발견되었는데, 이것은 분명히 박테리아에 있는 이 단백질의 고유 기능은 아니다.모든 박테리아에 보편적으로 존재하는 것은 아니지만 프로테오박테리아, 프로테오박테리아, 프로테오박테스, 스피로차테스, 테르모토개, 아퀴피카에, 그리고 아쿠아과 1종에서 발견되었다.(이 마지막 사례는 아마도 수평 유전자 전달의 경우일 것이다.)Hfq는 다양한 상호작용을 가진 플리토방성이며, 일반적으로 번역 규제와 관련이 있다.여기에는 mRNA에 대한 리보솜 결합 차단, 다A 꼬리에 결합해 mRNA의 분해 표시, 특정 mRNA에 결합해 번역을 제어하는 박테리아 소형 규제 RNA(DrsA RNA 등)와의 연관성 등이 포함된다.[20][21]두 번째 박테리아 LSm 단백질은 YlxS(YhbC라고도 함)로 토양세균 바실러스 소분열에서 처음 확인되었다.N단자 LSm 도메인을 가진 2개 영역 단백질이다.그 기능은 알려지지 않았지만, 아미노산 염기서열 호몰로겐은 현재까지 거의 모든 박테리아 게놈에서 발견되고 있으며, 필수적인 단백질일지도 모른다.[22]대장균에 있는 작은 전도성 기계 민감 채널 MscS의 중간 영역은 호모헵타메리카 링을 형성한다.[23]이 LSm 영역은 겉으로 드러나는 RNA 결합 기능을 가지고 있지 않지만, 호모헵타메리카 토루스는 이 막 단백질의 중심 채널의 일부분이다.null
진화 및 계통발생
LSm 호몰로그는 생명의 세 영역 모두에서 발견되며 심지어 모든 유기체에서도 발견될 수 있다.계산적 계통생성 방법은 계통생성 관계를 추론하는 데 사용된다.1차 구조물의 다중 시퀀스 정렬(아미노산 시퀀스), 3차 구조물의 구조 정렬(3차원 구조) 등 다양한 LSm 호몰로그램 간의 시퀀스 정렬이 이에 적합한 툴이다.LSm 단백질의 유전자가 모든 생물의 마지막 보편적 조상에 존재했다는 가설이다.[24]알려진 LSm 단백질의 기능에 기초하여, 이 원래의 LSm 단백질은 초기 생명에 대한 RNA 세계 가설의 일부로서 단백질을 합성하기 위한 RNA의 처리에 있어서 리볼란체를 보조했을지도 모른다.이 견해에 따르면, 이 유전자는 조상에서 후손으로 전달되었고, 빈번한 돌연변이와 유전자 중복 그리고 때때로 수평적인 유전자가 전이되었다.원칙적으로 이 과정은 마지막 보편적 조상(또는 그 이전)에 뿌리를 두고 오늘날 존재하는 LSm 유전자의 우주를 나타내는 팁을 가진 계통생식 나무로 요약할 수 있다.null
박테리아와 고고학에서 호모메릭 LSm 고리
알려진 LSm 단백질은 구조에 따라 박테리아 LSm 단백질(Hfq, YlxS, MscS)과 다른 모든 LSm 단백질의 두 번째 그룹으로 나뉘는데, 이는 가장 최근에 발표된 계통생성 나무에 따른 것이다.[25]3개의 고고학적 LSm 단백질(Sm1, sm2, sm3)도 eukaryote LSm 단백질과 구별되는 집단으로 군집한다.박테리아와 고고학적 LSm 단백질은 모두 균질 링에 중합되는데, 이것은 조상 조건이다.null
진핵생물의 이형 LSm 링
단일 eukaryote LSm 유전자의 일련의 유전자 복제는 알려진 eukaryote LSm 유전자를 대부분(전부는 아닐지라도) 만들었다.7개의 sm 단백질은 각각 다른 sm 단백질보다 해당 lsm 단백질에 대한 아미노산 염기서열 호몰로지(homology)가 더 크다.이는 조상의 LSm 유전자가 여러 번 복제되어 7개의 파라갈로그가 발생했음을 시사한다.이것들은 이후 서로 떨어져서 조상인 호모헵타머 LSm 고리가 이단 헵타머 고리가 되었다.진핵생물과 고고학에서 LSm 단백질의 알려진 기능을 바탕으로 조상 기능은 프리 리보솜 RNA, 프리트랜스퍼 RNA, 프리 RNase P를 처리하고 있었을지도 모른다.Then, according to this hypothesis, the seven ancestral eukaryote LSm genes duplicated again to seven pairs of Sm/LSm paralogs; LSm1/SmB, LSm2/SmD1, LSm3/SmD2, LSm4/SmD3, LSm5/SmE, LSm6/SmF and LSm7/SmG.7개의 LSm 유전자로 이루어진 이 두 그룹(및 그에 상응하는 두 종류의 LSm 링)은 SM 링(RNA가 필요함)과 LSM 링(RNA 없이 형성됨)으로 진화했다.또한 LSM1/LSM8 파라로그 쌍은 최소 15개의 LSM 단백질 유전자에 대해 마지막 공통 eukaryote 조상 이전에 발생한 것으로 보인다.SmD1/LSM10 파라로그 쌍과 SmD2/LSM11 파라로그 쌍은 동물, 곰팡이, 아메보조아(때로는 유니콘 클라드로 식별되기도 함)에만 존재하며 비콘 클라드(염색체, 굴착체, 식물, 리자리아)에는 없는 것으로 보인다.따라서, 이 두 유전자 중복은 진핵생물 혈통의 이러한 근본적인 분열을 앞섰다.SmB/SmN 파라로그 쌍은 이 LSm 유전자 복제의 연대를 나타내는 태반 포유류에서만 볼 수 있다.null
snRNP의 생물 발생
작은 핵 리보핵단백질(snRNPs)은 세포핵과 세포질 모두를 포함하는 밀접하게 조정되고 조절된 공정으로 조립된다.[26]null
참조
- ^ a b Reeves WH, Narain S, Satoh M (2003). "Henry Kunkel, Stephanie Smith, clinical immunology, and split genes". Lupus. 12 (3): 213–7. doi:10.1191/0961203303lu360xx. PMID 12708785. S2CID 33112464.
- ^ Tan EM, Kunkel HG (March 1966). "Characteristics of a soluble nuclear antigen precipitating with sera of patients with systemic lupus erythematosus". J. Immunol. 96 (3): 464–71. PMID 5932578.
- ^ Will CL, Lührmann R (June 2001). "Spliceosomal UsnRNP biogenesis, structure and function". Curr. Opin. Cell Biol. 13 (3): 290–301. doi:10.1016/S0955-0674(00)00211-8. hdl:11858/00-001M-0000-0012-F770-0. PMID 11343899.
- ^ He W, Parker R (June 2000). "Functions of Lsm proteins in mRNA degradation and splicing". Curr. Opin. Cell Biol. 12 (3): 346–50. doi:10.1016/S0955-0674(00)00098-3. PMID 10801455.
- ^ Törö I, Thore S, Mayer C, Basquin J, Séraphin B, Suck D (May 2001). "RNA binding in an Sm core domain: X-ray structure and functional analysis of an archaeal Sm protein complex". EMBO J. 20 (9): 2293–303. doi:10.1093/emboj/20.9.2293. PMC 125243. PMID 11331594.
- ^ Hermann H, Fabrizio P, Raker VA, Foulaki K, Hornig H, Brahms H, Lührmann R (May 1995). "snRNP Sm proteins share two evolutionarily conserved sequence motifs which are involved in Sm protein-protein interactions". EMBO J. 14 (9): 2076–88. doi:10.1002/j.1460-2075.1995.tb07199.x. PMC 398308. PMID 7744013.
- ^ Kambach C, Walke S, Young R, Avis JM, de la Fortelle E, Raker VA, Lührmann R, Li J, Nagai K (February 1999). "Crystal structures of two Sm protein complexes and their implications for the assembly of the spliceosomal snRNPs". Cell. 96 (3): 375–87. doi:10.1016/S0092-8674(00)80550-4. PMID 10025403. S2CID 17379935.
- ^ 국립 생명공학 정보 구조 데이터베이스 PDB 코드 1B34, 1D3B, 1I5L, 1KQ2, 1N9S, 1IB8
- ^ Khusial P, Plaag R, Zieve GW (September 2005). "LSm proteins form heptameric rings that bind to RNA via repeating motifs". Trends Biochem. Sci. 30 (9): 522–8. doi:10.1016/j.tibs.2005.07.006. PMID 16051491.
- ^ Urlaub H, Raker VA, Kostka S, Lührmann R (January 2001). "Sm protein-Sm site RNA interactions within the inner ring of the spliceosomal snRNP core structure". EMBO J. 20 (1–2): 187–96. doi:10.1093/emboj/20.1.187. PMC 140196. PMID 11226169.
- ^ Seto AG, Zaug AJ, Sobel SG, Wolin SL, Cech TR (September 1999). "Saccharomyces cerevisiae telomerase is an Sm small nuclear ribonucleoprotein particle". Nature. 401 (6749): 177–80. Bibcode:1999Natur.401..177S. doi:10.1038/43694. PMID 10490028. S2CID 4414530.
- ^ Beggs JD (June 2005). "Lsm proteins and RNA processing". Biochem. Soc. Trans. 33 (Pt 3): 433–8. doi:10.1042/BST0330433. PMID 15916535.
- ^ Kufel J, Allmang C, Petfalski E, Beggs J, Tollervey D (January 2003). "Lsm Proteins are required for normal processing and stability of ribosomal RNAs". J. Biol. Chem. 278 (4): 2147–56. doi:10.1074/jbc.M208856200. PMID 12438310.
- ^ Schümperli D, Pillai RS (October 2004). "The special Sm core structure of the U7 snRNP: far-reaching significance of a small nuclear ribonucleoprotein" (PDF). Cell. Mol. Life Sci. 61 (19–20): 2560–70. doi:10.1007/s00018-004-4190-0. PMID 15526162. S2CID 5780814.
- ^ Ma Y, Dostie J, Dreyfuss G, Van Duyne GD (June 2005). "The Gemin6-Gemin7 heterodimer from the survival of motor neurons complex has an Sm protein-like structure". Structure. 13 (6): 883–92. doi:10.1016/j.str.2005.03.014. PMID 15939020.
- ^ Chari A, Golas MM, Klingenhäger M, Neuenkirchen N, Sander B, Englbrecht C, Sickmann A, Stark H, Fischer U (2008-10-31). "An Assembly Chaperone Collaborates with the SMN Complex to Generate Spliceosomal SnRNPs". Cell. 135 (3): 497–509. doi:10.1016/j.cell.2008.09.020. hdl:11858/00-001M-0000-0010-93A3-A. PMID 18984161. S2CID 119444.
- ^ Albrecht M, Lengauer T (July 2004). "Novel Sm-like proteins with long C-terminal tails and associated methyltransferases". FEBS Lett. 569 (1–3): 18–26. doi:10.1016/j.febslet.2004.03.126. PMID 15225602. S2CID 37187215.
- ^ a b Zhang, Jiyuan; Guan, Xin; Shah, Kunal; Yan, Jiusheng (2021-08-06). "Lsm12 is an NAADP receptor and a two-pore channel regulatory protein required for calcium mobilization from acidic organelles". Nature Communications. 12 (1): 4739. doi:10.1038/s41467-021-24735-z. ISSN 2041-1723. PMC 8346516. PMID 34362892.
- ^ Mura C, Kozhukhovsky A, Gingery M, Phillips M, Eisenberg D (April 2003). "The oligomerization and ligand-binding properties of Sm-like archaeal proteins (SmAPs)". Protein Sci. 12 (4): 832–47. doi:10.1110/ps.0224703. PMC 2323858. PMID 12649441.
- ^ Schumacher MA, Pearson RF, Møller T, Valentin-Hansen P, Brennan RG (July 2002). "Structures of the pleiotropic translational regulator Hfq and an Hfq-RNA complex: a bacterial Sm-like protein". EMBO J. 21 (13): 3546–56. doi:10.1093/emboj/cdf322. PMC 126077. PMID 12093755.
- ^ Lease RA, Woodson SA (December 2004). "Cycling of the Sm-like protein Hfq on the DsrA small regulatory RNA". J. Mol. Biol. 344 (5): 1211–23. doi:10.1016/j.jmb.2004.10.006. PMID 15561140.
- ^ Yu L, Gunasekera AH, Mack J, Olejniczak ET, Chovan LE, Ruan X, Towne DL, Lerner CG, Fesik SW (August 2001). "Solution structure and function of a conserved protein SP14.3 encoded by an essential Streptococcus pneumoniae gene". J. Mol. Biol. 311 (3): 593–604. doi:10.1006/jmbi.2001.4894. PMID 11493012.
- ^ Bass RB, Strop P, Barclay M, Rees DC (November 2002). "Crystal structure of Escherichia coli MscS, a voltage-modulated and mechanosensitive channel" (PDF). Science. 298 (5598): 1582–7. Bibcode:2002Sci...298.1582B. doi:10.1126/science.1077945. PMID 12446901. S2CID 15945269.
- ^ Achsel T, Stark H, Lührmann R (March 2001). "The Sm domain is an ancient RNA-binding motif with oligo(U) specificity". Proc. Natl. Acad. Sci. U.S.A. 98 (7): 3685–9. Bibcode:2001PNAS...98.3685A. doi:10.1073/pnas.071033998. PMC 31112. PMID 11259661.
- ^ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (March 2006). "Toward automatic reconstruction of a highly resolved tree of life". Science. 311 (5765): 1283–7. Bibcode:2006Sci...311.1283C. CiteSeerX 10.1.1.381.9514. doi:10.1126/science.1123061. PMID 16513982. S2CID 1615592.
- ^ Kiss T (December 2004). "Biogenesis of small nuclear RNPs". J. Cell Sci. 117 (Pt 25): 5949–51. doi:10.1242/jcs.01487. PMID 15564372.