단백질 아이소폼
Protein isoform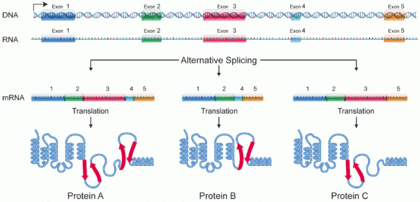
단백질 아이소폼 또는 "단백질 변종"[1]은 단일 유전자 또는 유전자 패밀리에서 유래하고 유전적 [2]차이의 결과인 매우 유사한 단백질 세트의 구성원이다.많은 것들이 같거나 유사한 생물학적 역할을 수행하지만, 일부 등소 형태는 독특한 기능을 가지고 있다.단백질 아이소폼 세트는 단일 유전자의 대체 스플라이싱, 가변 프로모터 사용 또는 기타 전사 후 수정으로부터 형성될 수 있다.일반적으로 번역 후 수정은 고려되지 않는다(단백질 형태 참조).RNA 스플라이싱 메커니즘을 통해 mRNA는 유전자의 다른 단백질 코드 세그먼트(exons) 또는 RNA로부터 다른 exons의 다른 부분을 선택하여 다른 mRNA 서열을 형성할 수 있는 능력을 가지고 있습니다.각각의 독특한 배열은 단백질의 특정한 형태를 생산한다.
아이소폼의 발견은 인간 게놈 프로젝트에 의해 밝혀진 적은 수의 단백질 코드 영역 유전자와 유기체에서 볼 수 있는 단백질의 큰 다양성 사이의 불일치를 설명할 수 있다: 같은 유전자에 의해 코드된 다른 단백질은 단백질의 다양성을 증가시킬 수 있다.RNA 수준의 Isoforms는 cDNA 전사 연구에 의해 쉽게 특징지어집니다.많은 인간 유전자들은 확인된 대체 접합 동형식을 가지고 있다.사람에게서 [1]최대 100,000개의 표현 시퀀스 태그(EST)가 식별될 수 있는 것으로 추정되었다.단백질 수준의 Isoforms는 일반적으로 단백질 표면에 위치한 전체 도메인 또는 짧은 루프의 [3]결실에서 나타날 수 있습니다.
정의.
으로 mRNA번역 isoforms의 풍성함을 반드시 연관되지 않는다 비록 그러한 과정은 인간의 proteome의 다양성에 영향을 미치는, 명확하지 않다 하나의 단일 유전자;[4][5]이 과정 mRNA의 대안적 결합에 의해 규제된다,는 능력을 구조 및 구성 모두에서 다른 여러 단백질 만들 수 있어요.와 함께단백질의 [6]풍부함번역된 아이소폼의 특이성은 단백질의 구조/기능뿐만 아니라 그것들이 [4][5]생산되는 세포 유형 및 발달 단계에 의해 도출된다.단백질에 여러 개의 서브유닛이 있고 각 서브유닛이 여러 개의 아이소폼을 가질 경우 특이성 결정이 더욱 복잡해진다.
예를 들어, 인간 세포에서 서로 다른 역할을 수행하는 효소인 5' AMP 활성화 단백질 키나제(AMPK)는 3개의 서브유닛을 가지고 [7]있다.
- 촉매 도메인인 α는 PRKAA1 및 PRKAA2에서 코드된 두 가지 동질 형태를 가진다.
- 조절 영역인 β는 PRKAB1과 PRKAB2에서 부호화된 β1과 β2의 두 가지 동질 형태를 가진다.
- 규제구역인 θ는 PRKAG1, PRKAG2, PRKAG3에서 부호화된 θ1, θ2, θ3의 3가지 아이소폼을 가진다.
인체 골격근에서 바람직한 형태는 α2β211이다.[7]그러나 인간의 간에서 가장 풍부한 형태는 α1β211이다.[7]
메커니즘
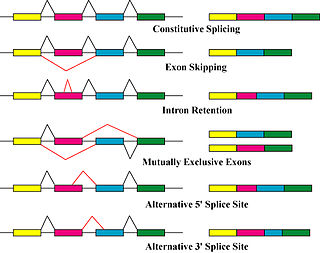
단백질 아이소폼을 생성하는 주요 메커니즘은 대체 스플라이싱과 가변 프로모터 사용이지만, 돌연변이 및 다형성과 같은 유전적 변화로 인한 변형은 때때로 별개의 [8]아이소폼으로 간주된다.
대체 스플라이싱은 mRNA 전사 아이소폼을 생성하는 주요 전사 후 수정 과정이며 단백질 [5]다양성에 기여할 수 있는 주요 분자 메커니즘이다.큰 리보핵단백질인 스플라이세오솜은 RNA 분열과 결찰을 담당하는 핵 내부의 분자 기계로, 비단백질 코드 세그먼트(내부)[9]를 제거합니다.
스플라이싱은 전사와 번역 사이에 발생하는 과정이기 때문에, 그 주된 효과는 주로 유전체학 기술을 통해 연구되어 왔다. 예를 들어 마이크로 어레이 분석과 RNA 시퀀싱은 대체 스플라이싱된 전사를 식별하고 [8]그 풍부성을 측정하기 위해 사용되었다.겔 전기영동과 질량분석법을 사용한 프로테오믹스 실험에서 전사량과 단백질 수 사이의 상관관계가 종종 낮으며 보통 [10]하나의 단백질 동형식이 우세하다는 것이 입증되었지만, 전사물의 풍부성은 종종 단백질 동형식의 풍부함에 대한 대용물로 사용된다.2015년 한 연구에 따르면 이러한 불일치의 원인은 근본적으로 [11]알려지지 않았지만 번역 후에 발생할 가능성이 높다.따라서 대체 스플라이싱이 변이와 질병 사이의 중요한 연결고리로 포함되기는 했지만, 주로 새로운 단백질 아이소폼을 [10]생성함으로써 작용한다는 결정적인 증거는 없다.
대체 스플라이싱은 일반적으로 스플라이싱 기계에 의해 대체 트랜스크립트가 의도적으로 생성되는 엄격하게 규제된 프로세스를 말합니다.하지만, 그러한 전사물은 또한 "노이즈 스플라이싱"이라고 불리는 과정의 스플라이싱 오류에 의해 생성되고, 잠재적으로 단백질 아이소폼으로도 번역됩니다.다중 엑소닉 유전자의 95%가 대체적으로 결합되어 있다고 생각되지만, 잡음 결합에 대한 한 연구는 다른 저농축성 전사체의 대부분이 노이즈임을 관찰하고 세포에 존재하는 대부분의 대체 전사체와 단백질 동질체는 [12]기능적으로 관련이 없다고 예측했다.
다른 전사 및 전사 후 조절 단계도 다른 단백질 [13]동소체를 생성할 수 있습니다.가변 프로모터 사용은 세포의 전사 기계(RNA 중합효소, 전사 인자 및 기타 효소)가 다른 프로모터(초기 결합 부위의 역할을 하는 유전자 근처의 DNA 영역)에서 전사를 시작할 때 발생하며, 결과적으로 약간 수정된 전사물과 단백질 아이소폼이 생성된다.
특성.
일반적으로 하나의 단백질 동질체는 다른 [14]종에서 직교(또는 기능적으로 유사한) 염기서열과의 유병률 및 유사성과 같은 기준에 기초하여 표준 염기서열로 라벨링된다.Isoforms는 대부분 유사한 시퀀스를 가지며 일부에서 대부분의 exons를 표준 시퀀스와 공유하기 때문에 유사한 기능 특성을 갖는 것으로 가정한다.그러나 일부 Isoforms는 (예를 들어 트랜스스플라이싱을 통해) 훨씬 더 큰 차이를 보이며 정규 시퀀스와 exon을 거의 또는 전혀 공유할 수 없습니다.또한, 그들은 다른 생물학적 영향을 가질 수 있다. 예를 들어, 극단적인 경우, 한 아이소폼의 기능은 세포 생존을 촉진하는 반면, 다른 아이소폼은 세포 사멸을 촉진할 수 있다. 또는 유사한 기본 기능을 가질 수 있지만 세포 내 위치 [15]파악에 차이가 있을 수 있다.그러나 2016년 한 연구는 1,492개의 유전자의 모든 동소체를 기능적으로 특징지었고 대부분의 동소체가 "기능적 동소체"로 작용한다는 것을 알아냈다.저자들은 대부분의 아이소폼의 기능이 [16]겹치지 않는 것을 관찰한 후 아이소폼이 별개의 단백질처럼 행동한다는 결론에 도달했다.이 연구는 체외 세포에 대해 수행되었기 때문에 발현된 인간 프로테옴의 동질체가 이러한 특징을 공유하는지 여부는 알려지지 않았다.또한 각 등각형의 기능은 일반적으로 개별적으로 결정되어야 하기 때문에 대부분의 식별 및 예측된 등각형은 여전히 알려지지 않은 기능을 가지고 있다.
관련 개념
글리코폼
글리코폼은 부착된 글리칸의 수 또는 종류에 따라만 다른 단백질의 아이소폼이다.당단백질은 종종 부착된 당류나 올리고당의 변화를 수반하는 여러 가지 당질 형태로 구성됩니다.이러한 변형은 글리코실화 과정 중의 생합성 차이 또는 글리코시드가수분해효소 또는 글리코실전달효소의 작용으로 인해 발생할 수 있다.글리코폼은 분리된 글리코폼의 상세한 화학분석을 통해 검출될 수 있으나 렉틴 친화성 크로마토그래피 및 렉틴 친화성 전기영동처럼 렉틴과의 미분반응을 통해 검출이 용이하다.당단백질의 대표적인 예는 오로소무코이드, 항트립신, 합토글로빈과 같은 혈중 단백질이다.다시아산을 포함한 신경세포 접착 분자, NCAM, PSA에서 비정상적인 당 형태 변화가 관찰된다.
예
- G-actin: 보존된 성질에도 불구하고, 그것은 다양한 수의 등소체를 가지고 있습니다(포유동물에서는 적어도 6개).
- 심근경색 진단에 도움이 되는 크레아틴인산화효소(creatine kinase)는 3가지 동질형태로 존재한다.
- 히알루론산 생성에 관여하는 효소인 히알루론산 합성효소는 포유동물 세포에 세 가지 동소체를 가지고 있다.
- UDP-글루쿠로노실트랜스퍼라아제는 많은 약물, 환경오염물질 및 독성 내인성 화합물의 해독 경로를 담당하는 효소 슈퍼패밀리로, 인간 [17]게놈에 코드된 16개의 알려진 동질 형태를 가지고 있다.
- G6PDA : 조직의 세포에서 활성 아이소폼의 정상비율은 G6PDG와 1:1로 동일하며, 이는 정확하게 과형성의 정상 아이소폼비이다.이러한 동질 형태들 중 하나만 종양 발생 중에 [18]발견됩니다.
모노아민의 산화를 촉매하는 효소 계열인 모노아민 산화효소는 MAO-A와 MAO-B의 두 가지 동소 형태로 존재한다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b Brett D, Pospisil H, Valcárcel J, Reich J, Bork P (January 2002). "Alternative splicing and genome complexity". Nature Genetics. 30 (1): 29–30. doi:10.1038/ng803. PMID 11743582. S2CID 2724843.
- ^ Schlüter H, Apweiler R, Holzhütter HG, Jungblut PR (September 2009). "Finding one's way in proteomics: a protein species nomenclature". Chemistry Central Journal. 3: 11. doi:10.1186/1752-153X-3-11. PMC 2758878. PMID 19740416.
- ^ Kozlowski, L.; Orlowski, J.; Bujnicki, J. M. (2012). "Structure Prediction for Alternatively Spliced Proteins". Alternative pre-mRNA Splicing. p. 582. doi:10.1002/9783527636778.ch54. ISBN 9783527636778.
- ^ a b Andreadis A, Gallego ME, Nadal-Ginard B (1987-01-01). "Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications". Annual Review of Cell Biology. 3 (1): 207–42. doi:10.1146/annurev.cb.03.110187.001231. PMID 2891362.
- ^ a b c Breitbart RE, Andreadis A, Nadal-Ginard B (1987-01-01). "Alternative splicing: a ubiquitous mechanism for the generation of multiple protein isoforms from single genes". Annual Review of Biochemistry. 56 (1): 467–95. doi:10.1146/annurev.bi.56.070187.002343. PMID 3304142.
- ^ Liu Y, Beyer A, Aebersold R (April 2016). "On the Dependency of Cellular Protein Levels on mRNA Abundance". Cell. 165 (3): 535–50. doi:10.1016/j.cell.2016.03.014. PMID 27104977.
- ^ a b c Dasgupta B, Chhipa RR (March 2016). "Evolving Lessons on the Complex Role of AMPK in Normal Physiology and Cancer". Trends in Pharmacological Sciences. 37 (3): 192–206. doi:10.1016/j.tips.2015.11.007. PMC 4764394. PMID 26711141.
- ^ a b Kornblihtt AR, Schor IE, Alló M, Dujardin G, Petrillo E, Muñoz MJ (March 2013). "Alternative splicing: a pivotal step between eukaryotic transcription and translation". Nature Reviews Molecular Cell Biology. 14 (3): 153–65. doi:10.1038/nrm3525. PMID 23385723. S2CID 54560052.
- ^ Lee Y, Rio DC (2015-01-01). "Mechanisms and Regulation of Alternative Pre-mRNA Splicing". Annual Review of Biochemistry. 84 (1): 291–323. doi:10.1146/annurev-biochem-060614-034316. PMC 4526142. PMID 25784052.
- ^ a b Tress ML, Abascal F, Valencia A (February 2017). "Alternative Splicing May Not Be the Key to Proteome Complexity". Trends in Biochemical Sciences. 42 (2): 98–110. doi:10.1016/j.tibs.2016.08.008. PMC 6526280. PMID 27712956.
- ^ Battle A, Khan Z, Wang SH, Mitrano A, Ford MJ, Pritchard JK, Gilad Y (February 2015). "Genomic variation. Impact of regulatory variation from RNA to protein". Science. 347 (6222): 664–7. doi:10.1126/science.1260793. PMC 4507520. PMID 25657249.
- ^ Pickrell JK, Pai AA, Gilad Y, Pritchard JK (December 2010). "Noisy splicing drives mRNA isoform diversity in human cells". PLOS Genetics. 6 (12): e1001236. doi:10.1371/journal.pgen.1001236. PMC 3000347. PMID 21151575.
- ^ Smith LM, Kelleher NL (March 2013). "Proteoform: a single term describing protein complexity". Nature Methods. 10 (3): 186–7. doi:10.1038/nmeth.2369. PMC 4114032. PMID 23443629.
- ^ Li HD, Menon R, Omenn GS, Guan Y (December 2014). "Revisiting the identification of canonical splice isoforms through integration of functional genomics and proteomics evidence" (PDF). Proteomics. 14 (23–24): 2709–18. doi:10.1002/pmic.201400170. PMC 4372202. PMID 25265570.
- ^ Sundvall M, Veikkolainen V, Kurppa K, Salah Z, Tvorogov D, van Zoelen EJ, Aqeilan R, Elenius K (December 2010). "Cell death or survival promoted by alternative isoforms of ErbB4". Molecular Biology of the Cell. 21 (23): 4275–86. doi:10.1091/mbc.E10-04-0332. PMC 2993754. PMID 20943952.
- ^ Yang X, Coulombe-Huntington J, Kang S, Sheynkman GM, Hao T, Richardson A, et al. (February 2016). "Widespread Expansion of Protein Interaction Capabilities by Alternative Splicing". Cell. 164 (4): 805–17. doi:10.1016/j.cell.2016.01.029. PMC 4882190. PMID 26871637.
- ^ Barre L, Fournel-Gigleux S, Finel M, Netter P, Magdalou J, Ouzzine M (March 2007). "Substrate specificity of the human UDP-glucuronosyltransferase UGT2B4 and UGT2B7. Identification of a critical aromatic amino acid residue at position 33". The FEBS Journal. 274 (5): 1256–64. doi:10.1111/j.1742-4658.2007.05670.x. PMID 17263731.
- ^ 병리학의 기초인 파토마


