플라비바이러스
Flavivirus플라비바이러스는 플라비바이러스과에 속하는 양스트랜드 RNA 바이러스의 속이다. 속에는 웨스트 나일 바이러스, 뎅기 바이러스, 진드기 매개 뇌염 바이러스, 황열병 바이러스, 지카 바이러스 및 뇌염을 유발할 수 있는 다른 여러 바이러스뿐만 아니라 세포 퓨전 바이러스(CFAV), 팜 크릭 바이러스(PCV), 파라마타 리버 바이러스(Parramatta River Virus)와 같은 곤충 고유의 향미료(ISFs)가 포함되어 있다.[3][4] 이중 숙주 향미균은 절지동물은 물론 척추동물을 감염시킬 수 있지만, 곤충 특유의 향미균은 유능한 절지동물로 제한된다.[5]
플라비루스는 황열병 바이러스의 이름을 따서 명명되었다. 플라비루스는 라틴어로 '황색'을 의미하고, 황열은 희생자들에게 황달의 원인이 되는 경향으로부터 차례로 명명되었다.[6]
플라미비루스는 몇 가지 공통적인 측면을 공유한다: 공통 크기(40~65nm), 대칭성(개발된, 이코사헤드랄 핵캡시드), 핵산(양성-감각, 1만~11,000 베이스 정도의 단일 가닥 RNA), 전자 현미경 아래에서의 외관.
이들 바이러스의 대부분은 주로 감염된 절지동물(모스키토 또는 진드기)에 물린 것에 의해 전염되기 때문에 아르보바이러스로 분류된다. 이러한 아르보바이러스의 대부분을 가진 인간 감염은 부수적인 것이다. 왜냐하면 인간은 바이러스 수명을 지속하는 데 필요한 절지동물을 재감염할 수 있을 만큼 충분히 높은 적자에 바이러스를 복제할 수 없기 때문이다. 즉, 인간은 막다른 숙주가 된다. 이에 대한 예외는 황열병 바이러스, 뎅기 바이러스, 지카 바이러스다. 이 세 가지 바이러스는 여전히 모기 벡터를 필요로 하지만 반드시 동물 숙주에 의존하지는 않을 정도로 인간에게 잘 적응되어 있다(그러나 그들은 중요한 동물 전염 경로를 계속 가지고 있다).
아르보바이러스의 다른 바이러스 전파 경로로는 감염된 동물의 사체 처리, 수혈, 성관계, 출산, 살균되지 않은 유제품 소비 등이 있다. 비인간 척추동물에서 중간 벡터 절지동물이 없는 인간으로의 전염은 대부분 낮은 확률로 발생한다. 예를 들어, 초기 황열병 검사에서 그 질병은 전염성이 없다는 것을 보여주었다.
플라비바이러스 계열의 알려진 비아르보바이러스는 절지동물이나 척추동물 중 하나에서 번식하지만 둘 다에서 번식하지 않으며, 그 중 하나의 홀수 구성원이 네마모드에 영향을 미친다.[7]
구조
플라미비루스는 포개지고 구면이며 사이비 T=3 대칭을 가진 이코사이드 기하학을 가지고 있다. 바이러스 입자 직경은 약 50nm이다.[8]
게놈
플라미비루스는 양성의 단일 가닥의 RNA 게놈을 가지고 있는데, 이 게놈은 분할되지 않고 길이가 약 10–11 kbp이다.[8] 일반적으로 게놈은 세 가지 구조 단백질(캡시드, prM, Envelope)과 일곱 가지 비구조적 단백질(NS1, NS2A, NS2B, NS3, NS4A, NS4B, NS5)을 암호화하고 있다.[9] 유전자 RNA는 cap-1 구조(me-GppA-me72)로 양성-스트랜드 유전 RNA의 5㎛ 끝에서 수정된다.[10]
라이프 사이클

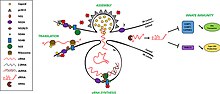
미각은 숙주 세포의 세포질에서 복제된다. 게놈은 다항아연화(폴리-A) 꼬리가 없는 것을 제외한 모든 면에서 세포 mRNA 분자를 모방한다. 이 특성은 복제하는 동안 바이러스가 세포 유령을 이용하여 구조 단백질과 비구조 단백질 모두를 합성할 수 있게 한다. 세포 리보솜은 RNA를 세포 mRNA와 유사한 방식으로 번역해 하나의 다단백질 합성을 이끌어 내기 때문에 플라비바이러스의 복제에 결정적이다.[9]
세포 RNA 캡 구조는 RNA 3인산효소의 작용을 통해 형성되며, 관릴전달효소, N7-메틸전달효소, 2 and-O 메틸전달효소 등이 있다. 그 바이러스는 이러한 활동을 비구조적 단백질로 암호화한다. NS3 단백질은 헬리코아제 영역 내에 RNA 3인산효소를 인코딩한다. 헬리카아제 ATP 가수분해 부지를 사용해 RNA의 5㎛ 끝에서 γ-인산염(phosphate)을 제거한다. 비구조 단백질 5(NS5)의 N-단자 영역은 성숙한 RNA 캡 구조를 형성하는 데 필요한 N7-메틸전달효소와 구알릴전달효소 활동을 모두 가지고 있다. RNA 결합 친화력은 ATP 또는 GTP의 존재에 의해 감소되고 S-adenosyl 메티오닌에 의해 강화된다.[10] 이 단백질은 또한 2′-O 메틸전달효소를 암호화한다.

일단 번역되면, 다단백질은 성숙한 폴리펩타이드 제품을 출시하기 위해 바이러스성 단백질과 숙주성 단백질의 결합에 의해 분해된다.[11] 그럼에도 불구하고, 세포 변환 후 수정은 다-A 꼬리의 존재에 따라 달라진다. 따라서 이 과정은 호스트에 의존하지 않는다. 대신에, 다단백질에는 바이러스 특이 효소인 첫 번째 펩타이드의 자동 방출 기능이 포함되어 있다. 그리고 나서 이 효소는 남은 다단백질을 개별 생산물에 분해할 수 있다. 분해된 제품 중 하나는 RNA 의존성 RNA 중합효소로서 음의 RNA 분자의 합성을 담당한다. 결과적으로, 이 분자는 유전체 프로게니 RNA의 합성을 위한 템플릿의 역할을 한다.
플라비바이러스 유전체 RNA 복제는 막실 구획의 거친 내소성 망막에서 발생한다. 새로운 바이러스 입자들이 이후에 조립된다. 이것은 봉투를 축적한 것과 세포 용해에도 책임이 있는 싹트기 과정 중에 발생한다.
G 단백질 결합 수용체 키나아제2(ADRBK1)는 플라비비비르과에서 여러 바이러스의 입력과 복제에 중요한 것으로 보인다.[12]
인간, 포유류, 모기, 진드기가 자연 숙주 역할을 한다. 전송 경로는 조노시스(Zunosis)와 물림이다.[8]
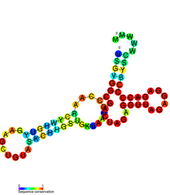
RNA 이차 구조 요소
플라비바이러스의 양성 감각 RNA 게놈에는 5'와 3'의 미역전 영역(UTR)이 들어 있다.
5'UTR
5'UTR은 뎅기 바이러스에서 95-101 뉴클레오티드가 길다.[13] 플라비바이러스 5'에는 보존된 두 가지 구조적 요소가 있다.UTR, 대형 스템 루프(SLA) 및 짧은 스템 루프(SLB) SLA는 측면 스템 루프와 작은 상단 루프가 있는 Y자 구조로 접힌다.[13][14] SLA는 촉진자 역할을 할 가능성이 높으며, 바이러스성 RNA 합성에 필수적이다.[15][16] SLB는 5' 사이의 상호작용에 관여한다.UTR과 3'바이러스 복제에 필수적인 바이러스 RNA의 사이클화를 초래하는 UTR.[17]
3'UTR
3'UTR의 길이는 일반적으로 0.3–0.5KB이며, 보존 상태가 양호하고 향미바이러스 계열로 제한되는 보존 상태가 높은 2차 구조를 다수 포함하고 있다. 대다수의 분석은 웨스트 나일 바이러스(WNV)를 사용하여 3의 기능을 연구해 왔다.UTR.
현재 3차 구조물은 8개소가 확인되었다.WNV의 UTR이며 (3'UTR과 함께 발견된 순서대로) SL-I, SL-III, SL-IV, DB1, DB2 및 CRE이다.[18][19] 이러한 이차 구조들 중 일부는 특징지어졌으며 바이러스 복제를 촉진하고 3차 구조물을 보호하는 데 중요하다.5' 내분비 소화에 따른 UTR. 누클레스 저항은 다운스트림 3'의 UTR RNA 파편을 열화로부터 보호하며 바이러스에 의한 세포질성 및 병생성에 필수적이다.
- SL-II
SL-II는 누클레스 저항성에 기여하는 것으로 제안되었다.[19] 5'에서 확인된 또 다른 헤어핀 루프와 관련이 있을 수 있다.일본뇌염바이러스(JEV) 게놈의 UTR.[20] JEV 헤어핀은 호스트 세포 감염 시 상당히 과대표시되며, 헤어핀 구조가 RNA 합성을 조절하는 역할을 할 수 있다고 제안되었다.
- SL-IV
이차 구조물은 3' 안에 위치한다.DB 요소 업스트림 플라비바이러스 게놈의 UTR. 보존된 구조물의 기능은 알려져 있지 않지만 리보누클레스 저항성에 기여할 것으로 생각된다.
- DB1/DB2
보존된 이 2차 구조물은 사이비 반복 원소라고도 한다. 그것들은 원래 뎅기 바이러스의 게놈 안에서 확인되었고, 3개의 바이러스 안에서 서로 인접하여 발견된다.그들은 플라비비라대 전역에 널리 보존되어 있는 것으로 보인다. 이러한 DB 요소는 3개의 나선형으로 구성된 2차 구조를 가지며 효율적인 번역을 보장하는 역할을 한다. DB1 삭제는 작지만 번역량이 크게 줄지만 DB2 삭제는 별 효과가 없다. DB1과 DB2를 모두 삭제하면 바이러스 게놈의 번역 효율이 25%[18]로 낮아졌다.
- 크레
CRE는 3'SL RNA 원소라고도 알려진 Cis 작용 복제 원소로, "복제 복합체"[21] 형성을 촉진하여 바이러스 복제에 필수적인 것으로 생각된다. 비록 이 RNA에 가성 구조의 존재에 대한 증거가 제시되었지만, 그것은 향미료에 걸쳐 잘 보존되지 않는 것으로 보인다.[22] 3' UTR의 미각 제거는 전염성 클론에게 치명적이라는 것이 밝혀졌다.
보존된 헤어핀 cHP
보존된 헤어핀(cHP) 구조는 나중에 몇몇 플라비바이러스 유전체에서 발견되었으며 캡시드 단백질의 직접 번역으로 여겨진다. 그것은 AUG 시작 코돈의 바로 하류에 위치해 있다.[23]
sfRNA 생산에서 RNA 2차 구조의 역할
서브게놈 플라비바이러스 RNA(subgenomic favivirus RNA, sfRNA)는 3'의 UTR의 연장선으로, 플라비바이러스 복제와 병원생식에 대한 역할을 하는 것으로 입증되었다.[24]sfRNA는 숙주세포 5'-3'의 터무니없는 바이러스 RNA(XRN1)에 의한 게놈 바이러스 RNA의 불완전 분해에 의해 생성된다.[25] XRN1은 바이러스성 RNA를 분해하므로, 5'와 3' UTR의 2차 구조에 의해 형성된 스템루프에서 정지한다.[26] 이 일시 정지는 sfRNA로 알려진 게놈 RNA의 분해되지 않은 파편을 초래한다. sfRNA는 농도에 의존하는 방식으로 플라비바이러스의 수명 주기에 영향을 미친다. sfRNA의 축적은 (1) 세포의 선천적인 면역반응의 반감을 유발하여 바이러스[27] (2) XRN1 및 디커 활동에 대한 숙주방어를 감소시켜 바이러스 복제 콤플렉스의 바이러스 RNA[28](3) 수정을 파괴하는 RNAi 경로를 수정하여 바이러스 재생을 증가시킨다.[29] 전체적으로, sfRNA는 숙주 방어를 손상시키고 향미료에 의한 감염을 촉진하는 다중 경로에 내포되어 있다.
진화

향미료는 벡터 매개 바이러스를 가진 것과 알려진 벡터가 없는 것의 두 개의 층으로 나눌 수 있다.[30] 벡터 클래드는 차례로 모기가 매개하는 클래드와 진드기가 매개하는 클래드로 세분될 수 있다. 이들 집단은 다시 나눌 수 있다.[31]
모기군은 두 가지로 나눌 수 있다: 한 가지 가지에는 사람 또는 가축에서 흔히 뇌질환과 관련된 신경방성 바이러스가 들어 있다. 이 가지에는 쿠렉스 종에 의해 퍼지는 경향이 있고 새 저수지가 있다. 두 번째 가지는 인간의 출혈성 질환과 관련된 비신경성 바이러스다. 이들은 벡터와 영장류 숙주로서 Aedes 종을 가지는 경향이 있다.
진드기 매개 바이러스는 또한 두 개의 뚜렷한 그룹을 형성한다. 하나는 바닷새와 관련이 있고 다른 하나는 진드기 매개 뇌염 복합 바이러스인데 주로 설치류와 관련이 있다.
알려진 벡터가 없는 바이러스는 세 그룹으로 나뉠 수 있다: 하나는 박쥐와 관련된 모기 매개 바이러스와 밀접하게 관련된 것이고, 다른 하나는 유전적으로 더 멀리 떨어져 있는 박쥐와 관련이 있고, 다른 하나는 설치류와 관련이 있다.
진드기 전달은 모기에 의한 그룹에서 유래되었을 가능성이 있어 보인다.[32]
바다거미 엔데이스 스피노사(Endeis Spinosa)[33]에서 플라비바이러스의 부분 게놈 하나가 발견됐다. 그 순서는 곤충 고유의 향미료에 포함된 것과 관련이 있다. 이 염기서열이 이 바이러스군의 진화에 어떻게 부합하는지는 현재 명확하지 않다.
이러한 바이러스들 중 몇 가지에 대해 발산 시간 추정이 이루어졌다.[34] 이 바이러스의 기원은 적어도 9400년에서 14,000년 전으로 보인다. 구세계와 신대륙의 뎅기 계통은 150년에서 450년 사이에 갈라졌다. 유럽과 극동 진드기 매개 뇌염 변종은 약 1087년(1610–649) 전에 갈라졌다. 유럽의 진드기 매개 뇌염과 질병 바이러스를 물리치는 것은 약 572년 전에 갈라졌다. 이 후자의 추정치는 역사적 기록과 일치한다. 건진 바이러스는 약 277년 전에 웨스트 나일 바이러스로부터 분리되었다. 이 시간은 유럽에서 호주가 정착한 것에 해당한다. 일본뇌염군은 2000~3000년 전 아프리카에서 진화했다가 초기 동남아로 퍼진 뒤 나머지 아시아로 이주한 것으로 보인다.
웨스트 나일 바이러스에 대한 계통학적 연구는 그것이 약 1000년 전에 뚜렷한 바이러스로 나타났다는 것을 보여주었다.[35] 이 초기 바이러스는 두 개의 뚜렷한 계통인 계통 1로 발전했고 그것의 다중 프로파일은 아프리카와 전 세계에서 전염병의 근원이다. 리니지2는 아프리카 조노시스라고 여겨졌다. 그러나, 2008년에 사하라 이남 아프리카와 마다가스카르의 말에서만 보던 혈통 2가 유럽에서 말에서 나타나기 시작했는데, 유럽에서 처음 알려진 발병은 2008년에 헝가리에서 18마리의 동물에게 영향을 미쳤다.[36] 리니지1 웨스트나일 바이러스는 2010년 남아프리카에서 암말과 그녀의 낙태된 태아에서 검출되었다. 이전에는 남아프리카의 말과 인간에게서만 2 웨스트나일 바이러스만이 검출되었다.[37] 텍사스의 범고래에서 2007년 치명적인 사건이 발생하면서 웨스트 나일 바이러스의 알려진 숙주 범위가 고래를 포함하도록 넓어졌다.[38]
옴스크 출혈열 바이러스는 지난 1000년 이내에 진화한 것으로 보인다.[39] 바이러스성 게놈은 A와 B라는 두 개의 층으로 나눌 수 있다. Clade A는 5개의 유전자형을 가지고 있고 Clade B는 1개의 유전자형을 가지고 있다. 이 집들은 약 700년 전에 갈라졌다. 이 분리는 쿠르간 지방에서 발생한 것으로 보인다. 클레이드 A는 그 후 230년 전에 C, D, E로 분할되었다. 클래드 C와 E는 각각 노보시비르스크 지방과 옴스크 지방에서 유래한 것으로 보인다. 이 바이러스에 매우 취약한 사향라트 온다트라 지베티쿠스는 1930년대에 이 지역에 도입되었다.
분류학
종
플라비바이러스속에는 53종이 정의되어 있다.[40]
- 아포이 바이러스
- 아로아바이러스
- 바가자바이러스
- 반지바이러스
- 부부우이바이러스
- 부칼라사박쥐바이러스
- 카카오포레바이러스
- 캐리 아일랜드 바이러스
- 카우본 리지 바이러스
- 다카르박쥐바이러스
- 뎅기 바이러스
- 에지 힐 바이러스
- 엔테베박쥐바이러스
- 가젯 굴리 바이러스
- 일허스 바이러스
- 이스라엘 칠면조 뇌염 바이러스
- 일본뇌염바이러스
- 주그라바이러스
- 주티아파바이러스
- 카담바이러스
- 케두구 바이러스
- 코코베라바이러스
- 쿠탕고바이러스
- 랴사누르 산림병바이러스
- 랑가트바이러스
- 루핑병바이러스
- 마반바이러스
- 모독바이러스
- 몬태나 근티백혈뇌염바이러스
- 머레이밸리뇌염바이러스
- 은타야바이러스
- 옴스크 출혈열 바이러스
- 프놈펜박쥐바이러스
- 포와산 바이러스
- 리오브라보바이러스
- 로열팜바이러스
- 사보야 바이러스
- 세인트루이스뇌염바이러스
- 살 비에자 바이러스
- 산펄리타바이러스
- 소마레즈 암초 바이러스
- 세픽바이러스
- 템부스 바이러스
- 진드기 매개 뇌염 바이러스
- 튤레니바이러스
- 우간다S바이러스
- 우수투바이러스
- 웨셀스브론바이러스
- 웨스트 나일 바이러스
- 야우데 바이러스
- 황열병바이러스
- 요코세바이러스
- 지카바이러스
벡터별로 정렬됨
백신
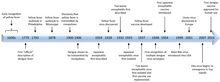
1937년에 도입된 매우 성공적인 황열병 17D 백신은 전염병 활동을 획기적으로 감소시켰다.
효과적인 불활성화 일본뇌염과 진드기 매개 뇌염 백신이 20세기 중반에 도입되었다. 용납할 수 없는 부작용은 생쥐 뇌가 활성화되지 않은 일본뇌염 백신을 보다 안전하고 효과적인 2세대 일본뇌염 백신으로 변화시키는 계기가 되었다. 이러한 질병들은 아시아의 거대한 인구인 북, 남, 동남에서 이 심각한 질병을 효과적으로 예방하기 위해 널리 사용될 수 있다.
뎅기 바이러스는 세계적으로 성공한 모기 벡터에 의한 전염으로 인해 매년 수백만 건의 감염을 일으킨다. 모기 방제에 실패하면서 여러 뎅기 백신이 다양한 개발 단계에 있다. 덩박시아라는 상표명으로 판매되는 CYD-TDV는 4개의 뎅기 바이러스의 구조적 유전자를 17D 황열등뼈에 이식하는 4가 치핵 백신이다.[45][46] 뎅박시아는 5개국에서 승인된다.[47]
참조
- ^ "Virus Taxonomy: 2018b Release". International Committee on Taxonomy of Viruses (ICTV). March 2019. Retrieved 16 March 2019. }}
- ^ Sirohi D, Chen Z, Sun L, Klose T, Pierson TC, Rossmann MG, Kuhn RJ (April 2016). "The 3.8 Å resolution cryo-EM structure of Zika virus". Science. 352 (6284): 467–470. Bibcode:2016Sci...352..467S. doi:10.1126/science.aaf5316. PMC 4845755. PMID 27033547.
- ^ Shi, P-Y (editor) (2012). Molecular Virology and Control of Flaviviruses. Caister Academic Press. ISBN 978-1-904455-92-9.CS1 maint: 추가 텍스트: 작성자 목록(링크)
- ^ McLean, Breeanna J.; Hobson-Peters, Jody; Webb, Cameron E.; Watterson, Daniel; Prow, Natalie A.; Nguyen, Hong Duyen; Hall-Mendelin, Sonja; Warrilow, David; Johansen, Cheryl A.; Jansen, Cassie C.; van den Hurk, Andrew F.; Beebe, Nigel W.; Schnettler, Esther; Barnard, Ross T.; Hall, Roy A. (2015). "A novel insect-specific flavivirus replicates only in Aedes-derived cells and persists at high prevalence in wild Aedes vigilax populations in Sydney, Australia". Virology. 486: 272–283. doi:10.1016/j.virol.2015.07.021. PMID 26519596.
- ^ Elrefaey, Ahmed ME; Abdelnabi, Rana; Rosales Rosas, Ana Lucia; Wang, Lanjiao; Basu, Sanjay; Delang, Leen (September 2020). "Understanding the Mechanisms Underlying Host Restriction of Insect-Specific Viruses". Viruses. 12 (9): 964. doi:10.3390/v12090964. PMC 7552076. PMID 32878245.
- ^ "황열병"에 대한 최초의 언급은 버지니아의 존 미첼이 1744년에 쓴 원고에 나타난다; 원고의 사본은 뉴욕의 의사인 Cadwallader Colden씨와 필라델피아의 벤자민 러쉬에게 보내졌다; 원고는 결국 1814년에 다시 인쇄되었다. 참조:
- Mitchell, John (written: 1744; reprinted: 1814) "Account of the Yellow fever which prevailed in Virginia in the years 1737, 1741, and 1742, in a letter to the late Cadwallader Colden, Esq. of New York, from the late John Mitchell, M.D.F.R.S. of Virginia", American Medical and Philosophical Register, 4: 181–215.
- ^ Jump up to: a b Bekal S, Domier LL, Gonfa B, McCoppin NK, Lambert KN, Bhalerao K (2014). "A novel flavivirus in the soybean cyst nematode". Journal of General Virology. 95 (Pt 6): 1272–1280. doi:10.1099/vir.0.060889-0. PMID 24643877.
- ^ Jump up to: a b c "Viral Zone". ExPASy. Retrieved 15 June 2015.
- ^ Jump up to: a b Rice, C.; Lenches, E.; Eddy, S.; Shin, S.; Sheets, R.; Strauss, J. (23 August 1985). "Nucleotide sequence of yellow fever virus: implications for flavivirus gene expression and evolution". Science. 229 (4715): 726–33. Bibcode:1985Sci...229..726R. doi:10.1126/science.4023707. PMID 4023707. Retrieved 14 November 2016.
- ^ Jump up to: a b Henderson BR, Saeedi BJ, Campagnola G, Geiss BJ (2011). Jeang K (ed.). "Analysis of RNA binding by the Dengue virus NS5 RNA capping enzyme". PLOS ONE. 6 (10): e25795. Bibcode:2011PLoSO...625795H. doi:10.1371/journal.pone.0025795. PMC 3192115. PMID 22022449.
- ^ Sun, G.; Larsen, C.; Baumgarth, N.; Klem, E; Scheuermann, R. (26 January 2017). "Comprehensive Annotation of Mature Peptides and Genotypes for Zika Virus". PLOS ONE. 12 (1): e0170462. Bibcode:2017PLoSO..1270462S. doi:10.1371/journal.pone.0170462. PMC 5268401. PMID 28125631.
- ^ Le Sommer C, Barrows NJ, Bradrick SS, Pearson JL, Garcia-Blanco MA (2012). Michael SF (ed.). "G protein-coupled receptor kinase 2 promotes flaviviridae entry and replication". PLOS Negl Trop Dis. 6 (9): e1820. doi:10.1371/journal.pntd.0001820. PMC 3441407. PMID 23029581.
- ^ Jump up to: a b Gebhard LG, Filomatori CV, Gamarnik AV (2011). "Functional RNA elements in the dengue virus genome". Viruses. 3 (9): 1739–56. doi:10.3390/v3091739. PMC 3187688. PMID 21994804.
- ^ Brinton MA, Dispoto JH (1988). "Sequence and secondary structure analysis of the 5'-terminal region of flavivirus genome RNA". Virology. 162 (2): 290–9. doi:10.1016/0042-6822(88)90468-0. PMID 2829420.
- ^ Filomatori CV, Lodeiro MF, Alvarez DE, Samsa MM, Pietrasanta L, Gamarnik AV (2006). "A 5' RNA element promotes dengue virus RNA synthesis on a circular genome". Genes Dev. 20 (16): 2238–49. doi:10.1101/gad.1444206. PMC 1553207. PMID 16882970.
- ^ Yu L, Nomaguchi M, Padmanabhan R, Markoff L (2008). "Specific requirements for elements of the 5' and 3' terminal regions in flavivirus RNA synthesis and viral replication". Virology. 374 (1): 170–85. doi:10.1016/j.virol.2007.12.035. PMC 3368002. PMID 18234265.
- ^ Alvarez DE, Lodeiro MF, Ludueña SJ, Pietrasanta LI, Gamarnik AV (2005). "Long-range RNA-RNA interactions circularize the dengue virus genome". J Virol. 79 (11): 6631–43. doi:10.1128/JVI.79.11.6631-6643.2005. PMC 1112138. PMID 15890901.
- ^ Jump up to: a b Chiu WW, Kinney RM, Dreher TW (July 2005). "Control of Translation by the 5′- and 3′-Terminal Regions of the Dengue Virus Genome". J. Virol. 79 (13): 8303–15. doi:10.1128/JVI.79.13.8303-8315.2005. PMC 1143759. PMID 15956576.
- ^ Jump up to: a b Pijlman GP, Funk A, Kondratieva N, et al. (December 2008). "A highly structured, nuclease-resistant, noncoding RNA produced by flaviviruses is required for pathogenicity". Cell Host Microbe. 4 (6): 579–91. doi:10.1016/j.chom.2008.10.007. PMID 19064258.
- ^ Lin KC, Chang HL, Chang RY (May 2004). "Accumulation of a 3′-Terminal Genome Fragment in Japanese Encephalitis Virus-Infected Mammalian and Mosquito Cells". J. Virol. 78 (10): 5133–8. doi:10.1128/JVI.78.10.5133-5138.2004. PMC 400339. PMID 15113895.
- ^ Zeng L, Falgout B, Markoff L (September 1998). "Identification of Specific Nucleotide Sequences within the Conserved 3′-SL in the Dengue Type 2 Virus Genome Required for Replication". J. Virol. 72 (9): 7510–22. doi:10.1128/JVI.72.9.7510-7522.1998. PMC 109990. PMID 9696848.
- ^ Shi PY, Brinton MA, Veal JM, Zhong YY, Wilson WD (April 1996). "Evidence for the existence of a pseudoknot structure at the 3' terminus of the flavivirus genomic RNA". Biochemistry. 35 (13): 4222–30. doi:10.1021/bi952398v. PMID 8672458.
- ^ Clyde K, Harris E (2006). "RNA Secondary Structure in the Coding Region of Dengue Virus Type 2 Directs Translation Start Codon Selection and Is Required for Viral Replication". J Virol. 80 (5): 2170–2182. doi:10.1128/JVI.80.5.2170-2182.2006. PMC 1395379. PMID 16474125.
- ^ Bidet, Katell; Garcia-Blanco, Mariano A. (1 September 2014). "Flaviviral RNAs: weapons and targets in the war between virus and host". Biochemical Journal. 462 (2): 215–230. doi:10.1042/BJ20140456. ISSN 0264-6021. PMID 25102029.
- ^ Chapman, Erich G.; Costantino, David A.; Rabe, Jennifer L.; Moon, Stephanie L.; Wilusz, Jeffrey; Nix, Jay C.; Kieft, Jeffrey S. (18 April 2014). "The Structural Basis of Pathogenic Subgenomic Flavivirus RNA (sfRNA) Production". Science. 344 (6181): 307–310. Bibcode:2014Sci...344..307C. doi:10.1126/science.1250897. ISSN 0036-8075. PMC 4163914. PMID 24744377.
- ^ Funk, Anneke; Truong, Katherine; Nagasaki, Tomoko; Torres, Shessy; Floden, Nadia; Melian, Ezequiel Balmori; Edmonds, Judy; Dong, Hongping; Shi, Pei-Yong (1 November 2010). "RNA Structures Required for Production of Subgenomic Flavivirus RNA". Journal of Virology. 84 (21): 11407–11417. doi:10.1128/JVI.01159-10. ISSN 0022-538X. PMC 2953152. PMID 20719943.
- ^ Chang, Ruey-Yi; Hsu, Ta-Wen; Chen, Yen-Lin; Liu, Shu-Fan; Tsai, Yi-Jer; Lin, Yun-Tong; Chen, Yi-Shiuan; Fan, Yi-Hsin (27 September 2013). "Japanese encephalitis virus non-coding RNA inhibits activation of interferon by blocking nuclear translocation of interferon regulatory factor 3". Veterinary Microbiology. 166 (1–2): 11–21. doi:10.1016/j.vetmic.2013.04.026. PMID 23755934.
- ^ Moon, Stephanie L.; Anderson, John R.; Kumagai, Yutaro; Wilusz, Carol J.; Akira, Shizuo; Khromykh, Alexander A.; Wilusz, Jeffrey (1 November 2012). "A noncoding RNA produced by arthropod-borne flaviviruses inhibits the cellular exoribonuclease XRN1 and alters host mRNA stability". RNA. 18 (11): 2029–2040. doi:10.1261/rna.034330.112. ISSN 1355-8382. PMC 3479393. PMID 23006624.
- ^ Clarke, B. D.; Roby, J. A.; Slonchak, A.; Khromykh, A. A. (3 August 2015). "Functional non-coding RNAs derived from the flavivirus 3′ untranslated region". Virus Research. Special Issue: Functions of the ends of positive strand RNA virus genomes. 206: 53–61. doi:10.1016/j.virusres.2015.01.026. PMID 25660582.
- ^ Kuno G, Chang GJ, Tsuchiya KR, Karabatsos N, Cropp CB (1998). "Phylogeny of the genus Flavivirus". J Virol. 72 (1): 73–83. doi:10.1128/JVI.72.1.73-83.1998. PMC 109351. PMID 9420202.
- ^ Gaunt MW, Sall AA, de Lamballerie X, Falconar AK, Dzhivanian TI, Gould EA (2001). "Phylogenetic relationships of flaviviruses correlate with their epidemiology, disease association and biogeography". J Gen Virol. 82 (8): 1867–1876. doi:10.1099/0022-1317-82-8-1867. PMID 11457992.
- ^ Cook S, Holmes EC (2006). "A multigene analysis of the phylogenetic relationships among the flaviviruses (Family: Flaviviridae) and the evolution of vector transmission". Arch Virol. 151 (2): 309–325. doi:10.1007/s00705-005-0626-6. PMID 16172840.
- ^ Conway MJ (2015). "Identification of a flavivirus sequence in a marine arthropod". PLOS ONE. 10 (12): e0146037. Bibcode:2015PLoSO..1046037C. doi:10.1371/journal.pone.0146037. PMC 4699914. PMID 26717191.
- ^ Moureau, Gregory; Cook, Shelley; Lemey, Philippe; Nougairede, Antoine; Forrester, Naomi L.; Khasnatinov, Maxim; Charrel, Remi N.; Firth, Andrew E.; Gould, Ernest A.; De Lamballerie, Xavier (2015). "New Insights into Flavivirus Evolution, Taxonomy and Biogeographic History, Extended by Analysis of Canonical and Alternative Coding Sequences". PLOS ONE. 10 (2): e0117849. Bibcode:2015PLoSO..1017849M. doi:10.1371/journal.pone.0117849. PMC 4342338. PMID 25719412.
- ^ Galli M, Bernini F, Zehender G (July 2004). "Alexander the Great and West Nile virus encephalitis". Emerging Infect. Dis. 10 (7): 1330–2, author reply 1332–3. doi:10.3201/eid1007.040396. PMID 15338540.
- ^ West, Christy (8 February 2010). "Different West Nile Virus Genetic Lineage Evolving?". The Horse. Retrieved 10 February 2010. 2009년 12월 5일–9일 헝가리 스젠트 이스tvan 대학의 오르소야 쿠타시(DVM)가 발표한 2009 미국 에콰인 실무자 협회에서 발표한 성명에서 인용된다.
- ^ Venter M, Human S, van Niekerk S, Williams J, van Eeden C, Freeman F (August 2011). "Fatal neurologic disease and abortion in mare infected with lineage 1 West Nile virus, South Africa". Emerging Infect. Dis. 17 (8): 1534–6. doi:10.3201/eid1708.101794. PMC 3381566. PMID 21801644.
- ^ St Leger J, Wu G, Anderson M, Dalton L, Nilson E, Wang D (2011). "West Nile virus infection in killer whale, Texas, USA, 2007". Emerging Infect. Dis. 17 (8): 1531–3. doi:10.3201/eid1708.101979. PMC 3381582. PMID 21801643.
- ^ Karan, Liudmila S.; Ciccozzi, Massimo; Yakimenko, Valerii V.; Presti, Alessandra Lo; Cella, Eleonora; Zehender, Gianguglielmo; Rezza, Giovanni; Platonov, Alexander E. (2014). "The deduced evolution history of Omsk hemorrhagic fever virus". Journal of Medical Virology. 86 (7): 1181–1187. doi:10.1002/jmv.23856. PMID 24259273. S2CID 36929638.
- ^ "International Committee on Taxonomy of Viruses (ICTV)". talk.ictvonline.org. Retrieved 16 November 2020.
- ^ Robin Y, Cornet M, Le Gonidec G, Chateau R, Heme G (1978). "[Kedougou virus (Ar D14701): a new Arbovirus ("Flavivirus") isolated in Senegal (author's transl)]". Ann Microbiol (Paris). 129 (2): 239–44. PMID 677616.CS1 maint: 여러 이름: 작성자 목록(링크)
- ^ Jansen van Vuren P, Parry R, Khromykh AA, Paweska JT (2021). "A 1958 Isolate of Kedougou Virus (KEDV) from Ndumu, South Africa, Expands the Geographic and Temporal Range of KEDV in Africa". Viruses. 13 (7). doi:10.3390/v13071368. PMC 8309962. PMID 34372574.CS1 maint: 여러 이름: 작성자 목록(링크)
- ^ van den Hurk, Andrew F.; Suen, Willy W.; Hall, Roy A.; O'Brien, Caitlin A.; Bielefeldt-Ohmann, Helle; Hobson-Peters, Jody; Colmant, Agathe M. G. (2016). "A newly discovered flavivirus in the yellow fever virus group displays restricted replication in vertebrates". Journal of General Virology. 97 (5): 1087–1093. doi:10.1099/jgv.0.000430. PMID 26878841. S2CID 43127614.
- ^ Jump up to: a b c d e Parry, Rhys; Asgari, Sassan (15 July 2019). "Discovery of Novel Crustacean and Cephalopod Flaviviruses: Insights into the Evolution and Circulation of Flaviviruses between Marine Invertebrate and Vertebrate Hosts". Journal of Virology. 93 (14). doi:10.1128/JVI.00432-19. PMC 6600200. PMID 31068424.
- ^ Thisyakorn, U. (2014). "Latest developments and future directions in dengue vaccines". Therapeutic Advances in Vaccines. 2 (1): 3–9. doi:10.1177/2051013613507862. PMC 3991153. PMID 24757522.
- ^ Yauch, Lauren E. (2014). Dengue Virus Vaccine Development. Advances in Virus Research. 88. pp. 315–372. doi:10.1016/B978-0-12-800098-4.00007-6. ISBN 9780128000984. PMID 24373316.
- ^ "WHO Questions and Answers on Dengue Vaccines". WHO.int. Retrieved 1 October 2016.
추가 읽기
- Kuno G, Chang GJ, Tsuchiya KR, Karabatsos N, Cropp CB (January 1998). "Phylogeny of the genus Flavivirus". J Virol. 72 (1): 73–83. doi:10.1128/JVI.72.1.73-83.1998. PMC 109351. PMID 9420202.
- Zanotto, P. M.; Gould, E. A.; Gao, G. F.; Harvey, P. H.; Holmes, E. C. (1996). "Population dynamics of flaviviruses revealed by molecular phylogenies". Proceedings of the National Academy of Sciences. 93 (2): 548–553. Bibcode:1996PNAS...93..548Z. doi:10.1073/pnas.93.2.548. PMC 40088. PMID 8570593.
- Kalitzky, Matthias (2006). Molecular Biology of the Flavivirus. Wymondham: Horizon Bioscience. ISBN 978-1-904933-22-9.
- Shi, Pei-Yong (2012). Molecular Virology and Control of Flaviviruses. Norfolk, UK: Caister Academic Press. ISBN 978-1-904455-92-9.
- Murray, Catherine L.; Jones, Christopher T.; Rice, Charles M. (2008). "Architects of assembly: roles of Flaviviridae non-structural proteins in virion morphogenesis". Nature Reviews Microbiology. 6 (9): 699–708. doi:10.1038/nrmicro1928. PMC 2764292. PMID 18587411.
외부 링크
- 미생물바이테스: 플라미비루스
- 노바티스 열대병연구소(NITD) – 노바티스 열대병연구소(NITD)의 뎅기열 연구
- Dengueinfo.org – 뎅기 바이러스 게놈 데이터 보관소
- 바이러스 영역: 플라비바이러스
- ViPR(Virus Pathogen Database and Analysis Resources) : 플라비비비르과
- 플라비바이러스 3'의 Rfam 엔트리에 들어감UTR 스템 루프 IV
- 플라비바이러스 DB 요소의 Rfam 항목
- Plavivirus 3' UTR cis 작동 복제 요소(CRE)에 대한 Rfam 항목
- 일본뇌염바이러스(JEV) 머리핀 구조 Rfam 진입