로제타@home
Rosetta@home | |
| 개발자 | 워싱턴 대학교 베이커 연구실; 로제타 커먼즈 |
|---|---|
| 초기 릴리즈 | 2005년 10월 6일 전() |
| 안정적 해제 | 로제타: 4.20 / 2020년 5월 1일; 전 로제타 미니: 3.78 / 2017년 10월 3일; 전 안드로이드용 로제타: 4.20 / 2020년 5월 1일; 전( |
| 개발여부 | 활동적인 |
| 운영 체제 | Windows, MacOS, Linux, Android |
| 플랫폼 | BOINC |
| 면허증 | 교육용 및 비영리용 전용 프리웨어,[1] 상업용 라이센스 제공[2] |
| 평균실적 | 487,946 GigaFLOPS[3] |
| 활성 사용자 | 36,726 |
| 총 사용자 수 | 1,363,584[4] |
| 활성 호스트 | 249,673 |
| 총 호스트 수 | 529,112 |
| 웹사이트 | boinc |
로제타@home은 워싱턴 대학교 베이커 연구소가 운영하는 BOINC(Berkeley Open Infrastructure for Network Computing) 플랫폼의 단백질 구조 예측을 위한 분산 컴퓨팅 프로젝트다.Rosetta@home은 2020년 9월 19일 현재 평균 487,946 GigaFLOPS의 약 5만 5천 개의 능동적 자발적 컴퓨터 처리로 단백질-단백질 도킹 예측과 새로운 단백질 설계를 목표로 한다.[5]Rosetta@home 비디오게임인 Poldit은 크라우드소싱 접근으로 이러한 목표에 도달하는 것을 목표로 한다.이 프로젝트의 많은 부분이 단백질학 방법의 정확성과 견고성을 향상시키기 위한 기초적인 연구를 지향하고 있지만, Rosetta@home은 말라리아, 알츠하이머병, 그리고 다른 병리학에 대한 연구도 하고 있다.[6]
모든 BOINC 프로젝트와 마찬가지로, Rosetta@home은 자원 봉사자들의 컴퓨터에서 나오는 유휴 컴퓨터 처리 자원을 사용하여 개별 작업장에 대한 계산을 수행한다.완료된 결과는 중앙 프로젝트 서버로 전송되어 검증되고 프로젝트 데이터베이스에 동화된다.이 프로젝트는 교차 플랫폼으로, 매우 다양한 하드웨어 구성으로 실행된다.사용자는 Rosetta@home 화면 보호기를 통해 개별 단백질 구조 예측의 진행 상황을 볼 수 있다.
질병 관련 연구 외에도, Rosetta@home 네트워크는 구조 생물정보학의 새로운 방법에 대한 시험 프레임워크의 역할을 한다.그런 방법은 Rosetta@home의 크고 다양한 자원봉사 컴퓨터 세트에서 충분히 개발되고 안정성이 입증된 후에 RosettaDock 또는 Human Proteome Polding Project와 Microbiome Immunity Project와 같은 다른 Rosetta 기반 애플리케이션에 사용된다.Rosetta@home에서 개발된 새로운 방법에 대한 두 가지 중요한 테스트는 단백질 구조 예측 기법의 비판적 평가(CASP)와 상호작용 예측의 비판적 평가(CAPRI) 실험, 단백질 구조 예측 및 단백질-단백질 도키에서 기술 상태를 평가하는 격년별 실험이다.각각 ng 예측.Rosetta@home은 지속적으로 가장 중요한 도킹 예측 변수들 중 하나이며, 이용 가능한 최고의 3차 구조 예측 변수들 중 하나이다.[7]
사스-CoV-2에 의한 COVID-19 대유행과의 싸움에 참가하려는 신규 사용자들이 몰리면서 로제타@home은 2020년 3월 28일 현재 컴퓨팅 파워를 1.7PetaFlops까지 끌어올렸다.[8][9]2020년 9월 9일, Rosetta@home 연구원들은 사스-CoV-2에 대항하는 10개의 강력한 항바이러스 후보들을 기술한 논문을 발표했다.Rosetta@home은 이 연구에 기여했고 이 항바이러스 후보자들은 2022년 초에 시작될지도 모르는 1단계 임상시험을 향해 가고 있다.[10][11][12][13]로제타@home 팀에 따르면, 로제타 자원봉사자들이 나노입자 백신 개발에 기여했다고 한다.[10]이 백신은 2021년 6월 임상 1상을 시작한 아이코사백스와 [14]SK바이오사이언스가 개발 중인 국내 임상 3상 허가를 받은 GBP510에 의해 IVX-411로 알려져 있다.[15][16]
단백질디자인연구소(IPD)에서 처음 만들어져 2019년 1월 논문에 발표된 암약 후보물질인 NL-201은 2021년 5월 IPD의 스핀오프(spin-off)인 네오루킨 테라퓨틱스의 지원으로 1단계 인간 임상시험을 시작했다.[17][18]Rosetta@home은 NL-201의 개발에 역할을 했고 단백질 디자인 검증에 도움이 되는 "전방 폴딩" 실험에 기여했다.[19]
컴퓨팅 플랫폼
Rosetta@home 애플리케이션과 BOINC 분산 컴퓨팅 플랫폼은 운영 체제 Windows, Linux 및 MacOS에서 사용할 수 있으며, BOINC는 FreeBSD와 같은 다른 여러 운영 체제에서도 실행된다.[20]로제타@home에 참여하려면 최소 500MHz의 클럭 속도, 200메가바이트의 여유 디스크 공간, 512메가바이트의 물리적 메모리, 인터넷 연결성을 갖춘 중앙처리장치(CPU)가 필요하다.[21]2016년 7월 20일 현재 로제타 미니 어플리케이션의 버전은 3.73이다.[22]현재 권장되고 있는 BOINC 프로그램 버전은 7.6.22.[20] 표준 하이퍼텍스트 전송 프로토콜(HTTP) (포트 80)은 사용자의 BOINC 클라이언트와 워싱턴 대학의 Rosetta@home 서버 간의 통신에 사용되며, 암호 교환 시 HTTPS (포트 443)가 사용된다.BOINC 클라이언트의 원격 및 로컬 제어는 포트 31416과 포트 1043을 사용하며, 방화벽 뒤에 있을 경우 특별히 차단을 해제해야 할 수 있다.[23]개별 단백질에 대한 데이터를 담은 워크유닛은 워싱턴 대학의 베이커 연구소에 위치한 서버에서 자원봉사자의 컴퓨터로 배포되며, 이 워크유닛은 할당된 단백질에 대한 구조 예측을 계산한다.주어진 단백질에 대한 중복된 구조 예측을 방지하기 위해 각 작업 단위는 무작위 시드 번호로 초기화된다.이것은 각 예측에 단백질의 에너지 경관을 따라 독특한 하강 궤적을 제공한다.[24]Rosetta@home의 단백질 구조 예측은 주어진 단백질의 에너지 환경에서 전지구적 최소값의 근사치들이다.그 전지구적 최소치는 단백질의 가장 에너지적으로 유리한 순응, 즉 그 토착 상태를 나타낸다.
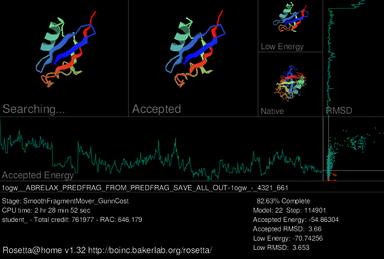
Rosetta@home GUI(Graphical User Interface)의 주요 특징은 시뮬레이션 단백질 접힘 과정 동안 현재 작업 유닛의 진행 상황을 보여주는 스크린세이버다.현재 화면 보호기의 왼쪽 상단에서 대상 단백질은 가장 낮은 에너지 구조를 찾는 과정에서 다른 모양(적합성)을 채택하는 것으로 나타난다.바로 오른쪽에 묘사된 것이 가장 최근에 받아들여진 구조다.오른쪽 상단에 현재 디코이의 가장 낮은 에너지 순응이 표시되며, 그 아래는 단백질의 진짜 또는 고유 구조(이미 결정된 경우)이다.세 개의 그래프가 스크린세이버에 포함되어 있다.중간 부근에는 수용된 모델의 열역학적 자유 에너지 그래프가 표시되며, 수용된 모델이 변함에 따라 변동한다.수용된 모형이 원시 모형과 구조적으로 얼마나 유사한지 측정한 RMSD(Root-Mean-Square Deviation, RMSD)의 그래프가 오른쪽 끝에 표시된다.승인된 에너지 그래프의 오른쪽과 RMSD 그래프 아래에서는 이 두 함수의 결과를 사용하여 에너지를 생성한다.모델이 점진적으로 정제됨에 따라 RMSD 그림.[25]
모든 BOINC 프로젝트와 마찬가지로 Rosetta@home은 사용자의 컴퓨터 백그라운드에서 호스트 운영 체제의 계정에 로그인하거나 로그인하기 전에 유휴 컴퓨터 전원을 사용하여 실행된다.이 프로그램은 다른 응용 프로그램이 필요로 하는 대로 CPU로부터 자원을 해방시켜 정상적인 컴퓨터 사용에 영향을 받지 않게 한다.많은 프로그램 설정은 프로그램이 사용할 수 있는 CPU 리소스의 최대 비율(지속 용량으로 실행되는 컴퓨터의 전력 소비 또는 열 생산을 제어하는 데 사용), 프로그램을 실행할 수 있는 시간 및 기타 등을 포함하여 사용자 계정 기본 설정을 통해 지정할 수 있다.[citation needed]
Rosetta@home 네트워크에서 실행되는 소프트웨어인 Rosetta는 Fortran에서 작성된 원래 버전에서 허용하는 것보다 더 쉬운 개발이 가능하도록 C++로 다시 작성되었다.이 새로운 버전은 객체 지향적이며, 2008년 2월 8일에 발매되었다.[22][26]로제타 코드의 개발은 로제타 커먼스가 한다.[27]이 소프트웨어는 학계에 자유롭게 허가되며 제약회사들이 유료로 이용할 수 있다.[27]
프로젝트 유의성
게놈 염기서열 분석 프로젝트의 확산으로 과학자들은 세포 내에서 기능을 수행하는 많은 단백질의 아미노산 염기서열, 즉 1차 구조를 유추할 수 있다.합리적인 약물 설계에 있어 단백질의 기능과 도움을 더 잘 이해하기 위해서, 과학자들은 단백질의 3차원 구조를 알 필요가 있다.
단백질 3D 구조는 현재 X선 결정학이나 핵자기공명(NMR) 분광학을 통해 실험적으로 결정된다.이 과정은 느리고(처음 단백질을 결정짓는 방법을 알아내는 데 몇 주 또는 심지어 몇 달이 걸릴 수 있다), 비용이 많이 든다(단백질당 미화 약 10만 달러).[28]불행히도, 새로운 시퀀스가 발견되는 속도는 구조 결정 속도를 훨씬 초과한다 – 국립생명공학정보센터(NCBI) 비중복(nr) 단백질 데이터베이스에서 이용할 수 있는 7,400,000개 이상의 단백질 시퀀스 중에서, 5만 2천 개 미만의 단백질의 3D 구조가 해결되어 프로테이에 축적되었다.n 단백질 구조 정보의 주요 보고인 데이터 뱅크.[29]로제타@home의 주요 목표 중 하나는 기존 방법과 같은 정확도로 단백질 구조를 예측하는 것이지만, 시간과 비용이 현저히 적게 드는 방식으로 예측하는 것이다.로제타@home은 X선 결정학, NMR 분광학 등 전통적 기법으로 분석하기 유난히 어려운 막단백질(예: G단백질결합수용체(GPCR))[30]의 구조와 도킹 결정법도 개발하지만 현대 의약품의 대다수를 대표한다.[citation needed]
단백질 구조 예측의 진척도는 전 세계 연구자들이 단백질 아미노산 염기서열에서 단백질 구조를 도출하려고 시도하는 2년마다 실시하는 단백질 구조 예측 기법의 임계평가(CASP) 실험에서 평가된다.때때로 경쟁적인 이 실험에서 높은 점수를 받은 그룹은 단백질 구조 예측에서 기술의 상태가 무엇인지에 대한 사실상의 표준 베어러로 간주된다.로제타@home의 기반이 되는 프로그램인 로제타는 2002년 CASP5 이후 사용되어 왔다.2004년 CASP6 실험에서, 로제타는 CASP 표적 T0281을 위해 제출된 모델에서 원자 수준의 분해능에 가까운 단백질 구조 예측을 최초로 생산함으로써 역사를 만들었다.[31]Ab initio 모델링은 구조 호몰로학의 정보를 사용하지 않고 단백질 내의 순서에 따른 호몰로학과 물리적 상호작용을 모델링하는 정보에 의존해야 하기 때문에 단백질 구조 예측의 특히 어려운 범주로 간주된다.Rosetta@home은 2006년부터 CASP에 사용되어 왔으며, CASP7의 모든 구조 예측 범주에서 최상위 예측 변수 중 하나였다.[32][33][34]이러한 높은 품질의 예측은 Rosetta@home 자원봉사자들이 이용할 수 있는 컴퓨팅 능력에 의해 가능해졌다.[35]컴퓨팅 파워가 증가하면 Rosetta@home은 순응 공간의 더 많은 영역(단백질이 가정할 수 있는 가능한 형태)을 샘플링할 수 있게 되는데, 레빈탈의 역설에 따르면, 단백질 길이에 따라 기하급수적으로 증가할 것으로 예측된다.[citation needed]
로제타@home은 단백질-단백질 도킹 예측에도 사용되는데, 복합 단백질, 즉 2분위 구조의 구조를 결정한다.이러한 유형의 단백질 상호작용은 항원-항원-항원-항원-항원 결합과 세포 수출입 등 많은 세포 기능에 영향을 미친다.이러한 상호작용을 결정하는 것은 약물 설계에 매우 중요하다.Rosetta는 CASP 게이지가 단백질 구조 예측에서 어떻게 진행되는지와 유사한 단백질 도킹장의 상태를 평가하는 CAPRI(Critical Assessment of Interactivity) 실험에서 사용된다.로제타@home의 프로젝트 자원봉사자들이 이용할 수 있게 만든 컴퓨팅 파워는 도킹 예측이 가장 정확하고 완벽한 CAPRI에서의 로제타의 성과에 주요 요인으로 꼽혀 왔다.[36]
2008년 초, 로제타는 자연에서 관찰된 적이 없는 함수를 가진 단백질을 계산적으로 설계하기 위해 사용되었다.[37]이것은 부분적으로 자연적인 형태에 비해 효소 활성도가 개선된 단백질의 계산적 설계를 원래 기술한 2004년도의 유명한 논문의 철회에서 영감을 받았다.[38]단백질이 어떻게 만들어졌는지를 기술한 데이비드 베이커 그룹의 2008년 연구 논문은 사용 가능한 컴퓨팅 자원에 대해 Rosetta@home을 인용했으며, 이 단백질 설계 방법에 대한 중요한 개념 증명서를 나타냈다.[37]이러한 유형의 단백질 설계는 약물 발견, 녹색 화학 및 생물 매개에 향후 적용될 수 있다.[37]
단백질 구조 예측, 도킹, 디자인에 관한 기초 연구 외에도, Rosetta@home은 즉각적인 질병 관련 연구에도 사용된다.[39]데이비드 베이커의 로제타@home 저널에 수많은 소규모 연구 프로젝트가 설명되어 있다.[40]2014년 2월 현재 포럼에서는 최근 출판물에 대한 정보와 작품에 대한 간략한 설명이 업데이트되고 있다.[41]포럼 실타래는 2016년 이후 더 이상 사용되지 않으며, 연구 관련 뉴스는 프로젝트의 종합 뉴스 섹션에서 확인할 수 있다.[42]
알츠하이머병
로제타 소프트웨어 제품군의 구성 요소인 로제타디자인은 아밀로이드와 유사한 섬유질을 만들 가능성이 가장 높은 아밀로이드 유발 단백질의 부위를 정확하게 예측하기 위해 사용되었다.[43]로제타디자인(RosettaDesign)[44]은 관심 단백질의 헥사펩타이드(6개의 아미노산-길이 조각)를 채취해 알려진 피브릴이 헥사프타드를 형성하는 구조와 유사한 구조와 가장 낮은 에너지 매치를 선택함으로써 무작위 단백질에 비해 섬유질이 형성될 확률이 두 배 높은 펩타이드(펩타이드)를 식별할 수 있었다.로제타@home은 알츠하이머병을 유발하는 것으로 가정된 섬유질 형성 단백질인 아밀로이드 베타 구조를 예측하기 위해 같은 연구에서 사용되었다.[45]비록 그것이 질병을 예방할 수 있을지는 알 수 없지만, 아직 발표되지 않은 결과들이 로제타가 고안한 단백질에 대해 생산되었다.[46]
탄저균
로제타의 또 다른 성분인 로제타독은 실험 방법과 함께 탄저균 독소를 구성하는 세 가지 단백질인 치명적 인자(LF), 부종 인자(EF), 보호 항원(PA) 사이의 상호작용을 모형화하는 데 사용되었다.[47][48][49]컴퓨터 모델은 LF와 PA 사이의 도킹을 정확하게 예측하여 LF-PA 복합체에 관련된 각 단백질의 영역을 규명하는데 도움을 주었다.이러한 통찰력은 결국 탄저균 백신을 개선시키는 연구에 사용되었다.[50][51]
헤르페스 심플렉스 바이러스 1
RosettaDock은 항체(임무노글로불린 G)와 감기감염 바이러스에 의해 발현된 표면 단백질인 헤르페스 심플렉스 바이러스 1(HSV-1) 사이의 도킹을 모형화하는 데 사용되어 항바이러스 항체를 저하시키는 역할을 하였다.RosettaDock이 예측한 단백질 복합체는 특히 얻기 어려운 실험 모델과 밀접하게 일치하여, 연구자들은 도킹 방법이 단백질-단백질 인터페이스 모델링으로 X선 결정학이 가지고 있는 몇 가지 문제를 해결할 수 있는 잠재력을 가지고 있다고 결론을 내렸다.[52]
HIV
빌 & 멜린다 게이츠 재단이 1,940만 달러의 보조금을 지원받은 연구의 일부로서, 로제타@[53]home은 인간 면역결핍 바이러스(HIV)를 위한 여러 개의 가능한 백신을 설계하는 데 사용되어 왔다.[54][55]
말라리아
세계보건대 과제와 관련된 연구에서 [56]로제타는 아노펠레스 감비아를 뿌리뽑거나 다른 방법으로 모기가 말라리아를 전염시킬 수 없게 만드는 새로운 단백질을 계산적으로 디자인하는데 이용되어 왔다.[57]단백질을 모델링하고 변형할 수 있는 능력-특히 DNA 상호작용은 호밍 엔도뉴클레아제처럼 유전자 치료에서 중요한 역할을 하는 로제타(Rosetta)와 같은 계산 단백질 설계 방법을 제공한다(가능한 암 치료법 포함).[39][58]
COVID-19
로제타 분자 모델링 스위트는 최근 사스-CoV-2 스파이크 단백질의 원자 규모 구조를 정확하게 예측하기 위해 실험실에서 측정하기 몇 주 전에 사용되었다.[59]2020년 6월 26일, 이 프로젝트는 연구실에서 사스-CoV-2 바이러스를 중화시키는 항바이러스 단백질을 만드는 데 성공했으며, 이러한 실험적인 항바이러스제가 동물 실험에 최적화되고 있다고 발표했다.[60]
후속편에서는 9월 9일 사이언스지에 10개의 사스-CoV-2 미니프로테인 억제제를 설명하는 논문이 발표되었다.이러한 억제제 중 두 가지인 LCB1과 LCB3는 SARS-CoV-2에 대해 어금니 및 질량 기준에서 개발되고 있는 최고의 단클론 항체보다 몇 배 더 강력하다.또한, 연구는 이러한 억제제가 높은 온도에서 활동을 유지하고 항체보다 20배 작으며, 따라서 잠재 중화 부위가 20배 더 많아져 국소 투여 약물의 잠재적 효능을 증가시킨다는 것을 시사한다.억제제의 작은 크기와 높은 안정성으로 인해 불침투가 가능한 젤 제형에 적합하거나 호흡기에 직접 투여할 분말로서 적합할 것으로 기대된다.연구원들은 앞으로 몇 달 안에 이러한 억제제를 치료법과 예방약으로 개발하기 위해 노력할 것이다.[11]2021년 7월 현재 이들 항바이러스 후보자는 2022년 초 임상시험을 시작할 것으로 예측됐으며, 빌&멜린다 게이츠 재단으로부터 임상 전·초기 임상시험을 위한 자금을 지원받았다.[13]동물 실험에서, 이러한 항바이러스 후보자들은 알파, 베타, 감마를 포함한 여러 가지 우려에 대해 효과적이었다.[13][61][62]
Rosetta@home은 200만개가 넘는 SARS-CoV-2 스파이크 바인딩 단백질을 검사하는데 도움을 주었고, 따라서 이 연구에 기여했다.[63][64]
단백질 디자인 연구소의 Rosetta@home 팀에 따르면, Rosetta@home 자원봉사자들은 항바이러스제 후보물질[11] 개발과 단백질 나노입자 백신 개발에 기여했다.[65]IVX-411 백신은 이미 아이코사백스가[14] 운영하는 임상 1상 시험 중이고, GBP510이라는 이름으로 다른 제조사에 허가받은 동일한 백신이 국내에서도 SK바이오사이언스가 운영하는 임상 3상 시험 허가를 받았다.[16][15]후보 항바이러스제 또한 임상 1상 시험으로 가고 있다.[10]
암
Rosetta@home 연구진은 수용기의 알파 서브유닛과 상호작용하지 않는 Neoleukin-2/15라는 IL-2 수용체 작용제를 설계했다.그러한 면역 신호 분자는 암 치료에 유용하다.자연적인 IL-2는 알파 서브 유닛과의 상호작용 때문에 독성을 가지고 있지만, 적어도 동물 모델에서는 설계한 단백질이 훨씬 더 안전하다.[17]Rosetta@home은 설계를 검증하는 데 도움이 되는 "전방 접기 실험"에 기여했다.[19]
데이비드 베이커는 뉴요커의 2020년 9월 특집 기사에서 Neoleukin-2/15가 인간 임상실험을 "올해는 더 늦을 것"이라고 말했다.네오루킨-2/15는 베이커 연구소의 스핀오프 회사인 네오루킨에 의해 개발되고 있다.[66]네오루킨은 2020년 12월, 네오루킨-2/15의 추가 개발인 NL-201 임상 1상을 시작하기 위해 식품의약품안전청에 '조사적 신약' 신청서를 제출할 것이라고 Neoleukin은 말했다.비슷한 신청서가 호주에서 제출되었고 네오루킨은 임상 1상에 120명의 참가자를 등록하기를 희망하고 있다.[67]1단계 인간 임상시험은 2021년 5월 5일에 시작되었다.[18]
개발이력 및 분기
원래 1998년 베이커 연구소에 의해 구조 예측에 대한 아비니티오 접근방식으로 소개된 로제타는 그 이후 여러 개발 흐름과 구별되는 서비스에 진출해 왔다.[68]로제타 플랫폼은 단백질의 아미노산 염기서열의 구조적인 "의미"를 해독하려고 시도하면서 로제타 스톤에서 그 이름을 유래했다.[69]로제타의 첫 등장 이후 7년여 만인 2005년 10월 6일 로제타@home 프로젝트가 공개되었다(즉,[22] 더 이상 베타 버전이 아니라고 발표).로제타의 초기 개발에 참여한 대학원생과 그 밖의 연구자들 중 많은 수가 그 후 다른 대학과 연구 기관으로 옮겨갔고, 그 후 로제타 프로젝트의 다른 부분을 강화했다.
로제타 디자인
로제타를 기반으로 한 단백질 설계에 대한 컴퓨팅 접근방식인 로제타디자인은 2000년 단백질 G의 접이식 경로 재설계 연구로 시작됐다.[70]2002년, RosettaDesign은 자연에 기록된 적이 없는 93-아미노산 긴 α/β 단백질인 Top7을 설계하기 위해 사용되었다.이 새로운 순응은 로제타가 X선 결정학에 의해 결정된 구조물의 1.2 å RMSD 이내로 예측했으며, 이는 비정상적으로 정확한 구조 예측을 나타낸다.[71]로제타와 로제타디자인은 사이언스지에 두 개의 긍정적인 문자를 촉발하는 이중적 접근법을 기술한 2002년 논문에 반영되고 240개 이상의 다른 과학 기사에 의해 인용되는 등, 그러한 길이의 새로운 단백질의 구조를 설계하고 정확하게 예측한 최초의 사람이 됨으로써 광범위한 인정을 받았다.[72][73][74]이 연구의 가시적 제품인 Top7은 2006년 10월에 RCSB PDB의 '이 달의 분자'로 특징지어졌다;[75] 각각의 코어(결과 60–79)의 중첩과 X선 결정 구조가 Rosetta@home 로고에 특징지어진다.[31]
데이비드 베이커의 연구실에서 박사 후 동료였던 브라이언 쿨먼은 현재 노스캐롤라이나 대학의 채플 힐 부교수로 온라인 서비스로 로제타디자인을 제공하고 있다.[76][77]
로제타독
로제타독은 2002년 제1차 CAPRI 실험에서 단백질-단백질 도킹 예측을 위한 베이커 실험실의 알고리즘으로 로제타 소프트웨어 제품군에 추가되었다.[78]그 실험에서 로제타독소는 연쇄상구균 화이성 엑소톡신 A와 T세포수용체 β-체인 사이의 도킹에 대해 높은 정확도를 예측했고, 포신 α-아밀라아제와 낙타 항체 사이의 복합체에 대한 중간 정확도를 예측했다.RosettaDock 방법은 7개 중에서 2개의 허용 가능한 정확한 예측만을 가능케 했지만, 이는 1차 CAPRI 평가에서 19개의 예측 방법 중 7위를 차지하기에 충분했다.[78]
워싱턴 대학교 재학 중 로제타독의 기초를 닦은 제프리 그레이가 존스 홉킨스 대학교에서 새로운 직책을 맡으면서 로제타독의 개발은 이후 CAPRI 라운드를 위해 두 갈래로 나뉘었다.베이커 연구소의 구성원들은 그레이가 없는 가운데 로제타독을 더욱 발전시켰다.두 버전은 사이드 체인 모델링, 디코이 선택 및 기타 영역에서 약간 차이를 보였다.[49][79]이러한 차이에도 불구하고 2차 CAPRI 평가에서 베이커 방법과 그레이 방법은 모두 30개의 예측 변수 그룹 중 각각 5위와 7위를 차지하며 좋은 성과를 거두었다.[80]제프리 그레이의 RosettaDock 서버는 비상업적 사용을 위한 무료 도킹 예측 서비스로 이용할 수 있다.[81]
2006년 10월, 로제타도크는 로제타@home에 통합되었다.이 방법은 단백질 백본만을 사용하여 빠르고 조잡한 도킹 모델 단계를 사용했다.그 뒤를 이어 서로 상대적인 상호작용을 하는 두 단백질의 방향과 단백질-단백질 인터페이스에서의 사이드체인 상호작용이 동시에 최적화되어 가장 낮은 에너지 순응을 찾아내는 느린 완전원자 미세화 단계가 이어졌다.[82]백본 유연성과 루프 모델링을 위해 수정된 폴드 트리 표현과 결합된 Rosetta@home 네트워크가 제공하는 엄청나게 향상된 컴퓨팅 파워는 3차 CAPRI 평가에서 63개의 예측 그룹 중 6위를 차지했다.[7][36]
로베타
로베타(Rosetta Beta) 서버는 베이커 연구소가 비상업적 초기화와 비교 모델링을 위해 제공하는 자동화된 단백질 구조 예측 서비스다.[83]2002년 CASP5 이후 2년마다 실시하는 CASP 실험에 자동화 예측 서버로 참여하여, 자동 서버 예측 부문에서 최고 성적을 거두었다.[84]Robetta는 이후 CASP6와 7에서 경쟁했는데, CASP6는 자동화된 서버와 인간 예측 변수 그룹들 사이에서 평균보다 더 좋은 성적을 거두었다.[34][85][86]COMEO3D 연속평가에 참여하기도 한다.
CASP6의 단백질 구조를 모델링할 때, Robetta는 먼저 VOLAST, PSI-BLAST, 3D-Jear를 사용하여 구조 호몰로그를 검색한 다음, Pfam 데이터베이스의 구조 패밀리에 그 순서를 매칭하여 목표 시퀀스를 개별 영역 또는 단백질의 독립적으로 접는 단위로 분석한다.그런 다음 구조 호몰로그가 있는 도메인은 "템플릿 기반 모델"(즉, 호몰로지 모델링) 프로토콜을 따른다.여기서 베이커 실험실의 사내 정렬 프로그램인 K*sync는 시퀀스 호몰로로그 그룹을 제작하며, 이들 각각은 로제타 데 노보 방법에 의해 모델화되어 디코이(가성 구조)를 제작한다.최종 구조물 예측은 저해상도 로제타 에너지 함수에 의해 결정되는 최저 에너지 모델을 취함으로써 선택된다.검출된 구조적 호몰로그가 없는 도메인의 경우, 최종 예측으로 생성된 디코이 집합에서 가장 낮은 에너지 모델을 선택하는 데노보 프로토콜을 따른다.그런 다음 이러한 도메인 예측을 함께 연결하여 단백질 내의 도메인 간, 3차 수준 상호작용을 조사한다.마지막으로, 사이드 체인 기여는 몬테카를로 순응 검색을 위한 프로토콜을 사용하여 모델링한다.[87]
CASP8에서 로베타는 로제타의 고해상도 올원자 미세화 방법을 사용하기 위해 증강되었는데,[88] 로베타의 부재가 CASP7의 로제타@home 네트워크보다 정확도가 떨어지는 주요 원인으로 꼽혔다.[35]CASP11에서는 GREMLIN이라는 관련 단백질의 잔류물을 공진화하여 단백질 접점 맵을 예측하는 방법이 추가되어 더 많은 노보 접기 성공이 가능해졌다.[89]
폴디트
2008년 5월 9일, 로제타@home 사용자들이 분산 컴퓨팅 프로그램의 인터랙티브 버전을 제안하자, 베이커 연구소는 로제타 플랫폼을 기반으로 한 온라인 단백질 구조 예측 게임인 폴디트를 공개 발매했다.[90]2008년[update] 9월 25일 현재, 폴디트는 59,000명 이상의 등록 사용자를 가지고 있다.[91]이 게임은 사용자에게 대상 단백질의 백본과 아미노산 사이드 체인을 보다 정력적으로 친화적인 순응으로 조작할 수 있는 일련의 제어장치(예: 흔들기, 흔들기, 재구축)를 제공한다.사용자는 솔리스트로서 개별적으로 또는 진화자로서 집합적으로 해결책을 작업할 수 있으며, 구조 예측을 개선할 때 어느 범주에서든 발생하는 지점을 파악할 수 있다.[92]
유사한 분산 컴퓨팅 프로젝트와의 비교
Rosetta@home의 연구 영역과 유사하지만 연구 접근방식이 다른 여러 분산형 컴퓨터 프로젝트가 있다.
폴딩@홈
단백질 연구에 관련된 모든 주요 분산 컴퓨팅 프로젝트 중 BOINC 플랫폼을 사용하지 않는 것은 Folding@home이 유일하다.[93][94][95]로제타@home과 폴딩@home 모두 알츠하이머병과 같은 단백질을 잘못 접는 질병을 연구하지만, 폴딩@home은 훨씬 더 독점적으로 한다.[96][97]폴딩@home은 단백질이 어떻게 그리고 왜 접히는지(또는 잠재적으로 잘못 접힌 후, 그 후에 종합하여 질병을 유발하는 것을 이해하기 위해 거의 독점적으로 모든 원자 분자 역학 모델을 사용한다.[98][99]즉, 폴딩@home의 강점은 단백질 접힘 과정을 모델링하는 것이고, Rosetta@home의 강점은 단백질 설계를 계산하고 단백질 구조와 도킹을 예측하는 것이다.
로제타@home의 결과 중 일부는 일부 폴딩@home 프로젝트의 근거로 사용된다.로제타는 가장 가능성이 높은 구조를 제공하지만 그것이 분자가 취하는 형태인지, 실행 가능한 형태인지는 확실하지 않다.이어 폴딩@home을 사용하여 Rosetta@home의 결과를 검증할 수 있으며, 추가된 원자 수준의 정보와 분자가 어떻게 모양을 변화시키는지에 대한 세부사항을 제공할 수 있다.[99][100]
두 프로젝트는 컴퓨팅 능력에서도 큰 차이를 보이며 다양성을 주관한다.중앙처리장치(CPU), 그래픽처리장치(GPU), (이전) PS3s의 호스트 베이스에서 평균 약 6,650테라FLOPS를 산출한 폴딩@home은 Rosetta@home보다 약 108배 높은 컴퓨팅 파워를 가지고 있다.[101][102]
월드 커뮤니티 그리드
세계 공동체 그리드의 하위 프로젝트인 인간 프로테오메 접기 프로젝트(HPF)의 1단계와 2단계 모두 로제타 프로그램을 사용하여 다양한 게놈의 구조적이고 기능적인 주석을 만들었다.[103][104]지금은 그것을 생물학자들을 위한 데이터베이스를 만드는 데 사용하고 있지만, 인간 프로테오메 접기 프로젝트의 수석 과학자 리처드 보니우는 박사학위를 취득하는 동안 데이비드 베이커의 실험실에서 로제타의 초기 개발에 적극적이었다.[105]HPF1, HPF2 및 Rosetta@home 간의 관계에 대한 자세한 내용은 Richard Bonneau의 웹사이트에서 확인할 수 있다.[106]
예측 변수@home
로제타@home과 마찬가지로 예측자@home은 단백질 구조 예측에 특화되었다.[107]Rosetta@home은 구조 예측을 위해 Rosetta 프로그램을 사용하는 반면, Optor@home은 dTASER 방법론을 사용했다.[108]2009년, 예측 변수@home은 폐쇄되었다.
BOINC의 다른 단백질 관련 분산 컴퓨팅 프로젝트로는 QMC@home, 도킹@home, SHIY@home, SIMAP, TANPAU 등이 있다. Ralph@home, Rosetta@home 알파 프로젝트로서, Rozetta@home은 BOINC에서 실행된다.[109]
자원봉사의 기부금
Rosetta@home은 연구를 위해 개별 프로젝트 구성원들이 기부한 컴퓨팅 파워에 의존한다.2020년[update] 3월 28일 현재 150개국에서 약 5만3천명의 사용자가 Rosetta@home의 활성 회원국으로, 총 1.7대 이상의 평균 성능을 위해 약 54,800대의 컴퓨터에서 유휴 프로세서 시간을 함께 제공했다.[102][110]
사용자에게는 기여도의 척도로 BOINC 크레딧이 부여된다.각 작업 단위에게 부여된 공제는 해당 작업 단위에서 생산된 미끼의 수에 해당 작업 단위에 대해 모든 컴퓨터 호스트가 제출한 미끼에 대한 평균 청구 크레딧을 곱한 것이다.이 사용자 지정 시스템은 표준 BOINC 클라이언트를 사용하는 사용자에게 부여된 신용과 최적화된 BOINC 클라이언트의 신용과 Windows 및 Linux 운영 체제에서 Rosetta@home을 실행하는 사용자 간의 신용 차이 간의 상당한 차이를 해결하기 위해 설계되었다.[111]Rosetta@home은 CPU 작업의 초당 크레딧이 대부분의 BOINC 프로젝트보다 낮다.[112]로제타@home은 총 신용도 면에서 BOINC 프로젝트 40여 개 중 13번째다.[113]
CASP 실험에 제출된 단백질 구조를 예측하는 Rosetta@home 사용자들은 그들의 결과와 관련하여 과학 간행물에서 인정받고 있다.[35]특정 작업 유닛의 최저 에너지 구조를 예측하는 사용자는 자신이 속한 팀과 함께 Rosetta@home 홈페이지에서 '오늘의 예측 변수'로 등장한다.[114]Rosetta@home 프로필을 만든 사용자 중에서 매일 무작위로 오늘의 사용자를 선택하여 홈페이지에도 올린다.[115]
참조
- ^ "Rosetta@home License Agreement". Boinc.bakerlab.org. Retrieved June 12, 2020.
- ^ "Rosetta: a molecular modeling software package". UW CoMotion – Collaborative Innovation Hub. Retrieved June 12, 2020.
- ^ "Rosetta@home".
- ^ "Rosetta@Home – Detailed stats BOINCstats/BAM!".
- ^ "Rosetta@home".
- ^ "What is Rosetta@home?". Rosetta@home forums. University of Washington. Archived from the original on September 13, 2008. Retrieved September 7, 2008.
- ^ a b Lensink MF, Méndez R, Wodak SJ (December 2007). "Docking and scoring protein complexes: CAPRI 3rd Edition". Proteins. 69 (4): 704–18. doi:10.1002/prot.21804. PMID 17918726. S2CID 25383642.
- ^ "Rosetta@home - Server Status "TeraFLOPS estimate"". Rosetta@home. March 25, 2020. Retrieved March 25, 2020.
- ^ "Rosetta@home Rallies a Legion of Computers Against the Coronavirus". HPCWire. March 24, 2020. Retrieved March 25, 2020.
- ^ a b c Rosetta@home (June 25, 2021). "The COVID-19 projects on our platform are headed into human clinical trials! Our amazing online volunteers have played a role in the development of a promising new vaccine as well as candidate antiviral treatments". Twitter. Retrieved June 26, 2021.
{{cite web}}: CS1 maint : url-status (링크) - ^ a b c Cao L, Goreshnik I, Coventry B, Case JB, Miller L, Kozodoy L, et al. (October 2020). "De novo design of picomolar SARS-CoV-2 miniprotein inhibitors". Science. 370 (6515): 426–431. Bibcode:2020Sci...370..426C. doi:10.1126/science.abd9909. PMC 7857403. PMID 32907861.
- ^ "Coronavirus update from David Baker. Thank you all for your contributions!". Rosetta@home. Rosetta@home. September 21, 2020. Retrieved September 23, 2020.
- ^ a b c "IPD Annual Report 2021" (PDF). Institute for Protein Design. July 14, 2021. Retrieved August 18, 2021.
{{cite web}}: CS1 maint : url-status (링크) - ^ a b "ANZCTR - Registration". anzctr.org.au. Retrieved July 9, 2021.
- ^ a b "S. Korea approves Phase III trial of SK Bioscience's COVID-19 vaccine". Reuters. August 10, 2021. Retrieved August 18, 2021.
- ^ a b Institute of Protein Design (August 10, 2021). "https://twitter.com/uwproteindesign/status/1425158796465213440". Twitter. Retrieved August 18, 2021.
{{cite web}}:외부 링크 위치title= - ^ a b Silva DA, Yu S, Ulge UY, Spangler JB, Jude KM, Labão-Almeida C, Ali LR, Quijano-Rubio A, Ruterbusch M, Leung I, Biary T, Crowley SJ, Marcos E, Walkey CD, Weitzner BD, Pardo-Avila F, Castellanos J, Carter L, Stewart L, Riddell SR, Pepper M, Bernardes GJ, Dougan M, Garcia KC, Baker D (January 2019). "De novo design of potent and selective mimics of IL-2 and IL-15". Nature. 565 (7738): 186–191. Bibcode:2019Natur.565..186S. doi:10.1038/s41586-018-0830-7. PMC 6521699. PMID 30626941.
- ^ a b "Neoleukin Therapeutics Announces Initiation of Phase 1 NL-201 Trial Neoleukin Therapeutics, Inc". investor.neoleukin.com. Retrieved June 22, 2021.
- ^ a b "Another publication in Nature describing the first de novo designed proteins with anti-cancer activity". Rosetta@home. January 14, 2020. Retrieved September 19, 2020.
- ^ a b "Download BOINC client software". BOINC. University of California. 2008. Retrieved December 1, 2008.
- ^ "Rosetta@home: Recommended System Requirements". Rosetta@home. University of Washington. 2008. Archived from the original on September 25, 2008. Retrieved October 7, 2008.
- ^ a b c "Rosetta@home: News archive". Rosetta@home. University of Washington. 2016. Retrieved July 20, 2016.
- ^ "Rosetta@home: FAQ (work in progress) (message 10910)". Rosetta@home forums. University of Washington. 2006. Retrieved October 7, 2008.
- ^ Kim DE (2005). "Rosetta@home: Random Seed (message 3155)". Rosetta@home forums. University of Washington. Retrieved October 7, 2008.
- ^ "Rosetta@home: Quick guide to Rosetta and its graphics". Rosetta@home. University of Washington. 2007. Archived from the original on September 24, 2008. Retrieved October 7, 2008.
- ^ Kim DE (2008). "Rosetta@home: Problems with minirosetta version 1.+ (Message 51199)". Rosetta@home forums. University of Washington. Retrieved September 7, 2008.
- ^ a b "Rosetta Commons". RosettaCommons.org. 2008. Archived from the original on September 15, 2008. Retrieved October 7, 2008.
- ^ Bourne PE, Helge W, eds. (2003). Structural Bioinformatics. Hoboken, NJ: Wiley-Liss. ISBN 978-0-471-20199-1. OCLC 50199108.
- ^ "Yearly Growth of Protein Structures". RCSB Protein Data Bank. 2021. Retrieved May 1, 2021.
- ^ Baker D (2008). "Rosetta@home: David Baker's Rosetta@home journal (message 55893)". Rosetta@home forums. University of Washington. Retrieved October 7, 2008.
- ^ a b "Rosetta@home: Research Overview". Rosetta@home. University of Washington. 2007. Archived from the original on September 25, 2008. Retrieved October 7, 2008.
- ^ Kopp J, Bordoli L, Battey JN, Kiefer F, Schwede T (2007). "Assessment of CASP7 predictions for template-based modeling targets". Proteins. 69 Suppl 8: 38–56. doi:10.1002/prot.21753. PMID 17894352. S2CID 31575350.
- ^ Read RJ, Chavali G (2007). "Assessment of CASP7 predictions in the high accuracy template-based modeling category". Proteins. 69 Suppl 8: 27–37. doi:10.1002/prot.21662. PMID 17894351. S2CID 33172629.
- ^ a b Jauch R, Yeo HC, Kolatkar PR, Clarke ND (2007). "Assessment of CASP7 structure predictions for template free targets". Proteins. 69 Suppl 8: 57–67. doi:10.1002/prot.21771. PMID 17894330. S2CID 38430899.
- ^ a b c Das R, Qian B, Raman S, et al. (2007). "Structure prediction for CASP7 targets using extensive all-atom refinement with Rosetta@home". Proteins. 69 Suppl 8: 118–28. doi:10.1002/prot.21636. PMID 17894356. S2CID 17470364.
- ^ a b Wang C, Schueler-Furman O, Andre I, et al. (December 2007). "RosettaDock in CAPRI rounds 6–12". Proteins. 69 (4): 758–63. doi:10.1002/prot.21684. PMID 17671979. S2CID 9028237.
- ^ a b c Jiang L, Althoff EA, Clemente FR, et al. (March 2008). "De novo computational design of retro-aldol enzymes". Science. 319 (5868): 1387–91. Bibcode:2008Sci...319.1387J. doi:10.1126/science.1152692. PMC 3431203. PMID 18323453.
- ^ Hayden EC (February 13, 2008). "Protein prize up for grabs after retraction". Nature. doi:10.1038/news.2008.569.
- ^ a b "Disease Related Research". Rosetta@home. University of Washington. 2008. Archived from the original on September 23, 2008. Retrieved October 8, 2008.
- ^ Baker D (2008). "Rosetta@home: David Baker's Rosetta@home journal". Rosetta@home forums. University of Washington. Retrieved September 7, 2008.
- ^ "Rosetta@home Research Updates". Boinc.bakerlab.org. Retrieved April 18, 2014.
- ^ "News archive". Rosetta@home. Retrieved May 10, 2019.
- ^ Kuhlman B, Baker D (September 2000). "Native protein sequences are close to optimal for their structures". Proceedings of the National Academy of Sciences of the United States of America. 97 (19): 10383–88. Bibcode:2000PNAS...9710383K. doi:10.1073/pnas.97.19.10383. PMC 27033. PMID 10984534.
- ^ Thompson MJ, Sievers SA, Karanicolas J, Ivanova MI, Baker D, Eisenberg D (March 2006). "The 3D profile method for identifying fibril-forming segments of proteins". Proceedings of the National Academy of Sciences of the United States of America. 103 (11): 4074–78. Bibcode:2006PNAS..103.4074T. doi:10.1073/pnas.0511295103. PMC 1449648. PMID 16537487.
- ^ Bradley P. "Rosetta@home forum: Amyloid fibril structure prediction". Rosetta@home forums. University of Washington. Retrieved September 7, 2008.
- ^ Baker D. "Rosetta@home forum: Publications on R@H's Alzheimer's work? (message 54681)". Rosetta@home forums. University of Washington. Retrieved October 8, 2008.
- ^ Wang C, Schueler-Furman O, Baker D (May 2005). "Improved side-chain modeling for protein–protein docking". Protein Science. 14 (5): 1328–39. doi:10.1110/ps.041222905. PMC 2253276. PMID 15802647.
- ^ Gray JJ, Moughon S, Wang C, et al. (August 2003). "Protein–protein docking with simultaneous optimization of rigid-body displacement and side-chain conformations". Journal of Molecular Biology. 331 (1): 281–99. doi:10.1016/S0022-2836(03)00670-3. PMID 12875852.
- ^ a b Schueler-Furman O, Wang C, Baker D (August 2005). "Progress in protein–protein docking: atomic resolution predictions in the CAPRI experiment using RosettaDock with an improved treatment of side-chain flexibility". Proteins. 60 (2): 187–94. doi:10.1002/prot.20556. PMID 15981249. S2CID 17672050.
- ^ Lacy DB, Lin HC, Melnyk RA, et al. (November 2005). "A model of anthrax toxin lethal factor bound to protective antigen". Proceedings of the National Academy of Sciences of the United States of America. 102 (45): 16409–14. Bibcode:2005PNAS..10216409L. doi:10.1073/pnas.0508259102. PMC 1283467. PMID 16251269.
- ^ Albrecht MT, Li H, Williamson ED, et al. (November 2007). "Human monoclonal antibodies against anthrax lethal factor and protective antigen act independently to protect against Bacillus anthracis infection and enhance endogenous immunity to anthrax". Infection and Immunity. 75 (11): 5425–33. doi:10.1128/IAI.00261-07. PMC 2168292. PMID 17646360.
- ^ Sprague ER, Wang C, Baker D, Bjorkman PJ (June 2006). "Crystal structure of the HSV-1 Fc receptor bound to Fc reveals a mechanism for antibody bipolar bridging". PLOS Biology. 4 (6): e148. doi:10.1371/journal.pbio.0040148. PMC 1450327. PMID 16646632.
- ^ Paulson, Tom (July 19, 2006). "Gates Foundation awards $287 million for HIV vaccine research". Seattle Post-Intelligencer. Retrieved September 7, 2008.
- ^ Liu Y, et al. (2007). "Development of IgG1 b12 scaffolds and HIV-1 env-based outer domain immunogens capable of eliciting and detecting IgG1 b12-like antibodies" (PDF). Global HIV Vaccine Enterprise. Archived from the original (PDF) on February 25, 2009. Retrieved September 28, 2008.
- ^ Baker D. "David Baker's Rosetta@home journal archives (message 40756)". Rosetta@home forums. University of Washington. Retrieved September 7, 2008.
- ^ "Homing Endonuclease Genes: New Tools for Mosquito Population Engineering and Control". Grand Challenges in Global Health. Retrieved September 7, 2008.
- ^ Windbichler N, Papathanos PA, Catteruccia F, Ranson H, Burt A, Crisanti A (2007). "Homing endonuclease mediated gene targeting in Anopheles gambiae cells and embryos". Nucleic Acids Research. 35 (17): 5922–33. doi:10.1093/nar/gkm632. PMC 2034484. PMID 17726053.
- ^ Ashworth J, Havranek JJ, Duarte CM, et al. (June 2006). "Computational redesign of endonuclease DNA binding and cleavage specificity". Nature. 441 (7093): 656–59. Bibcode:2006Natur.441..656A. doi:10.1038/nature04818. PMC 2999987. PMID 16738662.
- ^ "Rosetta's role in fighting coronavirus – Institute for Protein Design". Retrieved March 6, 2020.
- ^ "Coronavirus Research Update". Rosetta@home Official Twitter. Rosetta@Home. June 26, 2020. Retrieved June 27, 2020.
- ^ Case JB, Chen RE, Cao L, Ying B, Winkler ES, Johnson M, et al. (July 2021). "Ultrapotent miniproteins targeting the SARS-CoV-2 receptor-binding domain protect against infection and disease". Cell Host & Microbe. 29 (7): 1151–1161.e5. doi:10.1016/j.chom.2021.06.008. PMC 8221914. PMID 34192518.
- ^ Hunt AC, Case JB, Park YJ, Cao L, Wu K, Walls AC, et al. (July 2021). "Multivalent designed proteins protect against SARS-CoV-2 variants of concern". bioRxiv: 2021.07.07.451375. doi:10.1101/2021.07.07.451375. PMC 8282097. PMID 34268509.
- ^ "Big news out of @UWproteindesign: a new candidate treatment for #COVID19! More lab testing still needed. Thanks to all the volunteers who helped crunch data for this project!!". Rosetta@home Twitter. Rosetta@home Twitter. September 9, 2020. Retrieved September 19, 2020.
- ^ "De novo minibinders target SARS-CoV-2 Spike protein". Baker Lab. Baker Lab. September 9, 2020. Retrieved September 19, 2020.
- ^ Walls AC, Fiala B, Schäfer A, Wrenn S, Pham MN, Murphy M, et al. (November 2020). "Elicitation of Potent Neutralizing Antibody Responses by Designed Protein Nanoparticle Vaccines for SARS-CoV-2". Cell. 183 (5): 1367–1382.e17. doi:10.1016/j.cell.2020.10.043. PMC 7604136. PMID 33160446.
- ^ Hutson M (September 18, 2020). "Scientists Advance on One of Technology's Holy Grails". The New Yorker. Retrieved September 19, 2020.
- ^ "Neoleukin Therapeutics Announces Submission of Investigational New Drug Application for NL-201 De Novo Protein Immunotherapy Candidate for Cancer". Neoleukin Therapeutics. December 10, 2020. Retrieved December 10, 2020.
- ^ Simons KT, Bonneau R, Ruczinski I, Baker D (1999). "Ab initio protein structure prediction of CASP III targets using Rosetta". Proteins. Suppl 3: 171–76. doi:10.1002/(SICI)1097-0134(1999)37:3+<171::AID-PROT21>3.0.CO;2-Z. PMID 10526365.
- ^ "Interview with David Baker". Team Picard Distributed Computing. 2006. Archived from the original on February 18, 2009. Retrieved December 23, 2008.
- ^ Nauli S, Kuhlman B, Baker D (July 2001). "Computer-based redesign of a protein folding pathway". Nature Structural Biology. 8 (7): 602–05. doi:10.1038/89638. PMID 11427890. S2CID 18542707.
- ^ Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (November 2003). "Design of a novel globular protein fold with atomic-level accuracy". Science. 302 (5649): 1364–68. Bibcode:2003Sci...302.1364K. doi:10.1126/science.1089427. PMID 14631033. S2CID 1939390.
- ^ Jones DT (November 2003). "Structural biology. Learning to speak the language of proteins". Science. 302 (5649): 1347–48. doi:10.1126/science.1092492. PMID 14631028. S2CID 83109899.
- ^ von Grotthuss M, Wyrwicz LS, Pas J, Rychlewski L (June 2004). "Predicting protein structures accurately". Science. 304 (5677): 1597–99, author reply 1597–99. doi:10.1126/science.304.5677.1597b. PMID 15192202. S2CID 29787060.
- ^ "Articles citing: Kuhlman et al. (2003) 'Design of a novel globular protein fold with atomic-level accuracy'". ISI Web of Science. Retrieved July 10, 2008.
- ^ "October 2005 molecule of the month: Designer proteins". RCSB Protein Data Bank. Retrieved September 7, 2008.
- ^ "Kuhlman laboratory homepage". Kuhlman Laboratory. University of North Carolina. Retrieved September 7, 2008.
- ^ "RosettaDesign web server". Kuhlman Laboratory. University of North Carolina. Retrieved September 7, 2008.
- ^ a b Gray JJ, Moughon SE, Kortemme T, et al. (July 2003). "Protein–protein docking predictions for the CAPRI experiment". Proteins. 52 (1): 118–22. CiteSeerX 10.1.1.80.9354. doi:10.1002/prot.10384. PMID 12784377. S2CID 1186127.
- ^ Daily MD, Masica D, Sivasubramanian A, Somarouthu S, Gray JJ (2005). "CAPRI rounds 3–5 reveal promising successes and future challenges for RosettaDock". Proteins. 60 (2): 181–86. CiteSeerX 10.1.1.521.9981. doi:10.1002/prot.20555. PMID 15981262. S2CID 21137936. Archived from the original on June 30, 2012.
- ^ Méndez R, Leplae R, Lensink MF, Wodak SJ (2005). "Assessment of CAPRI predictions in rounds 3–5 shows progress in docking procedures". Proteins. 60 (2): 150–69. doi:10.1002/prot.20551. PMID 15981261. S2CID 24626361. Archived from the original on June 30, 2012.
- ^ "RosettaDock server". Rosetta Commons. Retrieved March 28, 2020.
- ^ "Protein–protein docking at Rosetta@home". Rosetta@home forums. University of Washington. Retrieved September 7, 2008.
- ^ "Robetta web server". Baker laboratory. University of Washington. Retrieved May 7, 2019.
- ^ Aloy P, Stark A, Hadley C, Russell RB (2003). "Predictions without templates: new folds, secondary structure, and contacts in CASP5". Proteins. 53 Suppl 6: 436–56. doi:10.1002/prot.10546. PMID 14579333. S2CID 22274928.
- ^ Tress M, Ezkurdia I, Graña O, López G, Valencia A (2005). "Assessment of predictions submitted for the CASP6 comparative modeling category". Proteins. 61 Suppl 7: 27–45. doi:10.1002/prot.20720. PMID 16187345. S2CID 24617067.
- ^ Battey JN, Kopp J, Bordoli L, Read RJ, Clarke ND, Schwede T (2007). "Automated server predictions in CASP7". Proteins. 69 Suppl 8: 68–82. doi:10.1002/prot.21761. PMID 17894354. S2CID 29879391.
- ^ Chivian D, Kim DE, Malmström L, Schonbrun J, Rohl CA, Baker D (2005). "Prediction of CASP6 structures using automated Robetta protocols". Proteins. 61 Suppl 7: 157–66. doi:10.1002/prot.20733. PMID 16187358. S2CID 8122486.
- ^ Baker D. "David Baker's Rosetta@home journal, message 52902". Rosetta@home forums. University of Washington. Retrieved September 7, 2008.
- ^ Ovchinnikov S, Kim DE, Wang RY, Liu Y, DiMaio F, Baker D (September 2016). "Improved de novo structure prediction in CASP11 by incorporating coevolution information into Rosetta". Proteins. 84 (Suppl 1): 67–75. doi:10.1002/prot.24974. PMC 5490371. PMID 26677056.
- ^ Baker D. "David Baker's Rosetta@home journal (message 52963)". Rosetta@home forums. University of Washington. Retrieved September 16, 2008.
- ^ "Foldit forums: How many users does Foldit have? Etc. (message 2)". University of Washington. Retrieved September 27, 2008.
- ^ "Foldit: Frequently Asked Questions". fold.it. University of Washington. Retrieved September 19, 2008.
- ^ "Project list – BOINC". University of California. Retrieved September 8, 2008.
- ^ Pande Group (2010). "High Performance FAQ". Stanford University. Archived from the original (FAQ) on September 21, 2012. Retrieved September 19, 2011.
- ^ 7im (April 2, 2010). "Re: Answers to: Reasons for not using F@H". Retrieved September 19, 2011.
- ^ Vijay Pande (August 5, 2011). "Results page updated – new key result published in our work in Alzheimer's Disease". Retrieved September 19, 2011.
- ^ Pande Group. "Folding@home Diseases Studied FAQ". Stanford University. Archived from the original (FAQ) on October 11, 2007. Retrieved September 12, 2011.
- ^ Vijay Pande (September 26, 2007). "How FAH works: Molecular dynamics". Retrieved September 10, 2011.
- ^ a b tjlane (June 9, 2011). "Re: Course grained Protein folding in under 10 minutes". Retrieved September 19, 2011.
- ^ jmn (July 29, 2011). "Rosetta@home and Folding@home: additional projects". Retrieved September 19, 2011.
- ^ Pande Group. "Client Statistics by OS". Stanford University. Retrieved October 18, 2011.
- ^ a b "Rosetta@home: Credit overview". boincstats.com. Retrieved March 28, 2020.
- ^ Malmström L, Riffle M, Strauss CE, et al. (April 2007). "Superfamily assignments for the yeast proteome through integration of structure prediction with the gene ontology". PLOS Biology. 5 (4): e76. doi:10.1371/journal.pbio.0050076. PMC 1828141. PMID 17373854.
- ^ Bonneau R (2006). "World Community Grid Message Board Posts: HPF -> HPF2 transition". Bonneau Lab, New York University. Retrieved September 7, 2008.
- ^ "List of Richard Bonneau's publications". Bonneau Lab, New York University. Archived from the original on July 7, 2008. Retrieved September 7, 2008.
- ^ Bonneau R. "World Community Grid Message Board Posts". Bonneau Lab, New York University. Archived from the original on July 4, 2008. Retrieved September 7, 2008.
- ^ "Predictor@home: Developing new application areas for P@H". The Brooks Research Group. Retrieved September 7, 2008.[데드링크]
- ^ Carrillo-Tripp M (2007). "dTASSER". The Scripps Research Institute. Archived from the original on July 6, 2007. Retrieved September 7, 2008.
- ^ "RALPH@home website". RALPH@home forums. University of Washington. Retrieved September 7, 2008.
- ^ "Rosetta@home". Retrieved March 19, 2020.
- ^ "Rosetta@home: The new credit system explained". Rosetta@home forums. University of Washington. 2006. Retrieved October 8, 2008.
- ^ "BOINCstats: Project Credit Comparison". boincstats.com. 2008. Archived from the original on September 13, 2008. Retrieved October 8, 2008.
- ^ "Credit divided over projects". boincstats.com. Retrieved February 19, 2015.
- ^ "Rosetta@home: Predictor of the day archive". Rosetta@home. University of Washington. 2008. Archived from the original on September 24, 2008. Retrieved October 8, 2008.
- ^ "Rosetta@home: Protein Folding, Design, and Docking". Rosetta@home. University of Washington. 2008. Retrieved October 8, 2008.
외부 링크
- 공식 웹사이트
- 베이커 랩 베이커 랩 웹 사이트
- 데이비드 베이커의 로제타@home 저널
- BOINC 플랫폼 개요 및 BOINC 설치 및 Rosetta@home에 첨부하기 위한 가이드 포함
- BOINCstats – Rosetta@home 상세 기여 통계
- Ralph@home 웹 사이트 Rosetta@home 알파 테스트 프로젝트
- David Baker와 실험실 구성원이 제공한 Rosetta@home의 YouTube 개요에 있는 Rosetta@home 비디오
- Rosetta 플랫폼 개발을 위한 Rosetta Commons Academy 공동 연구
- RosettaDesign 홈구장인 Kuhlman lab 웹페이지
온라인 로제타 서비스
- Rosetta Commons 목록
- Robetta 단백질 구조 예측 서버
- ROSIE 도킹, 설계 등 다기능 서버 세트
- RosettaDesign 단백질 설계 서버
- RosettaBackrub Flexib 백본/단백질 설계 서버