하플로그룹 R1a
Haplogroup R1a| 하플로그룹 R1a | |
|---|---|
 | |
| 원산지가능시간 | 2만2천년전에서[1] 2만5천년전에[2] |
| 원산지가능 | 동유럽, 남아시아, 중앙아시아 또는 서아시아 |
| 조상 | 하플로그룹R1 |
| 후예 | R1a-Z282, R1a-Z93 |
| 돌연변이 정의 |
|
| 최고 주파수 | 모집단별 R1a 빈도 목록 참조 |

하플로그룹 R1a 또는 하플로그룹 R-M420은 스칸디나비아와 중앙유럽에서 중앙아시아, 시베리아 남부 및 남아시아에 이르는 유라시아의 넓은 지역에 분포하는 인간 Y-염색체 DNA 하플로그룹입니다.[3][2]
한 유전학적 연구는 R1a가 25,000년[2] 전에 기원했다는 것을 나타내지만, 그것의 하위 분류 M417 (R1a1a1)은 5,800년 전에 다양해졌습니다.[4]인도유럽조어의 기원에 대한 논쟁에서 하위 분류의 기원지는 역할을 합니다.
SNP 돌연변이 R-M420은 R-M17(R1a1a) 이후 발견되었으며, 특히 R-M17로 이어지는 R-SRY10831.2(R1a1) 분기에 없는 비교적 드문 계통에 대한 새로운 파라그룹(R-M420*로 지정됨)을 구축했습니다.
오리진스
R1a원산지
R1a(M420)의 유전적 분화는 2만 5천[2] 년 전에 일어난 것으로 추정되는데, 이 시기는 마지막 빙하기의 최대 시기입니다.피터 A의 2014년 연구.Underhill et al. 은 유라시아 전역의 126명 이상의 인구 16,244명을 사용하여 "hg R1a의 지리적 기원으로서 중동, 아마도 현재의 이란 근처에 대한 설득력 있는 사례"가 있다고 결론 내렸습니다.[2]고대 DNA 기록에는 중석기 시대 동부 수렵채집인들(동유럽 출신)의 최초의 R1a가 기록되어 있으며,[5][6] 상부 구석기 고대 북유라시아인들 중 가장 초기의 R* 사례가 기록되어 있는데,[7] 이들은 주로 동부 수렵채집인들의 조상에서 유래했습니다.[8]
R1a1a1(M417)의 다양화와 고대 이주

Underhill et al. (2014)에 따르면, 하류 R1a-M417 서브클레이드는 약 5,800년 전에 "이란과 터키 동부 인근에서" Z282와 Z93으로 분화되었습니다.[4][note 1]비록 R1a가 슬라브어와 인도이란어와 같은 다양한 언어들 사이에서 Y염색체 하플로그룹으로 발생하지만, R1a1a의 기원에 대한 질문은 인도유럽조어족의 도시에 대한 현재의 논쟁과 관련이 있으며, 인더스 계곡 문명의 기원과도 관련이 있을 수 있습니다.R1a는 남아시아와 서아시아, 중유럽과 동유럽의 인도유럽어족 언어들과 강한 상관관계를 보여주고 있으며 어느 정도는 동유럽, 서아시아, 남아시아에서 스칸디나비아어가[10][3] 가장 널리 퍼져 있습니다.유럽에서는 특히 Z282가 유행하고 있고 아시아에서는 Z93이 우세합니다.1999년 T. Zerjal과 동료들은 Y-DNA R-M17과 인도유럽어족 언어의 확산 사이의 연관성을 처음으로 발견했습니다.[11]
R1a1a의 스텝분산액 제안
세미노 외. 2000년에는 우크라이나 기원과 후기 빙하기 최대 기간 동안 R1a1 하플로그룹의 후기 빙하기 확산을 제안했으며, 이후 쿠르간 문화가 유럽과 동쪽으로 확장되면서 확대되었습니다.[12]스펜서 웰스는 중앙아시아 기원을 제안하며, R1a1의 분포와 나이는 쿠르간족이 유라시아 스텝에서 확장하면서 확산한 것에 해당하는 고대 이주를 가리키고 있다고 제안합니다.[13]Pamjav et al. (2012)에 따르면, R1a1a는 유라시아 스텝 지대 또는 중동 및 코카서스 지역에서 다양화되었습니다.
내·중앙아시아는 R1a1-Z280 및 R1a1-Z93 계통의 중첩 영역입니다. 이는 남아시아와 중앙·동유럽 사이에 위치하고 있기 때문에 R1a1-M198의 초기 분화 영역이 유라시아 스텝 또는 중동·캅카스 지역 어딘가에서 발생했을 가능성을 시사합니다.[14]
2015년 3개의 유전학 연구는 인도유럽어족 우르헤마트에 관한 쿠르간의 김부타스 이론을 지지했습니다.그 연구들에 따르면, 현재 유럽에서 가장 흔한 하플로그룹 R1b와 R1a는 폰트-카스피해 스텝 지대에서 인도-유럽어족 언어들과 함께 확장되었을 것입니다. 그들은 또한 신석기 유럽인들에게는 존재하지 않았던 상염색체 성분을 현대 유럽인들에게 발견했습니다.인도유럽어 뿐만 아니라 부계 혈통인 R1b와 R1a와 함께 도입되었을 것입니다.[15][16][17]
Silva et al. (2017)은 남아시아의 R1a가 "중앙아시아의 단일 소스 풀에서 전파되었을 가능성이 가장 높으며, 인도 아대륙 내에 최소 3개 이상의 R1a 설립자 계층이 있으며, 다수의 도달 물결과 일치하는 것으로 보입니다."[18]라고 언급했습니다.마틴 P에 의하면.Silva et al. (2017)의 공동저자인 Richards는 인도에서 R1a가 널리 퍼졌다는 것은 "인도유럽어 사용자들을 인도로 데려왔을 가능성이 높은 중앙아시아에서 청동기 시대의 상당한 이주에 대한 매우 강력한 증거"였습니다.[19][note 2]
제안된 남아시아 기원
키비실트 외(2003)은 남아시아 또는 서아시아 중 하나를 제안한 반면, [20][note 3]미라발 외. (2009) 남아시아와 중앙아시아 모두에 대한 지원 참조.[10]센굽타 외.(2006)은 인도의 기원을 제안했습니다.[21]Thansee et al.(2006)은 남아시아 또는 중앙아시아를 제안했습니다.[22]사후 외.(2006)은 남아시아 또는 서아시아 중 하나를 제안했습니다.[23]Tangaraj et al. (2010)도 남아시아 기원을 제시한 바 있습니다.[24]Sharma et al.(2009)은 인도에서 R1a의 존재를 18,000년 이상에서 44,000년의 기원으로 이론화하고 있습니다.[1]
남아시아 인구는 R1a1a 및 [25][26][10][3][1][27]그 이후의 오래된 TMRCA 날짜 내에서 가장 높은 STR 다양성을 [citation needed]가지고 있으며, R1a1a는 더 높은 (Brahmin) 캐스트와 더 낮은 캐스트 모두에 존재하지만, 브라만 캐스트 중에서는 빈도가 더 높습니다.그럼에도 불구하고, R1a 하플로그룹의 가장 오래된 TMRCA 연대는 인도 중부 분델칸드 지역의 카스트로 예정된 사하라 부족에서 발생합니다.[1][27]이러한 발견으로부터 일부 연구자들은 R1a1a가 아프가니스탄, 발로치스탄, 펀자브, 카슈미르와 같은 북서 지역의 인도-유럽 이민자들로부터 유입된 유전자를 [26][1][note 4][note 5]제외하고 남아시아에서 기원했다고 결론지었습니다.[26][25][3]
그러나, 이러한 다양성과 그에 따른 오래된 TMRCA-데이트는 또한 역사적으로 높은 인구수로 설명될 수 있으며,[note 6] 이는 다양화와 미세 위성 변동의 가능성을 증가시킵니다.[19][18]Sengupta et al.에 의하면.(2006), "[R1a1과 R2]는 실제로 서남아시아 지역에서 인도 남부에 여러 번 도착했을 수도 있습니다."[25][note 7]그러나 센굽타는 이 기사에서도 다음과 같이 기술했습니다.
우리는 중앙아시아가 기존의 유전자 풀에 미치는 영향이 미미하다는 것을 발견했습니다.대부분의 인도 하플로그룹에서 누적된 미세 위성 변이의 나이는 10,000~15,000년을 초과하며, 이는 지역 분화의 고대성을 보여줍니다.따라서 우리의 데이터는 남아시아에서 관찰된 유전적 변이를 설명하기 위해 중앙아시아에서 뚜렷한 최근 유전자 입력을 호출하는 모델을 지원하지 않습니다.
그러나 R1a의 계통발생학적 재구성은 남아시아 기원을 뒷받침하지 않습니다.유전학자들 사이의 주류적인 입장은 하플로그룹 R1a가 남아시아에서 기원한 것이 아니며, 하플로그룹 R1a의 보유자들이 인더스 밸리 문명의 설립 이후 얼마 동안 서아시아에서 남아시아로 이동했다는 것입니다.[29][30]
제안된 얌나야 원산지


데이비드 앤서니는 얌나야 문화를 인도유럽어족 우르헤마트라고 생각합니다.[31][32]Haak et al. (2015)에 따르면, 기원전 2,500년경에 얌나야 문화에서 북쪽으로 대규모 이주가 일어났으며, 이는 Corded Ware 문화의 유전적 조상의 75%를 차지하며, R1a와 R1b가 "기원전 3,000년 이후 동쪽에서 유럽으로 전파되었을 수 있다"[33]고 언급했습니다.그러나 7개의 Yamnaya 샘플은 모두 R1b-M269 하위 클래스에 속했지만,[33] R1a1a는 Yamnaya 샘플에서 발견되지 않았습니다.이것은 코드드 웨어 문화의 R1a1a가 얌나야 문화가 아니었다면 어디서 왔는지에 대한 의문을 제기합니다.[34]
마크 하버(Marc Haber)에 따르면, 아프가니스탄에 하플로그룹 R1a-M458이 없는 것은 현대 중앙아시아 인구의 R1a 계통에 대한 폰틱-카스피해 스텝 기원을 지지하지 않는다고 합니다.[35]
레오 클라인(Leo Klejn)에 따르면, 얌나야에 하플로그룹 R1a가 없는 것은 (에놀리식 사마라(Eneolithic Samara)와 동부 사냥꾼 개터(Eastern Hunter Gatherer) 개체군에 존재함에도 불구하고) 유럽인들이 얌나야로부터 하플로그룹 R1a를 물려받을 가능성을 낮게 합니다.[36]
고고학자 Barry Cunliffe는 Yamnaya 표본에서 하플로그룹 R1a가 없는 것이 R1a가 Yamnaya 기원이라는 Haak의 제안의 주요한 약점이라고 말했습니다.[37]
Semenov & Bulat(2016)는 Corded Ware 문화에서 R1a1a의 Yamnaya 기원을 주장하고 있으며, 여러 출판물들이 Comb Ware 문화에서 R1a1의 존재를 지적하고 있다고 언급하고 있습니다.[38][note 8]
제안된 트랜스캅카스와 서아시아의 기원과 인더스 밸리 문명의 영향
Haak et al. (2015)은 Yamnaya 혈통의 일부가 중동에서 유래했으며 신석기 시대의 기술이 발칸반도에서 온 Yamnaya 문화에 도달했을 가능성이 있음을 발견했습니다.[note 9]뢰센 문화(기원전 4,600년–4,300년)[note 10]는 독일에 위치해 있었으며, R1a의 오래된 하위 계층인 코드드 웨어 문화보다 앞선 것으로 알려져 있습니다.
남아시아 유전자 조상의 일부는 서유라시아 인구에서 유래했으며, 일부 연구자들은 Z93이 이란을[40] 거쳐 인도로 건너와 인더스 밸리 문명 기간 동안 그곳으로 확장되었을 수 있다고 암시했습니다.[2][41]
Mascarenhas et al. (2015)은 Z93의 뿌리가 서아시아에 있음을 제안하였고, "Z93과 L342.2는 트랜스캅카스에서 남아시아로 동남쪽 방향으로 확장되었다"고 제안하였으며,[40]이러한 확장은 "우르크 4세 이후의 이른바 쿠라-아락시스의 이주로 절정에 이른 기원전 4천년 서아시아 인구의 동쪽 확장에 대한 고고학적 기록"과 양립할 수 있다는 점에 주목합니다.[40]그러나, Lazaridis는 그들의 Armenian Kura-Araxes 샘플인 Lazaridis et al. (2016)의 샘플 I1635가 Y-하플로그룹 R1b1-M415(xM269)([note 11]R1b1a1b-CTS3187로도 불림)[42][unreliable source?]을 운반했다고 지적했습니다.
Underhill et al.(2014)은 Z93의 다양화와 "인더스 밸리 내의 초기 도시화...[5,600년 전]에 발생했으며 R1a-M780(그림 3d[note 12])의 지리적 분포가 이를 반영할 수 있습니다."[2][note 13] Poznik et al. (2016)은 R1a-Z93에서 4,500-4,000년 전 경에 "돌발적인 팽창"이 일어났으며, 이는 "인더스 밸리 문명이 붕괴되기 몇 세기 전"이라고 언급했습니다.[41][note 14]
그러나 Narasimhan et al.(2018)에 의하면 인도의 R1a는 스텝목회주의자가 유력한 자원이라고 합니다.[44][note 15]
계통발생학
R1a 계보는 이제 세 가지 주요 수준의 분기를 가지고 있으며, 지배적이고 가장 잘 알려진 분기 내에서 가장 많은 수의 정의된 하위 계층인 R1a1a(비교적 최근의 문헌에서는 "R1a1"과 같은 다양한 이름으로 발견됨)를 가지고 있습니다.
위상
R1a의 토폴로지는 다음과 같습니다(괄호 안에 있는 코드는 비이소그 코드).[9][45][verification needed][46][2][47]Tatiana et al. (2014) "K-M526의 빠른 다양화 과정은 동남아시아에서 발생했을 가능성이 높으며, 그에 따라 하플로그룹 R과 Q의 조상들이 서쪽으로 확장되었습니다."[48]
- PP295/PF5866/S8(K2b2라고도 함).
- R(R-M207)[46][9]
- R*
- R1 (R-M173)
- R1*[46]
- R1a (M420)[46] (동유럽,아시아)[2]
- R1a*[9]
- R1a1[46] (M459/PF6235,[46] SRY1532.2/SRY10831.2[46])
- R1a1 (M459)[46][9]
- R1a1a(M17, M198)[46]
- R1a1a1 (M417, 7페이지)[46]
- R1a1a1a(CTS7083/L664/S298)[46]
- R1a1a1b (S224/Z645, S441/Z647)[46]
- R1a1a1b1 (PF6217/S339/Z283)[46]
- R1a1a1b2 (F992/S202/Z93)[46] [R1a1a2*] (Z93, M746)[49] (중앙아시아, 남아시아 및 서아시아)
- [R1a1a1c] (M64.2, M87, M204)[47]
- [R1a1a1d] (P98)[47]
- [R1a1a1d2a][50]
- [R1a1a1e] (PK5)[47]
- R1a1a1 (M417, 7페이지)[46]
- R1b (M343) (서유럽)
- R2 (인도)
하플로그룹R
R-M173 (R1)
R1a는 M420 돌연변이를 포함한 몇 가지 독특한 마커로 구별됩니다.하플로그룹 R-M173(이전 이름은 R1)의 하위 클래스입니다.R1a에는 자매 하위 분류인 하플로그룹 R1b-M343과 파라그룹 R-M173*이 있습니다.
R-M420 (R1a)
돌연변이 M420에 의해 정의된 R-M420에는 두 갈래가 있습니다: 대다수를 구성하는 돌연변이 SRY1532.2에 의해 정의된 R-SRY1532.2와 M420 양성이지만 SRY1532.2 음성으로 정의된 파라그룹 R-M420*. (2002년 계획에서 이 SRY1532.2 음성 소수는 파라그룹 R1*으로 분류된 비교적 드문 그룹의 일부였습니다.)M420과 동등한 것으로 이해되는 돌연변이에는 M449, M511, M513, L62, L63 등이 있습니다.[3][51]
Underhill 2009에서는 새로운 파라그룹 R-M420*의 분리된 샘플만 발견되었는데, 대부분 중동과 코카서스에서 1/121 오만인, 2/150 이란인, 아랍에미리트에서 1/164, 터키에서 3/612가 발견되었습니다.73개의 다른 유라시아 인구에서 7224명의 남성을 더 검사한 결과 이 범주의 징후는 없었습니다.[3]
이 그룹은 현재 R1a2(R-YP4141)로 알려져 있습니다.그 다음 R1a2a(R-YP5018) 및 R1a2b(R-YP4132) 두 개의 가지를 갖습니다.
R-SRY1532.2(R1a1)
R1a1은 SRY1532.2 또는 SRY10831.2에 의해 정의됩니다(항상 SRY10831.2, M448, L122, M459 및 M516을[3][52] 포함하는 것으로 이해됨).이 계통군은 M17과 M198이 지배적입니다.반면, 파라그룹 R-SRY1532.2*에는 M17 또는 M198 마커가 없습니다.
R-SRY1532.2* paragroup은 R1*보다 덜 드물지만, 한 번 이상의 설문조사를 통해 테스트되었지만 여전히 상대적으로 흔하지 않습니다.언더힐 등.(2009)는 노르웨이에서 1/51, 스웨덴에서 3/305, 그리스 마케도니아인 1/57, 이란인 1/150, 아르메니아인 2/734, 카바르디인 1/141을 보고했습니다.[3]사후 외. (2006)은 1/15 Himachal Pradesh Rajput 샘플에 대해 R-SRY1532.2*를 보고했습니다.[26]
R-M17/M198(R1a1a)
R1a1a와 관련된 SNP는 다음과 같습니다.
| SNP | 돌연변이 | Y 위치(NCBI36) | Y 위치(GRCh37) | RefSNP ID |
|---|---|---|---|---|
| M17 | INSG | 20192556 | 21733168 | rs3908 |
| M198 | C->T | 13540146 | 15030752 | rs2020857 |
| M512 | C->T | 14824547 | 16315153 | rs17222146 |
| M514 | C->T | 17884688 | 19375294 | rs 17315926 |
| M515 | T->A | 12564623 | 14054623 | rs17221601 |
| L168 | A->G | 14711571 | 16202177 | - |
| L449 | C->T | 21376144 | 22966756 | - |
| L457 | G->A | 14946266 | 16436872 | rs113195541 |
| L566 | C->T | - | - | - |
R-M417 (R1a1a1)
R1a1a1(R-M417)은 유럽(R1a1a1b1(R-Z282))[2]과 중앙 및 남아시아(R1a1a1b2(R-Z93)(R-Z93)(R-Z93)에서 각각 발견되는 두 가지 변형에서 가장 널리 발견되는 하위 클래스입니다.[2]
R-Z282 (R1a1a1b1a) (동유럽)
이 커다란 하위 분류군은 유럽에서 발견되는 R1a1a의 대부분을 포함하는 것으로 보입니다.[14]
- R1a1a1b1a [R1a1a1a* (Underhill (2014))] (R-Z282*)는 우크라이나 북부, 벨라루스 및 러시아에서 c. 20%[2]의 빈도로 발생합니다.
- R1a1a1b1a3[R1a1a1a1a1(언더힐(2014))](R-Z284)은 북서유럽에서 발생하고 노르웨이에서 c. 20%로 최고점을 찍습니다.[2]
- R1a1a1c (M64.2, M87, M204)는 분명히 희귀합니다: 그것은 이란 남부에서 타자를 친 117명의 남성들 중 한 명에게서 발견되었습니다.[53]
R-M458 (R1a1a1b1a1)

R-M458은 주로 슬라브계 SNP로, 자체 돌연변이를 특징으로 하며, 처음에는 클러스터 N이라고 불렸습니다.언더힐 등.(2009년) M458은 현대 유럽인들에게 대략 라인강 유역과 우랄산맥 사이에 존재하는 것을 발견했으며, "홀로세 초기, 7.9±2.6 KYA에 속하는 창설자 효과"로 추적했습니다. (지보토프스키 속도, 3배 과대평가됨)[3] M458은 14세기 Usedom의 한 무덤에서 나온 뼈 하나에서 발견되었으며,메클렌부르크포어포메른, 독일.[54]Underhill et al. 의 논문.(2009)는 또한 일부 북부 백인 인구에서 M458의 빈도가 놀랍도록 높다고 보고했습니다 (악 노가이[55] 중 18%, 카라 노가이 중 7.8%, 아바자 중 3.[56]4%).
R-L260 (R1a1a1b1a1a)
R1a1a1b1a1a1a1a1a1a1a(R-L260), 흔히 West Slavic 또는 Polish로 지칭되는, 더 큰 모그룹 R-M458의 하위클레이드이며, Powlowski et al. 2002에 의해 STR 클러스터로 처음 확인되었습니다.2010년에 자체 돌연변이(SNP)에 의해 확인된 하플로그룹임이 확인되었습니다.[57]폴란드 남성의 약 8%를 차지하며 폴란드에서 가장 흔한 하위 계층입니다.폴란드 밖에서는 덜 흔합니다.[58]폴란드 외에도 주로 체코와 슬로바키아에서 발견되며, "분명히 서슬라브어"로 여겨집니다.R-L260의 시조는 2000년에서 3000년 전, 즉 철기 시대에 살았던 것으로 추정되며, 1,500년 미만 전에 인구가 크게 증가했습니다.[59]
R-M334
Underhill 등은 R-M334 ([R1a1a1g1,[47] [R1a1a1g] (M458)[47] c.q. R1a1b1a1(M458))[46]의 서브클레이드를 발견하였습니다.(2009) 에스토니아인 한 명에게만 해당되며, 매우 최근에 설립된 소분류군을 정의할 수 있습니다.[3]
R1a1a1b1a2 (S466/Z280, S204/Z91)
R1a1a1b1a2b3* (Gwozdz의 클러스터 K)
R1a1a1b1a2b3* (M417+, Z645+, Z283+, Z282+, Z280+, CTS1211+, CTS3402, Y33+, CTS3318+, Y2613+) (Gwozdz의 클러스터 K)[45][verification needed]는 R-M17(xM458)인 STR 기반 그룹.이 클러스터는 폴란드에서 흔히 볼 수 있지만 폴란드에만 있는 것은 아닙니다.[59]
R1a1a1b1a2b3a (R-L365)
R1a1a1b1a2b3a(R-L365)[46]는 초기에 클러스터 G로 불렸습니다.[citation needed]
R1a1a1b2 (R-Z93) (아시아)
| 지역 | 사람 | N | R-M17 | R-M434 | ||
|---|---|---|---|---|---|---|
| 번호 | 빈도(%) | 번호 | 빈도(%) | |||
| 파키스탄 | 발록 | 60 | 9 | 15% | 5 | 8% |
| 파키스탄 | 막라니 | 60 | 15 | 25% | 4 | 7% |
| 중동 | 오만 | 121 | 11 | 9% | 3 | 2.5% |
| 파키스탄 | 신디 | 134 | 65 | 49% | 2 | 1.5% |
| 표에는 60개의 유라시아 모집단 표본에서 파생된 N = 3667의 양의 집합만 나와 있습니다. | ||||||
이 큰 하위 분류는 아시아에서 발견되는 R1a1a의 대부분을 포함하는 것으로 보이며, 인도유럽인의 이주(스키타이인, 인도아리아인의 이주 등)와 관련이 있습니다.[14]
- R-Z93* 또는 R1a1a1b2*(언더힐(2014))은 러시아의 남시베리아 알타이 지역에서 가장 흔하며(>30%), 키르기스스탄(6%) 및 모든 이란 인구(1~8%)[2]에서 발생합니다.
- R-Z2125는 키르기스스탄과 아프가니스탄 파슈툰에서 가장 높은 빈도로 발생합니다(>40%).10% 이상의 빈도로 다른 아프가니스탄 민족과 코카서스 및 이란의 일부 인구에서도 관찰됩니다.[2]
- R-M560은 매우 희귀하며 부루샤스키 화자 2명(파키스탄 북부), 하자라어 1명(아프가니스탄), 이란계 아제르바이잔인 1명 등 4개 샘플에서만 관찰됐습니다.[2]
- R-M780은 남아시아에서 높은 빈도로 발생합니다.인도, 파키스탄, 아프가니스탄 그리고 히말라야 산맥.터키는 R1a (12.1%) 하위 라인 연령을 공유합니다.[60]슬로바키아 출신의 로마는 R1a의[61] 3%를 공유합니다. 이 그룹은 또한 일부 이란 인구에서 3% 이상 발생하고 크로아티아와 헝가리 출신의 로마에서도 30% 이상 발생합니다.[2]
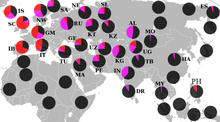
R1a1a의 지리적 분포
프리 히스토리
중석기 유럽에서 R1a는 EHG(Eastern Hunter-Gatherers)의 특징입니다.[62]러시아 아르한겔스크주 라차 호수 근처 페샤니차에 묻힌 베레티 문화의 남성 EHG. 기원전 10,700년 부계 하플로그룹 R1a5-YP1301과 모계 하플로그룹 U4a의 운반자로 밝혀졌습니다.[63][64][62] 카렐리야 c. 기원전 8,800년부터 기원전 7950년까지의 중석기 남성이 하플로그룹 R1a를 운반하는 것으로 밝혀졌습니다.[65]Deriivka c. 기원전 7000년에서 기원전 6700년 사이에 묻힌 중석기 남성은 부계 하플로그룹 R1a와 모계 U5a2a를 운반했습니다.[17]기원전 5,500년에서 5,000년경 카렐리야 출신의 EHG로 여겨졌던 또 다른 남성은 하플로그룹 R1a를 옮겼습니다.[15]쿠드루쿨라 c. 기원전 5,900년에서 기원전 3,800년 사이의 빗 도자기 문화의 남성이 R1a와 모체 U2e1의 운반자로 결정되었습니다.[66]고고학자 데이비드 앤서니에 따르면, 부계 R1a-Z93은 더 이상 존재하지 않는 콜호즈 "알렉산드리아" 근처의 오스콜 강에서 발견되었습니다. 기원전 4000년, "락타아제 지속성(13910-T)에 대한 유전적 적응을 보여주는 가장 초기의 알려진 표본"입니다.[67] R1a는 그것이 우세한 코드웨어 문화에서 발견되었습니다.[68][69][70]조사된 청동기 시대의 파티아노보 문화의 남성들은 전적으로 R1a, 특히 하위 계급인 R1a-Z93에 속합니다.[62][63][71]
Haplogroup R1a has later been found in ancient fossils associated with the Urnfield culture;[72] as well as the burial of the remains of the Sintashta,[16] Andronovo,[73] the Pazyryk,[74] Tagar,[73] Tashtyk,[73] and Srubnaya cultures, the inhabitants of ancient Tanais,[75] in the Tarim mummies,[76] and the aristocracy of Xiongnu.[77]2005년 에라우(독일 작센안할트) 근처에서 발견된 기원전 2600년경의 고고학 유적에서 Y-SNP 표지 SRY10831.2 양성 반응을 보인 아버지와 그의 두 아들의 유골.을라우 유적에 대한 Y 검색 번호는 2C46S입니다.따라서 조상의 종족은 유럽에 적어도 4600년 전에 존재했으며, 이는 널리 퍼져있는 코드드 웨어 문화의 한 장소와 관련이 있습니다.[68]
유럽
유럽에서 R1a1 하위 분류는 중부와 동유럽 혈통의 사람들 사이에서 가장 높은 수준으로 발견되며, 체코인, 헝가리인, 폴란드인, 슬로바키아인, 서부 우크라이나인, 루신인, 벨라루스인, 몰도바인, 러시아인 사이에서 35%에서 65%의 결과를 보입니다.[78][79][12]발트해에서 R1a1a의 주파수는 리투아니아(45%)에서 에스토니아(약 30%)[80][81][82][12][83]로 감소합니다.
스칸디나비아 혈통의 사람들에게 중요한 존재감이 있으며, 노르웨이와 아이슬란드에서 가장 높은 수준을 보이고 있으며, 남성의 20~30%가 R1a1a에 속해 있습니다.[84][85]바이킹과 노르만족은 또한 R1a1a 혈통을 더 멀리 가져갔을 수도 있으며, 브리튼 제도, 카나리아 제도, 시칠리아에 있는 작은 존재의 적어도 일부를 차지했습니다.[86][87]동독에서 하플로그룹 R1a1a는 평균 20~30% 사이이며, Rostock의 최고점은 31.3%입니다.[88]
남유럽에서는 R1a1a가 흔하지 않지만, 스페인 북부의 파스 계곡, 베네치아 지역, 이탈리아의 칼라브리아와 같은 주머니에서 상당한 수준이 발견되었습니다.[89][better source needed]The Balkans shows wide variation between areas with significant levels of R1a1a, for example 36–39% in Slovenia,[90] 27–34% in Croatia,[81][91][92][93][94] and over 30% in Greek Macedonia, but less than 10% in Albania, Kosovo and parts of Greece south of Olympus gorge.[95][82][12]
R1a는 사실상 스칸디나비아의 Z284 서브클레이드로만 구성되어 있습니다.슬로베니아의 주요 하위 분류는 Z282(Z280 및 M458)이지만, Z284 하위 분류는 슬로베니아인의 표본에서 발견되었습니다.터키에서는 Z93을 무시할 정도로 표현하고 있으며, 12,1%[60][2]의 서슬라브족과 헝가리인들은 M458의 높은 빈도와 Z280의 낮은 Z92를 특징으로 합니다.수백 명의 슬로베니아인 샘플과 체코인은 Z280의 Z92 하위 클래스가 부족한 반면, 폴란드인, 슬로바키아인, 크로아티아인, 헝가리인은 Z92의 빈도가 매우 낮습니다.[2]발트족, 동슬라브족, 세르비아인, 마케도니아인, 불가리아인, 루마니아인은 Z280>M458과 Z92의 우세한 점유율까지 높은 비율을 보이고 있습니다.[2]발트족과 동슬라브족은 더 자세한 하위 분류 체계에서 동일한 하위 분류군과 유사한 빈도를 가지고 있습니다.[96][97]러시아의 유전학자 올레크 발라노프스키는 동슬라브족과 서슬라브족의 유전학에서 슬라브 이전의 기질이 우세하다고 추측했습니다.그에 따르면 동슬라브족과 발트족이 다른 인구들과 대조되는 공통적인 유전적 구조는 동슬라브족과 서슬라브족의 슬라브 이전 기질이 발트어 화자들로 가장 크게 구성되었다는 설명을 제시할 수 있습니다.한 때는 고고학적 및 지명학적 문헌에 따르면 유라시아 스텝의 문화에서 슬라브족을 앞섰습니다.[note 16]
아시아
중앙아시아
Zerjal et al. (2002)는 타지키스탄의 타직족 표본의 64%와 키르기스스탄의 키르기스스탄 표본의 63%에서 R1a1a를 발견했습니다.[98]
Haber et al. (2012)은 Nuristanis 표본 60% (3/5), Pashtun 표본 51.0% (25/49), Tajik 표본 30.4% (17/56), 우즈베키스탄 표본 17.6% (3/17), 하자라스 표본 6.7% (4/60) 및 유일하게 표본이 된 투르크멘 개인을 포함하여 아프가니스탄에서 온 표본의 26.0% (53/204)에서 R1a1a-M17을 발견했습니다.[99]
Di Cristofaro et al. (2013)은 아프가니스탄에서 온 한 쌍의 파슈툰 샘플 중 56.3%(49/87)에서 R1a1a-M198/M17을 발견했습니다(바글란에서 온 파슈툰 샘플 중 20/34 또는 58.8%, 쿤두즈에서 온 파슈툰 샘플 중 29/53 또는 54.7% 포함). 아프가니스탄에서 온 우즈베키스탄인 샘플 중 29.1%(37/127).자우잔에서 온 우즈벡인 표본의 8%, 사르에폴에서 온 우즈벡인 표본의 8/28 또는 28.6%, 발흐에서 온 우즈벡인 표본의 1/5 또는 20%), 아프가니스탄에서 온 타지크인 표본의 27.5%(39/142)(발흐에서 온 타지크인 표본의 22/54 또는 40.7% 포함, 타하르에서 온 타지크인 표본의 9/35 또는 25.7% 포함, 4/16 또는 25).사만간의 타직 표본의 0%, 바다흐샨의 타직 표본의 4/37 또는 10.8%), 자우잔의 투르크멘 표본의 16.2%(12/74), 아프가니스탄의 하자라 표본의 9.1%(7/77). (바미얀의 하자라 표본의 7/69 또는 10.1%, 발흐의 하자라 표본의 0/8 또는 0% 포함).[100]
Malyarchuk et al. (2013)은 타지키스탄의 Tajik 샘플의 30.0% (12/40)에서 R1a1-SRY10831.2를 발견하였습니다.[101]
Ashirbekov et al. (2017)은 카자흐스탄 출신 카자흐스탄인 샘플 세트의 6.03% (78/1294)에서 R1a-M198을 발견했습니다.R1a-M198은 수안 표본의 13/41 = 31.7%, 오샤크티 표본의 8/29 = 27.6%, Qozha 표본의 6/30 = 20.0%, Qypshaq 표본의 4/29 = 13.8%, Tore 표본의 1/8 = 12.5%, Jetyru 표본의 9/86 = 10.5%, 4/50 = 8.5%의 연구 빈도로 관찰되었습니다.Argyn 시료 0%, Shanyshqly 시료 1/13 = 7.7%, Alimuly 시료 8/122 = 6.6%, Alban 시료 3/46 = 6.5%.보고되지 않은 부족 소속 카자흐인 표본의 5/42 = 11.9%에서도 R1a-M198이 관찰되었습니다.
남아시아
남아시아에서, R1a1a는 종종 다수의 인구학적 그룹에서 관찰되었습니다.[26][25]
인도에서는 동쪽으로는 서벵골 브라만족(72%),[25] 서쪽으로는 구자라트 로하나스(60%), 북쪽으로는 펀자브/하리아나 카트리스(67%),[3] 남쪽으로는 카르나타카 메다르스(39%)[103]에서 이 하플로그룹의 높은 빈도가 관측됩니다.이는 또한 인도 남부 부족의 인도인들에게 널리 퍼져 있음을 암시하는 첸추족(26%)과 안드라프라데시주의 발미키스족, 코타족([104]22.58%), 타밀나두족의 칼라르족 등 여러 남인도 드라비다어를 사용하는 아디바시스에서도 발견되었습니다.[20]
이 외에도 마니푸르인(50%)[3]과 펀자브인(47%)[20]과 같이 지역적으로 다양한 집단에서 높은 비율을 보이는 연구가 있습니다.
파키스탄에서는 남쪽으로 신드주에 있는 모한나 부족의 71%, 북쪽으로 길기트발티스탄의 발트족의 46%에서 발견됩니다.[3]스리랑카의 싱할라인들 중 23%는 R1a1a(R-SRY1532) 양성으로 나타났습니다.[105]네팔 테라이 지역의 치트완 지역 힌두교도는 69%[106]로 나타났습니다.
동아시아
R1a1a의 빈도는 야쿠트족과 같은 일부 튀르크어를 사용하는 집단에서 비교적 낮지만, 보난족, 둥샹족, 살라족, 위구르족과 같은 중국 북서부의 특정 튀르크어 또는 몽골어를 사용하는 집단에서 높은 수준(19~28%)을 보이고 있습니다.[13][107][108]
2018년에 발표된 중국 논문에서는 신장(于田县达里雅布依乡) 위톈현 다리아보이/다리아보이 마을의 케리얄릭 위구르 표본의 38.5%(15/39)에서 R1a-Z94가, 신장(阿瓦提县乌鲁却勒镇) 아왓현 호리콜읍의 돌란위구르 표본의 28.9%(22/76)에서 R1a-Z93이, 6.1a-Z93이 발견되었습니다.신장(尉犁县喀尔曲尕乡) 율리현 카르쿠가 / 카르추하 마을의 로플릭 위구르 표본의 3%(4/64).R1a(xZ93)는 76개의 돌란 위구르족 중 하나에서만 관측되었습니다.[109]다리아 보이이 마을은 타클라마칸 사막의 케리야 강에 의해 형성된 외딴 오아시스에 위치해 있습니다.2011년 Y-DNA 연구에 따르면 윈난성에서 온 남부 후이족 표본의 10%, 시짱(티베트 자치구)에서 온 티베트인 표본의 1.6%, 신장에서 온 시베족 표본의 1.6%, 닝샤에서 온 북부 후이족 표본의 3.2%, 신장에서 온 하자크(카자흐족) 표본의 9.4%, 그리고 24.0%, 22.2%, 35.2%, 29%의 비율로 Y-dna R1a1을 발견했습니다.신장에서 온 위구르인 4개 표본에서 2%, 내몽골에서 온 몽골인 표본에서 9.1%가 나왔습니다.닝샤에서 온 북부 후이족 표본의 1.5%에서도 R1의 다른 하위 분류가 발견되었습니다.[110]같은 연구에서 윈난성 한족 6개, 광시성 한족 1개, 구이저우성 한족 5개, 광둥성 한족 2개, 푸젠성 한족 2개, 저장성 한족 2개, 상하이성 한족 1개, 장시성 한족 1개, 후난성 한족 2개,후베이성 한 표본 1건, 쓰촨성 한 표본 2건, 충칭성 한 표본 1건, 산둥성 한 표본 3건, 간쑤성 한 표본 5건, 지린성 한 표본 3건, 헤이룽장성 한 표본 2건.[111]한 표본에서 조사한 바오안(보난)의 하플로그룹 다양성은 0.8946±0.0305로 가장 많았으며, 신장의 타직(Tajiks)의 45.2%, 동샹(東 and)의 54.3%, 타타르(Tatars)의 60.6%, 키르기스의 68.9%에서 R1a1-M17이 검출되었습니다.0.7602±0.0546의 값입니다.[112]
동부 시베리아에서 R1a1a는 캄차칸족과 추코트칸족을 포함한 특정 원주민 집단에서 발견되며, 이텔만어는 22%[113]에 달합니다.
서아시아
R1a1a는 요르단과 같은 지역에서는 거의 존재하지 않는 것부터 쿠웨이트와 이란의 지역에서는 훨씬 높은 수준에 이르기까지 서아시아의 대부분 지역에서 다양한 형태로 발견되었습니다.쿠웨이트의 시마르(샴마르) 베두인 부족은 43%[114][115][116]로 중동에서 가장 높은 빈도를 보이고 있습니다.
Wells 2001은 이란 서부 지역에서 이란인들은 낮은 R1a1a 수준을 보이는 반면, 이란 동부 지역의 남성들은 최대 35%의 R1a1a 수준을 가지고 있다고 지적했습니다.Nasidze 외. 2004년 테헤란과 이스파한에서 온 이란 남성의 약 20%에서 R1a1a를 발견했습니다.레게이로 2006년 이란에 대한 연구에서 남쪽이 북쪽보다 훨씬 더 높은 빈도를 기록했습니다.
새로운 연구에 따르면 이란 서부 쿠르디스탄 지방에서 채취된 쿠르드족 표본 중 R-M17*이 20.3%, 서아제르바이잔의 아제르바이잔인 중 19%, 마잔다란 지방의 북 이란 마잔다란인 중 9.7%, 길란 지방의 길락인 중 9.4%, 페르시아인 중 12.8%, 야즈드의 조로아스터인 중 17.6%로 나타났습니다.이스파한의 페르시아인 중 2%, 호라산의 페르시아인 중 20.[117]3%, 아프로-이란인 중 16.7%, 케슈미 '게슈미' 18.4%, 호르모즈간의 페르시아 반다리인 중 21.4%, 시스탄과 발루체스탄 주의 발루치인 중 25%.
Di Cristofaro et al. (2013)은 이란 샘플 세트의 9.68% (18/186)에서 하플로그룹 R1a를 발견했지만, 테헤란에서 온 이란인 샘플의 0% (0/18)에서 호라산에서 온 이란인 샘플의 25% (5/20), 그리고 출처가 불분명한 이란인 샘플의 27% (3/11)에 이르기까지 큰 차이를 보였습니다.R1a-SRY1532.2(xM198, M17)에 속하는 것으로 보고된 길란(n=27)의 이란인 샘플에서 한 명을 제외한 모든 이란인 R1a 개체가 M198 및 M17 돌연변이를 옮겼습니다.
Malyarchuk et al. (2013)은 이란 동부 Khorasan 지방과 Kermansha 지방에서 수집된 페르시아인 표본의 20.8%(16/77)에서 R1a1-SRY10831.2를 발견했지만, 이란 서부 Kermanshah 지방에서 수집된 25명의 쿠르드인 표본에서는 이 하플로그룹의 구성원을 찾지 못했습니다.[101]
반면에 이 서아시아 지역들의 북쪽으로는 다시 한 번 불균등한 방식으로 코카서스에서 R1a1a 수준이 증가하기 시작합니다.몇몇 연구된 개체군들은 R1a1a의 징후를 보이지 않고 있으며, 이 지역에서 지금까지 발견된 가장 높은 수치는 카라차이-발카르어 화자들의 것으로 보이며, 그 중 지금까지 실험된 남성의 약 1/4이 하플로그룹 R1a1a에 속해 있습니다.[3]
로마니족
로마인의 R1a-Z93 부계 유전자도 발견되었습니다.[118]
R1a의 과거 이름
R1a에 일반적으로 사용되는 역사적 명명 시스템은 자주 변경되었기 때문에 출판된 여러 출처에서 일관성이 없었습니다. 이에 대한 설명이 필요합니다.
2002년 Y 염색체 컨소시엄(YCC)은 하플로그룹(YCC 2002)에 대한 새로운 명명 시스템을 제안했고, 현재는 표준이 되었습니다.이 시스템에서 "R1"과 "R1a" 형식의 이름은 가계도에서 위치를 표시하는 것을 목표로 하는 "생리학적" 이름입니다.SNP 돌연변이의 이름은 클래스 또는 하플로그룹의 이름을 지정하는 데 사용할 수도 있습니다.예를 들어, 현재 M173이 R1의 정의적 돌연변이이므로, R1도 "돌연변이" 클래스 이름인 R-M173입니다.트리에서 새로운 분기가 발견되면 일부 계통발생학적 이름이 변경되지만 정의상 모든 변이 이름은 동일하게 유지됩니다.
돌연변이 M17에 의해 정의된 광범위하게 존재하는 하플로그룹은 "Eu19"와 같은 다양한 이름으로 알려져 있었습니다(Semino et al. 2000) 이전의 명명 시스템에서.2002년 YCC 제안은 돌연변이 SRY1532.2에 의해 정의된 하플로그룹에 R1a라는 이름을 부여했습니다.여기에는 Eu19(즉, R-M17)가 서브클레이드로 포함되어 있어, Eu19는 R1a1으로 명명되었습니다.참고로, SRY1532.2는 SRY10831.2로도[citation needed] 알려져 있습니다.2009년 M420의 발견으로 인해 이들 계통명이 재배분되었습니다.(언더힐 등) 2009 및 ISOGG 2012) R1a는 이제 M420 변이에 의해 정의됩니다: 이 업데이트된 트리에서, SRY1532.2에 의해 정의된 하위 클래스는 R1a에서 R1a1로, Eu19(R-M17)는 R1a1에서 R1a1a로 이동했습니다.
ISOGG 참조 웹페이지에 기록된 최신 업데이트는 하나의 주요 지점인 R-M417을 포함한 R-M17의 지점을 포함합니다.
| 2002년 계획 제안 (YCC 2002) | 2009년 계획(언더힐 외). 2009) | 2011년[citation needed] 1월 기준 ISOGG 트리 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|
참고 항목
Y-DNA R-M207 서브클레이드
Y-DNA 백본 트리
메모들
- ^ 가계도에 따르면,[who?] 그들은 5,000년 전에 다양해졌습니다.[9]
- ^ 참고 항목:
- ^ 키비실트 외(2003):"이전에 인도-아리아 침공으로 추정되는 하플로그룹 R1a는 펀자브에서 가장 높은 빈도로 발견되었지만 첸추 부족에서도 비교적 높은 빈도(26%)로 발견되었습니다.이 발견은 유럽과 중앙 아시아에 비해 인도와 이란에서 R1a와 관련된 짧은 연쇄 반복 다양성이 더 높다는 것과 함께, 남아시아와 서아시아가 이 하플로그룹의 근원일지도 모른다는 것을 암시합니다."[20]
- ^ 사후 외. "... 인도의 롤라 계통 사이에서 유전적 변이가 극적으로 낮아질 것으로 기대해야 합니다.사실, 그 반대가 사실입니다: 중앙 아시아(및 동유럽)의 R1a를 배경으로 한 STR 하플로타입 다양성은 이미 인도(6)보다 낮은 것으로 나타났습니다.오히려 (대부분의 경우 R2와 R*가 없는) 중앙아시아 유럽 인구 전체에서 R1*과 Rla의 높은 발병률은 유전자 흐름이 반대 방향으로 더 간결하게 설명되며, 아마도 남아시아 또는 서아시아에서 초기 설립자 효과가 있을 것입니다.[28]
- ^ 샤르마 외. (2009):"브라만스에서 Y-하플로그룹 R1a1*의 가장 높은 빈도(최대 72.22%)를 기록한 특이한 관찰은 이 카스트 그룹의 창시자 혈통으로서의 존재를 암시했습니다.또한 다양한 부족 집단에서 R1a1*의 관찰, 조상에서 Y-하플로그룹 R1a*의 존재 및 530명의 인도인 풀링 데이터셋의 확장된 계통발생학적 분석,224명의 파키스탄인들과 276명의 중앙아시아인들과 276명의 유라시아인들이 R1a1 하플로그룹을 가지고 인도의 R1a1 혈통의 고유한 기원과 인도의 브라만 부족과의 관계를 지지했습니다.그러나 R1a1*의 고해상도를 위해 새로운 Y-염색체 이진 마커를 발견하고 현재의 결론을 확인하는 것이 중요합니다."[1]
- ^ 대부분의 역사동안, 인도 아대륙은 세계에서 가장 인구가 많은 지역이었습니다.
- ^ 센굽타 외.(2006):"유라시아 전역에 걸쳐 HG R1a1-M17의 지리적 분포가 광범위하고 이진 표식에 의해 정의된 정보적 세분화가 없는 현재의 상황은 HG R1a1-M17의 지리적 기원을 불확실하게 합니다.그러나 R1a1-M17 분산의 등고선 지도는 인도 북서부 지역에서 가장 높은 분산을 보여줍니다.L1의 역사가 R1a1 및 R2 대표자의 일부 또는 전부와 얼마나 다른지에 대한 의문이 남아 있습니다.이러한 불확실성은 드라비다어를 사용하는 남부 부족의 북서쪽에서 발생한 HGs R1a1과 R2의 침입이 최근의 단일 사건으로 인한 것이라는 이전의 결론을 무력화시킵니다.[R1a1과 R2]는 실제로 서남아시아 지역에서 인도 남부에 여러 번 도착할 수 있었고, 일부 에피소드는 다른 에피소드보다 훨씬 일찍 도착할 수 있었습니다.인도에 중석기 시대(케네디 2000)가 존재하는 것과 관련하여 상당한 고고학적 증거가 존재하며, 이들 중 일부는 플라이스토세 후기에 북서쪽에서 아대륙으로 유입되었을 수 있습니다.인도의 R1a1의 높은 분산(표 12), R1a1 마이크로위성 분산 라인의 공간 주파수 분포(그림 4), 그리고 확장 시간(표 11)이 이러한 관점을 뒷받침합니다."[25]
- ^ Semenov & Bulat(2016)는 다음과 같은 간행물을 참조합니다.
- Haak, Wolfgang (2015). "Massive migration from the steppe is a source for Indo-European languages in Europe". Nature. 522 (7555): 207–211. arXiv:1502.02783. Bibcode:2015Natur.522..207H. bioRxiv 10.1101/013433. doi:10.1038/NATURE14317. PMC 5048219. PMID 25731166.
- Mathieson, Iain (2015). "Eight thousand years of natural selection in Europe". bioRxiv 10.1101/016477.
- 체쿠노바 е м, 야르트세바 NV, 체쿠노프 м к, м주르케비치 аUpper Podvin'e 고고학 기념관의 원주민 및 인골유해 유전자형의 첫 번째 결과. // IV호 정착촌의 고고학-II 수천 BC:문화와 자연 환경 그리고 기후 리듬의 연대표.국제회의 의사록, 러시아 북서부 말뚝박기 정착지 50년 연구를 중심으로2014년 11월 13일부터 15일까지 상트페테르부르크.
- Jones, ER; Gonzalez-Fortes, G; Connell, S; Siska, V; Eriksson, A; Martiniano, R; McLaughlin, RL; Gallego Llorente, M; Cassidy, LM; Gamba, C; Meshveliani, T; Bar-Yosef, O; Müller, W; Belfer-Cohen, A; Matskevich, Z; Jakeli, N; Higham, TF; Currat, M; Lordkipanidze, D; Hofreiter, M; Manica, A; Pinhasi, R; Bradley, DG (2015). "Upper Palaeolithic genomes reveal deep roots of modern Eurasians". Nat Commun. 6: 8912. Bibcode:2015NatCo...6.8912J. doi:10.1038/ncomms9912. PMC 4660371. PMID 26567969.
- ^ 그러나 Haak et al. 은 또한 "초기 농부들에 의해 도입된 것과는 다른 유형의 근동 조상"을 명시적으로 언급하고 있습니다.[clarification needed][39]
- ^ 가계도 DNA에 의하면 L664는 4,700 ybp, 즉 기원전 2,700 ybp를 형성했습니다.[9]
- ^ 라자리디스, 트위터, 2016년 6월 18일: "I1635 (아르메니아_EBA)는 R1b1-M415(xM269)입니다.우리는 반드시 개정안에 포함시키겠습니다.알아주신 분들께 감사드립니다!#나는 지문을 사랑합니다."[unreliable source?]
동일한 주제에 대한 토론도 참조하십시오. - ^ Y-happlogroup R1a의 깊은 기원에 대한 주요 신규 기사, Dieneke's Anthropology Blog에서 M780 분포 지도 참조 (Underhill et al. 2014)[43]
- ^ Family Tree DNA에 의하면, M780은 4700 ybp를 형성했습니다.[9]이 연대는 기원전 2800년에서 2600년 사이에 얌나야 문화가 인도-이란인들이 기원한 신타슈타 문화의 전신인 폴타브카 문화의 지역으로 동쪽으로 이동한 것과 일치합니다.M780은 고전적인 베다 사회의 중심지인 갠지스 계곡에 집중되어 있습니다.
- ^ Poznik et al. (2016)은 생성 시간을 30년으로 계산하고, 생성 시간을 20년으로 계산하면 다른 결과가 나옵니다.
- ^ "스텝이..."MLBA [중후기 청동기 시대] 클러스터는 남아시아에서 스텝 혈통에 대한 그럴듯한 원천이며, 오늘날 남아시아에서 흔히 볼 수 있는 Z93 아형의 하플로그룹 R1a 또한 Y 염색체 증거에 의해 뒷받침되고 있습니다 [Underhill et al. (2014), Silva et al. (2017)]은 스텝에서 높은 빈도를 보였습니다.MLBA(68%)(16), 스텝에서는 드물지만_EMBA [초기에서 중기 청동기 시대] (자료에 없음)"[44]
- ^ Балановский (2015), p. 208 (in Russian) Прежде всего, это преобладание в славянских популяциях дославянского субстрата — двух ассимилированных ими генетических компонентов – восточноевропейского для западных и восточных славян и южноевропейского для южных славян...Можно с осторожностью предположить, что ассимилированный субстратмог быть представлен по преимуществу балтоязычными популяциями. Действительно, археологические данные указыва ют на очень широкое распространение балтских групп перед началом расселения славян. Балтскийсубстрату славян (правда, наряду с финно-угорским) выявляли и антропологи. Полученные нами генетические данные — и на графиках генетических взаимоотношений, и по доле общих фрагментов генома — указывают, что современные балтские народы являются ближайшими генетически ми соседями восточных славян. При этом балты являются и лингвистически ближайшими род ственниками славян. И можно полагать, что к моменту ассимиляции их генофонд не так сильно отличался от генофонда начавших свое широкое расселение славян. Поэтому если предположить,что расселяющиеся на восток славяне ассимилировали по преимуществу балтов, это может объяснить и сходство современных славянских и балтских народов друг с другом, и их отличия от окружающих их не балто-славянских групп Европы...В работе высказывается осторожное предположение, что ассимилированный субстрат мог быть представлен по преимуществу балтоязычными популяциями. Действительно, археологические данные указывают на очень широкое распространение балтских групп перед началом расселения славян. Балтский субстрат у славян (правда, наряду с финно-угорским) выявляли и антропологи. Полученные в этой работе генетические данные — и на графиках генетических взаимоотношений, и по доле общих фрагментов генома — указывают, что современные балтские народы являются ближайшими генетическими соседями восточных славян.
참고문헌
- ^ a b c d e f 샤르마 외. 2009.
- ^ a b c d e f g h i j k l m n o p q r s t u 언더힐 외. 2014.
- ^ a b c d e f g h i j k l m n o p q r 언더힐 등. 2009.
- ^ a b 언더힐 외. 2014, 페이지 130.
- ^ Saag, Lehti; Vasilyev, Sergey V.; Varul, Liivi; Kosorukova, Natalia V.; Gerasimov, Dmitri V.; Oshibkina, Svetlana V.; Griffith, Samuel J.; Solnik, Anu; Saag, Lauri; D'Atanasio, Eugenia; Metspalu, Ene (January 2021). "Genetic ancestry changes in Stone to Bronze Age transition in the East European plain". Science Advances. 7 (4): eabd6535. Bibcode:2021SciA....7.6535S. doi:10.1126/sciadv.abd6535. PMC 7817100. PMID 33523926.
- ^ Haak, Wolfgang; Lazaridis, Iosif; Patterson, Nick; Rohland, Nadin; Mallick, Swapan; Llamas, Bastien; Brandt, Guido; Nordenfelt, Susanne; Harney, Eadaoin; Stewardson, Kristin; Fu, Qiaomei (February 10, 2015). "Massive migration from the steppe is a source for Indo-European languages in Europe". bioRxiv: 013433. doi:10.1101/013433. S2CID 196643946.
- ^ Raghavan, Maanasa; Skoglund, Pontus; Graf, Kelly E.; Metspalu, Mait; Albrechtsen, Anders; Moltke, Ida; Rasmussen, Simon; Stafford Jr, Thomas W.; Orlando, Ludovic; Metspalu, Ene; Karmin, Monika (January 2014). "Upper Palaeolithic Siberian genome reveals dual ancestry of Native Americans". Nature. 505 (7481): 87–91. Bibcode:2014Natur.505...87R. doi:10.1038/nature12736. PMC 4105016. PMID 24256729.
- ^ Narasimhan, Vagheesh M.; Patterson, Nick; Moorjani, Priya; Rohland, Nadin; Bernardos, Rebecca; Mallick, Swapan; Lazaridis, Iosif; Nakatsuka, Nathan; Olalde, Iñigo; Lipson, Mark; Kim, Alexander M. (September 6, 2019). "The formation of human populations in South and Central Asia". Science. 365 (6457): eaat7487. doi:10.1126/science.aat7487. PMC 6822619. PMID 31488661.
Y chromosome haplogroup types R1b or R1a not represented in Iran and Turan in this period ...
- ^ a b c d e f g h i j "R1a tree". YFull.
- ^ a b c 미라발 외. 2009.
- ^ Zerjal, T.; et al. (1999). "The use of Y-chromosomal DNA variation to investigate population history: recent male spread in Asia and Europe". In Papiha, S. S.; Deka, R. & Chakraborty, R. (eds.). Genomic diversity: applications in human population genetics. New York: Kluwer Academic/Plenum Publishers. pp. 91–101. ISBN 978-0-3064-6295-5.
- ^ a b c d 세미노 외. 2000.
- ^ a b 웰스 2001.
- ^ a b c Pamjav et al. 2012.
- ^ a b 하크 외 2015.
- ^ a b Allentoft 등. 2015.
- ^ a b Mathiesson et al. 2015.
- ^ a b Silva et al. 2017.
- ^ a b Joseph, Tony (June 16, 2017). "How genetics is settling the Aryan migration debate". The Hindu.
- ^ a b c d Kivisild et al. 2003.
- ^ Sengupta S, Zhivotovsky LA, King R, Mehdi SQ, Edmonds CA, Chow CE, et al. (February 2006). "Polarity and temporality of high-resolution y-chromosome distributions in India identify both indigenous and exogenous expansions and reveal minor genetic influence of Central Asian pastoralists". American Journal of Human Genetics. 78 (2): 202–221. doi:10.1086/499411. PMC 1380230. PMID 16400607."남아시아의 사회적 계층과 언어에 미치는 상당한 문화적 영향이 유목민 중앙아시아 목축가들의 도래에 기인하지만, 유전자 데이터(미토콘드리아와 Y 염색체)는 남아시아의 부족과 카스테스의 유전적 기원에 대해 극적으로 상반된 추론을 산출했습니다.우리는 남아시아의 지리적, 사회적, 언어적으로 대표적인 민족 집단의 69개의 유익한 Y-염색체 이진 마커와 10개의 마이크로위성 마커에 대한 고해상도 데이터를 사용하여 이러한 갈등을 해결하고자 했습니다.우리는 중앙아시아가 기존의 유전자 풀에 미치는 영향이 미미하다는 것을 발견했습니다.대부분의 인도 하플로그룹에서 누적된 미세 위성 변이의 나이는 10,000~15,000년을 초과하며, 이는 지역 분화의 고대성을 보여줍니다.따라서 우리의 데이터는 남아시아에서 관찰된 유전적 변이를 설명하기 위해 중앙아시아에서 뚜렷한 최근 유전자 입력을 호출하는 모델을 지원하지 않습니다.R1a1 및 R2 하플로그룹은 최근 단일 이력과 일치하지 않는 인구 통계학적 복잡성을 나타냅니다.고주파 R1A1 하플로그룹 염색체의 관련 미세위성 분석은 인더스 밸리와 반도 인도 지역의 독립적인 최근 역사를 나타냅니다."
- ^ Thanseem I, Thangaraj K, Chaubey G, Singh VK, Bhaskar LV, Reddy BM, et al. (August 2006). "Genetic affinities among the lower castes and tribal groups of India: inference from Y chromosome and mitochondrial DNA". BMC Genetics. 7: 42. doi:10.1186/1471-2156-7-42. PMC 1569435. PMID 16893451.
- ^ Sahoo S, Singh A, Himabindu G, Banerjee J, Sitalaximi T, Gaikwad S, et al. (January 2006). "A prehistory of Indian Y chromosomes: evaluating demic diffusion scenarios". Proceedings of the National Academy of Sciences of the United States of America. 103 (4): 843–848. Bibcode:2006PNAS..103..843S. doi:10.1073/pnas.0507714103. PMC 1347984. PMID 16415161.
- ^ Thangaraj K, Naidu BP, Crivellaro F, Tamang R, Upadhyay S, Sharma VK, et al. (December 2010). Cordaux R (ed.). "The influence of natural barriers in shaping the genetic structure of Maharashtra populations". PLOS ONE. 5 (12): e15283. Bibcode:2010PLoSO...515283T. doi:10.1371/journal.pone.0015283. PMC 3004917. PMID 21187967.
- ^ a b c d e f 센굽타 2006.
- ^ a b c d e 사후 외 2006.
- ^ a b Thangaraj et al. 2010.
- ^ 사후 외. 2006, 페이지 845-846.
- ^ Pande, A. (2022). Birth controlled: Selective reproduction and neoliberal eugenics in South Africa and India. Governing Intimacies in the Global South. Manchester University Press. p. 3-PA1976-3-PA1979. ISBN 978-1-5261-6053-9. Retrieved July 16, 2023. "주류의 관점은 R1a가 인도의 '외부'를 기원하고 인더스 밸리 문명 이후에 도착한 이주자 집단('아리아인')에 의해 인도로 옮겨진다는 것입니다."
- ^ Lalueza-Fox, C. (2022). Inequality: A Genetic History. MIT Press. p. 81-82. ISBN 978-0-262-04678-7. Retrieved July 16, 2023. "몇 년 전, 국내 과학자들은 R1a Y 염색체의 존재가 외국의 유전자 흐름에 기인한 것이 아니라 이 혈통이 아대륙에서 생겨나서 거기서 퍼졌다는 견해를 지지했습니다.그러나 이 하플로그룹의 계통발생학적 재건은 이 견해를 지지하지 않았습니다."
- ^ 앤서니 2007.
- ^ 앤서니 & 링게 2015.
- ^ a b Haak et al. 2015, p. 5.
- ^ Semenov & Bulat 2016.
- ^ Haber et al. 2012"R1a1a7-M458은 아프가니스탄에 존재하지 않았으며, 이는 R1a1a-M17이 이전에 생각했던 [47] 폰틱 스텝[3]에서 인도유럽어족 언어를 중앙아시아와 인도로 가져오는 확장을 지원하지 않음을 시사합니다."
- ^ Klejn, Leo S. (April 22, 2017). "The Steppe Hypothesis of Indo-European Origins Remains to be Proven". Acta Archaeologica. 88 (1): 193–204. doi:10.1111/j.1600-0390.2017.12184.x. ISSN 0065-101X. "Y-염색체에 관해서는 Haak, Lazaridis et al. (2015)에서 사마라의 Yamnaya가 R-M269에 속하지만 서유럽에서 흔히 볼 수 있는 분류군에는 속하지 않는 Y-염색체를 가지고 있음이 이미 지적되었습니다 (보충 46쪽).또한 코드드 웨어(R1a 중심)와 달리 얌나야에서는 단 한 개의 R1a도 없습니다."
- ^ Koch, John T.; Cunliffe, Barry (2016). Celtic from the West 3: Atlantic Europe in the Metal Ages. Oxbow Books. p. 634. ISBN 978-1-78570-228-0.
- ^ Semenov & Bulat 2016, p. 41.
- ^ Haak et al. 2015, p. 4.
- ^ a b c Mascarenhas et al. 2015, p. 9.
- ^ a b 포즈니크 외. 2016, p. 5.
- ^ 아람의 영어 블로그, 고대 근동에서 온 YDNA
- ^ "Dienekes' Anthropology Blog: Major new article on the deep origins of Y-haplogroup R1a (Underhill et al. 2014)". March 27, 2014. Retrieved December 20, 2019.[unre신뢰할 수 있는 출처?]
- ^ a b Narasimhan 외 2018.
- ^ a b c "About Us". Family Tree DNA. Retrieved December 20, 2019.
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa "ISOGG 2017 Y-DNA Haplogroup R". isogg.org. Retrieved December 20, 2019.
- ^ a b c d e f g h i j k "Haplogroup R (Y-DNA) - SNPedia". www.snpedia.com. Retrieved December 20, 2019.
- ^ Karafet et al. 2014.
- ^ a b c d e f g h i j k l m 언더힐 외. 2014, 페이지 125.
- ^ "R1a in Yamnaya". Eurogenes Blog. March 21, 2016. Archived from the original on May 5, 2018. Retrieved December 20, 2019.
- ^ "Y-DNA Haplogroup R and its Subclades". International Society of Genetic Genealogy (ISOGG). Retrieved January 8, 2011.
- ^ Krahn, Thomas. "Draft Y-Chromosome Tree". Family Tree DNA. Archived from the original on May 26, 2013. Retrieved December 7, 2012.
- ^ 레게이루 2006.
- ^ Freder, Janine (2010). Die mittelalterlichen Skelette von Usedom: Anthropologische Bearbeitung unter besonderer Berücksichtigung des ethnischen Hintergrundes [Anthropological investigation in due consideration of the ethnical background] (Thesis) (in German). Freie Universität Berlin. p. 86. doi:10.17169/refubium-8995.
- ^ https://cyberleninka.ru/article/n/tyurki-kavkaza-sravnitelnyy-analiz-genofondov-po-dannym-o-y-hromosome "высокая частота R1a среди кубанских ногайцев (субветвь R1a1a1g-M458 забирает 18%"
- ^ https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2987245/bin/ejhg2009194x4.pdf
- ^ Gwozdz, Peter (August 6, 2018). "Polish Y-DNA Clades".
- ^ 폴로우스키 외 2002.
- ^ a b Gwozdz 2009.
- ^ a b Kars, M. E.; Başak, A. N.; Onat, O. E.; Bilguvar, K.; Choi, J.; Itan, Y.; Çağlar, C.; Palvadeau, R.; Casanova, J. L.; Cooper, D. N.; Stenson, P. D.; Yavuz, A.; Buluş, H.; Günel, M.; Friedman, J. M.; Özçelik, T. (2021). "The genetic structure of the Turkish population reveals high levels of variation and admixture". Proceedings of the National Academy of Sciences of the United States of America. 118 (36): e2026076118. Bibcode:2021PNAS..11826076K. doi:10.1073/pnas.2026076118. PMC 8433500. PMID 34426522.
- ^ Petrejcíková, EVA; Soták, Miroslav; Bernasovská, Jarmila; Bernasovský, Ivan; Sovicová, Adriana; Bôziková, Alexandra; Boronová, Iveta; Švícková, Petra; Gabriková, Dana; MacEková, Sona (2009). "Y-haplogroup frequencies in the Slovak Romany population". Anthropological Science. 117 (2): 89–94. doi:10.1537/ase.080422.
- ^ a b c Saag et al. 2020, p. 5.
- ^ a b Saag et al. 2020, p. 29, 표 1
- ^ Saag et al. 2020, 부가자료 2, 4열
- ^ Fu et al. 2016.
- ^ Saag et al. 2017.
- ^ 앤서니 2019, 16, 17쪽
- ^ a b 하크 외 2008.
- ^ Brandit et al. 2013.
- ^ Malmström et al. 2019, p. 2.
- ^ Saag et al. 2020, 부가자료 2, 5-49열
- ^ Schweitzer, D. (March 23, 2008). "Lichtenstein Cave Data Analysis" (PDF). dirkschweitzer.net. Archived from the original (PDF) on August 14, 2011. Silz의 영어 요약 (2006).
- ^ a b c 키서 등. 2009.
- ^ Ricaut 등. 2004.
- ^ 코르니엔코, I.V.; Vodolazhsky D.I. "Использование нерекомбинантных маркеров Y-хромосомы в исследованиях древних популяций (на примере поселения Танаис)" [The use of non-recombinant markers of the Y-chromosome in the study of ancient populations (on the example of the settlement of Tanais)].Материалы Донских антропологических чтений [Materials of the Don Anthropological Readings].Rostov-on-Don: Rostov 연구소 종양학, 2013
- ^ 리춘샹 외 2010. 2010
- ^ Kim et al. 2010.
- ^ Balanovsky et al.
- ^ Behar et al. 2003.
- ^ 2005년 Kasperaviciute, Kucinskas & Stoneking.
- ^ a b Battaglia et al. 2008.
- ^ a b 로서 외. 2000.
- ^ 탐베츠 외. 2004.
- ^ Bowden et al. 2008.
- ^ Dupuy et al. 2005.
- ^ Passarino et al. 2002.
- ^ 카펠리 외 2003.
- ^ Kayser et al. 2005.
- ^ 스코자리 외 2001.
- ^ Underhill, Peter A. (January 1, 2015). "The phylogenetic and geographic structure of Y-chromosome haplogroup R1a". European Journal of Human Genetics. 23 (1): 124–131. doi:10.1038/ejhg.2014.50. PMC 4266736. PMID 24667786.
- ^ L. Barać; et al. (2003). "Y chromosomal heritage of Croatian population and its island isolates" (PDF). European Journal of Human Genetics. 11 (7): 535–42. doi:10.1038/sj.ejhg.5200992. PMID 12825075. S2CID 15822710.
- ^ S. Rootsi; et al. (2004). "Phylogeography of Y-Chromosome Haplogroup I Reveals Distinct Domains of Prehistoric Gene Flow in Europe" (PDF). American Journal of Human Genetics. 75 (1): 128–137. doi:10.1086/422196. PMC 1181996. PMID 15162323.
- ^ M. Peričić; et al. (2005). "High-resolution phylogenetic analysis of southeastern Europe traces major episodes of paternal gene flow among Slavic populations". Molecular Biology and Evolution. 22 (10): 1964–75. doi:10.1093/molbev/msi185. PMID 15944443.
- ^ M. Peričić; et al. (2005). "Review of Croatian Genetic Heritage as Revealed by Mitochondrial DNA and Y Chromosomal Lineages". Croatian Medical Journal. 46 (4): 502–513. PMID 16100752.
- ^ 페리치 외 2005.
- ^ "Untitled". pereformat.ru (in Russian).
- ^ "Untitled". www.rodstvo.ru.
- ^ Zerjal et al. 2002.
- ^ Haber et al. 2012.
- ^ a b Di Christofaro et al. 2013
- ^ a b Malyarchuk et al. 2013.
- ^ Ashirbekov et al. 2017.
- ^ 샤 2011.
- ^ 2012년 아룬쿠마르.
- ^ Toomas Kivisild; Siiri Rootsi; Mait Metspalu; Ene Metspalu; Juri Parik; Katrin Kaldma; Esien Usanga; Sarabjit Mastana; Surinder S. Papiha; Richard Villems. "The Genetics of Language and Farming Spread in India" (PDF). In P. Bellwwood; C. Renfrew (eds.). Examining the farming/language dispersal hypothesis. McDonald Institute Monographs. Cambridge University. pp. 215–222. Retrieved December 20, 2019.
- ^ 포르나리노 외. 2009.
- ^ Wang et al. 2003.
- ^ Zhou et al. 2007.
- ^ 류슈후 외 2018.
- ^ Zhong 등 2011.
- ^ Zhong, Hua; Shi, Hong; Qi, Xue-Bin; Duan, Zi-Yuan; Tan, Ping-Ping; Jin, Li; Su, Bing; Ma, Runlin Z. (2011). "Extended Y Chromosome Investigation Suggests Postglacial Migrations of Modern Humans into East Asia via the Northern Route". Molecular Biology and Evolution. 28 (1): 717–727. doi:10.1093/molbev/msq247. PMID 20837606.
- ^ Shou, Wei-Hua; Qiao, Wn-Fa; Wei, Chuan-Yu; Dong, Yong-Li; Tan, Si-Jie; Shi, Hong; Tang, Wen-Ru; Xiao, Chun-Jie (2010). "Y-chromosome distributions among populations in Northwest China identify significant contribution from Central Asian pastoralists and lesser influence of western Eurasians". Journal of Human Genetics. 55 (5): 314–322. doi:10.1038/jhg.2010.30. PMID 20414255. S2CID 23002493.
- ^ Lell et al. 2002.
- ^ 모하마드 외. 2009.
- ^ Nasidze 외. 2004.
- ^ Nasidze 외 2005.
- ^ Grugni et al. 2012.
- ^ "R1a1a conquers the world...in a few pulses?". Discover Magazine.
원천
- Allentoft, Morten E.; Sikora, Martin; Sjögren, Karl-Göran; Rasmussen, Simon; Rasmussen, Morten; Stenderup, Jesper; Damgaard, Peter B.; Schroeder, Hannes; et al. (2015). "Population genomics of Bronze Age Eurasia". Nature. 522 (7555): 167–172. Bibcode:2015Natur.522..167A. doi:10.1038/nature14507. PMID 26062507. S2CID 4399103.
- Anthony, David W. (2007), The Horse The Wheel And Language. How Bronze-Age Riders From the Eurasian Steppes Shaped The Modern World, Princeton University Press
- Anthony, David (Spring–Summer 2019). "Archaeology, Genetics, and Language in the Steppes: A Comment on Bomhard". Journal of Indo-European Studies. 47 (1–2). Retrieved January 9, 2020.
- Anthony, David; Ringe, Don (2015), "The Indo-European Homeland from Linguistic and Archaeological Perspectives", Annual Review of Linguistics, 1: 199–219, doi:10.1146/annurev-linguist-030514-124812
- Shah, A. M.; Tamang, R.; Moorjani, P.; Rani, D. S.; Govindaraj, P.; Kulkarni, G.; Bhattacharya, T.; Mustak, M. S.; Bhaskar, L. V. K. S.; Reddy, A. G.; Gadhvi, D.; Gai, P. B.; Chaubey, G.; Patterson, N.; Reich, D.; Tyler-Smith, C.; Singh, L.; Thangaraj, K. (2011). "Indian Siddis: African Descendants with Indian Admixture". The American Journal of Human Genetics. 89 (1): 154–61. doi:10.1016/j.ajhg.2011.05.030. PMC 3135801. PMID 21741027.
- ArunKumar, G; Soria-Hernanz, DF; Kavitha, VJ; Arun, VS; Syama, A; Ashokan, KS (2012). "Population Differentiation of Southern Indian Male Lineages Correlates with Agricultural Expansions Predating the Caste System". PLOS ONE. 7 (11): e50269. Bibcode:2012PLoSO...750269A. doi:10.1371/journal.pone.0050269. PMC 3508930. PMID 23209694.
- Ashirbekov, E. E.; et al. (2017). "Distribution of Y-Chromosome Haplogroups of the Kazakh from the South Kazakhstan, Zhambyl, and Almaty Regions" (PDF). Reports of the National Academy of Sciences of the Republic of Kazakhstan. 6 (316): 85–95. Archived from the original (PDF) on September 23, 2021. Retrieved June 4, 2020.
- Balanovsky O, Rootsi S, Pshenichnov A, Kivisild T, Churnosov M, Evseeva I, Pocheshkhova E, Boldyreva M, et al. (2008). "Two Sources of the Russian Patrilineal Heritage in Their Eurasian Context". American Journal of Human Genetics. 82 (1): 236–250. doi:10.1016/j.ajhg.2007.09.019. PMC 2253976. PMID 18179905.
- Балановский, О. П. (November 30, 2015). Генофонд Европы (in Russian). KMK Scientific Press. ISBN 9785990715707.
- Battaglia V, Fornarino S, Al-Zahery N, Olivieri A, Pala M, Myres NM, King RJ, Rootsi S, et al. (2008). "Y-chromosomal evidence of the cultural diffusion of agriculture in southeast Europe". European Journal of Human Genetics. 17 (6): 820–30. doi:10.1038/ejhg.2008.249. PMC 2947100. PMID 19107149.
- Behar D, Thomas MG, Skorecki K, Hammer MF, Bulygina E, Rosengarten D, Jones AL, Held K, et al. (2003). "Multiple Origins of Ashkenazi Levites: Y Chromosome Evidence for Both Near Eastern and European Ancestries" (PDF). American Journal of Human Genetics. 73 (4): 768–779. doi:10.1086/378506. PMC 1180600. PMID 13680527.
- Bowden GR, Balaresque P, King TE, Hansen Z, Lee AC, Pergl-Wilson G, Hurley E, Roberts SJ, et al. (2008). "Excavating Past Population Structures by Surname-Based Sampling: The Genetic Legacy of the Vikings in Northwest England". Molecular Biology and Evolution. 25 (2): 301–309. doi:10.1093/molbev/msm255. PMC 2628767. PMID 18032405.
- Brandit, G.; et al. (The Genographic Consortium) (2013). "Ancient DNA Reveals Key Stages in the Formation of Central European Mitochondrial Genetic Diversity". Science. 342 (6155): 257–261. Bibcode:2013Sci...342..257B. doi:10.1126/science.1241844. PMC 4039305. PMID 24115443.
- Capelli C, Redhead N, Abernethy JK, Gratrix F, Wilson JF, Moen T, Hervig T, Richards M, et al. (2003). "A Y Chromosome Census of the British Isles" (PDF). Current Biology. 13 (11): 979–84. doi:10.1016/S0960-9822(03)00373-7. PMID 12781138. S2CID 526263. 에도
- Chunxiang Li; Hongjie Li; Yinqiu Cui; Chengzhi Xie; Dawei Cai; Wenying Li; Victor H Mair; Zhi Xu; et al. (2010). "Evidence that a West-East admixed population lived in the Tarim Basin as early as the early Bronze Age". BMC Biology. 8 (1): 15. doi:10.1186/1741-7007-8-15. PMC 2838831. PMID 20163704.
- Di Cristofaro J, Pennarun E, Mazières S, Myres NM, Lin AA, Temori SA, Metspalu M, Metspalu E, et al. (2013). "Afghan Hindu Kush: Where Eurasian Sub-Continent Gene Flows Converge". PLOS ONE. 8 (10). e76748. Bibcode:2013PLoSO...876748D. doi:10.1371/journal.pone.0076748. PMC 3799995. PMID 24204668.
- Dupuy BM, Stenersen M, Lu TT, Olaisen B (2005). "Geographical heterogeneity of Y-chromosomal lineages in Norway" (PDF). Forensic Science International. 164 (1): 10–19. doi:10.1016/j.forsciint.2005.11.009. PMID 16337760.
- Fornarino, Simona; Pala, Maria; Battaglia, Vincenza; Maranta, Ramona; Achilli, Alessandro; Modiano, Guido; Torroni, Antonio; Semino, Ornella; et al. (2009). "Mitochondrial and Y-chromosome diversity of the Tharus (Nepal): a reservoir of genetic variation". BMC Evolutionary Biology. 9: 154. doi:10.1186/1471-2148-9-154. PMC 2720951. PMID 19573232.
- Fu, Qiaomei; et al. (May 2, 2016). "The genetic history of Ice Age Europe". Nature. 534 (7606): 200–205. Bibcode:2016Natur.534..200F. doi:10.1038/nature17993. hdl:10211.3/198594. PMC 4943878. PMID 27135931.
- Grugni V, Battaglia V, Kashani BH, Parolo S, Al-Zahery N, Achilli A, Olivieri A, Gandini F, Houshmand M, Sanati MH, Torroni A, Semino O (2012). "Ancient Migratory Events in the Middle East: New Clues from the Y-Chromosome Variation of Modern Iranians". PLOS ONE. 7 (7). e41252. Bibcode:2012PLoSO...741252G. doi:10.1371/journal.pone.0041252. PMC 3399854. PMID 22815981.
- Gwozdz (2009). "Y-STR Mountains in Haplospace, Part II: Application to Common Polish Clades" (PDF). Journal of Genetic Genealogy. 5 (2).
- Haak, W.; Brandt, G.; Jong, H. N. d.; Meyer, C.; Ganslmeier, R.; Heyd, V.; Hawkesworth, C.; Pike, A. W. G.; et al. (2008). "Ancient DNA, Strontium isotopes, and osteological analyses shed light on social and kinship organization of the Later Stone Age". Proceedings of the National Academy of Sciences. 105 (47): 18226–18231. Bibcode:2008PNAS..10518226H. doi:10.1073/pnas.0807592105. PMC 2587582. PMID 19015520.
- Haak, Wolfgang; Lazaridis, Iosif; Patterson, Nick; Rohland, Nadin; Mallick, Swapan; Llamas, Bastien; Brandt, Guido; Nordenfelt, Susanne; et al. (2015). "Massive migration from the steppe is a source for Indo-European languages in Europe". Nature. 522 (7555): 207–211. arXiv:1502.02783. Bibcode:2015Natur.522..207H. bioRxiv 10.1101/013433. doi:10.1038/NATURE14317. PMC 5048219. PMID 25731166.
- Haber M, Platt DE, Ashrafian Bonab M, Youhanna SC, Soria-Hernanz DF, Martínez-Cruz B, Douaihy B, Ghassibe-Sabbagh M, et al. (2012). "Afghanistan's ethnic groups share a Y-chromosomal heritage structured by historical events". PLOS ONE. 7 (3). e34288. Bibcode:2012PLoSO...734288H. doi:10.1371/journal.pone.0034288. PMC 3314501. PMID 22470552.
- Karafet, Tatiana M.; Mendez, Fernando L.; Sudoyo, Herawati; Lansing, J. Stephen; Hammer, Michael F. (2014). "Improved phylogenetic resolution and rapid diversification of Y-chromosome haplogroup K-M526 in Southeast Asia". Nature. 23 (3): 369–373. doi:10.1038/ejhg.2014.106. PMC 4326703. PMID 24896152.
- Kasperaviciūte, D.; Kucinskas, V.; Stoneking, M. (2005). "Y Chromosome and Mitochondrial DNA Variation in Lithuanians". Annals of Human Genetics. 68 (5): 438–452. doi:10.1046/j.1529-8817.2003.00119.x. PMID 15469421. S2CID 26562505.
- Kayser M, Lao O, Anslinger K, Augustin C, Bargel G, Edelmann J, Elias S, Heinrich M, et al. (2005). "Significant genetic differentiation between Poland and Germany follows present-day political borders, as revealed by Y-chromosome analysis" (PDF). Human Genetics. 117 (5): 428–443. doi:10.1007/s00439-005-1333-9. PMID 15959808. S2CID 11066186. Archived from the original (PDF) on March 4, 2009.
- Keyser, Christine; Bouakaze, Caroline; Crubézy, Eric; Nikolaev, Valery G.; Montagnon, Daniel; Reis, Tatiana; Ludes, Bertrand (2009). "Ancient DNA provides new insights into the history of south Siberian Kurgan people". Human Genetics. 126 (3): 395–410. doi:10.1007/s00439-009-0683-0. PMID 19449030. S2CID 21347353.
- Kim, Kijeong; Brenner, Charles H.; Mair, Victor H.; Lee, Kwang-Ho; Kim, Jae-Hyun; Gelegdorj, Eregzen; Batbold, Natsag; Song, Yi-Chung; et al. (2010). "A western Eurasian male is found in 2000-year-old elite Xiongnu cemetery in Northeast Mongolia". American Journal of Physical Anthropology. 142 (3): 429–440. doi:10.1002/ajpa.21242. PMID 20091844.
- Kivisild, T; Rootsi, S; Metspalu, M; Mastana, S; Kaldma, K; Parik, J; Metspalu, E; Adojaan, M; et al. (2003). "The Genetic Heritage of the Earliest Settlers Persists Both in Indian Tribal and Caste Populations". AJHG. 72 (2): 313–32. doi:10.1086/346068. PMC 379225. PMID 12536373.
- Lazaridis, Iosif; et al. (2016). "Genomic insights into the origin of farming in the ancient Near East". Nature. 536 (7617): 419–424. Bibcode:2016Natur.536..419L. doi:10.1038/nature19310. PMC 5003663. PMID 27459054.
- Lell JT, Sukernik RI, Starikovskaya YB, Su B, Jin L, Schurr TG, Underhill PA, Wallace DC (2002). "The Dual Origin and Siberian Affinities of Native American Y Chromosomes" (PDF). American Journal of Human Genetics. 70 (1): 192–206. doi:10.1086/338457. PMC 384887. PMID 11731934. Archived from the original (PDF) on April 22, 2003.
- Liu Shu-hu; Nizam Yilihamu; Rabiyamu Bake; Abdukeram Bupatima; Dolkun Matyusup (2018). "A study of genetic diversity of three isolated populations in Xinjiang using Y-SNP". Acta Anthropologica Sinica. 37 (1): 146–156.
- Carlos Quiles (September 10, 2018). "A study of genetic diversity of three isolated populations in Xinjiang using Y-SNP". Indo-European.
- Malmström, Helena; Günther, Torsten; Svensson, Emma M.; Juras, Anna; Fraser, Magdalena; Munters, Arielle R.; Pospieszny, Łukasz; Tõrv, Mari; et al. (October 9, 2019). "The genomic ancestry of the Scandinavian Battle Axe Culture people and their relation to the broader Corded Ware horizon". Proceedings of the Royal Society B. 286 (1912). doi:10.1098/rspb.2019.1528. PMC 6790770. PMID 31594508.
- Malyarchuk, Boris; Derenko, Miroslava; Wozniak, Marcin; Grzybowski, Tomasz (2013). "Y-chromosome variation in Tajiks and Iranians". Annals of Human Biology. 40 (1): 48–54. doi:10.3109/03014460.2012.747628. PMID 23198991. S2CID 2752490.
- Mascarenhas, Desmond D.; Raina, Anupuma; Aston, Christopher E.; Sanghera, Dharambir K. (2015). "Genetic and Cultural Reconstruction of the Migration of an Ancient Lineage". BioMed Research International. 2015: 651415. doi:10.1155/2015/651415. PMC 4605215. PMID 26491681.
- Mathieson, Iain; Lazaridis, Iosif; Rohland, Nadin; Mallick, Swapan; Patterson, Nick; Alpaslan Roodenberg, Songul; Harney, Eadaoin; Stewardson, Kristin; et al. (2015). "Eight thousand years of natural selection in Europe". bioRxiv 10.1101/016477.
- Mirabal, Sheyla; Regueiro, M; Cadenas, AM; Cavalli-Sforza, LL; Underhill, PA; Verbenko, DA; Limborska, SA; Herrera, RJ; et al. (2009). "Y-Chromosome distribution within the geo-linguistic landscape of northwestern Russia". European Journal of Human Genetics. 17 (10): 1260–1273. doi:10.1038/ejhg.2009.6. PMC 2986641. PMID 19259129.
- Mohammad T, Xue Y, Evison M, Tyler-Smith C (2009). "Genetic structure of nomadic Bedouin from Kuwait". Heredity. 103 (5): 425–433. doi:10.1038/hdy.2009.72. PMC 2869035. PMID 19639002.
- Narasimhan, Vagheesh M.; Anthony, David; Mallory, James; Reich, David (2018). "The Genomic Formation of South and Central Asia". bioRxiv 10.1101/292581.
- Nasidze I, Ling EY, Quinque D, Dupanloup I, Cordaux R, Rychkov S, Naumova O, Zhukova O, et al. (2004). "Mitochondrial DNA and Y-Chromosome Variation in the Caucasus" (PDF). Annals of Human Genetics. 68 (Pt 3): 205–221. doi:10.1046/j.1529-8817.2004.00092.x. PMID 15180701. S2CID 27204150. Archived from the original (PDF) on October 30, 2004.
- Nasidze I, Quinque D, Ozturk M, Bendukidze N, Stoneking M (2005). "MtDNA and Y-chromosome Variation in Kurdish Groups" (PDF). Annals of Human Genetics. 69 (Pt 4): 401–412. doi:10.1046/j.1529-8817.2005.00174.x. PMID 15996169. S2CID 23771698. Archived from the original (PDF) on August 23, 2009.
- Pamjav, Horolma; Fehér, Tibor; Németh, Endre; Pádár, Zsolt (2012), "Brief communication: new Y-chromosome binary markers improve phylogenetic resolution within haplogroup R1a1", American Journal of Physical Anthropology, 149 (4): 611–615, doi:10.1002/ajpa.22167, PMID 23115110, S2CID 4820868
- Passarino G, Cavalleri GL, Lin AA, Cavalli-Sforza LL, Børresen-Dale AL, Underhill (2002). "Different genetic components in the Norwegian population revealed by the analysis of mtDNA and Y chromosome polymorphisms". European Journal of Human Genetics. 10 (9): 521–529. doi:10.1038/sj.ejhg.5200834. PMID 12173029.
- Pathak, Ajai K.; Kadian, Anurag; Kushniarevich, Alena; Montinaro, Francesco; Mondal, Mayukh; Ongaro, Linda; Singh, Manvendra; Kumar, Pramod; et al. (December 6, 2018). "The Genetic Ancestry of Modern Indus Valley Populations from Northwest India". The American Journal of Human Genetics. 103 (6): 918–929. doi:10.1016/j.ajhg.2018.10.022. PMC 6288199. PMID 30526867.
- Pawlowski, R; Dettlaff-Kakol, A; MacIejewska, A; Paszkowska, R; Reichert, M; Jezierski, G (2002). "Population genetics of 9 Y-chromosome STR loci w Northern Poland". Arch. Med. Sadowej Kryminol. 52 (4): 261–277. PMID 14669672.
- Pericić M, Lauc LB, Klarić IM, Rootsi S, Janićijević B, Rudan I, Terzić R, Colak I, et al. (2005). "High-resolution phylogenetic analysis of southeastern Europe traces major episodes of paternal gene flow among Slavic populations". Mol. Biol. Evol. 22 (10): 1964–75. doi:10.1093/molbev/msi185. PMID 15944443.
- Poznik GD, et al. (2016). "Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences". Nature Genetics. 48 (6): 593–599. doi:10.1038/ng.3559. hdl:11858/00-001M-0000-002A-F024-C. PMC 4884158. PMID 27111036.
- Regueiro, M; Cadenas, AM; Gayden, T; Underhill, PA; Herrera, RJ (2006). "Iran: Tricontinental Nexus for Y-Chromosome Driven Migration". Hum Hered. 61 (3): 132–143. doi:10.1159/000093774. PMID 16770078. S2CID 7017701.
- Ricaut F, Keyser-Tracqui C, Bourgeois I, Crubézy E, Ludes B (2004). "Genetic Analysis of a Scytho-Siberian Skeleton and Its Implications for Ancient Central Asian Migrations". Human Biology. 76 (1): 109–25. doi:10.1353/hub.2004.0025. PMID 15222683. S2CID 35948291.
- Rosser ZH, Zerjal T, Hurles ME, Adojaan M, Alavantic D, Amorim A, Amos W, Armenteros M, et al. (2000). "Y-Chromosomal Diversity in Europe Is Clinal and Influenced Primarily by Geography, Rather than by Language". American Journal of Human Genetics. 67 (6): 1526–1543. doi:10.1086/316890. PMC 1287948. PMID 11078479.
- Saag, Lehti; Varul, Liivi; Scheib, Christiana Lyn; Stenderup, Jesper; Allentoft, Morten E.; Saag, Lauri; Pagani, Luca; Reidla, Maere; et al. (July 24, 2017). "Extensive Farming in Estonia Started through a Sex-Biased Migration from the Steppe". Current Biology. Cell Press. 27 (14): 2185–2193. doi:10.1016/j.cub.2017.06.022. PMID 28712569.
- Saag, Lehti; Vasilyev, Sergey V.; Varul, Liivi; Kosorukova, Natalia V.; Gerasimov, Dmitri V.; Oshibkina, Svetlana V.; Griffith, Samuel J.; Solnik, Anu; et al. (July 3, 2020). "Genetic ancestry changes in Stone to Bronze Age transition in the East European plain". bioRxiv 10.1101/2020.07.02.184507.
- Sahoo, S; Singh, A; Himabindu, G; Banerjee, J; Sitalaximi, T; Gaikwad, S; Trivedi, R; Endicott, P; et al. (2006). "A prehistory of Indian Y chromosomes: Evaluating demic diffusion scenarios". Proceedings of the National Academy of Sciences. 103 (4): 843–848. Bibcode:2006PNAS..103..843S. doi:10.1073/pnas.0507714103. PMC 1347984. PMID 16415161.
- Scozzari R, Cruciani F, Pangrazio A, Santolamazza P, Vona G, Moral P, Latini V, Varesi L, et al. (2001). "Human Y-Chromosome Variation in the Western Mediterranean Area: Implications for the Peopling of the Region" (PDF). Human Immunology. 62 (9): 871–84. CiteSeerX 10.1.1.408.4857. doi:10.1016/S0198-8859(01)00286-5. PMID 11543889.
- Semenov, Alexander S.; Bulat, Vladimir V. (2016), "Ancient Paleo-DNA of Pre-Copper Age North-Eastern Europe: Establishing the Migration Traces of R1a1 Y-DNA Haplogroup", European Journal of Molecular Biotechnology, 11 (1): 40–54, doi:10.13187/ejmb.2016.11.40, S2CID 172131289
- Semino, O; Passarino, G; Oefner, PJ; Lin, AA; Arbuzova, S; Beckman, LE; De Benedictis, G; Francalacci, P; et al. (2000). "The Genetic Legacy of Paleolithic Homo sapiens sapiens in Extant Europeans: A Y Chromosome Perspective" (PDF). Science. 290 (5494): 1155–1159. Bibcode:2000Sci...290.1155S. doi:10.1126/science.290.5494.1155. PMID 11073453. Archived from the original (PDF) on November 25, 2003.
- Sengupta, S; Zhivotovsky, LA; King, R; Mehdi, SQ; Edmonds, CA; Chow, CE; Lin, AA; Mitra, M; et al. (2006). "Polarity and Temporality of High-Resolution Y-Chromosome Distributions in India Identify Both Indigenous and Exogenous Expansions and Reveal Minor Genetic Influence of Central Asian Pastoralists". American Journal of Human Genetics. 78 (2): 202–21. doi:10.1086/499411. PMC 1380230. PMID 16400607.
- Sharma, S; Rai, E; Sharma, P; Jena, M; Singh, S; Darvishi, K; Bhat, AK; Bhanwer, AJ; et al. (2009). "The Indian origin of paternal haplogroup R1a1(*)substantiates the autochthonous origin of Brahmins and the caste system". Journal of Human Genetics. 54 (1): 47–55. doi:10.1038/jhg.2008.2. PMID 19158816.
- Schilz, Felix (2006). Molekulargenetische Verwandtschaftsanalysen am prähistorischen Skelettkollektiv der Lichtensteinhöhle [Molecular genetic kinship analysis on the prehistoric skeleton collective of the Lichtenstein Cave] (PDF) (Dissertation) (in German). Göttingen: Mathematisch-Naturwissenschaftlichen Fakultäten der Georg-August-Universität.
- Silva, Marina; et al. (2017). "A genetic chronology for the Indian Subcontinent points to heavily sex-biased dispersals". BMC Evolutionary Biology. 17 (1): 88. doi:10.1186/s12862-017-0936-9. PMC 5364613. PMID 28335724.
- Tambets K, Rootsi S, Kivisild T, Help H, Serk P, Loogväli EL, Tolk HV, Reidla M, et al. (2004). "The Western and Eastern Roots of the Saami—the Story of Genetic 'Outliers' Told by Mitochondrial DNA and Y Chromosomes". American Journal of Human Genetics. 74 (4): 661–682. doi:10.1086/383203. PMC 1181943. PMID 15024688.
- Thangaraj, Kumarasamy; Naidu, B. Prathap; Crivellaro, Federica; Tamang, Rakesh; Upadhyay, Shashank; Sharma, Varun Kumar; Reddy, Alla G.; Walimbe, S. R.; et al. (2010). Cordaux, Richard (ed.). "The Influence of Natural Barriers in Shaping the Genetic Structure of Maharashtra Populations". PLOS ONE. 5 (12): e15283. Bibcode:2010PLoSO...515283T. doi:10.1371/journal.pone.0015283. PMC 3004917. PMID 21187967.
- Underhill, PA; Myres, NM; Rootsi, S; Metspalu, M; Zhivotovsky, LA; King, RJ; Lin, AA; Chow, CE; et al. (November 4, 2009). "Separating the post-Glacial coancestry of European and Asian Y chromosomes within haplogroup R1a". European Journal of Human Genetics (published April 2010). 18 (4): 479–84. doi:10.1038/ejhg.2009.194. PMC 2987245. PMID 19888303.
- Underhill, Peter A.; et al. (March 26, 2014). "The phylogenetic and geographic structure of Y-chromosome haplogroup R1a". European Journal of Human Genetics (published January 2015). 23 (1): 124–131. doi:10.1038/ejhg.2014.50. PMC 4266736. PMID 24667786. "PDF" (PDF). Archived from the original (PDF) on August 16, 2016. Retrieved June 12, 2016.
- Wang, Wei; Wise, Cheryl; Baric, Tom; Black, Michael L.; Bittles, Alan H. (2003). "The origins and genetic structure of three co-resident Chinese Muslim populations: The Salar, Bo'an and Dongxiang". Human Genetics. 113 (3): 244–52. doi:10.1007/s00439-003-0948-y. PMID 12759817. S2CID 11138499.
- Wells, R.S. (2001), "The Eurasian Heartland: A continental perspective on Y-chromosome diversity", Proceedings of the National Academy of Sciences of the USA, 98 (18): 10244–10249, Bibcode:2001PNAS...9810244W, doi:10.1073/pnas.171305098, PMC 56946, PMID 11526236
- Yan, Shi; Wang, Chuan-Chao; Zheng, Hong-Xiang; Wang, Wei; Qin, Zhen-Dong; Wei, Lan-Hai; Wang, Yi; Pan, Xue-Dong; et al. (August 29, 2014). "Y Chromosomes of 40% Chinese Descend from Three Neolithic Super-Grandfathers". PLOS ONE. 9 (8). e105691. arXiv:1310.3897. Bibcode:2014PLoSO...9j5691Y. doi:10.1371/journal.pone.0105691. PMC 4149484. PMID 25170956.
- Y Chromosome Consortium "YCC" (2002). "A Nomenclature System for the Tree of Human Y-Chromosomal Binary Haplogroups". Genome Research. 12 (2): 339–348. doi:10.1101/gr.217602. PMC 155271. PMID 11827954.
- Zerjal, Tatiana; Wells, R. Spencer; Yuldasheva, Nadira; Ruzibakiev, Ruslan; Tyler-Smith, Chris (2002). "A Genetic Landscape Reshaped by Recent Events: Y-Chromosomal Insights into Central Asia". The American Journal of Human Genetics. 71 (3): 466–82. doi:10.1086/342096. PMC 419996. PMID 12145751.
- Zhong H, Shi H, Qi XB, Duan Y, Tan PP, Jin L, SU B, Ma RZ (January 2011). "Extended Y chromosome investigation suggests postglacial migrations of modern humans into East Asia via the northern route". Molecular Biology and Evolution. 28 (1): 717–27. doi:10.1093/molbev/msq247. PMID 20837606.
- Zhou, Ruixia; An, Lizhe; Wang, Xunling; Shao, Wei; Lin, Gonghua; Yu, Weiping; Yi, Lin; Xu, Shijian; et al. (2007). "Testing the hypothesis of an ancient Roman soldier origin of the Liqian people in northwest China: a Y-chromosome perspective". Journal of Human Genetics. 52 (7): 584–91. doi:10.1007/s10038-007-0155-0. PMID 17579807.
추가열람
- Gimbutas (1970). Indo-European and Indo-Europeans. Philadelphia, PA: Univ. of Pennsylvania Press. pp. 155–195.
- Patrizia Malaspina; Andrey I. Kozlov; Fulvio Cruciani; Piero Santolamazza; Nejat Akar; Dimiter Kovatchev; Marina G. Kerimova; Juri Parik; Richard Villems; Rosana Scozzari; Andrea Novelletto (2003). "Analysis of Y-chromosome variation in modern populations at the European-Asian border" (PDF). In K. Boyle; C. Renfrew; M. Levine (eds.). Ancient interactions: east and west in Eurasia. McDonald Institute Monographs. Cambridge: Cambridge University Press. pp. 309–313.
- Parpola, Asko (2015). The Roots of Hinduism. The early Aryans and the Indus Civilisation. Oxford University Press.
- Sharma, S. (2007). "1344/T: The Autochthonous Origin and a Tribal Link of Indian Brahmins: Evaluation Through Molecular Genetic Markers". The American Society of Human Genetics 57th Annual Meeting October 23–27, 2007; San Diego, California (PDF) (Abstract). p. 273. Archived from the original (PDF) on June 26, 2008.
- Trautmann, Thomas (2005). The Aryan Debate. Oxford University Press.
- Varzari, Alexander (2006). Population History of the Dniester-Carpathians: Evidence from Alu Insertion and Y-Chromosome Polymorphisms (PDF) (Dissertation). München: Ludwig-Maximilians-Universität.
- Wells, Spencer (2002). The Journey of Man: A Genetic Odyssey. Princeton University Press. ISBN 978-0-691-11532-0.
저널 기사 |
|---|
|
외부 링크
- DNA 트리
- FTDNA R1a Y-염색체 하플로그룹 프로젝트
- R1a1a1 및 하위 클래스 Y-DNA 프로젝트 – 배경 가계도 DNA R1a1a1
- TMRCA
- 여러가지
- 덴마크 데메스 지역 DNA 프로젝트: Y-DNA 하플로그룹 R1a 2008년 8월 20일 웨이백 머신에서 보관
- 유로진스 블로그, 폴토프카 아웃트라이어
- 세상을 감동시킨 차딕인 슈폴러 제이다의 Y-DNA 지문인 아보타이누 온라인


