리보핵산가수분해효소T
Ribonuclease T| 리보핵산가수분해효소T | |||||||||
|---|---|---|---|---|---|---|---|---|---|
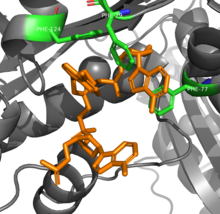
 PDB ID 3NH1에서 [1]DNA(주황색)와 복합체인 리보핵산가수분해효소 T이합체. | |||||||||
| 식별자 | |||||||||
| 기호. | 하지 않다 | ||||||||
| 팜 | PF00929 | ||||||||
| 인터프로 | IPR013520 | ||||||||
| 스마트 | SM00479 | ||||||||
| |||||||||
리보핵산가수분해효소 T(RNase T, exonuclease T, exo T)는 박테리아에서 [2]전달 RNA 및 리보솜 RNA의 성숙 및 DNA 복구 [3]경로에 관여하는 리보핵산가수분해효소이다.DnaQ 계열의 엑소뉴클라아제이며, 단일가닥 핵산의 3' 말단에 비프로세서적으로 작용한다.RNase T는 기질의 [1][2]3' 말단에서 시토신을 구별하는 극단적인 배열 특이성으로 DNA와 RNA를 모두 절단할 수 있다.
구조 및 메커니즘
RNAse T는 RNA와 DNA의 3' 말단에서 뉴클레오티드의 제거를 촉매한다.그것은 RNA의 3' 말단에 있는 시토신 잔기뿐만 아니라 이중 가닥 DNA와 RNA에 의해 억제된다.RNA의 3' 말단에 있는 두 개의 세포신은 RNAse T의 활성을 [3]완전히 제거하는 것으로 보인다.그러나 이러한 시토신 효과는 ssDNA에서 덜 관찰된다.ssDNA의 이러한 배열 특이성의 결여와 이중화 영역에 가까운 ssDNA에 작용하는 그것의 능력으로 인해,[4] DNA 복제를 위한 무딘 끝을 만드는 데 사용되어 왔다.구조적으로 RNAse T는 반평행 이합체로[5][6] 존재하며 기능하려면 [7]2가 캐티온이 필요합니다.
RNAse T는 핵염기 사이에 끼어 있는 여러 방향족 잔류물을 통해 RNA 소화에서 배열 특이성을 달성할 수 있다.4개의 페닐알라닌 잔기와 3' 말단의 2개의 뉴클레오티드 사이의 γ-γ 상호작용은 뉴클레오티드의 식별에 따라 다르며,[8] 이는 효소의 형태와 활성을 변화시킨다.추가적인 글루탐산 잔기는 다른 염기가 아닌 시토신에 수소 결합으로 회전하여 특이성을 [9]더욱 높인다.
기능.
더 큰 DEDD 계열의 엑토리보핵산가수분해효소인 RNAse T는 tRNA의 성숙과 5S[11] 및 23S[12] rRNA 도메인의 성숙에 중요한 역할을 한다.구체적으로는 RNAse T는 tRNA 말기의 3' CCA 배열에서 3' AMP 잔기를 분리하여 3' CC [13]배열에서 정지하기 위한 RNAse T의 배열 특이성을 설명한다.또한 RNAse T는 벌지 DNA의 [3]3' 끝을 절단함으로써 DNA 수복에 관여할 수 있다.
대장균은 RNAse T 없이도 생존할 수 있지만, 대장균의 부재로 인해 수명 주기가 느려지고 [14]기아에 대한 반응이 약해진다.또한 대장균에서 RNAse T의 존재는 UV [15]손상에 대한 내성의 증가와 관련이 있다.다른 리본큘라아제들은 RNAse T 기능을 수행할 수 있지만, RNAse T가 DNA와 RNA를 이중가닥 영역 근처에서 분해하는 데 더 효과적이라는 사실은 대안이 덜 [16]효과적이라는 것을 의미한다.RNAse T의 명백한 유용성에도 불구하고, 효소는 감마프로테오박테리아에서만 [17]발견된다.
대장균에서 RNAse T는 rnt 유전자에 의해 부호화되어 감마프로테오박테리아가 [16][17]출현하는 동안 중합효소 III의 교정 서브유닛에서 분리되었다고 가정된다.
레퍼런스
- ^ a b Hsiao YY, Duh Y, Chen YP, Wang YT, Yuan HS (September 2012). "How an exonuclease decides where to stop in trimming of nucleic acids: crystal structures of RNase T-product complexes". Nucleic Acids Research. 40 (16): 8144–54. doi:10.1093/nar/gks548. PMC 3439924. PMID 22718982.
- ^ a b Zuo Y, Deutscher MP (August 2002). "The physiological role of RNase T can be explained by its unusual substrate specificity". The Journal of Biological Chemistry. 277 (33): 29654–61. doi:10.1074/jbc.M204252200. PMID 12050169.
- ^ a b c Hsiao YY, Fang WH, Lee CC, Chen YP, Yuan HS (March 2014). "Structural insights into DNA repair by RNase T--an exonuclease processing 3' end of structured DNA in repair pathways". PLOS Biology. 12 (3): e1001803. doi:10.1371/journal.pbio.1001803. PMC 3942315. PMID 24594808.
- ^ Zuo Y, Deutscher MP (October 1999). "The DNase activity of RNase T and its application to DNA cloning". Nucleic Acids Research. 27 (20): 4077–82. doi:10.1093/nar/27.20.4077. PMC 148676. PMID 10497273.
- ^ Li Z, Zhan L, Deutscher MP (January 1996). "Escherichia coli RNase T functions in vivo as a dimer dependent on cysteine 168". The Journal of Biological Chemistry. 271 (2): 1133–7. doi:10.1074/jbc.271.2.1133. PMID 8557641.
- ^ Zuo Y, Zheng H, Wang Y, Chruszcz M, Cymborowski M, Skarina T, et al. (April 2007). "Crystal structure of RNase T, an exoribonuclease involved in tRNA maturation and end turnover". Structure. 15 (4): 417–28. doi:10.1016/j.str.2007.02.004. PMC 1907377. PMID 17437714.
- ^ Deutscher MP, Marlor CW (June 1985). "Purification and characterization of Escherichia coli RNase T". The Journal of Biological Chemistry. 260 (11): 7067–71. doi:10.1016/S0021-9258(18)88888-3. PMID 3888994.
- ^ Duh Y, Hsiao YY, Li CL, Huang JC, Yuan HS (December 2015). "Aromatic residues in RNase T stack with nucleobases to guide the sequence-specific recognition and cleavage of nucleic acids". Protein Science. 24 (12): 1934–41. doi:10.1002/pro.2800. PMC 4815224. PMID 26362012.
- ^ Hsiao YY, Yang CC, Lin CL, Lin JL, Duh Y, Yuan HS (April 2011). "Structural basis for RNA trimming by RNase T in stable RNA 3'-end maturation". Nature Chemical Biology. 7 (4): 236–43. doi:10.1038/nchembio.524. PMID 21317904.
- ^ Li Z, Deutscher MP (August 1996). "Maturation pathways for E. coli tRNA precursors: a random multienzyme process in vivo". Cell. 86 (3): 503–12. doi:10.1016/s0092-8674(00)80123-3. PMID 8756732.
- ^ Li Z, Deutscher MP (July 1995). "The tRNA processing enzyme RNase T is essential for maturation of 5S RNA". Proceedings of the National Academy of Sciences of the United States of America. 92 (15): 6883–6. Bibcode:1995PNAS...92.6883L. doi:10.1073/pnas.92.15.6883. PMC 41434. PMID 7542780.
- ^ Li Z, Pandit S, Deutscher MP (January 1999). "Maturation of 23S ribosomal RNA requires the exoribonuclease RNase T". RNA. 5 (1): 139–46. doi:10.1017/s1355838299981669. PMC 1369746. PMID 9917073.
- ^ Deutscher MP, Marlor CW, Zaniewski R (July 1984). "Ribonuclease T: new exoribonuclease possibly involved in end-turnover of tRNA". Proceedings of the National Academy of Sciences of the United States of America. 81 (14): 4290–3. Bibcode:1984PNAS...81.4290D. doi:10.1073/pnas.81.14.4290. PMC 345573. PMID 6379642.
- ^ Kelly KO, Deutscher MP (October 1992). "The presence of only one of five exoribonucleases is sufficient to support the growth of Escherichia coli". Journal of Bacteriology. 174 (20): 6682–4. doi:10.1128/jb.174.20.6682-6684.1992. PMC 207653. PMID 1400219.
- ^ Viswanathan M, Lanjuin A, Lovett ST (March 1999). "Identification of RNase T as a high-copy suppressor of the UV sensitivity associated with single-strand DNA exonuclease deficiency in Escherichia coli". Genetics. 151 (3): 929–34. doi:10.1093/genetics/151.3.929. PMC 1460521. PMID 10049912.
- ^ a b Bechhofer DH, Deutscher MP (June 2019). "Bacterial ribonucleases and their roles in RNA metabolism". Critical Reviews in Biochemistry and Molecular Biology. 54 (3): 242–300. doi:10.1080/10409238.2019.1651816. PMC 6776250. PMID 31464530.
- ^ a b Zuo Y, Deutscher MP (March 2001). "Exoribonuclease superfamilies: structural analysis and phylogenetic distribution". Nucleic Acids Research. 29 (5): 1017–26. doi:10.1093/nar/29.5.1017. PMC 56904. PMID 11222749.