리보핵산가수분해효소T1
Ribonuclease T1| 리보핵산가수분해효소T1 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 식별자 | |||||||||
| EC 번호 | 3.1.27.3 | ||||||||
| CAS 번호 | 9026-12-4 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
| 리보핵산가수분해효소T1 | |||||||
|---|---|---|---|---|---|---|---|
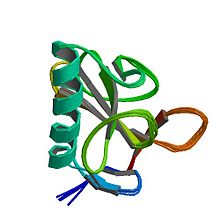 Aspergillus oryzae의 [1]리보핵산가수분해효소1 T. | |||||||
| 식별자 | |||||||
| 기호. | rntA | ||||||
| PDB | 1 YGW | ||||||
| 유니프로트 | P00651 | ||||||
| 기타 데이터 | |||||||
| EC 번호 | 3.1.27.3 | ||||||
| |||||||
리보 뉴 클레이스 T1(EC3.1.27.3, guanyloribonuclease, 아스 페르질 oryzae 리보 핵산 가수 분해 효소, RN아제 N1, RN아제 N2, 리보 핵산 가수 분해 효소 N3, 리보 핵산 가수 분해 효소 U, 리보 핵산 가수 분해 효소 F1, 리보 핵산 가수 분해 효소 Ch, 리보 핵산 가수 분해 효소 PP1, 리보 핵산 분해 효소 SA는 RN아제 F1, 리보 핵산 가수 분해 효소 C2, binase, RN아제 사우,guanyl-specific RN아제, RN아제 G, RN아제 T1, 리보 핵산 가수 분해 효소 guaninenucleotido-2'-transferase(cyclizing.),리보핵산가수분해효소 N3, 리보핵산가수분해효소 N1)는 구아닌 잔기(즉, 말단 3') 이후에 단일 가닥 RNA를 분해하는 균류 엔도핵산가수분해효소이다.구아닌에 대한 특이성 때문에 RNase T는1 염기서열 분석 전에 변성 RNA를 소화하는데 종종 사용된다.바나제 및 RNase A와 같은 다른 리보핵산가수분해효소들과 유사하게, 리보핵산가수분해효소1 T는 접이식 [2]연구에서 인기가 있었다.
구조적으로 리보핵산가수분해효소1 T는 긴 알파나선(약 5회전)을 덮는 4가닥 반팔레알 베타 시트를 가진 작은 α+β 단백질(104 아미노산)이다.RNase1 T는 Cys2-Cys10 및 Cys6-Cys103의 두 가지 이황화 결합을 가지며, 이 중 후자는 접힘 [3]안정성에 더 기여합니다. 두 이황화물의 완전한 감소는 일반적으로 단백질을 펼치지만 고염 [4]농도로 접힘을 복구할 수 있습니다.
RNase1 T는 또한 4개의 프로라인을 가지며, 그 중 2개(Pro39 및 Pro55)는 X-Pro 펩타이드 결합의 시스 이성질체를 가진다.이러한 프로라인의 비원어민 이성질체는 10°C 및 pH [6]5에서 7,000초(거의 2시간)의 시간 척도로 접히면서 형태적 접힘을 [5]극적으로 지연시킬 수 있습니다.
레퍼런스
- ^ PDB: 1ygw;Pfeiffer S, Karimi-Nejad Y, Rüterjans H (1997). "Limits of NMR structure determination using variable target function calculations: ribonuclease T1, a case study". Journal of Molecular Biology. 266 (2): 400–423. doi:10.1006/jmbi.1996.0784. PMID 9047372.
- ^ Pace CN, Heinemann U, Hahn U, Saenger W (1991). "Ribonuclease T1: Structure, Function, and Stability". Angewandte Chemie. 30 (4): 343–360. doi:10.1002/anie.199103433.
- ^ Pace CN, Grimsley GR, Thomson JA, Barnett BJ (1988). "Conformational stability and activity of ribonuclease T1 with zero, one, and two intact disulfide bonds". Journal of Biological Chemistry. 263 (24): 11820–11825. PMID 2457027.
- ^ Oobatake M, Takahashi S, Ooi T (1979). "Conformational stability of ribonuclease T1. II. Salt-induced renaturation". Journal of Biochemistry. 86: 65–70.
- ^ Mayr LM, Odefey CO, Schutkowski M, Schmid FX (1996). "Kinetic analysis of the unfolding and refolding of ribonuclease T1 by a stopped-flow double-mixing technique". Biochemistry. 35 (17): 5550–5561. doi:10.1021/bi953035y. PMID 8611546.
- ^ Mullins LS, Pace CN, Raushel FM (1997). "Conformational stability of ribonuclease T1 measured by hydrogen-deuterium exchange". Protein Science. 6 (7): 1387–1395. doi:10.1002/pro.5560060702. PMC 2143755. PMID 9232639.
외부 링크