단백질분해효소 PA군
PA clan of proteases| 단백질분해효소 PA군 | |
|---|---|
 | |
| 식별자 | |
| 기호. | 없음 |
| 빠맘 클랜 | CL0124 |
| 인터프로 | IPR009003 |
| SCOP2 | 50494 / SCOPe / SUPFAM |
| 막질 | 319 |
PA 클랜(혼합 친핵성 단백질, 슈퍼 패밀리 A)은 구조적 호몰로지로 식별되는 공통 조상을 가진 단백질 분해효소 중 가장 큰 그룹이다.구성원은 키모트립신 유사 접힘 및 유사한 단백질 분해 메커니즘을 가지지만 10% 미만의 동일성을 가질 수 있다.씨족은 시스테인과 세린 프로테아제(다른 [1][2]친핵체)를 모두 포함하고 있다.PA 클랜 프로테아제는 식물,[3] 동물,[3] 곰팡이,[3] 유박테리아,[4] 고세균[5][6] 및 [2]바이러스에서 발견됩니다.
PA 클랜을 포함한 단백질 분해효소의 여러 패밀리에 의한 가수분해를 위한 촉매 삼합체의 일반적인 사용은 수렴 [7]진화의 한 예를 나타낸다.PA 클랜 내의 촉매 삼합체 차이 또한 [2]효소에서 활성 부위의 발산 진화의 한 예이다.
역사
1960년대에, 몇몇 단백질 분해 효소의 배열 유사성은 그것들이 진화적으로 [8]관련이 있다는 것을 보여주었다.이것들은 키모트립신 유사 세린 단백질[9] 분해효소(현재는 S1 계열로 불린다)로 그룹화 되었다.1970~80년대 X선 결정학으로 이들 단백질 분해효소 등의 구조가 해결되면서 Tobco Ech Virus 단백질 분해효소 등 몇몇 바이러스 단백질 분해효소들은 뚜렷한 배열 유사성이 없고 심지어 다른 [2][10][11]친핵성 물질에도 불구하고 구조적 호몰로지를 보였다.구조적 호몰로지에 기초하여 슈퍼 패밀리가 정의되었고 나중에 (MEROPS 분류 시스템에 의해) PA 클랜으로 명명되었다.더 많은 구조가 해결됨에 따라 PA 클랜 슈퍼패밀리에 더 많은 프로테아제 [12][13]패밀리가 추가되었다.
어원학
P는 혼합 친핵 단백질 분해 효소를 의미한다.A는 이러한 클랜이 최초로 식별되었음을 나타냅니다(PB,[1] PC, PD 및 PE 클랜도 존재합니다).
구조.
10%의 배열 정체성을 유지함에도 불구하고 바이러스, 원핵생물 및 진핵생물에서 분리된 PA 클랜 구성원은 구조적 호몰로지를 보여주며 구조적 유사성(DALI [3]등)에 의해 정렬될 수 있다.
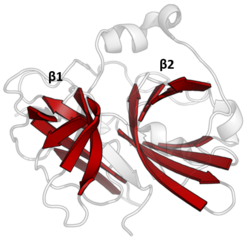
이중 β배럴
PA클랜단백질가수분해효소는 모두 2개의 β-배럴의 핵심 모티브를 산-히스티딘-핵소성 촉매삼합체 모티브에 의해 이루어지는 공유 촉매작용과 공유한다.배럴은 효소의 핵심 골격으로 이들을 고정시키는 소수성 잔류물과 함께 서로 수직으로 배열됩니다.3중 잔류물은 두 배럴 사이에 분할되어 촉매 작용이 그들의 [14]경계면에서 이루어지도록 한다.
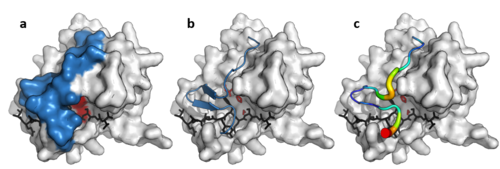
바이러스단백질가수분해효소루프
이중 β-배럴 코어 외에도, 일부 바이러스 단백질 분해효소(TEV 단백질 분해효소 등)는 기질을 완전히 덮고 결합 터널을 만드는 뚜껑을 형성하는 길고 유연한 C-말단 루프를 가지고 있습니다.이 터널은 기질펩타이드(P6~P1')의 각 측쇄가 상보적인 부위(S6~S1')에 결합되어 효소와 [11]기질 사이의 큰 접촉면적에 의해 특이성이 부여되는 일련의 타이트한 결합 포켓을 포함한다.반대로, 트립신과 같이 이 루프가 없는 세포단백질가수분해효소는 더 넓은 특이성을 가지고 있다.
진화와 기능
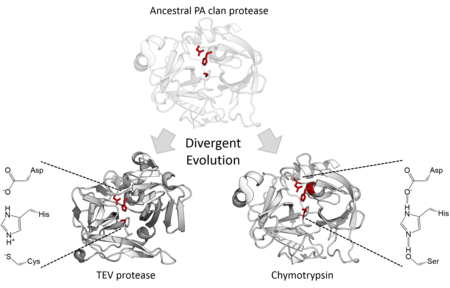
촉매 활성
구조적 호몰로지는 PA 클랜 구성원들이 같은 그룹의 공통 조상의 후손이라는 것을 보여준다.PA 클랜 단백질 분해효소는 2단계 친핵 촉매 작용을 [7]하는 촉매 삼합체를 사용하지만, 친핵체로 세린을 사용하는 반면 시스테인을 사용하는 [2]가족도 있다.따라서 슈퍼 패밀리는 진화 역사 동안 효소의 핵심 촉매 잔기가 다른 [15]패밀리로 전환되었기 때문에 발산 효소 진화의 극단적인 예이다.이들의 구조적 유사성 외에도, 유도 진화는 시스테인 단백질 분해 효소를 활성 세린 단백질 분해 [16]효소로 전환할 수 있는 것으로 나타났다.모든 세포 PA 클랜 단백질 분해효소는 세린 단백질 분해효소이지만, 바이러스 단백질 [7]분해효소에는 세린 및 시스테인 단백질 분해효소 계열이 모두 있다.대부분은 엔도펩티다아제이며, [17][18]예외적으로 S46 계열의 엑소펩티다아제이다.
생물학적 역할과 기질 특이성
PA 클랜 프로테아제들은 핵심 촉매기계에서 발산되는 것 외에도 기능적으로도 광범위한 진화를 보인다.PA클랜의 구성원은 진핵생물, 원핵생물 및 바이러스에서 발견될 수 있으며 광범위한 기능을 포함한다.포유동물에서 일부는 혈액 응고에 관여하기 때문에(예: 트립신), 높은 기질 특이성과 광범위한 기질 특이성을 지닌 소화(예: 트립신)를 가지고 있다.몇몇 뱀 독은 Pit viper hemotoxin과 같은 PA 클랜 단백질 분해효소이며 희생자의 혈액 응고를 방해합니다.또한 황색포도상구균과 같은 박테리아는 숙주의 조직을 소화하고 손상시키는 각질성 독소를 분비한다.많은 바이러스들은 그들의 게놈을 하나의 거대한 폴리단백질로 표현하고 이것을 기능 단위(예: 소아마비, 노로바이러스, TEV 단백질 분해효소)[19][20]로 나누기 위해 PA 클랜 단백질 분해효소를 사용합니다.
슈퍼패밀리에는 또한 여러 개의 유사 효소가 있으며, 여기서 촉매 삼극체 잔기는 돌연변이되어 결합 [21]단백질로 기능한다.예를 들어 헤파린결합단백질 아즈로시딘은 친핵체 대신 글리신, 히스티딘 [22]대신 세린을 가진다.
가족들
PA클랜(P=혼합핵인자의 효소) 내에서 패밀리는 촉매성 친핵인자(C=시스테인단백질가수분해효소, S=세린단백질가수분해효소)에 의해 지정된다.PA 클랜 전체에 대한 시퀀스 호몰로지가 없음에도 불구하고, PA 클랜 내의 개별 패밀리는 시퀀스 유사성에 의해 식별될 수 있다.
| 가족 | 예 | 알려진 구조? |
|---|---|---|
| C03 | 소아마비형 피코르나인3C(폴리오바이러스) | 네. |
| C04 | 담배에칭바이러스단백질가수분해효소(에칭바이러스) | 네. |
| C24 | 토끼출혈성질환바이러스3C양펩티드가수분해효소(토끼출혈성질환바이러스) | 아니요. |
| C30 | 돼지 전염성 위장염 바이러스형 주펩티드가수분해효소(전염성 위장염 바이러스) | 네. |
| C37 | 칼리시비린(사우스햄프턴바이러스) | 네. |
| C62 | 질관련바이러스3C양펩티드가수분해효소(질관련바이러스) | 아니요. |
| C74 | 페스티바이러스NS2펩티드가수분해효소(소바이러스설사바이러스1) | 아니요. |
| C99 | iflavirus 처리펩티드가수분해효소(Ectropis obliqua picorna 유사바이러스) | 아니요. |
| C107 | 알파메소니바이러스3C양펩티드가수분해효소(카발리바이러스) | 아니요. |
| S01 | 키모트립신A(Bos 황소자리) | 네. |
| S03 | 토가비린(신드비스 바이러스) | 네. |
| S06 | IgA특이세린엔도펩티드가수분해효소(Neisseria gonorhoae) | 네. |
| S07 | 플라비비비린(황열 바이러스) | 아니요. |
| S29 | 간염C바이러스 | 네. |
| S30 | 포티바이러스 P1펩티드가수분해효소(플럼독스바이러스) | 아니요. |
| S31 | 페스티바이러스NS3폴리단백질펩티드가수분해효소(소바이러스설사바이러스1) | 아니요. |
| S32 | 말동맥바이러스세린펩티드가수분해효소(말동맥염바이러스) | 네. |
| S39 | 소베모바이러스펩티드가수분해효소(두발모틀바이러스) | 네. |
| S46 | 디펩티딜펩티드가수분해효소7(Porphyromonas gingivalis) | 아니요. |
| S55 | SpoIVB펩티드가수분해효소(세브틸리스 바실러스) | 아니요. |
| S64 | Ssy5펩티드가수분해효소(사카로미세스세레비시아에) | 아니요. |
| S65 | 피코르나인양시스테인펩티드가수분해효소(브레다-1 토로바이러스) | 아니요. |
| S75 | 흰뱀바이러스세린펩티드가수분해효소(흰뱀바이러스) | 아니요. |
「 」를 참조해 주세요.
레퍼런스
- ^ a b Rawlings ND, Barrett AJ, Bateman A (January 2012). "MEROPS: the database of proteolytic enzymes, their substrates and inhibitors". Nucleic Acids Research. 40 (Database issue): D343-50. doi:10.1093/nar/gkr987. PMC 3245014. PMID 22086950.
- ^ a b c d e Bazan JF, Fletterick RJ (November 1988). "Viral cysteine proteases are homologous to the trypsin-like family of serine proteases: structural and functional implications". Proceedings of the National Academy of Sciences of the United States of America. 85 (21): 7872–6. Bibcode:1988PNAS...85.7872B. doi:10.1073/pnas.85.21.7872. PMC 282299. PMID 3186696.
- ^ a b c d Laskar A, Rodger EJ, Chatterjee A, Mandal C (May 2012). "Modeling and structural analysis of PA clan serine proteases". BMC Research Notes. 5: 256. doi:10.1186/1756-0500-5-256. PMC 3434108. PMID 22624962.
- ^ Barbosa JA, Saldanha JW, Garratt RC (July 1996). "Novel features of serine protease active sites and specificity pockets: sequence analysis and modelling studies of glutamate-specific endopeptidases and epidermolytic toxins". Protein Engineering. 9 (7): 591–601. doi:10.1093/protein/9.7.591. PMID 8844831.
- ^ "MEROPS - Archaeal S01 proteases".
- ^ Ruiz-Perez F, Nataro JP (March 2014). "Bacterial serine proteases secreted by the autotransporter pathway: classification, specificity, and role in virulence". Cellular and Molecular Life Sciences. 71 (5): 745–70. doi:10.1007/s00018-013-1355-8. PMC 3871983. PMID 23689588.
- ^ a b c Buller AR, Townsend CA (February 2013). "Intrinsic evolutionary constraints on protease structure, enzyme acylation, and the identity of the catalytic triad". Proceedings of the National Academy of Sciences of the United States of America. 110 (8): E653-61. Bibcode:2013PNAS..110E.653B. doi:10.1073/pnas.1221050110. PMC 3581919. PMID 23382230.
- ^ de Haën C, Neurath H, Teller DC (February 1975). "The phylogeny of trypsin-related serine proteases and their zymogens. New methods for the investigation of distant evolutionary relationships". Journal of Molecular Biology. 92 (2): 225–59. doi:10.1016/0022-2836(75)90225-9. PMID 1142424.
- ^ Lesk AM, Fordham WD (May 1996). "Conservation and variability in the structures of serine proteinases of the chymotrypsin family". Journal of Molecular Biology. 258 (3): 501–37. doi:10.1006/jmbi.1996.0264. PMID 8642605.
- ^ Gorbalenya AE, Blinov VM, Donchenko AP (January 1986). "Poliovirus-encoded proteinase 3C: a possible evolutionary link between cellular serine and cysteine proteinase families". FEBS Letters. 194 (2): 253–7. doi:10.1016/0014-5793(86)80095-3. PMID 3000829. S2CID 23268152.
- ^ a b Phan J, Zdanov A, Evdokimov AG, Tropea JE, Peters HK, Kapust RB, Li M, Wlodawer A, Waugh DS (December 2002). "Structural basis for the substrate specificity of tobacco etch virus protease". The Journal of Biological Chemistry. 277 (52): 50564–72. doi:10.1074/jbc.M207224200. PMID 12377789.
- ^ Allaire M, Chernaia MM, Malcolm BA, James MN (May 1994). "Picornaviral 3C cysteine proteinases have a fold similar to chymotrypsin-like serine proteinases". Nature. 369 (6475): 72–6. Bibcode:1994Natur.369...72A. doi:10.1038/369072a0. PMID 8164744. S2CID 4312593.
- ^ Snijder EJ, Wassenaar AL, van Dinten LC, Spaan WJ, Gorbalenya AE (March 1996). "The arterivirus nsp4 protease is the prototype of a novel group of chymotrypsin-like enzymes, the 3C-like serine proteases". The Journal of Biological Chemistry. 271 (9): 4864–71. doi:10.1074/jbc.271.9.4864. PMID 8617757.
- ^ Dougherty WG, Parks TD, Cary SM, Bazan JF, Fletterick RJ (September 1989). "Characterization of the catalytic residues of the tobacco etch virus 49-kDa proteinase". Virology. 172 (1): 302–10. doi:10.1016/0042-6822(89)90132-3. PMID 2475971.
- ^ Laskar A, Rodger EJ, Chatterjee A, Mandal C (May 2012). "Modeling and structural analysis of PA clan serine proteases". BMC Research Notes. 5 (1): 256. doi:10.1186/1756-0500-5-256. PMC 3434108. PMID 22624962.
- ^ Shafee T, Gatti-Lafranconi P, Minter R, Hollfelder F (September 2015). "Handicap-Recover Evolution Leads to a Chemically Versatile, Nucleophile-Permissive Protease". ChemBioChem. 16 (13): 1866–1869. doi:10.1002/cbic.201500295. PMC 4576821. PMID 26097079.
- ^ Suzuki Y, Sakamoto Y, Tanaka N, Okada H, Morikawa Y, Ogasawara W (March 2014). "Identification of the catalytic triad of family S46 exopeptidases, closely related to clan PA endopeptidases". Scientific Reports. 4: 4292. doi:10.1038/srep04292. PMC 3944710. PMID 24598890.
- ^ Sakamoto Y, Suzuki Y, Iizuka I, Tateoka C, Roppongi S, Fujimoto M, Inaka K, Tanaka H, Masaki M, Ohta K, Okada H, Nonaka T, Morikawa Y, Nakamura KT, Ogasawara W, Tanaka N (May 2014). "S46 peptidases are the first exopeptidases to be members of clan PA". Scientific Reports. 4: 4977. doi:10.1038/srep04977. PMC 4021333. PMID 24827749.
- ^ Salvesen G (2013). Rawlings N (ed.). Handbook of proteolytic enzymes. Boston: Academic Press. ISBN 9780123822192.
- ^ Polgár L (October 2005). "The catalytic triad of serine peptidases". Cellular and Molecular Life Sciences. 62 (19–20): 2161–72. doi:10.1007/s00018-005-5160-x. PMID 16003488. S2CID 3343824.
- ^ Todd AE, Orengo CA, Thornton JM (October 2002). "Sequence and structural differences between enzyme and nonenzyme homologs". Structure. 10 (10): 1435–51. doi:10.1016/s0969-2126(02)00861-4. PMID 12377129.
- ^ Iversen LF, Kastrup JS, Bjørn SE, Rasmussen PB, Wiberg FC, Flodgaard HJ, Larsen IK (April 1997). "Structure of HBP, a multifunctional protein with a serine proteinase fold". Nature Structural Biology. 4 (4): 265–8. doi:10.1038/nsb0497-265. PMID 9095193. S2CID 19949043.