유전적 연관성
Genetic linkage유전자 연계는 성생식의 감수분열 단계에서 염색체에서 서로 가까운 DNA 염기서열이 함께 유전되는 경향이다.물리적으로 서로 가까운 두 개의 유전자 마커는 염색체 교차 중에 다른 염색체 상에 분리될 가능성이 낮으며, 따라서 멀리 떨어져 있는 마커보다 더 관련이 있다고 한다.즉, 두 유전자가 염색체에 가까울수록, 그들 사이의 재결합 가능성은 낮아지고, 함께 유전될 가능성이 높아진다.다른 염색체 상의 마커는 완전히 연결되어 있지 않지만, 잠재적 유해 대립 유전자의 투과성은 다른 대립 유전자의 존재에 의해 영향을 받을 수 있으며, 이러한 다른 대립 유전자는 특정 잠재적으로 유해 유전자가 [1]위치한 다른 염색체에 위치할 수 있다.
Gregor Mendel의 독립적 분류의 법칙에 대한 가장 두드러진 예외는 유전적 연관성이다.연계성을 입증하는 첫 번째 실험은 1905년에 수행되었다.그 당시에는 어떤 특성들이 함께 유전되는 경향이 있는 이유는 알려지지 않았다.이후 연구는 유전자가 물리적 거리에 의해 연관된 물리적 구조라는 것을 밝혀냈다.
유전자 결합의 전형적인 단위는 센티모건(cM)이다. 두 마커 사이의 거리가 1cmM이면 마커들이 평균 100개의 감수성 생성물 당 1회, 즉 50개의 감수성 생성물 당 1회 다른 염색체로 분리된다는 것을 의미한다.
검출
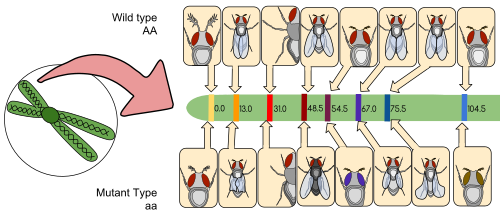
그레고르 멘델의 독립적 분류의 법칙은 모든 특성이 다른 모든 특성과는 독립적으로 유전된다고 말합니다.그러나 멘델의 작품이 재발견된 직후, 이 규칙에 대한 예외가 발견되었다.1905년 영국의 유전학자 윌리엄 베이츠슨, 에디스 레베카 손더스, 레지날드 푸넷은 멘델과 [2][3]비슷한 실험을 통해 완두콩 식물을 교배시켰다.그들은 스위트피에서의 특성 유전에 관심이 있었고 두 가지 유전자, 즉 꽃 색깔에 대한 유전자 (P, 보라색, p, 빨간색)와 꽃가루 알갱이의 모양에 영향을 미치는 유전자 (L, 긴, 그리고 l, 둥근 모양)를 연구하고 있었다.그들은 순수한 선 PPLL과 ppl을 교차시킨 후 결과 PpLl 선을 자가 교차시켰습니다.
멘델 유전학에 따르면, 예상되는 표현형은 PL:PL:pL:pl의 9:3:3:1 비율로 발생할 것이다.놀랍게도, 그들은 PL과 PL의 빈도가 증가하고 PL과 pL의 빈도가 감소하는 것을 관찰했다.
| 표현형 및 유전자형 | 관찰된 | 9:3:3:1의 비율로 예상 |
|---|---|---|
| 퍼플, 롱(P_L_) | 284 | 216 |
| 보라색, 원형(P_ll) | 21 | 72 |
| 레드, 롱(ppL_) | 21 | 72 |
| 빨강, 둥근(ppl) | 55 | 24 |
그들의 실험은 P와 L 대립 유전자와 P와 L 대립 유전자의 연관성을 밝혀냈다.L과 함께 발생하는 P, l과 함께 발생하는 p의 빈도는 재조합 P, pL의 빈도보다 크다.재조합 빈도는 역교차보다 F2 교차로 [4]계산하기가 더 어렵지만, 위 표의 관측된 자손 수와 예상 자손 수 사이의 적합성이 결여되어 있어 50% 미만임을 알 수 있다.이는 두 요인이 어떤 방식으로 상호작용하여 다른 두 표현형의 외관을 마스킹함으로써 이러한 차이를 만든다는 것을 나타냈다.이것은 어떤 특징들은 염색체에서 서로 근접하기 때문에 서로 관련이 있다는 결론으로 이어졌다.
연계성에 대한 이해는 토마스 헌트 모건의 연구로 확대되었다.연결된 유전자 사이의 교차량이 다르다는 모건의 관찰은 교차 빈도가 염색체의 유전자를 분리하는 거리를 나타낼 수 있다는 생각을 하게 했다.건널목의 빈도를 나타내는 센티모간은 그를 기리기 위해 붙여진 이름이다.
링크 맵
연결 지도(유전자 지도라고도 함)는 각 염색체를 따라 특정한 물리적 거리가 아닌 재조합 빈도로 서로 상대적인 유전자 또는 유전자 표지의 위치를 보여주는 종 또는 실험 집단을 위한 표이다.연계 지도는 토마스 헌트 모건의 제자인 알프레드 스터테반트에 의해 처음 개발되었다.
연계 지도는 상동 염색체 교차 중 마커 간의 재조합 빈도에 기초한 지도이다.두 유전자 마커 사이의 재조합 빈도(분리)가 클수록, 그들은 더 멀리 떨어져 있는 것으로 가정된다.반대로 마커 간의 재결합 빈도가 낮을수록 마커 간의 물리적 거리는 줄어듭니다.역사적으로, 원래 사용된 마커는 코드 DNA 배열에서 파생된 검출 가능한 표현형(효소 생성, 눈 색깔)이었다. 결국, 마이크로 위성이나 제한 단편 길이 다형(RFLP)을 생성하는 것과 같은 확인되거나 가정된 비코드 DNA 배열이 사용되었다.
연계 지도는 연구자들이 이미 알려진 표지들의 유전적 연관성을 검사함으로써 다른 유전자와 같은 다른 표지들을 찾을 수 있도록 돕는다.링크 맵의 개발 초기 단계에서 데이터는 링크되어 있는 것으로 알려진 유전자 세트인 링크 그룹을 조립하기 위해 사용된다.지식이 발전함에 따라 그룹이 전체 [6]염색체를 덮을 때까지 그룹에 더 많은 마커를 추가할 수 있습니다.잘 연구된 유기체의 경우 연결 그룹은 염색체와 일대일로 대응한다.
연결 지도는 물리적 지도(방사선 감소 하이브리드 지도 등)나 유전자 지도가 아니다.
연계 분석
연계분석은 가족을 통해 질병 표현형과 공격리되는 염색체 세그먼트를 찾는 유전학적 방법으로 지방영양증 유전자의 [7][8]부피를 결정하는 데 사용되어 온 분석 기법이다.그것은 2진수 및 [8]양적 특성 모두의 유전자를 매핑하는 데 사용될 수 있다.연계 분석은 파라메트릭(표현형과 유전적 유사성 사이의 관계를 알고 있는 경우) 또는 비파라메트릭일 수 있다.파라메트릭 연계 분석은 LOD 점수를 통해 질병에 중요한 유전자가 유전 마커와 연결될 확률이 연구되는 전통적인 접근법이다. LOD 점수는 질병과 마커가 공분리되는 특정 혈통이 연결의 존재에 기인하는 확률을 평가한다.또는 운에 맡깁니다.비모수 연결 분석은 차례로 대립 유전자가 그 자체로 하강하여 동일할 확률을 연구합니다.
파라메트릭 연계 분석
Newton [9]Morton이 개발한 LOD 점수(확률의 대수(기준 10))는 인간, 동물 및 식물 모집단의 연계 분석에 자주 사용되는 통계 검정이다.LOD 점수는 두 개의 위치가 실제로 연결되어 있는 경우 검정 데이터를 얻을 수 있는 가능성을 순전히 우연에 의해 동일한 데이터를 관측할 수 있는 가능성과 비교합니다.LOD 점수가 양수이면 연계성이 있는 것을 선호하지만, 음수이면 연계성이 낮다는 것을 나타냅니다.컴퓨터화된 LOD 점수 분석은 Mendelian 특성(또는 특징과 표식자 또는 두 표식자) 사이의 연관성을 결정하기 위해 복잡한 가족 혈통을 분석하는 간단한 방법이다.
이 방법은 Strachan 및 Read에서 자세히 설명합니다.[1] 간단히 다음과 같이 동작합니다.
- 혈통을 확립하다
- 재조합 빈도를 여러 번 추정하다
- 각 견적에 대한 LOD 점수 계산
- LOD 점수가 가장 높은 견적이 최선의 견적으로 간주됩니다.
LOD 점수는 다음과 같이 계산됩니다.
NR은 비반생 자손의 수를 나타내고, R은 재조합 자손의 수를 나타낸다.분모에 0.5가 사용되는 이유는 완전히 연결되지 않은 대립 유전자(예: 별도의 염색체 상의 대립 유전자)가 독립적인 분류로 인해 재조합될 확률이 50%이기 때문이다.γ는 재조합 분율, 즉 연구된 유전자 표지와 질병과 관련된 추정 유전자 사이에서 재조합이 일어난 출생 분율이다.즉, R / (NR + R)과 같습니다.
관례상 LOD 점수가 3.0보다 크면 연결이 우연히 발생하지 않았음을 1000:1의 확률로 나타내므로 연관성에 대한 증거로 간주됩니다.한편, -2.0 미만의 LOD 점수는 연계성을 배제하는 증거로 간주됩니다.단일 혈통에서 LOD 점수 3을 얻을 가능성은 매우 낮지만, 테스트의 수학적 특성은 LOD 점수를 합산하여 여러 혈통의 데이터를 결합할 수 있습니다.LOD 점수가 3이면 p-값이 [10]약 0.05이므로 다중 검정 보정([11]예: Bonferroni 보정)이 필요하지 않습니다.
제한 사항
Linkage 분석은 힘 인간의 양적 형질 유전자좌를 그리는 것을 줄이(QTL)[12]는 동안 연결 분석 성공적으로 헌팅턴 병과 같은 희귀한 장애에 기여한 유전자 변형을 식별할 수 있는 데 사용될 때, perf지 않았어 1오차율을 증가시킬 수 있고 이론적 방법론적 한계가 많이 있다.orm심장병이나 다른 형태의 [13]암과 같은 더 흔한 질환에 적용되었을 때 잘 나타난다.일반적인 질환에 영향을 미치는 유전적 메커니즘이 일부 희귀 [14]질환을 일으키는 메커니즘과 다르다는 설명이다.
재결합 빈도
재조합 빈도는 유전자 연계의 척도로 유전자 연계 지도 작성에 사용된다.재조합 빈도())는 감수분열 시 두 유전자 사이에서 단일 염색체 교차가 일어나는 빈도이다.centimorgan(cM)은 1%의 재조합 빈도를 나타내는 단위이다.이 방법으로 우리는 재조합 빈도에 따라 두 위치 사이의 유전적 거리를 측정할 수 있다.이것은 실제 거리를 충분히 추정해 볼 수 있다.이중 교차로는 재결합이 되지 않습니다.이 경우 크로스오버가 발생했는지 여부를 알 수 없습니다.분석하려는 궤적이 매우 가까운 경우(7cm 미만) 이중 교차는 거의 발생하지 않습니다.거리가 길어지면 이중 교차 가능성이 높아집니다.이중 교차 가능성이 증가함에 따라 우리는 체계적으로 두 위치 사이의 유전적 거리를 과소평가한다.
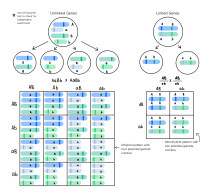
감수분열 중에 염색체는 무작위로 생식체로 분류되어 한 유전자의 대립 유전자의 분리가 다른 유전자의 대립 유전자와는 독립적이다.이것은 멘델의 제2법칙에 명시되어 있으며 독립적 분류의 법칙으로 알려져 있습니다.다른 염색체에 있는 유전자에 대해서는 독립적 분류의 법칙이 항상 적용되지만, 같은 염색체에 있는 유전자에 대해서는 항상 해당되지 않는다.
독립적 어소시에이션의 예로서, 순수 교배된 호모이지고트 부모주와 유전자형 AABB의 교배를 고려한다.A, A, B, B는 유전자 A, B의 대립 유전자를 나타낸다.이러한 호모 접합성 부모 균주를 교차시키면 유전자형 AaBb를 가진 이중 헤테로 접합자인 F1 세대 자손을 낳는다.F1 자손 AaBb는 감수분열 시 유전자 B의 대립 유전자와는 독립적으로 분류되기 때문에 동일한 주파수(25%)로 AB, Ab, aB, Ab인 배우자를 생성한다.4개의 배우자 중 2개(50%)(Ab와 aB)는 부모 세대에는 존재하지 않았습니다.이 생식체들은 재조합 생식체를 나타냅니다.재조합 배우자는 원래의 이배체 세포를 구성하는 두 개의 반수체 배우자와는 다른 배우자입니다.이 예에서는 4개의 생식체 중 2개가 재조합 생식체였기 때문에 재조합 빈도는 50%이다.
두 유전자가 서로 다른 염색체에 있거나 같은 염색체에 광범위하게 떨어져 있을 때 재조합 빈도는 50%가 될 것이다.이것은 독립적인 분류의 결과이다.
두 유전자가 같은 염색체 위에 가까이 있으면 독립적으로 분류되지 않고 연결된다고 한다.다른 염색체에 위치한 유전자는 독립적으로 분류되어 재조합 빈도가 50%인 반면, 연결된 유전자는 재조합 빈도가 50% 미만이다.
연결의 예로 William Bateson과 Reginald Punnett의 [citation needed]고전적인 실험을 생각해 보십시오.그들은 스위트피에서의 특성 유전에 관심이 있었고 두 가지 유전자, 즉 꽃 색깔에 대한 유전자 (P, 보라색, p, 빨간색)와 꽃가루 알갱이의 모양에 영향을 미치는 유전자 (L, 긴, 그리고 l, 둥근 모양)를 연구하고 있었다.그들은 순수한 선 PPLL과 ppl을 교차시킨 후 결과 PpLl 선을 자가 교차시켰습니다.멘델 유전학에 따르면, 예상되는 표현형은 PL:PL:pL:pl의 9:3:3:1 비율로 발생할 것이다.놀랍게도, 그들은 PL과 PL의 빈도가 증가하고 PL과 pL의 빈도가 감소하는 것을 관찰했다(아래 표 참조).
| 표현형 및 유전자형 | 관찰된 | 9:3:3:1의 비율로 예상 |
|---|---|---|
| 퍼플, 롱(P_L_) | 284 | 216 |
| 보라색, 원형(P_ll) | 21 | 72 |
| 레드, 롱(ppL_) | 21 | 72 |
| 빨강, 둥근(ppl) | 55 | 24 |
그들의 실험은 P와 L 대립 유전자와 P와 L 대립 유전자의 연관성을 밝혀냈다.L과 함께 발생하는 P 및 L과 함께 발생하는 p의 빈도는 재조합 P 및 pL의 빈도보다 크다.재조합 빈도는 역교차보다 F2 교차로 [4]계산하기가 더 어렵지만, 위 표의 관측된 자손 수와 예상 자손 수 사이의 적합성이 결여되어 있어 50% 미만임을 알 수 있다.
이 경우 자손은 하나의 염색체에 연결된 두 개의 지배적 대립 유전자를 받았다(커플링 또는 시스 배열로 참조).그러나 교차 후, 일부 자손은 두 번째 특성(예: 원형)에 대한 열성 대립 유전자에 연결된 한 가지 특징(예: 보라색)에 대한 지배적 대립 유전자를 가진 하나의 부모 염색체를 받을 수 있으며, 다른 부모 염색체(예: 빨간색과 긴색)에 대해서는 그 반대일 수 있다.이를 반발 또는 트랜스 어레인지먼트라고 합니다.여기서 표현형은 여전히 보라색이고 길지만 열성 부모와의 테스트 교배는 두 개의 교차 표현형의 훨씬 더 많은 비율을 가진 자손을 낳을 것이다.이 예에서는 그러한 문제가 발생하지 않을 수 있지만, 일부 작물의 질병 내성을 위해 번식할 때 바람직하지 않은 반발 연계가 나타난다.
이중 헤테로 접합체 내 대립 유전자의 두 가지 가능한 배열인 시스와 트랜스(trans)를 배우자상이라고 하며, 단계화는 주어진 개인에게 둘 중 어떤 것이 존재하는지 결정하는 과정이다.
두 개의 유전자가 같은 염색체에 위치할 때, 유전자 사이의 재조합을 생성하는 교차의 가능성은 두 유전자 사이의 거리와 관련이 있습니다.따라서 재조합 빈도의 사용은 연계 지도 또는 유전자 지도를 개발하기 위해 사용되어 왔다.
하지만, 재조합 빈도는 연결된 두 유전자 사이의 거리를 과소평가하는 경향이 있다는 것에 주목하는 것이 중요하다.이것은 두 유전자가 더 멀리 떨어져 있을 때, 그들 사이에 교차하는 횟수가 두 배 또는 짝수일 확률도 증가하기 때문이다.두 유전자 사이의 두 배 또는 짝수 교차는 그들이 같은 배우자에 공분할되어 예상되는 재조합 자손 대신에 부모 자손을 낳게 됩니다.앞에서 설명한 바와 같이 Kosambi 변환과 Haldane 변환은 여러 크로스오버에 [15][16]대해 수정을 시도합니다.
유전자 내 유전부위 연계
1950년대 초반에는 염색체 내 유전자가 유전자 재조합에 의해 분리되지 않고 끈에 구슬처럼 배열된 별개의 실체라는 견해가 지배적이었다.1955년부터 1959년까지 벤저는 박테리오파지 T4의 rII 돌연변이를 사용하여 유전자 재조합 실험을 수행했다.그는 재조합 테스트에 기초하여 돌연변이 부위가 선형 [17][18]순서로 매핑될 수 있다는 것을 발견했다.이 결과는 그 유전자가 독립적으로 돌연변이를 일으킬 수 있는 많은 부위의 DNA 길이에 해당하는 선형 구조를 가지고 있다는 핵심 아이디어에 대한 증거를 제공했다.
Edgar 등은 [19]rII 돌연변이 간의 재조합 빈도가 엄격히 첨가되지 않음을 보여주는 박테리오파지 T4의 r 돌연변이에 대한 매핑 실험을 수행했다.2개의 rII 돌연변이(a x d)의 교차로부터의 재결합 빈도는 일반적으로 인접한 내부 하위 간격(a x b) + (b x c) + (c x d)의 재결합 빈도의 합보다 작다.엄격히 가법적이지는 않지만 유전자 재조합의 기본 분자 메커니즘을 반영하는 체계적인 관계가 관찰되었다[20].
재조합 빈도 변화
염색체의 재조합은 감수분열 과정에서 필수적인 과정이지만, 유기체와 종 간에 교차하는 빈도는 매우 다양합니다.성적 이형성 재조합 속도는 이형성이라고 불리며, 남성과 여성 사이의 일반적인 비율보다 더 자주 관찰된다.포유동물에서 암컷은 수컷에 비해 재결합률이 높은 경우가 많다.비율의 차이에 영향을 미치는 독특한 선택 작용 또는 감수성 동인이 있다는 이론이 있다.비율의 차이는 또한 난형성과 정자 [citation needed]형성에 있어 감수 분열의 매우 다른 환경과 조건을 반영할 수 있다.
재조합 빈도에 영향을 미치는 유전자
DNA의 처리에 관여하는 단백질을 코드하는 유전자의 돌연변이는 종종 재조합 빈도에 영향을 미친다.박테리오파지 T4에서는 복제 DNA 중합효소[제네 생성물 43(gp43)]의 발현을 감소시키는 돌연변이가 재조합(감소결합)을 몇 [21][22]배 증가시킨다.재조합의 증가는 템플릿 스위치와 같은 재조합 이벤트인 결함 있는 DNA 중합효소에 의한 복제 오류, 즉 복사 선택 재조합 이벤트 때문일 수 있습니다.[23]DNA 합성에 사용되는 두 효소인 DNA 리가아제(gp30)[24][22]와 dCMP 히드록시메틸라아제(gp42)[21][22]의 발현을 감소시키는 돌연변이에 의해서도 재조합이 증가한다.
핵산가수분해효소 기능을 가진 단백질을 코드하는 유전자 돌연변이([24][22]gp46 및 gp47)에 의해 재조합이 감소(연계 증가)되며, 박테리오파지 ubsX 유전자의 DNA결합단백질 돌연변이(gp32)[22]도 [25]재조합을 실질적으로 감소시킨다.uvsX 유전자는 [26]재조합에 중추적인 역할을 하는 대장균의 잘 연구된 recA 유전자와 유사하다.
감수 분열 지표
매우 큰 혈통이나 전체 유전자 염기서열 분석과 같은 매우 조밀한 유전자 마커 데이터를 사용하여 재조합을 정확하게 찾을 수 있다.이러한 유형의 유전자 분석에서 감수분열 지표는 혈통에서 각 감수분열별로 게놈의 각 위치에 할당된다.이 표시기는 부모 염색체의 어떤 복사가 그 위치에서 전달된 배우자에 기여하는지를 나타냅니다.예를 들어, 부모 염색체의 '첫 번째' 복제에서 나온 대립 유전자가 전달되면, 그 감수 분열에 '0'이 할당될 수 있다.만약 부모 염색체의 '두 번째' 복제에서 나온 대립 유전자가 전달된다면, '1'은 감수 분열에 할당될 것이다.부모의 두 대립 유전자는 각각 두 명의 조부모로부터 나왔다.이 지표들은 질병에 책임이 있는 유전자를 식별하는데 사용되는 동일한 IBD 상태 또는 유전 상태를 결정하는 데 사용됩니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Cooper, DN; Krawczak, M; Polychronakos, C; Tyler-Smith, C; Kehrer-Sawatzki, H (October 2013). "Where genotype is not predictive of phenotype: towards an understanding of the molecular basis of reduced penetrance in human inherited disease". Human genetics. 132 (10): 1077–130. doi:10.1007/s00439-013-1331-2.
- ^ Lobo, Ingrid; Shaw, Kenna. "Discovery and Types of Genetic Linkage". Scitable. Nature Education. Retrieved 21 January 2017.
- ^ Bateson, W; Saunders, ER; Punnett, RC (18 May 1904). Reports to the Evolution committee of the Royal Society. London: Harrison and Sons, Printers. Retrieved 21 January 2017.
- ^ a b Fisher, RA; Balmukand, B (July 1928). "The estimation of linkage from the offspring of selfed heterozygotes". Journal of Genetics. 20 (1): 79–92. doi:10.1007/BF02983317. S2CID 27688031.
- ^ Mader, Sylvia (2007). Biology Ninth Edition. New York: McGraw-Hill. p. 209. ISBN 978-0-07-325839-3.
- ^ Griffiths, AJF (2000). An Introduction to Genetic Analysis (7th ed.). W. H. Freeman.
- ^ Lanktree, Matthew B.; Johansen, Christopher T.; Joy, Tisha R.; Hegele, Robert A. (2010), "A Translational View of the Genetics of Lipodystrophy and Ectopic Fat Deposition", in Bouchard, Claude (ed.), Progress in Molecular Biology and Translational Science, Genes and Obesity, vol. 94, Academic Press, pp. 159–196, doi:10.1016/b978-0-12-375003-7.00006-6, ISBN 9780123750037, PMID 21036325
- ^ a b Cantor, Rita M. (2013), "Analysis of Genetic Linkage", in Rimoin, David; Pyeritz, Reed; Korf, Bruce (eds.), Emery and Rimoin's Principles and Practice of Medical Genetics (6th ed.), Academic Press, pp. 1–9, doi:10.1016/b978-0-12-383834-6.00010-0, ISBN 9780123838346
- ^ Morton NE (1955). "Sequential tests for the detection of linkage". American Journal of Human Genetics. 7 (3): 277–318. PMC 1716611. PMID 13258560.
- ^ Nyholt, Dale R (August 2000). "All LODs Are Not Created Equal". American Journal of Human Genetics. 67 (2): 282–288. doi:10.1086/303029. PMC 1287176. PMID 10884360.
- ^ Risch, Neil (June 1991). "A Note on Multiple Testing Procedures in Linkage Analysis". American Journal of Human Genetics. 48 (6): 1058–1064. PMC 1683115. PMID 2035526.
- ^ Ferreira, Manuel A. R. (2004-10-01). "Linkage Analysis: Principles and Methods for the Analysis of Human Quantitative Traits". Twin Research and Human Genetics. 7 (5): 513–530. doi:10.1375/twin.7.5.513. ISSN 2053-6003. PMID 15527667. S2CID 199001341.
- ^ Gusella, James F.; Frontali, Marina; Wasmuth, John J.; Collins, Francis S.; Lehrach, Hans; Myers, Richard; Altherr, Michael; Allitto, Bernice; Taylor, Sherry (1992-05-01). "The Huntington's disease candidate region exhibits many different haplotypes". Nature Genetics. 1 (2): 99–103. doi:10.1038/ng0592-99. ISSN 1546-1718. PMID 1302016. S2CID 25472459.
- ^ Mark J. Daly; Hirschhorn, Joel N. (2005-02-01). "Genome-wide association studies for common diseases and complex traits". Nature Reviews Genetics. 6 (2): 95–108. doi:10.1038/nrg1521. ISSN 1471-0064. PMID 15716906. S2CID 2813666.
- ^ Griffiths, AJF; Miller, JH; Suzuki, DT (2000). "Accurate calculation of large map distances, Derivation of a mapping function". An Introduction to Genetic Analysis (7th ed.). New York: W. H. Freeman. ISBN 978-0-7167-3520-5.
- ^ Griffiths, AJF; Miller, JH; Suzuki, DT (2000). "Accurate calculation of large map distances, Figure 6-4". An Introduction to Genetic Analysis (7th ed.). New York: W. H. Freeman. ISBN 978-0-7167-3520-5. 지도 단위와의 이상적인 1-1 등가 재조합 빈도 비율(RF%)과 비교한 지도 함수 그래프.
- ^ 벤저 S.박테리오파지의 유전자 영역의 미세한 구조.Proc Natl Acad Sci US 1955; 41(6): 344-354.doi: 10.1073/pnas.41.6.344
- ^ 벤저 S.유전자 미세 구조의 토폴로지.Proc Natl Acad Sci U S. A. 1959;45 (11) : 1607-1620.doi : 10.1073 / pnas . 45 . 11 . 1607
- ^ Edgar RS, Feyman RP, Klein S, Lielausis I, Steinberg CM.박테리오파지 T4D의 돌연변이 r개에 대한 매핑 실험.유전학.1962;47:179~186.PMC 1210321.PMID 13889186
- ^ 피셔 KM, 번스타인 H파지 T4D의 RIIA 시스트론 간격의 가산도.유전학.1965;52(6):1127–1136. PMC 1210971.PMID 5882191
- ^ a b 번스타인 H.파지 T4D의 DNA-중합효소 및 디옥시티딜레이트 히드록시메틸라아제 돌연변이 결함 재조합에 대한 영향.유전학.1967;56(4):755-769
- ^ a b c d e 버거 H, 워렌 AJ, 프라이 KET4D 박테리오파지의 호박 돌연변이로 인한 유전자 재조합의 변화.JVIrol. 1969;3(2):171-175.doi:10.1128/JVI.3.2.171-175.1969
- ^ 번스타인 H. 유전자 내 재조합 메커니즘에 관한 연구.I. 박테리오파지 T4의 rII 영역.(1962) 이론생물학 저널.1962; 3, 335-353.https://doi.org/10.1016/S0022-5193(62)80030-7
- ^ a b 번스타인 H. T4 파지의 복구 및 재조합.I. 재조합에 영향을 미치는 유전자콜드 스프링 하버 심프 수량 Biol. 1968;33:325-331.doi:10.1101/sqb.1968.033.01.037
- ^ 햄릿 NV, 버거 H. 돌연변이가 박테리오파지 T4에서 유전자 재조합과 DNA의 수복을 변화시킨다.바이러스학.1975;63 (2):539-567.doi:10.1016/0042-6822(75)90326-8
- ^ 후지사와 H, 요네사키 T, 미나가와 T.T4 재조합 유전자 uvsX의 배열 및 대장균의 recA 유전자와의 비교.핵산 규격 1985;13(20):7473-7481.doi:10.1093/nar/13.20.7473
| 라이브러리 리소스 정보 유전자 매핑 |
- Griffiths AJF; Miller JH; Suzuki DT; Lewontin RC; et al. (1993). "Chapter 5". An Introduction to Genetic Analysis (5th ed.). New York: W.H. Freeman and Company. ISBN 978-0-7167-2285-4.
- Poehlman JM; Sleper DA (1995). "Chapter 3". Breeding Field Crops (4th ed.). Iowa: Iowa State Press. ISBN 978-0-8138-2427-7.