피코나바이러스
Picornavirus| 피코르나바이러스과 | |
|---|---|
 | |
| 폴리오 바이러스의 전자 현미경 사진 | |
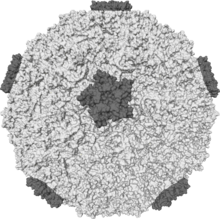 | |
| 단백질 스파이크를 나타내는 인간 라이노 바이러스의 등각면 | |
| 바이러스 분류 | |
| (순위 미지정): | 바이러스 |
| 영역: | 리보비리아 |
| 왕국: | 오르토나비라과 |
| 문: | 피수비리코타 |
| 클래스: | 피소니비리세테스 |
| 주문: | 피코르나비랄레스 |
| 패밀리: | 피코르나바이러스과 |
| 속[1] | |
피코나바이러스는 어류,[2] 포유류, 조류를 포함한 척추동물을 감염시키는 관련되지 않은 RNA 바이러스 그룹이다.그것들은 30 nm의 정십면체 캡시드를 가진 작고, 양성의, 단일 가닥의 RNA 바이러스의 큰 패밀리를 나타내는 바이러스입니다.이 패밀리의 바이러스는 감기, 소아마비, 뇌수막염, 간염, [3][4][5][6]마비를 포함한 다양한 질병을 일으킬 수 있다.
피코나비루스는 피코나비루스과, 피코나비루스목, 리보비리아 왕국을 구성한다.이 과에는 158종이 있으며 68속으로 분류된다.대표적인 예로는 엔테로바이러스(라이노바이러스와 폴리오바이러스 포함), 아프토바이러스, 심장바이러스,[1][7] 간바이러스가 있다.
어원학
"피코나바이러스"라는 이름은 이중 어원을 가지고 있다.첫째, 이 이름은 "폴리오바이러스, 에테르에 대한 불감증, 콕사키바이러스, 고아바이러스, 라이노바이러스, 리보핵산"의 약자인 피코르나에서 유래했다.둘째, 이 이름은 매우 작은 RNA [8]바이러스의 그룹을 설명하기 위해 rna와 결합된 매우 작은 측정 단위(10과−12 동일)를 나타내는 pico-에서 유래했다.
역사
최초로 발견된 동물 바이러스는 구제역 바이러스(1897년)였다.피코나바이러스과에 [5]속하는 아프토바이러스속의 원형종이다.플라크 분석은 폴리오바이러스를 사용하여 개발되었습니다.배양에서의 바이러스 복제의 발견도 1949년에 폴리오바이러스와 함께 이루어졌습니다.배양된 [9]세포에서 감염 바이러스가 생성된 것은 이번이 처음이었다.폴리단백질 합성, 내부 리보솜 진입부위, 미캡 mRNA는 모두 폴리오바이러스 감염세포를 연구해 발견됐으며 폴리오바이러스 복제는 동물에서 RNA바이러스로 만든 첫 감염 DNA 복제였다.라이노바이러스와 함께 폴리오바이러스는 X선 결정학에 의해 구조가 결정된 최초의 동물 바이러스였다.RNA의존성 RNA중합효소는 피코나바이러스속인 멘고바이러스에서 [10]발견되었다.
바이러스학
구조.

피코나바이러스는 20면체 캡시드와 [4]함께 포개져 있지 않다.캡시드는 촘촘히 채워진 20면체 구조로 60개의 프로토머를 배열한 것입니다.각 프로토머는 VP(바이러스 단백질) 1, 2, 3, 4로 알려진 4개의 폴리펩타이드로 구성됩니다. VP2 및 VP4 폴리펩타이드는 서로 다른 캡시드 성분을 제공하기 위해 분해된 VP0로 알려진 하나의 프로토머에서 유래합니다.이십면체는 삼각수 3을 가지고 있다고 하는데, 이것은 이십면체 구조에서 캡시드를 구성하는 60개의 삼각형 각각이 모서리에 서브유닛이 있는 세 개의 작은 삼각형으로 분할된다는 것을 의미합니다.
많은 피코나바이러스는 이코사면체의 12개의 꼭지점 주위에 형성된 깊은 틈을 가지고 있다.캡시드의 바깥쪽 표면은 VP1, VP2, VP3의 영역으로 구성됩니다.각 정점 주위에는 VP1과 VP3의 C 흰자리가 늘어선 협곡이 있다.캡시드의 내부 표면은 VP4와 VP1. J. Esposito and Freederick A의 N개미니로 구성되어 있습니다.Murphy는 X선 결정학과 극저온 전자 [9]현미경을 사용하여 협곡이라고 불리는 구개 구조를 시연합니다.
탈수의 종류와 정도에 따라, 바이러스 입자의 [7]지름은 약 30-32 nm이다.바이러스 게놈은 길이가 약 2500nm로 인산기가 일으키는 RNA의 음전하 균형을 맞추기 위해 나트륨 이온 등의 물질과 함께 캡시드 안에 촘촘히 포장되어 있다.
게놈
피코나바이러스는 볼티모어의 바이러스 분류 체계에서 IV 그룹으로 분류되는데, 이는 단일 가닥의 양성 감각의 RNA 게놈을 포함하고 있기 때문이다.그들의 게놈 길이는 [7]6.7에서 10.1 사이이다.대부분의 양성 감각의 RNA 게놈처럼 유전 물질만 감염됩니다; 비록 바이러스 입자에 포함된 것보다 훨씬 덜 치명적이지만, RNA는 세포에 감염되었을 때 감염성을 높일 수 있습니다.게놈 RNA는 RNA 중합효소에 의한 전사의 프라이머로 사용되는 단백질이 5' 말단에 있기 때문에 특이하다.이 프라이머는 VPg 게놈이라고 불리며 범위는 2kb에서 3kb 사이입니다.VPg에는 티로신 잔류물이 3' 끝에 포함되어 있습니다.RNA의 [9][11]5' 말단과 공유 결합하기 위한 –OH 공급원으로서 티로신.
게놈은 분할되어 있지 않고 양성(mRNA 포유동물과 같은 감각, 5'에서 3'까지 읽힌다)이다.포유류의 mRNA와 달리, 피코나바이러스는 5'의 캡을 가지고 있지 않지만 VPg로 알려진 바이러스 코드 단백질을 가지고 있다.그러나 포유류의 mRNA와 마찬가지로 게놈의 끝에는 폴리(A) 꼬리가 있다.피코나바이러스 게놈의 양단에서 미번역영역(UTR)이 발견된다.5' UTR은 길이가 약 500–1200 뉴클레오티드(nt)로 30–650nt인 3' UTR에 비해 일반적으로 더 길다.5' UTR은 번역에서, 3'은 음의 스트랜드 합성에 중요하다고 생각되지만, 5' 말단은 바이러스의 독성에 대한 역할도 할 수 있다.나머지 게놈은 5' 말단 구조단백질과 3' 말단 비구조단백질을 단일 폴리단백질로 코드한다.
폴리단백질은 L-1ABCD-2ABCD-3ABCD로 구성되며, 각 문자는 단백질을 나타내지만, 이 배치에는 변이가 존재한다.
1A, 1B, 1C, 1D 단백질은 각각 캡시드 단백질 VP4, VP2, VP3, VP1이다.바이러스 코드 단백질 분해 효소는 분열을 수행하는데, 그 중 일부는 분자 내이다.먼저 폴리단백질을 절단하여 P1, P2, P3를 생성한다.P1은 프로캅시드를 형성하는 단백질인 VP0, VP3, VP1로 분해되기 전에 N 말단에서 미리스틸화됩니다.VP0은 나중에 분해되어 VP2 및 VP4가 생성됩니다.다른 분열 생성물로는 3B(VPg), 2C(ATPase), 3D(RNA 중합효소)[9][12]가 있습니다.
레플리케이션
RNA 원소
피코나바이러스의 게놈 RNA는 여러 개의 RNA 원소를 가지고 있으며, 음성 및 양성 가닥 RNA 합성에 모두 필요하다.복제에는 CRE(cis-acting Replication Element)가 필요합니다.CRE를 포함하는 스템루프 구조는 위치와 무관하지만 식별되면 바이러스 유형 간의 위치에 따라 변화합니다.또한 바이러스 RNA의 3' 말단 원소는 피코르나바이러스의 RNA 복제에 유의하고 효율적이다.피코르나바이러스의 3' 말단에는 감염에 필요한 폴리(A) 관이 포함되어 있다.그러나 RNA 합성은 이 지역에서 일어나는 것으로 가정된다.폴리오바이러스의 3' 말단 NCR은 음성 가닥 합성에 필요하지 않지만 양성 가닥 합성에 중요한 요소이다.또한 RNA 복제 및 폴리오바이러스 번역 개시를 위해 2차 구조 요소를 포함하는 5' 말단 NCR이 필요하다.내부 리보솜 진입 부위는 캡의존적인 번역 개시를 가능하게 하는 RNA 구조이며, 메신저 [13]RNA의 중간에서 번역을 개시할 수 있다.
라이프 사이클
바이러스 입자는 세포 표면 수용체에 결합한다.세포표면수용체는 피코나바이러스의 혈청형별로 특징지어진다.예를 들어 폴리오바이러스 수용체는 당단백질 CD155로 인간과 다른 영장류 종에 대한 특별한 수용체이다.이러한 이유로, 1990년대에 세포 표면에 CD155 수용체를 가진 트랜스제닉 마우스가 개발되기 전까지는 많은 실험실에서 폴리오바이러스를 만들 수 없었다.이 동물들은 감염될 수 있고 복제와 [9]병인에 대한 연구에 사용될 수 있다.결합은 바이러스 캡시드 단백질의 구조 변화를 일으키고 미리스트산이 방출된다.그 산은 RNA가 [14]주입되는 세포막에 구멍을 형성한다.
일단 세포 내에 들어가면 RNA는 코팅을 풀고 바이러스 RNA의존성 RNA중합효소를 사용하여 형성된 이중사슬 RNA 중간체를 통해 (+) 가닥 RNA 게놈이 복제된다.숙주 세포 리보솜에 의한 번역은 통상대로 5' G캡에 의해 개시되는 것이 아니라 내부 리보솜 진입 부위에 의해 개시된다.바이러스의 라이프 사이클은 매우 빨라 평균 8시간 이내에 모든 복제 과정이 완료됩니다.그러나 최초 감염 후 30분 이내에 세포단백질 합성은 거의 0으로 감소한다. 기본적으로 세포단백질의 고분자 합성은 차단된다.바이러스 단백질이 합성되기 시작하고 감염 후 시간이 약 3시간이 되면 점차 확산되기 시작하는 세포질에 액포가 나타나기 전에 다음 1~2시간 동안 핵에서 염색질과 균질성의 상실이 발생한다.이 시간이 지나면 세포 플라즈마 막이 투과성을 갖게 되고, 4~6시간이 지나면 바이러스 입자가 모여 세포질에서 볼 수 있다.약 8시간이 지나면 세포는 사실상 사멸하고, 바이러스 입자를 방출하기 위해 용해된다.
단일 단계 성장 곡선과 같은 실험의 실험 데이터를 통해 피코나바이러스의 복제를 매우 자세히 관찰할 수 있었습니다.전체 복제는 숙주 세포질 내에서 일어나고 감염은 핵을 포함하지 않는 세포와 악티노마이신 D로 처리된 세포에서도 일어날 수 있다.
번역은 -1 리보솜 프레임시프트, 바이러스 시작 및 리보솜 건너뛰기에 의해 이루어집니다.바이러스는 용융과 바이로포린에 의해 숙주 세포에서 빠져나간다.척추동물은 자연 숙주의 역할을 한다.전염 경로는 분변-구강,[4] 접촉, 섭취 및 공기로 전달되는 입자입니다.
바이러스단백질(VPG)
피코나바이러스는 세포 mRNA와 같은 7-메틸구아노신 캡 대신 5' 말단에 공유적으로 연결된 바이러스 단백질(VPG)을 가지고 있다.바이러스 RNA 중합효소는 프라이머로 VPg를 사용한다.프라이머로서의 VPg는 양의 가닥과 음의 가닥 RNA 합성을 모두 사용합니다.피코나바이러스 복제는 VPg의 요리딜화에 의해 시작된다.티로신 [3]잔기의 수산기에서 요릴화된다.VPg 프라이머 메커니즘은 RNA 바이러스의 피코르나바이러스(장기 바이러스, 기타), 추가 바이러스군(포티 바이러스, 코모 바이러스, 칼리시 바이러스 등) 및 피코르나바이러스 유사 슈퍼그룹(코로나 바이러스, 노타 바이러스 등)에 의해 사용된다.이 메커니즘은 구제역을 일으키는 동물 병원체인 아프토바이러스뿐만 아니라 장바이러스(폴리오바이러스, 콕사키 바이러스 등 많은 인간 병원체를 포함한다)에 대해서도 가장 잘 연구되고 있다.
이 그룹에서 프라이머 의존성 RNA 합성은 VPg에[15] 연결된 작은 22~25-아미노산 길이의 바이러스 단백질을 사용하여 중합효소 활성을 시작하고 프라이머는 RNA [16]템플릿의 5' 말단에 공유 결합된다.요오드릴화는 VPg의 세 번째 위치에 있는 티로신 잔류물에서 발생합니다.RNA 스템루프 구조인 CRE는 VPg의 요릴릴화를 위한 템플릿으로 작용하여 VPgPUpUOH를 합성한다.CRE-RNA 구조 내의 돌연변이는 VPg 요릴화를 방지하고 VPg 배열 내의 돌연변이는 Rdrp 촉매 [17]활성을 심각하게 감소시킬 수 있다.VPg의 티로신 수산화기는 CRE- 및 VPgpUpUOH에 의존하지 않는 방법으로 음가닥 RNA 합성을 프라이밍할 수 있지만, 양가닥 RNA 합성을 위해서는 CRE 의존 VPgpUpUOH 합성이 절대적으로 필요하다.CRE 의존성 VPg 유리딜화는 바이러스 RNA 복제 및 CRE 의존성 VPgPUOH 합성에 필요한 UTP의 K를m¬ 낮추고, 특히 UTP 농도가 [18]제한적일 때 효율적인 음성 가닥 RNA 합성에 필요하다.VPgpUpUOH 프라이머는 RdRp에 의한 뉴클레오티드 염기의 첨가로 지속될 수 있는 RNA 템플릿의 3' 말단으로 전달된다.구제역[19] 바이러스와 콕사키 바이러스[20] B3의 VPgs에 대한 부분 결정 구조는 피코나바이러스의 작은 VPgs에 대한 바이러스 중합효소 상에 두 개의 부위가 있을 수 있음을 시사한다.폴리오바이러스 VPg[21] 및 VPGPU의[22] NMR 용액구조는 유리딜화가 VPg의 구조를 안정화시키고, 그 외에는 용액이 상당히 유연하다는 것을 보여준다.두 번째 부위는 요릴화에 사용될 수 있으며, 그 후 VPGPU가 RNA 합성을 시작할 수 있다.이제 [23]막 구조가 드러나기 시작한 칼리시바이러스의 VPg 프라이머는 피코나바이러스보다 훨씬 크다.요오드릴화와 프라이밍을 위한 메커니즘은 이 모든 그룹에서 상당히 다를 수 있다.
VPg우리딜화는 전구체 단백질의 사용을 포함할 수 있으며, 이는 전장 RNA의 생산을 위해 양 또는 음의 가닥 RNA의 3' 말단에서 이우리딜릴화된 VPg 함유 전구체의 위치를 결정하기 위한 가능한 메커니즘의 결정을 가능하게 한다.VPg우리딜화 효율의 결정 요인은 형성 및/붕괴 또는 재작성을 시사한다.고용된 [24]VPg 공여자에 따라 시험관내 속도 제한 단계로 요오드화 제품의 리스.전구체 단백질은 또한 VPg-CRE 특이성과 [25]안정성에 영향을 미친다.VPg가 결합하는 상부 RNA 스템 루프는 VPg 및 Pol의 유지 및 모집에 큰 영향을 미칩니다.CRE의 스템루프는 부분적으로 풀리기 때문에 선행 컴포넌트가 VPg 및 Pol4를 바인드 및 채용할 수 있습니다.CRE 루프는 개시 구성요소가 결합하는 정의된 합의 시퀀스를 가지고 있지만, 지지 스템에 대한 합의 시퀀스는 존재하지 않으며, 이는 CRE의 구조적 안정성만이 [26]중요하다는 것을 의미한다.
- 2개의 3CD(VPG 복합체) 분자는 상부 스템에 접하는 3C 도메인(VPG 도메인)과 하부 스템에 접하는 3D 도메인(VPG 도메인)으로 CRE에 결합합니다.
- 3C 이합체는 스템을 형성하는 단일 가닥과 보다 안정적인 상호작용을 형성하여 RNA 스템을 엽니다.
- 3Dpol은 3Dpol의 뒷부분 서브도메인과 3CD의 한쪽 또는 양쪽 3C 서브도메인의 표면 사이의 물리적 상호작용에 의해 이 복합체에 모집되어 유지된다.
VPg는 또한 이동 단백질(MP)에 의한 바이러스 게놈의 특정 인식에 중요한 역할을 할 수 있는데, 이동 단백질은 식물 바이러스가 하나의 감염된 세포에서 이웃 [27]세포로 이동할 수 있도록 암호화하는 비구조적 단백질이다.MP와 VPg는 세포에서 세포로 바이러스 RNA를 전달하기 위한 특이성을 제공하기 위해 상호작용합니다.에너지 요구 사항을 충족시키기 위해 MP는 또한 세포 내 ATP 효소인 P10과 상호작용합니다.
질병.
피코나바이러스는 다양한 질병을 일으킨다.피코나바이러스 계열의 장바이러스는 그들의 이름에 반영되어 있는 장내 기관에 감염된다.라이노바이러스는 주로 코와 목에 감염된다.장바이러스는 37°C에서 복제되는 반면, 라이노바이러스는 33°C에서 더 잘 자란다. 이는 코의 온도가 낮기 때문이다.장바이러스는 산성 조건에서도 안정적이기 때문에 위산에 노출되어도 살아남을 수 있다.반대로, 라이노바이러스는 산성이기 때문에 (낮은 pH 조건에 의해 비활성화되거나 파괴됨) 라이노바이러스는 코와 목구멍으로 감염이 제한된다.
분류법
다음과 같은 속들이 [1]인식되고 있습니다.
- 알리바이러스
- 아일루리바이러스
- 암피바이러스
- 아나티바이러스
- 아프토바이러스
- 아쿠아마바이러스
- 아비헤파토바이러스
- 아비시바이러스
- 부스피바이러스
- 보피바이러스
- 케실리바이러스
- 심장바이러스
- 코사바이러스
- 크라헬리바이러스
- 크로히바이러스
- 다니피바이러스
- 디케이피바이러스
- 디레사피바이러스
- 엔테로바이러스
- 엘보바이러스
- 펠리피바이러스
- 피피바이러스
- 갈리바이러스
- 그루헬리바이러스
- 그루소피바이러스
- 하르카바이러스
- 헤미피바이러스
- 간바이러스
- 훈니바이러스
- 코부바이러스
- 쿤사키바이러스
- 림니피바이러스
- 리부피바이러스
- 루도피바이러스
- 말라가시바이러스
- 마르수피바이러스
- 메가바이러스
- 미스키바이러스
- 모사바이러스
- 무피바이러스
- 미로피바이러스
- 오리바이러스
- 오시바이러스
- 파라보바이러스
- 파레코바이러스
- 파시바이러스
- 패시바이러스
- 페마피바이러스
- 포에시바이러스
- 포타미피바이러스
- 피고세피바이러스
- 라보바이러스
- 라피바이러스
- 라지다피바이러스
- 로헬리바이러스
- 로자바이러스
- 사코부바이러스
- 살리바이러스
- 사펠로바이러스
- 세네카바이러스
- 산바바이러스
- 시시니바이러스
- 시마피바이러스
- 테초바이러스
- 토치바이러스
- 토토리바이러스
- 트레모바이러스
- 트로피바이러스
「 」를 참조해 주세요.
레퍼런스
- ^ a b c "Virus Taxonomy: 2020 Release". International Committee on Taxonomy of Viruses (ICTV). March 2021. Retrieved 20 May 2021.
- ^ Altan E, Kubiski SV, Boros Á, Reuter G, Sadeghi M, Deng X, Creighton EK, Crim MJ, Delwart E (2019). "A highly divergent picornavirus infecting the Gut Epithelia of Zebrafish (Danio rerio) in research institutions worldwide". Zebrafish. 16 (3): 291–299. doi:10.1089/zeb.2018.1710. PMID 30939077. S2CID 92999901.
- ^ a b Ryu WS (2016). "Chapter 11 – Picornavirus". Molecular virology of human pathogenic viruses. Korea: Academic Press. pp. 153–64. doi:10.1016/b978-0-12-800838-6.00011-4. ISBN 978-0-12-800838-6.
- ^ a b c "Viral Zone". ExPASy. Retrieved 15 June 2015.
- ^ a b Martinez-Salas E, Saiz M, Sobrino F (2008). "Foot-and-Mouth Disease Virus". In Mettenleiter TC, Sobrino F (eds.). Animal Viruses: Molecular Biology. Norfolk, UK: Caister Academic Press. ISBN 978-1-904455-22-6.
- ^ Lau SK, Woo PC, Lai KK, Huang Y, Yip CC, Shek CT, Lee P, Lam CS, Chan KH, Yuen KY (September 2011). "Complete genome analysis of three novel picornaviruses from diverse bat species". Journal of Virology. 85 (17): 8819–28. doi:10.1128/JVI.02364-10. PMC 3165794. PMID 21697464.
- ^ a b c "Picornaviridae - Picornaviridae - Picornavirales". International Committee on Taxonomy of Viruses (ICTV). Retrieved 12 June 2020.
- ^ "Picornaviridae". International Committee on Taxonomy of Viruses (ICTV). October 2017. Retrieved 5 February 2019.
- ^ a b c d e Carter JB, Saunders VA (2007). "Picornaviruses (and other plus-strand RNA viruses)". Virology: Principles and applications. Chichester, England: John Wiley & Sons. pp. 160–65. ISBN 978-0-470-02386-0.
- ^ Knipe DM, Howley P (2013). Fields Virology. Lippincott Williams & Wilkins. ISBN 978-1-4698-3066-7.
- ^ Zabel P, Moerman M, Lomonossoff G, Shanks M, Beyreuther K (July 1984). "Cowpea mosaic virus VPg: sequencing of radiochemically modified protein allows mapping of the gene on B RNA". The EMBO Journal. 3 (7): 1629–34. doi:10.1002/j.1460-2075.1984.tb02021.x. PMC 557569. PMID 16453534.
- ^ Acheson NH (2011). Fundamentals of Molecular Virology (2nd ed.). John Wiley & Sons, Inc. ISBN 978-0470900598.
- ^ Daijogo S, Semler BL (2011). "Mechanistic intersections between picornavirus translation and RNA replication". Advances in Virus Research. 80: 1–24. doi:10.1016/B978-0-12-385987-7.00001-4. ISBN 9780123859877. PMID 21762819.
- ^ "Pathology, Microbiology and Immunology - School of Medicine Columbia University of South Carolina".
- ^ Flanegan JB, Baltimore D (September 1977). "Poliovirus-specific primer-dependent RNA polymerase able to copy poly(A)". Proceedings of the National Academy of Sciences of the United States of America. 74 (9): 3677–80. Bibcode:1977PNAS...74.3677F. doi:10.1073/pnas.74.9.3677. PMC 431685. PMID 198796.
- ^ Ambros V, Baltimore D (August 1978). "Protein is linked to the 5' end of poliovirus RNA by a phosphodiester linkage to tyrosine". The Journal of Biological Chemistry. 253 (15): 5263–66. doi:10.1016/S0021-9258(17)30361-7. PMID 209034.
- ^ Gu C, Zeng T, Li Y, Xu Z, Mo Z, Zheng C (October 2009). "Structure-function analysis of mutant RNA-dependent RNA polymerase complexes with VPg". Biochemistry. Biokhimiia. 74 (10): 1132–41. doi:10.1134/S0006297909100095. PMID 19916926. S2CID 24968119.
- ^ Steil BP, Barton DJ (October 2008). "Poliovirus cis-acting replication element-dependent VPg Uridylylation lowers the Km of the initiating nucleoside triphosphate for viral RNA replication". Journal of Virology. 82 (19): 9400–08. doi:10.1128/JVI.00427-08. PMC 2546976. PMID 18653453.
- ^ Ferrer-Orta C, Arias A, Agudo R, Pérez-Luque R, Escarmís C, Domingo E, Verdaguer N (February 2006). "The structure of a protein primer-polymerase complex in the initiation of genome replication". The EMBO Journal. 25 (4): 880–88. doi:10.1038/sj.emboj.7600971. PMC 1383552. PMID 16456546.
- ^ Gruez A, Selisko B, Roberts M, Bricogne G, Bussetta C, Jabafi I, et al. (October 2008). "The crystal structure of coxsackievirus B3 RNA-dependent RNA polymerase in complex with its protein primer VPg confirms the existence of a second VPg binding site on Picornaviridae polymerases". Journal of Virology. 82 (19): 9577–90. doi:10.1128/JVI.00631-08. PMC 2546979. PMID 18632861.
- ^ Schein CH, Oezguen N, Volk DE, Garimella R, Paul A, Braun W (July 2006). "NMR structure of the viral peptide linked to the genome (VPg) of poliovirus". Peptides. 27 (7): 1676–84. doi:10.1016/j.peptides.2006.01.018. PMC 1629084. PMID 16540201.
- ^ Schein CH, Oezguen N, van der Heden van Noort GJ, Filippov DV, Paul A, Kumar E, Braun W (August 2010). "NMR solution structure of poliovirus uridylyated peptide linked to the genome (VPgpU)". Peptides. 31 (8): 1441–48. doi:10.1016/j.peptides.2010.04.021. PMC 2905501. PMID 20441784.
- ^ Leen EN, Kwok KY, Birtley JR, Simpson PJ, Subba-Reddy CV, Chaudhry Y, et al. (May 2013). "Structures of the compact helical core domains of feline calicivirus and murine norovirus VPg proteins" (PDF). Journal of Virology. 87 (10): 5318–30. doi:10.1128/JVI.03151-12. PMC 3648151. PMID 23487472.
- ^ Pathak HB, Oh HS, Goodfellow IG, Arnold JJ, Cameron CE (November 2008). "Picornavirus genome replication: roles of precursor proteins and rate-limiting steps in oriI-dependent VPg uridylylation". The Journal of Biological Chemistry. 283 (45): 30677–88. doi:10.1074/jbc.M806101200. PMC 2576561. PMID 18779320.
- ^ Shen M, Wang Q, Yang Y, Pathak HB, Arnold JJ, Castro C, Lemon SM, Cameron CE (November 2007). "Human Rhinovirus Type 14 Gain-of-Function Mutants for oriI Utilization Define Residues of 3C(D) and 3Dpol That Contribute to Assembly and Stability of the Picornavirus VPg Uridylylation Complex". J. Virol. 81 (22): 12485–95. doi:10.1128/JVI.00972-07. PMC 2169002. PMID 17855535.
- ^ Yang Y, Rijnbrand R, McKnight KL, Wimmer E, Paul A, Martin A, Lemon SM (August 2002). "Sequence requirements for viral RNA replication and VPg uridylylation directed by the internal cis-acting replication element (CRE) of human rhinovirus type 14". Journal of Virology. 76 (15): 7485–94. doi:10.1128/JVI.76.15.7485-7494.2002. PMC 136355. PMID 12097561.
- ^ Roy Chowdhury S, Savithri HS (January 2011). Pfeffer S (ed.). "Interaction of Sesbania mosaic virus movement protein with VPg and P10: implication to specificity of genome recognition". PLOS ONE. 6 (1): e15609. Bibcode:2011PLoSO...615609R. doi:10.1371/journal.pone.0015609. PMC 3016346. PMID 21246040.
추가 정보
- Kahn CM, Line S, eds. (2005). "Porcine Enteroviral Encephalomyelitis". The Merck Veterinary Manual (9th ed.). Merck. ISBN 978-0-911910-50-6.
- Thompson JR, Dasgupta I, Fuchs M, Iwanami T, Karasev AV, Petrzik K, Sanfaçon H, Tzanetakis I, van der Vlugt R, Wetzel T, Yoshikawa N, et al. (ICTV Report Consortium) (April 2017). "ICTV Virus Taxonomy Profile: Secoviridae". The Journal of General Virology. 98 (4): 529–531. doi:10.1099/jgv.0.000779. PMC 5657025. PMID 28452295.
- Thompson JR (2020). "Secoviruses (Secoviridae)". Reference Module in Life Sciences. pp. 692–702. doi:10.1016/B978-0-12-809633-8.21253-3. ISBN 978-0-12-809633-8. S2CID 241194454.
외부 링크
- Büchen-Osmond C, ed. (2006). "ICTVdB – The Universal Virus Database, version 4". ICTVdB Management, Mailman School of Public Health. New York: Columbia University. Archived from the original on 10 March 2010.
- "Picornaviridae". Medical Subject Headings (MeSH). National Library of Medicine. Retrieved 3 September 2007.
- "Picornavirus". The Pirbright Institute. Retrieved 3 September 2007.
- ICTV 온라인 리포트 피코르나바이러스과
- ICTV 국제 바이러스 분류 위원회 마스터 종 목록
- ICTV 국제 바이러스 분류 위원회 (공식 사이트)
- Picornavirus – 설명, 레플리케이션, 질병
- NCBI Taxonomy 브라우저의 Picornavirus
- 국제 바이러스 분류 위원회의 피코르나바이러스과 분류
- 동물바이러스
- 바이러스 영역:피코르나바이러스과
- 바이러스 병원체 데이터베이스 및 분석 자원(ViPR): Picornaviridae
- ICTV





