커뮤니티 구조
Community structure| 시리즈의 일부 | ||||
| 네트워크 과학 | ||||
|---|---|---|---|---|
| 네트워크 타입 | ||||
| 그래프 | ||||
| ||||
| 모델 | ||||
| ||||
| ||||
복잡한 네트워크의 연구에서 네트워크의 노드가 (잠재적으로 중복될 수 있는) 노드 세트로 쉽게 그룹화되어 각 노드 세트가 내부적으로 촘촘히 연결되어 있는 경우 네트워크는 커뮤니티 구조를 갖는다고 한다.특히 중복되지 않는 커뮤니티 검출의 경우, 네트워크는 내부적으로는 고밀도 접속을 가지는 노드 그룹과 그룹간의 접속이 희박한 노드 그룹으로 자연스럽게 분할되는 것을 의미합니다.그러나 중복되는 지역 사회도 허용된다.보다 일반적인 정의는 노드 쌍이 같은 커뮤니티의 멤버일 경우 연결 가능성이 높고 커뮤니티를 공유하지 않을 경우 연결 가능성이 낮다는 원칙에 기초하고 있습니다.관련성이 있지만 다른 문제는 커뮤니티 검색입니다.커뮤니티 검색에서는 특정 정점이 속한 커뮤니티를 찾는 것이 목표입니다.
특성.
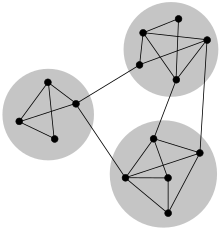
컴퓨터 및 정보 네트워크, 소셜 네트워크 및 생물 네트워크와 같은 네트워크 연구에서, 작은 세계의 재산, 두꺼운 꼬리 모양의 학위 분포, 그리고 군집화 등 많은 다른 특징들이 공통적으로 발생하는 것으로 밝혀졌다.또 다른 공통적인 특징은 공동체 [1][2][3][4][5]구조이다.네트워크의 맥락에서 커뮤니티 구조는 오른쪽의 그림 예시와 같이 네트워크의 다른 부분보다 내부적으로 더 촘촘하게 연결되어 있는 네트워크 내의 노드 그룹이 발생하는 것을 의미합니다.이러한 접속의 불균형은 네트워크 내에 특정 자연분할이 있음을 나타냅니다.
커뮤니티는 종종 정점 집합의 파티션으로 정의됩니다.즉, 그림에서와 같이 각 노드가 하나의 커뮤니티에 배치됩니다.이것은 유용한 단순화로 대부분의 커뮤니티 검출 방법에서는 이런 유형의 커뮤니티 구조를 찾을 수 있습니다.그러나 경우에 따라서는 정점이 둘 이상의 커뮤니티에 있는 것이 더 나은 표현이 될 수 있다.이것은 각 꼭지점이 한 사람을 나타내는 소셜 네트워크에서 일어날 수 있고, 그 커뮤니티는 다른 그룹의 친구들을 대표한다: 가족을 위한 커뮤니티, 동료들을 위한 커뮤니티, 같은 스포츠 클럽의 친구들을 위한 커뮤니티 등.다음에 설명하는 커뮤니티 검출에 CLI를 사용하는 것은 이러한 중복되는 커뮤니티 구조가 어떻게 발견될 수 있는지를 보여주는 하나의 예에 불과합니다.
네트워크에 따라서는 의미 있는 커뮤니티 구조가 없는 경우가 있습니다.랜덤 그래프나 BarabaaSi-Albert 모델 등 많은 기본 네트워크 모델에서는 커뮤니티 구조가 표시되지 않습니다.
중요성
커뮤니티 구조는 실제 네트워크에서는 매우 일반적입니다.소셜 네트워크는 공통 위치, 관심사, 직업 [5][6]등에 기반한 커뮤니티 그룹(사실상 이 용어의 기원)을 포함합니다.
네트워크 내에서 기반이 되는 커뮤니티 구조를 찾는 것은 여러 가지 이유로 중요합니다.커뮤니티는 개별 커뮤니티가 네트워크 내의 메타노드처럼 작용하기 때문에 대규모 네트워크 맵을 작성할 수 있게 되어 연구가 [7]쉬워집니다.
커뮤니티가 시스템의 기능 유닛에 대응하는 경우가 많기 때문에 개별 커뮤니티도 네트워크에 의해 대표되는 시스템의 기능을 조명합니다.대사 네트워크에서 그러한 기능군은 주기 또는 경로에 해당하는 반면, 단백질 상호작용 네트워크에서, 군집은 생물학적 세포 내에서 유사한 기능을 가진 단백질에 대응한다.마찬가지로 인용 네트워크는 연구 [1]주제별로 커뮤니티를 형성한다.네트워크내에서 이러한 서브 구조를 특정할 수 있으면, 네트워크의 기능과 토폴로지가 서로 어떻게 영향을 주는지를 파악할 수 있습니다.그러한 통찰력은 스펙트럼 클러스터링과 [8]같은 그래프의 일부 알고리즘을 개선하는 데 유용할 수 있다.
중요한 것은 커뮤니티가 네트워크의 평균 속성과는 매우 다른 속성을 갖는 경우가 많다는 것입니다.따라서 보통 평균 속성에만 집중하면 네트워크 내에서 중요하고 흥미로운 많은 기능이 누락됩니다.예를 들어, 특정 소셜 네트워크에서는 사교적인 그룹과 말수가 적은 그룹이 [7]동시에 존재할 수 있습니다.
커뮤니티의 존재는 일반적으로 네트워크에서 발생하는 루머 확산이나 전염병 확산과 같은 다양한 과정에도 영향을 미칩니다.따라서 이러한 프로세스를 올바르게 이해하기 위해서는 커뮤니티를 검출하고 다양한 환경에서 커뮤니티가 확산 프로세스에 어떻게 영향을 미치는지 연구하는 것이 중요하다.
마지막으로 커뮤니티 검출이 네트워크 과학에서 발견한 중요한 응용 프로그램은 누락된 링크의 예측과 네트워크 내의 잘못된 링크의 식별입니다.측정 프로세스 중에 여러 가지 이유로 일부 링크가 관찰되지 않을 수 있습니다.마찬가지로 일부 링크는 측정 오류로 인해 데이터에 잘못 입력될 수 있습니다.이러한 경우 모두 커뮤니티 검출 알고리즘에 의해 적절히 처리됩니다.이는 특정 노드 [9]쌍 간에 엣지의 존재 가능성을 할당할 수 있기 때문입니다.
커뮤니티를 찾기 위한 알고리즘
임의의 네트워크 내에서 커뮤니티를 찾는 것은 계산상 어려운 작업이 될 수 있습니다.네트워크내에 커뮤니티가 있는 경우는, 통상은 불명확하고, 커뮤니티의 규모나 밀도가 일정하지 않은 경우가 많습니다.그러나 이러한 어려움에도 불구하고, 지역사회를 찾는 몇 가지 방법이 개발되어 다양한 [4]수준의 성공을 거두고 있다.
최소 컷법
네트워크를 여러 부분으로 분할하는 가장 오래된 알고리즘 중 하나는 최소 컷 방식(및 비율 컷이나 정규화된 컷 등의 변형)입니다.이 방법에서는 예를 들어 프로세서노드 간의 통신을 최소화하기 위해 병렬 컴퓨팅 로드밸런싱에 사용됩니다.
최소 컷 방법에서는, 네트워크는, 그룹간의 엣지수를 최소한으로 억제하도록, 통상은 거의 같은 사이즈의 소정의 수의 부품으로 분할된다.이 메서드는 원래 의도된 어플리케이션의 대부분에서 잘 동작하지만 커뮤니티가 구조 내에 암묵적으로 존재하는지 여부에 관계없이 커뮤니티를 검출하고 고정된 [10]수만을 검출하기 때문에 일반적인 네트워크에서의 커뮤니티 구조를 검출하는 데는 적합하지 않습니다.
계층 클러스터링
네트워크에서 커뮤니티 구조를 찾는 또 다른 방법은 계층 클러스터링입니다.이 방법에서는 노드 쌍 간의 유사성 유형을 수량화하는 유사성 측정을 정의합니다.일반적으로 사용되는 측정치에는 코사인 유사도, Jaccard 지수 및 인접 매트릭스 행 간의 Hamming 거리가 포함됩니다.그런 다음 이 척도에 따라 유사한 노드를 커뮤니티로 그룹화합니다.그룹화를 수행하기 위한 몇 가지 공통 스킴이 있습니다.가장 간단한 것은 단일 링크 클러스터링입니다.이 두 그룹은 다른 그룹의 모든 노드 쌍이 주어진 임계값보다 낮은 유사성을 갖는 경우에만 별개의 커뮤니티로 간주되며, 모든 그룹 내의 모든 노드가 simi를 갖는 완전한 링크 클러스터링입니다.문턱값보다 크다.중요한 단계는 집적 클러스터링을 정지하기 위한 임계값을 결정하는 방법이며, 이는 최적에 가까운 커뮤니티 구조를 나타냅니다.공통 전략은 네트워크의 글로벌 속성을 감시하는1개 또는 여러 메트릭을 구축하는 것으로, 클러스터화의 특정 단계에서 피크 상태가 됩니다.이 방향의 흥미로운 접근법은 볼록 [11]합계를 통해 결합된 다양한 유사성 또는 차이성 측정의 사용이다.다량을 담긴 밀도에 갈 때 유사성 메트릭 가장자리 사이의(는 중복되는 지역 사회의 정의 허가)[12] 때는 similarit 확대 정의되어 있는 제안되고 있는 파티션을 밀도, 같은 성단 사이의 존경과 감시 모서리의 밀도의 또 다른 근사는 계산.y는 나는노드 간에 정의되어 길드와 같은 커뮤니티의 대체 정의를 검토할 수 있다(즉, 동일한 인접 라우터에 관해 유사한 수의 링크를 공유하는 노드 그룹, 그러나 반드시 [13]그 자체가 연결되어 있는 것은 아니다.이러한 방법은 다차원 네트워크를 고려하도록 확장할 수 있습니다.예를 들어 링크 [13]유형이 다른 노드를 가진 네트워크를 취급하는 경우 등입니다.
지르반-뉴먼 알고리즘
커뮤니티를 찾기 위해 일반적으로 사용되는 또 다른 알고리즘은 Girvan-Newman [1]알고리즘입니다.이 알고리즘은 커뮤니티 간에 존재하는 네트워크 내의 엣지를 식별하여 커뮤니티 자체만 남기고 삭제합니다.식별은 그래프 이론적 측정 간성 중심성을 사용하여 수행되며, 이는 에지가 다수의 노드 쌍 사이에 있는 경우 큰 각 에지에 숫자를 할당합니다.
Girvan-Newman 알고리즘은 합리적인 품질의 결과를 반환하며 많은 표준 소프트웨어 패키지로 구현되었기 때문에 인기가 있습니다.그러나 n개의 정점과 m개의 엣지로 이루어진 네트워크에서는 O(mn2)의 시간이 걸리기 때문에 수천 개 이상의 [14]노드를 가진 네트워크에서는 실행할 수 없습니다.
모듈화의 최대화
기존의 단점에도 불구하고 커뮤니티 검출에 가장 널리 사용되는 방법 중 하나는 모듈러형 [14]최대화입니다.모듈성은 커뮤니티에 대한 네트워크의 특정 분할의 품질을 측정하는 유익함수입니다.모듈러형 최대화 방식은 네트워크의 가능한 분할을 검색하여 특히 모듈러형이 높은1개 또는 여러 개를 검색함으로써 커뮤니티를 검출합니다.가능한 모든 구분에 대한 철저한 탐색은 보통 다루기 어렵기 때문에 실용적인 알고리즘은 속도와 [15][16]정확도 사이의 다른 균형을 제공하는 다양한 접근법과 함께 탐욕 알고리즘, 시뮬레이션 어닐링 또는 스펙트럼 최적화와 같은 대략적인 최적화 방법을 기반으로 한다.널리 사용되는 모듈러성 최대화 접근방식은 Louvain 메서드입니다.이 접근방식은 [17][18]현재 커뮤니티 상태에 대한 동요를 고려하여 글로벌모듈성을 더 이상 개선할 수 없을 때까지 로컬 커뮤니티를 반복적으로 최적화합니다.Extremal Ensemble Learning(EEL) 패러다임의 한 예인 RenEEL 방식을 활용하는 알고리즘은 현재 최고의 모듈식 최대화 [19]알고리즘이다.[20]
그 모듈화 최적화 종종, 네트워크(해상도 limit[21])의 크기에 따라 클러스터 일부 규모보다 작은 것을 감지할 수 있다. 반면에 모듈화 값의 풍경 파티션의 높은 modularit과 함께 커다란 축퇴에 의해 특징 지어진다 실패한다고 알려져 왔다. 모듈화 최적화의 유용성, 의심스럽다.y, c절대 최대값으로 지는 것은 서로 [22]매우 다를 수 있습니다.
통계적 추론
통계적 추론에 기초한 메서드는 커뮤니티 구조를 부호화하는 네트워크 데이터에 생성 모델을 적합시키려고 시도합니다.대안과 비교하여 이 접근방식의 전체적인 장점은 보다 원칙적인 성격과 통계적으로 중요한 문제를 본질적으로 다룰 수 있는 능력이다.문헌의 대부분의 방법은 혼합 멤버쉽,[24][25] 정도 [26]보정 및 계층 [27]구조를 포함한 변형뿐만 아니라 확률 블록[23] 모델에 기초한다.모델 선택은 최소 설명[28][29] 길이(또는 이에 상당하는 베이지안 모델[30] 선택) 및 우도비 [31]테스트와 같은 원칙적인 접근방식을 사용하여 수행될 수 있다.현재 믿음 전파와[32][33] 응집체 몬테 [34]카를로를 포함하여 확률적 블록 모델의 효율적인 추론을 수행하기 위해 많은 알고리즘이 존재한다.
객관적인 함수가 주어진 네트워크를 클러스터화하려는 접근법과는 달리, 이 클래스의 방법은 네트워크의 대규모 구조를 설명하는 역할을 할 뿐만 아니라 데이터를 일반화하고 네트워크에서 [35][36]누락되거나 스플리어스한 링크의 발생을 예측하는 데 사용할 수 있는 생성 모델을 기반으로 합니다.
Clique 기반 메서드
클리크는 각 노드가 클리크 내의 다른 모든 노드에 연결되어 있는 서브그래프입니다.노드를 이것보다 긴밀하게 접속할 수 없기 때문에 그래프 내의 CLI 검출과 이러한 중복에 대한 분석을 바탕으로 네트워크 내의 커뮤니티 검출에 많은 접근방식이 있는 것은 놀라운 일이 아닙니다.노드가 여러 clique의 멤버가 될 수 있기 때문에 이러한 방식에서는 노드가 여러 커뮤니티의 멤버가 될 수 있으므로 "오버랩 커뮤니티 구조"가 됩니다.
한 가지 방법은 "최대 집단"을 찾는 것입니다.즉, 다른 clique의 서브그래프가 아닌 clique를 찾는 것입니다.이를 찾기 위한 전형적인 알고리즘은 Bron-Kerbosch 알고리즘이다.이러한 오버랩은 커뮤니티를 정의하는 데 여러 가지 방법으로 사용할 수 있습니다.가장 간단한 방법은 최소 크기(노드 수)보다 큰 최대 CLI만 고려하는 것입니다.그런 다음 이들 집단을 결합하면 구성 요소(연결 해제된 부분)가 [37]커뮤니티를 정의하는 하위 그래프가 정의됩니다.이러한 접근방식은 UCInet과 같은 소셜 네트워크 분석 소프트웨어에서 구현되는 경우가 많습니다.
대체 접근법은 고정 kk의 클리어를 사용하는 것입니다.이러한 중복은 k k - 정규 하이퍼그래프의 유형 "Clique 그래프"[38]로 알려진 선 그래프의 일반화 구조(k k}인 경우)를 정의하는 데 사용할 수 있다.클리크 그래프에는 원래 그래프에서 클리크를 나타내는 정점이 있으며, 클리크 그래프의 엣지는 원래 그래프에서 클리크의 오버랩을 기록합니다.(각 노드를 커뮤니티에 할당하는) 이전 커뮤니티 검출 방식을 CLI 그래프에 적용하면 각 커뮤니티가 커뮤니티에 할당됩니다.이를 사용하여 CLI 내 노드의 커뮤니티 멤버십을 결정할 수 있습니다.노드도 여러 개의 CLI에 속할 수 있기 때문에 여러 커뮤니티의 멤버가 될 수 있습니다.예를 들어 clique percolation[39] 메서드는 커뮤니티를 k kcliques의 percolation 클러스터로 정의합니다.이를 위해 네트워크 내의 - cliks를 찾습니다k개 - 의 완전한 서브그래프입니다.그런 다음\- cliques가k - 1개 를 하는 경우 인접하도록 합니다. 즉, clique 그래프에서 가장자리를 정의하는 데 사용됩니다.커뮤니티는 k kclique의 결합으로 정의되며, 여기서 kclique 인접을 통해 k k\clique에서 kclique에 도달할 수 있습니다.커뮤니티는 clique 그래프에서 연결된 컴포넌트일 뿐입니다.노드가 동시에 여러 개의 다른 kclique 침투 클러스터에 속할 수 있으므로 커뮤니티가 서로 오버랩될 수 있습니다.
커뮤니티 알고리즘을 찾는 테스트 방법
어떤 것이 커뮤니티 구조를 더 잘 탐지할 수 있는지를 탐지하는 알고리즘의 평가는 여전히 미해결 문제이다.기존 구조의 네트워크 분석에 기초해야 합니다.전형적인 예로는 '4개의 그룹' 테스트를 들 수 있습니다.이 테스트에서는 네트워크가 4개의 동등한 크기의 그룹(보통 각각 32개의 노드)으로 분할되며 검출 알고리즘에 대해 다소 까다로운 구조를 작성하기 위해 그룹 내 및 그룹 간의 연결 확률이 달라집니다.이러한 벤치마크 그래프는 Condon과 Karp의 심어진 L-파티션[40] 모델 또는 커뮤니티 구조를 포함하는 랜덤 네트워크 모델의 일반적인 클래스인 "stochastic block model"의 특수한 경우이다.노드 정도와 커뮤니티 크기의 이종 분포를 포함하는 4개 그룹 벤치마크의 연장선인 LFR[41] 벤치마크와 같이 다양한 그룹 규모와 중요하지 않은 정도 분포를 허용하는 다른 보다 유연한 벤치마크가 제안되어 지역사회 감지 [43][44]방법에 대한 보다 엄격한 테스트가 되었다.
일반적으로 사용되는 컴퓨터 생성 벤치마크는 잘 정의된 커뮤니티의 네트워크로부터 시작됩니다.그 후, 이 구조는 링크의 재배선 또는 삭제에 의해 저하되어 알고리즘이 원래의 파티션을 검출하는 것이 점점 더 어려워집니다.최종적으로 네트워크는 본질적으로 랜덤인 지점에 도달합니다.이런 종류의 벤치마크는 "오픈"이라고 할 수 있습니다.이러한 벤치마크의 성과는 상호 정보 표준화 또는 정보의 변동과 같은 조치를 통해 평가된다.이들은 알고리즘에 의해 얻어진 솔루션을 원래의 커뮤니티 구조와 비교하여 두 파티션의 유사성을 평가합니다.
검출 가능성
최근 몇 년 동안, 커뮤니티 검출 문제에 단계 전이가 존재함을 보여주는 다양한 그룹에 의해 다소 놀라운 결과가 얻어졌으며, 커뮤니티 내부와 커뮤니티 간의 연결 밀도가 점점 더 같거나 (커뮤니티 구조가 변화함에 따라) 둘 다 작아지는 것을 보여준다.o가 약해지거나 네트워크가 너무 희박해집니다). 갑자기 커뮤니티가 검출되지 않게 됩니다.어떤 의미에서, 에지의 유무는 여전히 엔드 포인트의 커뮤니티 멤버쉽과 상관관계가 있기 때문에 커뮤니티 자체는 여전히 존재한다. 그러나 우연보다 노드에 더 나은 라벨을 붙이거나 심지어 Erdos-Renyi 모델과 같은 null 모델에 의해 생성된 그래프와 구별하는 것은 이론적으로 불가능하다.사회 구조 없이 말이죠.이러한 전환은 커뮤니티를 검출하는 데 사용되는 알고리즘의 유형과 무관하며, 이는 최적의 베이지안 추론(즉, 계산 [45][46][47]자원에 관계없이)에도 네트워크에서 커뮤니티를 검출하는 우리의 능력에 근본적인 한계가 있음을 의미한다.
의 , 의 동일한 크기의 그룹을 갖는 확률적 블록 모델을 고려하고, p {\와 }}}}을 각각 그룹 내부 및 그룹 간의 연결 확률로 . in> out { p _ { \ { } > p _ { \ { out} 、그룹 내 링크 밀도가 그룹 간 링크 밀도보다 높기 때문에 네트워크는 커뮤니티 구조를 가집니다.희박한 경우 p { 은 O로 스케일링되므로 평균도가 일정합니다.
- in /n {\{in}}= / {\}=
그 후 다음과 [46]같은 경우에는 커뮤니티를 검출할 수 없게 됩니다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c M. Girvan; M. E. J. Newman (2002). "Community structure in social and biological networks". Proc. Natl. Acad. Sci. USA. 99 (12): 7821–7826. arXiv:cond-mat/0112110. Bibcode:2002PNAS...99.7821G. doi:10.1073/pnas.122653799. PMC 122977. PMID 12060727.
- ^ S. Fortunato (2010). "Community detection in graphs". Phys. Rep. 486 (3–5): 75–174. arXiv:0906.0612. Bibcode:2010PhR...486...75F. doi:10.1016/j.physrep.2009.11.002. S2CID 10211629.
- ^ F. D. Malliaros; M. Vazirgiannis (2013). "Clustering and community detection in directed networks: A survey". Phys. Rep. 533 (4): 95–142. arXiv:1308.0971. Bibcode:2013PhR...533...95M. doi:10.1016/j.physrep.2013.08.002. S2CID 15006738.
- ^ a b M. A. Porter; J.-P. Onnela; P. J. Mucha (2009). "Communities in Networks" (PDF). Notices of the American Mathematical Society. 56: 1082–1097, 1164–1166.
- ^ a b Fani, Hossein; Bagheri, Ebrahim (2017). "Community detection in social networks". Encyclopedia with Semantic Computing and Robotic Intelligence. Vol. 1. pp. 1630001 [8]. doi:10.1142/S2425038416300019. S2CID 52471002.
- ^ Hamdaqa, Mohammad; Tahvildari, Ladan; LaChapelle, Neil; Campbell, Brian (2014). "Cultural Scene Detection Using Reverse Louvain Optimization". Science of Computer Programming. 95: 44–72. doi:10.1016/j.scico.2014.01.006.
- ^ a b M.E.J.Neman (2006). "Finding community structure in networks using the eigenvectors of matrices". Phys. Rev. E. 74 (3): 1–19. arXiv:physics/0605087. Bibcode:2006PhRvE..74c6104N. doi:10.1103/PhysRevE.74.036104. PMID 17025705. S2CID 138996.
- ^ Zare, Habil; P. Shooshtari; A. Gupta; R. Brinkman (2010). "Data reduction for spectral clustering to analyze high throughput flow cytometry data". BMC Bioinformatics. 11 (1): 403. doi:10.1186/1471-2105-11-403. PMC 2923634. PMID 20667133.
- ^ Aaron Clauset; Cristopher Moore; M.E.J. Newman (2008). "Hierarchical structure and the prediction of missing links in networks". Nature. 453 (7191): 98–101. arXiv:0811.0484. Bibcode:2008Natur.453...98C. doi:10.1038/nature06830. PMID 18451861. S2CID 278058.
- ^ M. E. J. Newman (2004). "Detecting community structure in networks". Eur. Phys. J. B. 38 (2): 321–330. Bibcode:2004EPJB...38..321N. doi:10.1140/epjb/e2004-00124-y. hdl:2027.42/43867. S2CID 15412738.
- ^ Alvarez, Alejandro J.; Sanz-Rodríguez, Carlos E.; Cabrera, Juan Luis (2015-12-13). "Weighting dissimilarities to detect communities in networks". Phil. Trans. R. Soc. A. 373 (2056): 20150108. Bibcode:2015RSPTA.37350108A. doi:10.1098/rsta.2015.0108. ISSN 1364-503X. PMID 26527808.
- ^ Ahn, Y.-Y.; Bagrow, J.P.; Lehmann, S. (2010). "Link communities reveal multi-scale complexity in networks". Nature. 466 (7307): 761–764. arXiv:0903.3178. Bibcode:2010Natur.466..761A. doi:10.1038/nature09182. PMID 20562860. S2CID 4404822.
- ^ a b Pascual-García, Alberto; Bell, Thomas (2020). "functionInk: An efficient method to detect functional groups in multidimensional networks reveals the hidden structure of ecological communities". Methods Ecol Evol. 11 (7): 804–817. doi:10.1111/2041-210X.13377. S2CID 214033410.
- ^ a b M. E. J. Newman (2004). "Fast algorithm for detecting community structure in networks". Phys. Rev. E. 69 (6): 066133. arXiv:cond-mat/0309508. Bibcode:2004PhRvE..69f6133N. doi:10.1103/PhysRevE.69.066133. PMID 15244693. S2CID 301750.
- ^ L. Danon; J. Duch; A. Díaz-Guilera; A. Arenas (2005). "Comparing community structure identification". J. Stat. Mech. 2005 (9): P09008. arXiv:cond-mat/0505245. Bibcode:2005JSMTE..09..008D. doi:10.1088/1742-5468/2005/09/P09008. S2CID 14798969.
- ^ R. Guimera; L. A. N. Amaral (2005). "Functional cartography of complex metabolic networks". Nature. 433 (7028): 895–900. arXiv:q-bio/0502035. Bibcode:2005Natur.433..895G. doi:10.1038/nature03288. PMC 2175124. PMID 15729348.
- ^ V.D. Blondel; J.-L. Guillaume; R. Lambiotte; E. Lefebvre (2008). "Fast unfolding of community hierarchies in large networks". J. Stat. Mech. 2008 (10): P10008. arXiv:0803.0476. Bibcode:2008JSMTE..10..008B. doi:10.1088/1742-5468/2008/10/P10008. S2CID 334423.
- ^ "Lightning-fast Community Detection in Social Media: A Scalable Implementation of the Louvain Algorithm". 2013. S2CID 16164925.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ J. Guo; P. Singh; K.E. Bassler (2019). "Reduced network extremal ensemble learning (RenEEL) scheme for community detection in complex networks". Scientific Reports. 9 (14234): 14234. arXiv:1909.10491. Bibcode:2019NatSR...914234G. doi:10.1038/s41598-019-50739-3. PMC 6775136. PMID 31578406.
- ^ "RenEEL-Modularity". GitHub. 31 October 2019.
- ^ S. Fortunato; M. Barthelemy (2007). "Resolution limit in community detection". Proceedings of the National Academy of Sciences of the United States of America. 104 (1): 36–41. arXiv:physics/0607100. Bibcode:2007PNAS..104...36F. doi:10.1073/pnas.0605965104. PMC 1765466. PMID 17190818.
- ^ B. H. Good; Y.-A. de Montjoye; A. Clauset (2010). "The performance of modularity maximization in practical contexts". Phys. Rev. E. 81 (4): 046106. arXiv:0910.0165. Bibcode:2010PhRvE..81d6106G. doi:10.1103/PhysRevE.81.046106. PMID 20481785. S2CID 16564204.
- ^ Holland, Paul W.; Kathryn Blackmond Laskey; Samuel Leinhardt (June 1983). "Stochastic blockmodels: First steps". Social Networks. 5 (2): 109–137. doi:10.1016/0378-8733(83)90021-7. ISSN 0378-8733.
- ^ Airoldi, Edoardo M.; David M. Blei; Stephen E. Fienberg; Eric P. Xing (June 2008). "Mixed Membership Stochastic Blockmodels". J. Mach. Learn. Res. 9: 1981–2014. ISSN 1532-4435. PMC 3119541. PMID 21701698. Retrieved 2013-10-09.
- ^ Ball, Brian; Brian Karrer; M. E. J. Newman (2011). "Efficient and principled method for detecting communities in networks". Physical Review E. 84 (3): 036103. arXiv:1104.3590. Bibcode:2011PhRvE..84c6103B. doi:10.1103/PhysRevE.84.036103. PMID 22060452. S2CID 14204351.
- ^ Karrer, Brian; M. E. J. Newman (2011-01-21). "Stochastic blockmodels and community structure in networks". Physical Review E. 83 (1): 016107. arXiv:1008.3926. Bibcode:2011PhRvE..83a6107K. doi:10.1103/PhysRevE.83.016107. PMID 21405744. S2CID 9068097.
- ^ Peixoto, Tiago P. (2014-03-24). "Hierarchical Block Structures and High-Resolution Model Selection in Large Networks". Physical Review X. 4 (1): 011047. arXiv:1310.4377. Bibcode:2014PhRvX...4a1047P. doi:10.1103/PhysRevX.4.011047. S2CID 5841379.
- ^ Martin Rosvall; Carl T. Bergstrom (2007). "An information-theoretic framework for resolving community structure in complex networks". Proceedings of the National Academy of Sciences of the United States of America. 104 (18): 7327–7331. arXiv:physics/0612035. Bibcode:2007PNAS..104.7327R. doi:10.1073/pnas.0611034104. PMC 1855072. PMID 17452639.
- ^ P. Peixoto, T. (2013). "Parsimonious Module Inference in Large Networks". Phys. Rev. Lett. 110 (14): 148701. arXiv:1212.4794. Bibcode:2013PhRvL.110n8701P. doi:10.1103/PhysRevLett.110.148701. PMID 25167049. S2CID 2668815.
- ^ P. Peixoto, T. (2019). "Bayesian stochastic blockmodeling". Advances in Network Clustering and Blockmodeling. pp. 289–332. arXiv:1705.10225. doi:10.1002/9781119483298.ch11. ISBN 9781119224709. S2CID 62900189.
- ^ Yan, Xiaoran; Jacob E. Jensen; Florent Krzakala; Cristopher Moore; Cosma Rohilla Shalizi; Lenka Zdeborová; Pan Zhang; Yaojia Zhu (2012-07-17). "Model Selection for Degree-corrected Block Models". Journal of Statistical Mechanics: Theory and Experiment. 2014 (5): P05007. arXiv:1207.3994. Bibcode:2014JSMTE..05..007Y. doi:10.1088/1742-5468/2014/05/P05007. PMC 4498413. PMID 26167197.
- ^ Gopalan, Prem K.; David M. Blei (2013-09-03). "Efficient discovery of overlapping communities in massive networks". Proceedings of the National Academy of Sciences. 110 (36): 14534–14539. Bibcode:2013PNAS..11014534G. doi:10.1073/pnas.1221839110. ISSN 0027-8424. PMC 3767539. PMID 23950224.
- ^ Decelle, Aurelien; Florent Krzakala; Cristopher Moore; Lenka Zdeborová (2011-12-12). "Asymptotic analysis of the stochastic block model for modular networks and its algorithmic applications". Physical Review E. 84 (6): 066106. arXiv:1109.3041. Bibcode:2011PhRvE..84f6106D. doi:10.1103/PhysRevE.84.066106. PMID 22304154. S2CID 15788070.
- ^ Peixoto, Tiago P. (2014-01-13). "Efficient Monte Carlo and greedy heuristic for the inference of stochastic block models". Physical Review E. 89 (1): 012804. arXiv:1310.4378. Bibcode:2014PhRvE..89a2804P. doi:10.1103/PhysRevE.89.012804. PMID 24580278. S2CID 2674083.
- ^ Guimerà, Roger; Marta Sales-Pardo (2009-12-29). "Missing and spurious interactions and the reconstruction of complex networks". Proceedings of the National Academy of Sciences. 106 (52): 22073–22078. arXiv:1004.4791. Bibcode:2009PNAS..10622073G. doi:10.1073/pnas.0908366106. PMC 2799723. PMID 20018705.
- ^ Clauset, Aaron; Cristopher Moore; M. E. J. Newman (2008-05-01). "Hierarchical structure and the prediction of missing links in networks". Nature. 453 (7191): 98–101. arXiv:0811.0484. Bibcode:2008Natur.453...98C. doi:10.1038/nature06830. ISSN 0028-0836. PMID 18451861. S2CID 278058.
- ^ M.G. Everett; S.P. Borgatti (1998). "Analyzing Clique Overlap Connections". Connections. 21: 49.
- ^ T.S. Evans (2010). "Clique Graphs and Overlapping Communities". J. Stat. Mech. 2010 (12): P12037. arXiv:1009.0638. Bibcode:2010JSMTE..12..037E. doi:10.1088/1742-5468/2010/12/P12037. S2CID 2783670.
- ^ G. Palla; I. Derényi; I. Farkas; T. Vicsek (2005). "Uncovering the overlapping community structure of complex networks in nature and society". Nature. 435 (7043): 814–818. arXiv:physics/0506133. Bibcode:2005Natur.435..814P. doi:10.1038/nature03607. PMID 15944704. S2CID 3250746.
- ^ Condon, A.; Karp, R. M. (2001). "Algorithms for graph partitioning on the planted partition model". Random Struct. Algor. 18 (2): 116–140. CiteSeerX 10.1.1.22.4340. doi:10.1002/1098-2418(200103)18:2<116::AID-RSA1001>3.0.CO;2-2.
- ^ A. Lancichinetti; S. Fortunato; F. Radicchi (2008). "Benchmark graphs for testing community detection algorithms". Phys. Rev. E. 78 (4): 046110. arXiv:0805.4770. Bibcode:2008PhRvE..78d6110L. doi:10.1103/PhysRevE.78.046110. PMID 18999496. S2CID 18481617.
- ^ a b Fathi, Reza (April 2019). "Efficient Distributed Community Detection in the Stochastic Block Model". arXiv:1904.07494 [cs.DC].
- ^ M. Q. Pasta; F. Zaidi (2017). "Leveraging Evolution Dynamics to Generate Benchmark Complex Networks with Community Structures". arXiv:1606.01169 [cs.SI].
- ^ Pasta, M. Q.; Zaidi, F. (2017). "Topology of Complex Networks and Performance Limitations of Community Detection Algorithms". IEEE Access. 5: 10901–10914. doi:10.1109/ACCESS.2017.2714018.
- ^ Reichardt, J.; Leone, M. (2008). "(Un)detectable Cluster Structure in Sparse Networks". Phys. Rev. Lett. 101 (78701): 1–4. arXiv:0711.1452. Bibcode:2008PhRvL.101g8701R. doi:10.1103/PhysRevLett.101.078701. PMID 18764586. S2CID 41197281.
- ^ a b Decelle, A.; Krzakala, F.; Moore, C.; Zdeborová, L. (2011). "Inference and Phase Transitions in the Detection of Modules in Sparse Networks". Phys. Rev. Lett. 107 (65701): 1–5. arXiv:1102.1182. Bibcode:2011PhRvL.107f5701D. doi:10.1103/PhysRevLett.107.065701. PMID 21902340. S2CID 18399723.
- ^ Nadakuditi, R.R; Newman, M.E.J. (2012). "Graph Spectra and the Detectability of Community Structure in Networks". Phys. Rev. Lett. 108 (188701): 1–5. arXiv:1205.1813. Bibcode:2012PhRvL.108r8701N. doi:10.1103/PhysRevLett.108.188701. PMID 22681123. S2CID 11820036.





 동일한 크기의 그룹을
동일한 크기의 그룹을 


 스케일링되므로 평균도가 일정합니다.
스케일링되므로 평균도가 일정합니다.



