점 수용 돌연변이
Point accepted mutationPAM이라고도 알려진 점 수용 돌연변이는 단백질의 일차 구조에서 단일 아미노산을 다른 단일 아미노산으로 대체하는 것으로, 자연 선택 과정에 의해 수용된다.이 정의는 유기체의 DNA에 있는 모든 점 돌연변이를 포함하지는 않는다.특히 무성 돌연변이는 점으로 인정된 돌연변이가 아니며, 치명적이거나 다른 방법으로 자연선택에 의해 거부된 돌연변이도 아니다.
PAM 행렬은 각 열과 행이 20개의 표준 아미노산 중 하나를 나타내는 행렬이다.생물정보학에서 PAM 행렬은 단백질에 대한 시퀀스 정렬을 점수화하기 위한 대체 행렬로 사용되기도 한다.PAM 매트릭스의 각 항목은 이 두 아미노산이 우연한 관계로 정렬되는 것이 아니라 특정 진화 간격 동안 하나 이상의 점 수용 돌연변이를 통해 해당 열의 아미노산으로 대체될 가능성을 나타낸다.다른 PAM 매트릭스는 단백질 순서의 진화에서 다른 시간의 길이에 대응한다.
생물학적 배경
살아있는 유기체의 모든 복제 세포의 유전적 지시사항은 DNA 안에 포함되어 있다.[1]세포의 수명 동안, 이 정보는 세포 메커니즘에 의해 기록되고 복제되어 단백질을 생산하거나 세포 분열 동안 딸 세포에 대한 지침을 제공하며, 이러한 과정 동안 DNA가 변형될 가능성이 존재한다.[1][2]이것은 돌연변이라고 알려져 있다.분자 수준에서, DNA가 복제되기 전에 이러한 변화들 중 대부분(전부는 아니지만)을 수정하는 규제 시스템이 있다.[2][3]
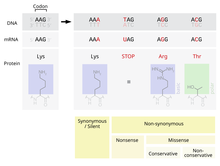
발생할 수 있는 돌연변이 중 하나는 점 돌연변이라고 알려진 단일 뉴클레오티드 대체다.만약 점 돌연변이가 유전자의 표현된 영역인 엑손 안에서 일어난다면, 이것은 그 유전자에 의해 생성된 단백질의 특정한 아미노산을 지정하는 코돈을 변화시킬 것이다.[2]유전자 코드의 중복성에도 불구하고, 이 돌연변이가 변환 중에 생성되는 아미노산을 변화시킬 가능성이 있으며, 그 결과 단백질의 구조가 바뀔 것이다.
단백질의 기능성은 그 구조에 따라 크게 좌우된다.[4]단백질에서 단일 아미노산을 바꾸는 것은 이 기능을 수행하는 능력을 감소시키거나, 돌연변이가 단백질이 수행하는 기능을 변화시킬 수도 있다.[2]이와 같은 변화는 세포의 중요한 기능에 심각한 영향을 미쳐 잠재적으로 세포와 극단적인 경우 유기체가 죽게 할 수 있다.[5]반대로, 그 변화는 세포가 비록 다르지만 계속 기능하도록 할 수 있고, 돌연변이는 유기체의 자손에게 전달될 수 있다.이러한 변화가 자손에게 유의미한 신체적 불이익을 주지 않는다면, 이 돌연변이가 모집단 내에서 지속될 가능성이 존재한다.기능의 변화가 유리해질 가능성도 존재한다.어느 경우든 자연선택 과정을 거치는 동안 포인트 돌연변이가 유전자 풀에 받아들여졌다.
유전자 코드에 의해 번역된 20개의 아미노산은 그들의 사이드 체인의 물리적, 화학적 특성에 의해 크게 달라진다.[4]그러나 이러한 아미노산은 유사한 물리화학 특성을 가진 그룹으로 분류할 수 있다.[4]아미노산을 같은 범주의 다른 범주로 대체하는 것은 다른 범주의 아미노산으로 대체하는 것보다 단백질의 구조와 기능에 더 작은 영향을 미칠 가능성이 있다.결과적으로 점 돌연변이의 수용은 돌연변이에 대체되는 아미노산과 대체 아미노산에 크게 좌우된다.PAM 행렬은 정렬 중 단백질의 유사성을 평가할 때 이러한 다양한 합격률을 설명하는 수학적 도구다.
용어.
수용된 지점 돌연변이라는 용어는 처음에 돌연변이 현상을 설명하기 위해 사용되었다.그러나 가독성 때문에 APM보다 PAM이라는 약어가 선호되었기 때문에 점 수용 돌연변이라는 용어가 더 규칙적으로 사용된다.[6]PAMn 매트릭스의 n 값은 100개의 아미노산당 돌연변이의 수를 나타내기 때문에 돌연변이의 백분율로 비유할 수 있기 때문에 허용 돌연변이 비율이라는 용어가 사용되기도 한다.
포인트 허용 돌연변이(PAM), 포인트 허용 돌연변이 행렬(PAM 행렬) 및 PAMn 매트릭스를 구분하는 것이 중요하다.'점 수용 돌연변이'라는 용어는 돌연변이 사건 자체를 가리킨다.그러나 'PAM 매트릭스'는 무작위적 우연이 아닌 일련의 돌연변이 사건으로 인해 두 아미노산이 정렬될 가능성을 나타내는 점수를 포함하는 행렬의 한 계열을 가리킨다.'PAM 매트릭스'는 100개의 아미노산당 이벤트가 발생할 수 있을 만큼 긴 시간 프레임에 해당하는 PAM 매트릭스다.
PAM 매트릭스 구축
PAM 매트릭스는 1978년 마가렛 데이호프에 의해 소개되었다.[7]이러한 행렬의 계산은 밀접하게 연관된 71개 단백질 계열의 계통생성 나무에서 관찰된 1572개의 돌연변이에 기초하였다.연구 대상 단백질은 전임자와 유사성이 높다는 점을 근거로 선정했다.포함된 단백질 정렬은 최소 85%의 정체성을 표시하도록 요구되었다.[6][8]결과적으로, 정렬된 불일치는 동일한 위치에 여러 개 있는 것이 아니라 단일 돌연변이가 발생한 결과라고 가정하는 것이 타당하다.
각 PAM 매트릭스에는 20개의 행과 20개의 열이 있다. 하나는 유전 코드에 의해 번역된 20개의 아미노산을 각각 나타낸다.PAM 매트릭스의 각 셀의 값은 돌연변이가 이후 칼럼 아미노산과 정렬되기 전의 행 아미노산의 확률과 관련이 있다.[6][7][8]이 정의에서 PAM 행렬은 대체 행렬의 예다.
계통생성 나무의 데이터 수집
단백질 계열의 계통생성 나무의 각 가지에 대해 관찰된 불일치 횟수를 기록하고 관련 아미노산 2개를 기록하였다.[7]이러한 계수들은 행렬 의 주요 아래 항목으로 사용되었다 단백질 샘플의 대부분은 현재 살아 있는 유기체로부터 나오기 때문에(확장종) 돌연변이의 '방향'을 결정할 수 없다.즉, 돌연변이 이전에 존재하는 아미노산은 돌연변이 후 이를 대체한 아미노산과 구별할 수 없다.이 때문에 매트릭스 은(는) 대칭으로 가정하고, 주 대각선 위 의 항목은 이 기준으로 계산된다. 의 대각선을 따르는 항목은 돌연변이와 일치하지 않으므로 채워지지 않은 상태로 둘 수 있다.
이러한 계수 외에도, 아미노산의 돌연변이와 빈도에 대한 데이터를 얻었다.[6][7]아미노산의 돌연변이는 관여하는 돌연변이의 수와 정렬에서 발생하는 횟수의 비율이다.[7]돌연변이는 아미노산이 얼마나 쉽게 돌연변이를 일으킬 수 있는지를 측정한다.아미노산 중 가장 변이성이 높은 것은 작은 극성측 체인으로 아미노산인 아스파라긴(Asparagine은 아미노산 중에서 가장 변이성이 높은 것으로 밝혀졌다.[7]시스틴과 트립토판은 가장 변이성이 적은 아미노산으로 밝혀졌다.[7]시스틴과 트립토판을 위한 사이드 체인은 덜 흔한 구조를 가지고 있다: 시스틴의 사이드 체인은 다른 시스틴 분자와 이황화 결합에 참여하는 유황을 포함하고 있으며 트립토판의 사이드 체인은 크고 향기롭다.[4]여러 개의 작은 극지방 아미노산이 있기 때문에, 이러한 양극단은 대체 아미노산들 사이에서 그들의 물리적, 화학적 성질이 더 흔하다면, 아미노산이 받아들일 수 있게 변이될 가능성이 더 높다는 것을 암시한다.[6][8]
돌연변이 행렬의 구성
th 아미노산의 경우 ) 및 값은 그 변이성과 주파수다.아미노산의 주파수는 1로 합치도록 정규화된다. th 아미노산의 총 발생 횟수가 (j ) 이고 이(가) 모든 아미노산의 총 발생 횟수인 경우
돌연변이를 아미노산 발생에 대한 돌연변이의 비율으로서의 돌연변이의 정의에 기초한다.
또는
돌연변이 행렬 은(는) 항목 , j) 이() i th 아미노산으로 될 확률을 나타내도록 구성된다.비대각 입력은 방정식으로[7] 계산된다.
여기서 은(는) 비례의 상수다.그러나 이 방정식은 대각선 항목을 계산하지 않는다.매트릭스 의 각 열에는 아미노산에 대해 가능한 20가지 결과가 각각 나열되어 있는데, 아미노산은 다른 19개 아미노산 중 하나로 변이되거나 변하지 않을 수 있다.19개 돌연변이의 각 확률을 나열하는 비대각선 입력값을 알고 있으며, 이 20개 결과의 확률의 합은 1이어야 하므로, 이 마지막 확률은 다음과 같이 계산할 수 있다.
로[7] 단순화하는.
대각선 입력의 계산 비대각 항목 돌연변이 행렬에 대한 식 대체:
및 의 값은 i 의 값에 따라 변경되지 않는 상수이기 때문에
따라서 취소를 통해
특히 유의한 결과는 비대각 입구의 경우
즉, 돌연변이 행렬의 모든 항목에 대해
비례 상수 선택
에 포함된 확률은 단백질 시퀀스가 변이될 수 있는 시간의 일부 알 수 없는 함수에 따라 달라진다.이 관계를 결정하려고 시도하는 대신 기간 동안 M 값을 계산하고, 더 긴 시간 동안의 행렬은 돌연변이가 마르코프 체인 모델을 따른다고 가정하여 계산한다.[9][10]PAM 행렬의 기본 시간 단위는 100개의 아미노산당 1개의 돌연변이가 발생하는 데 필요한 시간이며, 때로는 시간의 'PAM 단위' 또는 'PAM 단위'라고도 한다.[6]이것은 정확히 PAM1 매트릭스에 의해 가정된 돌연변이의 지속시간이다.
상수 은(는) 변하지 않는 아미노산의 비율을 조절하는 데 사용된다.최소한 85%의 유사성을 가진 단백질의 정렬만 사용함으로써 관찰된 돌연변이가 중간 상태 없이 직접적이었다고 합리적으로 가정할 수 있었다.이는 유사도가 100%에 가까웠다면 공통 인자에 의해 이러한 계수를 축소하면 돌연변이 계수의 정확한 추정치를 제공할 수 있다는 것을 의미한다.또한 PAM의n 은(는) 100개의 아미노산당 돌연변이 아미노산 수와 같다는 뜻이기도 하다.
PAM1 매트릭스에 대한 돌연변이 매트릭스를 찾기 위해, 일련의 아미노산의 99%를 보존해야 한다는 요건이 부과된다. () ( j, ) n은 보존 j{\ j 수와 같으므로 보존된 아미노산의 총 수는
후 99%의 ID를 생성하기 위해 to 을(를) 선택해야 하는 값은 그 다음 방정식에 의해 주어진다.
이 값은 PAM1 매트릭스의 돌연변이 매트릭스에서 사용할 수 있다.
PAMn 매트릭스 구축
단백질 돌연변이의 마르코프 체인 모델은 PAMn 의 돌연변이 행렬을 단순한 관계에 의한 PAM1 매트릭스 1}에 관련시킨다.
PAMn 매트릭스는 th 아미노산을 th 아미노산으로 대체하는 점 수용 돌연변이의 확률과 이러한 아미노산이 우연히 정렬될 확률의 비율로 구성된다.PAMn 매트릭스의 항목은 다음[11][12] 방정식으로 제공된다.
구스필드 책에서 M(, ) 및 , ) 은(는) th 아미노산이 th 아미노산으로 변질될 확률과 관련이 있다.[11]이것은 PAM 행렬의 입력에 대한 다른 방정식의 기원이다.
PAMn 매트릭스를 사용하여 두 단백질의 정렬 점수를 매길 때 다음과 같은 가정을 한다.
- 이 두 단백질이 연관되어 있다면, 이들을 분리하는 진화적 간격은 100개의 아미노산당 포인트 허용 돌연변이가 발생하는 데 걸리는 시간이다.
와 j{\ th 아미노산의 정렬을 고려할 때, 점수는 관련 단백질 또는 무작위 확률로 인한 정렬의 상대적 가능성을 나타낸다.
- 단백질과 관련이 있다면, 일련의 점 수용된 돌연변이가 원래의 아미노산을 대체하기 위해 돌연변이가 일어났을 것이다. th 아미노산이 원본이라고 가정하자.단백질에 아미노산이 풍부하다는 점에 기초하여 th 아미노산이 원산이 될 은 () f이다. 이 아미노산의 어떤 특정한 단위를 감안할 때 된 시간 간격에서 i{\i} th 아미노산으로 대체될 은 M n이다, ) 따라서 정렬의 확률은 logarithm 내의 분자인f() (, 이다.
- 단백질이 관련이 없는 경우, 두 개의 정렬된 아미노산이 th 및 th 아미노산인 이벤트는 독립적이어야 한다.이러한 사건의 확률은 ( ) 과 f이며 이는 선형 확률은 로그의 분모인( i) ( f임을 의미한다.
- 따라서 이 방정식의 로그는 점 수용 돌연변이로 인해 정렬 가능성이 더 높으면 양의 입력으로, 우연으로 인해 정렬 가능성이 더 높으면 음의 입력으로 이어진다.
PAM 매트릭스의 속성
PAM 행렬의 대칭성
돌연변이 확률 매트릭스 이(가) 대칭이 아닌 반면, 각 PAM 매트릭스는 다음과 같다.[6][7]이 다소 놀라운 속성은 돌연변이 확률 매트릭스에 대해 언급된 관계의 결과물이다.
실제로 이 관계는 M 의 모든 양의 정수 검정력에 대해 유지된다
양의 정수 매트릭스 힘에 대한 속성 일반화 이러한 일반화는 수학적 유도를 통해 증명될 수 있다. M 의 경우
이 값은 양의 k {\ k에 대해
매트릭스 제품 + 1= 의 확장
매트릭스 에 대해 가정된 속성 사용
그리고 M M 행렬의 속성 사용
이 경우 결과가 = 을(를) 유지한다는 것만 처음에 알려져 있다 그러나 위의 주장은 속성이 k= }을를) 유지한다는 것을 보여준다 이 새로운 지식은 속성이 을(를) 유지한다는 것을 나타내며, 이 반복된다.의 정수 k k
결과적으로 PAMn 매트릭스의 항목은 다음과 같이 대칭적이다.
돌연변이 아미노산의 수와 돌연변이 수 관련
값은 100개의 아미노산당 발생하는 돌연변이의 수를 나타내지만, 이 값은 거의 접근할 수 없고 종종 추정되기도 한다.그러나 두 단백질을 비교할 때 대신 을(를) 계산하기 쉬운데, 이는 100개의 아미노산당 돌연변이 아미노산 수입니다.돌연변이의 무작위적 특성에도 불구하고 이러한 값은 다음과 같이[13] 근사적으로 연관될 수 있다.
과( n {\ 사이의 관계 파생 단백질 1차 구조의 돌연변이는 그 순서를 따라 어디에서나 발생할 수 있다.아미노산 위치 간 돌연변이의 분포가 균일하다고 가정할 경우 콤비네이터학에서 흔히 발생하는 문제인 "balls into bins"의 분포와 유사하다. 볼(즉, 돌연변이)이 빈(아미노산 위치)에 분포하는 경우, 적어도 하나의 볼을 포함하는 빈의 수, 은 다음과[14] 같은 평균을 가진 분포를 가진다.
100개의 아미노산당 돌연변이 이 n 개일 경우
그리고 100개의 아미노산당 개의 돌연변이 아미노산이 있다면 대략 다음과 같다.
이제 및 은(는)
일반적인 단백질에 대해 합리적으로 만들 수 있는 가정인 의 큰 값에 대해 이 표현은 대략 다음과 같다.
이러한 추정치의 유효성은 매트릭스 의 작용에 따라 변하지 않는 아미노산의 수를 세어 확인할 수 있다 PAMn 매트릭스의 시간 간격에 대한 변경되지 않은 아미노산의 총 수는 다음과 같다.
그래서 변하지 않은 아미노산의 비율은
예 - PAM250
PAM250은 시퀀스 비교를 위해 일반적으로 사용되는 점수 매트릭스다.구조상 PAM 매트릭스는 대칭이어야 하므로 매트릭스의 하위 절반만 계산하면 된다.20개의 아미노산은 각각 3개의 모호한 아미노산이 추가되어 매트릭스 상단과 측면으로 표시된다.아미노산은 알파벳 순으로 나열되거나 그룹으로 분류된다.이 그룹들은 아미노산들 사이에서 공유되는 특성들이다.[7]
생물정보학에서 사용
계통생성 나무의 산란 시간 결정
분자시계 가설은 특정 단백질에서 아미노산 치환율이 단백질 계열마다 다를 수 있지만 시간이 지남에 따라 거의 일정할 것이라고 예측한다.[13]이것은 단백질의 아미노산당 돌연변이의 수가 시간에 따라 대략적으로 선형적으로 증가함을 시사한다.
두 단백질이 갈라진 시간을 결정하는 것은 유전체학에서 중요한 일이다.화석 기록은 흔히 지구의 진화 역사의 연대표에 있는 사건들의 위치를 정립하기 위해 사용되지만, 이 원천의 적용은 제한적이다.그러나 단백질 계열의 분자시계가 진드기를 내는 속도, 즉 아미노산당 돌연변이의 수가 증가하는 속도가 알려져 있다면, 이 수의 돌연변이를 알면 분리의 날짜를 찾을 수 있을 것이다.
오늘날 살고 있는 유기체로부터 채취한 두 개의 관련 단백질에 대한 분리의 날짜가 모색된다고 가정해보자.두 단백질은 모두 분진일로부터 인정된 돌연변이를 축적해 왔으며, 따라서 이들을 분리하는 아미노산당 총 돌연변이의 수는 대략 두 배 정도로 공통의 조상으로부터 분리된다.관련된 것으로 알려진 두 단백질을 정렬하기 위해 다양한 PAM 매트릭스를 사용하는 경우, 최상의 점수를 얻는 PAMn 매트릭스에서 의 값은 두 단백질을 분리하는 아미노산당 돌연변이와 가장 일치할 가능성이 높다.이 값을 절반으로 줄이고 수용된 돌연변이가 단백질 계열에 축적되는 비율로 나누면 이 두 단백질이 공통의 조상으로부터 분리되는 시간을 추정할 수 있다.즉[13] myr에서 분기의 시간은
여기서 은(는) 아미노산당 돌연변이의 수이고, 은 아미노산 사이트당 돌연변이의 수용된 누적 비율이다.
블라스트에 사용
PAM 행렬은 DNA 시퀀스 또는 단백질 시퀀스를 비교하여 정렬의 품질을 판단할 때 점수 매트릭스로도 사용된다.이 형태의 채점 시스템은 블라스트를 포함한 광범위한 정렬 소프트웨어에 의해 활용된다.[15]
PAM과 BLOSUM 비교
비록 PAM 로그-odd 매트릭스가 블라스트와 함께 사용된 첫 번째 점수 매트릭스였지만, PAM 매트릭스는 주로 BLOSUM 매트릭스로 대체되었다.비록 두 행렬이 유사한 채점 결과를 내지만, 그들은 다른 방법론을 사용하여 생성되었다.BLOSUM 행렬은 서로 다른 정도로 분산된 정렬된 블록의 아미노산 차이에서 직접 생성되었다. PAM 행렬은 긴 시간 수정에 밀접하게 관련된 시퀀스에 기초한 진화 정보의 외삽화를 반영한다.[16]PAM과 BLOSUM 매트릭스에 대한 정보 채점이 매우 다른 방식으로 생성되었기 때문에 매트릭스와 관련된 숫자는 근본적으로 다른 의미를 갖는다; PAM 매트릭스의 숫자는 더 많은 다른 단백질들 간의 비교를 위해 증가하지만 BLOSUM 매트릭스의 수는 감소한다.[17]그러나 모든 아미노산 대체 매트릭스는 상대 엔트로피를 사용하여 정보 이론적[18] 프레임워크에서 비교할 수 있다.
| PAM 매트릭스 | 등가 BLOSUM 행렬 | 상대 엔트로피(비트) |
|---|---|---|
| PAM100 | 블로섬90 | 1.18 |
| PAM120 | 블로섬89 | 0.98 |
| PAM160 | 블로섬60 | 0.70 |
| PAM200 | 블로섬52 | 0.51 |
| PAM250 | 블로섬45 | 0.36 |
참고 항목
참조
- ^ a b Campbell NA, Reece JB, Meyers N, Urry LA, Cain ML, Wasserman SA, Minorsky PV, Jackson RB (2009). "The Molecular Basis of Inheritance". Biology (8th ed.). Pearson Education Australia. pp. 307–325. ISBN 9781442502215.
- ^ a b c d Campbell NA, Reece JB, Meyers N, Urry LA, Cain ML, Wasserman SA, Minorsky PV, Jackson RB (2009). "From Gene to Protein". Biology: Australian Version (8th ed.). Pearson Education Australia. pp. 327–350. ISBN 9781442502215.
- ^ Pal JK, Ghaskadbi SS (2009). "DNA Damage, Repair and Recombination". Fundamentals of Molecular Biology (1st ed.). Oxford University Press. pp. 187–203. ISBN 9780195697810.
- ^ a b c d Campbell NA, Reece JB, Meyers N, Urry LA, Cain ML, Wasserman SA, Minorsky PV, Jackson RB (2009). "The Structure and Function of Large Biological Molecules". Biology: Australian Version (8th ed.). Pearson Education Australia. pp. 68–89. ISBN 9781442502215.
- ^ Lobo I (January 2008). "Mendelian Ratios and Lethal Genes". Nature Education. 1 (1): 138.
- ^ a b c d e f g Pevsner J (2009). "Pairwise Sequence Alignment". Bioinformatics and Functional Genomics (2nd ed.). Wiley-Blackwell. pp. 58–68. ISBN 978-0-470-08585-1.
- ^ a b c d e f g h i j k Dayhoff MO, Schwartz RM, Orcutt BC (1978). "A model of Evolutionary Change in Proteins". Atlas of protein sequence and structure (volume 5, supplement 3 ed.). Washington, DC.: National Biomedical Research Foundation. pp. 345–358. ISBN 978-0-912466-07-1.
{{cite book}}: CS1 maint: 날짜 및 연도(링크) - ^ a b c Wing-Kin S (2010). Algorithms in Bioinformatics: A Practical Introduction. CRC Press. pp. 51–52. ISBN 978-1-4200-7033-0.
- ^ Kosiol C, Goldman N (2005). "Different versions of the Dayhoff rate matrix". Molecular Biology and Evolution. 22 (2): 193–9. doi:10.1093/molbev/msi005. PMID 15483331.
- ^ Liò P, Goldman N (1998). "Models of molecular evolution and phylogeny". Genome Research. 8 (12): 1233–44. doi:10.1101/gr.8.12.1233. PMID 9872979.
- ^ a b Gusfield D (1997). Algorithms on String, Trees, and Sequences -Computer Science and Computational Biology. Cambridge University Press. pp. 383–384. ISBN 978-0521585194.
- ^ Boeckenhauer H, Bongartz D (2010). Algorithmic Aspects of Bioinformatics. Springer. pp. 94–96. ISBN 978-3642091001.
- ^ a b c Pevsner J (2009). "Molecular Phylogeny and Evolution". Bioinformatics and Functional Genomics (2nd ed.). Wiley-Blackwell. pp. 221–227. ISBN 978-0-470-08585-1.
- ^ Motwani R, Raghavan P (1995). Randomized Algorithms. Cambridge University Press. p. 94. ISBN 978-0521474658.
- ^ "The Statistics of Sequence Similarity Scores". National Centre for Biotechnology Information. Retrieved 20 October 2013.
- ^ Henikoff S, Henikoff JG (1992). "Amino acid substitution matrices from protein blocks". Proceedings of the National Academy of Sciences of the United States of America. 89 (22): 10915–10919. Bibcode:1992PNAS...8910915H. doi:10.1073/pnas.89.22.10915. PMC 50453. PMID 1438297.
- ^ Saud O (2009). "PAM and BLOSUM Substitution Matrices". Birec. Archived from the original on 9 March 2013. Retrieved 20 October 2013.
- ^ a b Altschul SF (June 1991). "Amino acid substitution matrices from an information theoretic perspective". Journal of Molecular Biology. 219 (3): 555–65. doi:10.1016/0022-2836(91)90193-A. PMC 7130686. PMID 2051488.
외부 링크
- http://www.inf.ethz.ch/personal/gonnet/DarwinManual/node148.html
- http://www.bioinformatics.nl/tools/pam.html PAM 매트릭스를 신속하게 계산하는 경우.
- http://web.expasy.org/docs/relnotes/relstat.html 스위스 프로트 단백질 기술 자료의 가장 최근의 통계.6.1절은 가장 최신의 아미노산 주파수를 포함한다.






 (가) 모든 아미노산의 총 발생 횟수인 경우
(가) 모든 아미노산의 총 발생 횟수인 경우






 (는) 비례의 상수다.그러나 이 방정식은 대각선 항목을 계산하지 않는다.매트릭스
(는) 비례의 상수다.그러나 이 방정식은 대각선 항목을 계산하지 않는다.매트릭스 












































 아미노산 사이트당 돌연변이의 수용된 누적 비율이다.
아미노산 사이트당 돌연변이의 수용된 누적 비율이다. 
