단백질분해효소억제제(생물학)
Protease inhibitor (biology)생물학 및 생화학에서, 단백질 분해효소 억제제 또는 역단백질 [1]분해효소는 단백질 분해효소(단백질 분해에 도움을 주는 효소)의 기능을 억제하는 분자입니다.자연적으로 발생하는 많은 단백질 분해효소 억제제는 [2]단백질이다.
의학에서 단백질분해효소 억제제는 종종 알파1-안티트립신(A1AT, 이러한 [3]이유로 약칭 PI)과 교환 가능하게 사용된다.A1AT는 실제로 질병, 즉 알파-1 항트립신 결핍에 가장 자주 관여하는 단백질 분해효소 억제제이다.
분류
단백질분해효소 억제제는 억제하는 단백질 분해효소의 유형 또는 작용 메커니즘에 의해 분류될 수 있다.2004년 롤링스와 동료들은 아미노산 [4]배열 수준에서 검출 가능한 유사성에 기초한 단백질 분해효소 억제제 분류를 도입했다.이 분류는 처음에 구조에 따라 26개의 관련 슈퍼 패밀리(또는 클랜)로 분류할 수 있는 48개의 억제제 패밀리를 식별했다.MEROPS 데이터베이스에 따르면 현재 81개의 억제제 패밀리가 있다.이 패밀리는 I 뒤에 숫자가 붙는 이름으로 명명됩니다. 예를 들어 I14에는 히루딘 유사 억제제가 포함되어 있습니다.
단백질분해효소별
단백질 분해 효소의 종류는 다음과 같습니다.
메커니즘별
억제제 메커니즘의 작용 등급은 다음과 같다.
가족들
억제제 I4
이것은 세르핀이라고 불리는 단백질 분해효소 자살 억제제입니다.그것은 여러 시스테인 및 세린 단백질 분해효소 계열의 억제제를 포함한다.그들의 작용 메커니즘은 목표물의 촉매 삼합체를 비활성화하는 큰 구조 변화를 겪는 것에 의존한다.
억제제 I9
| 펩티드가수분해효소억제I9 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
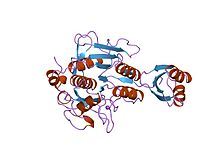 서브틸리신 bpn' 산문(77잔기)과 돌연변이 서브틸리신 bpn'(266잔기).결정 ph 4.6. 결정화 온도 20 c 회절 온도-160 c | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I9 | ||||||||
| 팜 | PF05922 | ||||||||
| 인터프로 | IPR010259 | ||||||||
| 메롭스 | I9 | ||||||||
| SCOP2 | 1gns/SCOPe/SUPFAM | ||||||||
| |||||||||
프로테이나아제 프로펩타이드 억제제(일명 활성화 펩타이드)는 펩티다아제 프로효소 또는 자이모겐의 접힘 및 활성 조절을 담당한다.프로세그먼트는 효소에 도킹하여 기질결합부위를 보호함으로써 효소의 억제를 촉진한다.그러한 프로피드 몇 개는 종종 낮은 배열 [5][6]동일성에도 불구하고 유사한 위상을 공유한다.프로페타이드 영역에는 (베타/알파/베타)x2 토폴로지를 가진 2개의 알파 헬리크와 4개의 베타 스트랜드가 있는 오픈샌드위치 안티팔레알-알파/안티팔레알-베타 폴드가 있다.펩티드가수분해효소 억제제 I9 패밀리는 MEROPS 패밀리 S8A, 서브틸리신에 속하는 펩티드가수분해효소의 N 말단에 프로페타이드 도메인을 포함한다.프로펩타이드는 단백질 분해 분해 분해에 의해 제거되며, 제거는 효소를 활성화시킨다.
인히비터 I10
| 세린엔도펩티드가수분해효소억제제 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 두 개의 에스테르 결합을 포함하는 단백질 분해효소 억제제인 마리노스타틴 용액 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I10 | ||||||||
| 팜 | PF12559 | ||||||||
| 인터프로 | IPR022217 | ||||||||
| |||||||||
이 과는 마이크로비리딘과 마리노스타틴을 모두 포함한다.두 경우 모두 전장 사전펩타이드의 번역 후 수정 후 활성 억제제가 되는 C 말단인 것으로 보인다.분자를 원형으로 만들어 억제 배열을 제공하는 주요 12-잔류 영역 내의 에스테르 결합이다.
인히비터 I24
| PinA펩티드가수분해효소억제제 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | 억제제_I24 | ||||||||
| 팜 | PF10465 | ||||||||
| 인터프로 | IPR019506 | ||||||||
| 메롭스 | I24 | ||||||||
| |||||||||
이 패밀리는 엔도펩티드가수분해효소 La를 억제하는 PinA를 포함한다.그것은 La 호모테트라머에 결합하지만 ATP 결합 부위나 La의 활성 부위에는 간섭하지 않는다.
억제제 I29
| 카테프신프로펩티드억제 도메인(I29) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
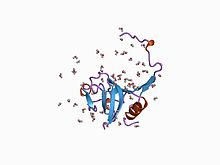 시스테인단백질가수분해효소프로폼결정구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I29 | ||||||||
| 팜 | PF08246 | ||||||||
| 인터프로 | IPR013201 | ||||||||
| |||||||||
MEROPS 펩티드가수분해효소 억제제 패밀리 I29에 속하는 억제제 I29 도메인은 MEROPS 펩티드가수분해효소 서브패밀리 C1A에 속하는 다양한 펩티드가수분해효소 전구체의 N 말단에서 발견되며 카테프신 L, 파파인 및 프로카리케인을 [7]포함한다.기판 결합 사이트를 통과하는 알파 나선 도메인을 형성하여 액세스를 차단합니다.단백질 분해 분해에 의한 이 영역의 제거는 효소의 활성화를 초래한다.이 도메인은 또한 하나 이상의 [8]복사본에서 살라린과 같은 다양한 시스테인 펩티드가수분해효소 억제제에서 발견된다.
인히비터 I34
| 사카로펩신 억제제 I34 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 ia3 돌연변이 억제제와 복합된 단백질 분해효소 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I34 | ||||||||
| 팜 | PF10466 | ||||||||
| 인터프로 | IPR019507 | ||||||||
| 메롭스 | I34 | ||||||||
| |||||||||
사카로펩신 억제제 I34는 아스파라긴펩티드가수분해효소 사카로펩신에 매우 특이적이다.사카로펩신이 없을 때는 대부분 구조화되어 [9]있지 않지만, 그 존재 하에서 억제제는 펩티드가수분해효소의 활성부위분열에서 Asn2에서 Met32까지 거의 완벽한 알파나선을 형성하는 구조변화를 겪는다.
인히비터 I36
| 펩티드가수분해효소억제제I36 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
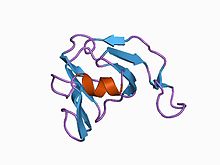 스트렙토미세스 메탈로프로테이나아제 억제제 smpi의 3차원 구조, 스트렙토미세스 니그레센스 tk-23, nmr, 최소 평균 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I36 | ||||||||
| 팜 | PF03995 | ||||||||
| 빠맘 클랜 | CL0333 | ||||||||
| 인터프로 | IPR007141 | ||||||||
| 메롭스 | I36 | ||||||||
| SCOP2 | 1 bhu / SCOPe / SUPFAM | ||||||||
| |||||||||
펩티다아제 억제제 패밀리 I36 도메인은 Streptomyces 종으로 제한된 소수의 단백질에서만 발견된다.모두 아마도 두 개의 이황화 결합을 형성하는 네 개의 보존된 시스테인을 가지고 있다.Streptomyces nigrescens의 이러한 단백질 중 하나는 잘 특성화된 금속단백질가수분해효소 억제제 SMPI이다.[10][11]
SMPI의 구조가 결정되었다.SMPI는 2개의 이황화 브릿지와 함께 102개의 아미노산 잔기를 가지며, MEROPS 펩티드가수분해효소 M4에 속하는 서몰리신 등의 금속단백질가수분해효소를 특이적으로 억제한다. SMPI는 각각 4개의 역평행 베타스트랜드로 구성된 2개의 베타시트로 구성되어 있다.이 구조는 내부 대칭이 2배인 그리스 키 베타 배럴인 두 개의 그리스 키 모티브로 간주할 수 있다.SMPI에서 발견되는 하나의 독특한 구조적 특징은 SMPI의 억제 활성에 관여하는 것으로 알려진 두 번째 그리스 키 모티브의 첫 번째와 두 번째 스트랜드 사이의 연장이다.배열 유사성이 없는 경우 SMPI 구조는 안렌즈 크리스털린의 양쪽 도메인, 칼슘 센서 단백질-S의 양쪽 도메인 및 단일 도메인 효모 킬러 독소와 뚜렷한 유사성을 보인다.효모 킬러 독소 구조는 베타 감마 결정의 각 도메인과 구조적으로 유사하기 때문에 2-도메인 베타 감마 결정체 단백질의 전구체로 생각되었다.따라서 SMPI는 베타 감마 크리스탈린 슈퍼패밀리의 2개 도메인 단백질이 [12]진화한 것으로 여겨지는 조상 접힘에 해당하는 단일 도메인 단백질 구조의 또 다른 예를 제공한다.
억제제 I42
| 샤신족펩티드가수분해효소억제제I42 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 트리파노소마크루지시스테인단백질가수분해효소억제제가신용액구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I42 | ||||||||
| 팜 | PF09394 | ||||||||
| 인터프로 | IPR018990 | ||||||||
| 메롭스 | I42 | ||||||||
| |||||||||
억제제 패밀리 I42는 파파인 유사 시스테인 단백질 [13]분해효소의 가역억제제인 샤가신을 포함한다.샤가신은 인간 CD8alpha에 [14][15]대한 상동성을 가진 면역글로불린 접힘의 독특한 변형인 베타 배럴 구조를 가지고 있다.
인히비터 I48
| 펩티드가수분해효소억제제 클리토사이핀 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | 억제제_I48 | ||||||||
| 팜 | PF10467 | ||||||||
| 인터프로 | IPR019508 | ||||||||
| 메롭스 | I48 | ||||||||
| |||||||||
억제제족 I48은 시스테인단백질가수분해효소를 결합 및 억제하는 클리토사이핀을 포함한다.그것은 알려진 다른 시스테인 단백질 분해효소 억제제와는 유사하지 않지만 [16]버섯에서 추출된 렉틴과 유사한 단백질군과 유사합니다.
억제제 I53
| 트롬빈억제제 마다닌 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | 억제제_I53 | ||||||||
| 팜 | PF11714 | ||||||||
| 인터프로 | IPR021716 | ||||||||
| 메롭스 | I53 | ||||||||
| |||||||||
이 패밀리의 구성원은 펩티드가수분해효소 억제제 마다닌 단백질이다.이 단백질들은 진드기 [17]침에서 분리되었다.
억제제 I67
| 브로멜레인 억제제 VI | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 파인애플 줄기의 브롬레인 억제제 vi의 nmr 구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I67 | ||||||||
| 팜 | PF11405 | ||||||||
| 인터프로 | IPR022713 | ||||||||
| 메롭스 | I67 | ||||||||
| |||||||||
브로멜레인 억제제 VI는 억제제 I67 패밀리에서 11-잔류 및 41-잔류 체인으로 이루어진 이중사슬 억제제이다.
인히비터 I68
| 카르복시펩티드가수분해효소억제제I68 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 인간 카르복시펩티드가수분해효소 b와 복합체 내 진드기 카르복시펩티드가수분해효소 억제제의 결정구조 | |||||||||
| 식별자 | |||||||||
| 기호. | 억제제_I68 | ||||||||
| 팜 | PF10468 | ||||||||
| 인터프로 | IPR019509 | ||||||||
| 메롭스 | I68 | ||||||||
| |||||||||
카르복시펩티드가수분해효소 억제제 I68군은 틱카르복시펩티드가수분해효소 억제제군을 나타낸다.
억제제 I78
| 펩티드가수분해효소억제제I78 패밀리 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| 기호. | 억제제_I78 | ||||||||
| 팜 | PF11720 | ||||||||
| 빠맘 클랜 | CL0367 | ||||||||
| 인터프로 | IPR021719 | ||||||||
| 메롭스 | I78 | ||||||||
| |||||||||
펩티드가수분해효소 억제제 I78 패밀리는 아스페르길루스 엘라스타아제 억제제를 포함한다.
컴파운드
- 아프로티닌
- 베스타틴
- 칼파인 억제제 I 및 II
- 키모스타틴
- E-64
- 루펩틴(N-아세틸-L-류실-L-류실-L-아르긴날)
- 알파-2-마크로글로불린
- 페파블락 SC
- 펩스타틴
- PMSF(불화페닐메탄술포닐)
- 째깍째깍
- 트립신 억제제
「 」를 참조해 주세요.
레퍼런스
- ^ "antiprotease". The Free Dictionary.
- ^ Roy, Mrinalini; Rawat, Aadish; Kaushik, Sanket; Jyoti, Anupam; Srivastava, Vijay Kumar (2022-08-01). "Endogenous cysteine protease inhibitors in upmost pathogenic parasitic protozoa". Microbiological Research. 261: 127061. doi:10.1016/j.micres.2022.127061. ISSN 0944-5013.
- ^ 오밈단백질가수분해효소억제ITOR 1, PI
- ^ Rawlings ND, Tolle DP, Barrett AJ (March 2004). "Evolutionary families of peptidase inhibitors". Biochem. J. 378 (Pt 3): 705–16. doi:10.1042/BJ20031825. PMC 1224039. PMID 14705960.
- ^ Tangrea MA, Bryan PN, Sari N, Orban J (July 2002). "Solution structure of the pro-hormone convertase 1 pro-domain from Mus musculus". J. Mol. Biol. 320 (4): 801–12. doi:10.1016/S0022-2836(02)00543-0. PMID 12095256.
- ^ Jain SC, Shinde U, Li Y, Inouye M, Berman HM (November 1998). "The crystal structure of an autoprocessed Ser221Cys-subtilisin E-propeptide complex at 2.0 A resolution". J. Mol. Biol. 284 (1): 137–44. doi:10.1006/jmbi.1998.2161. PMID 9811547.
- ^ Groves MR, Taylor MA, Scott M, Cummings NJ, Pickersgill RW, Jenkins JA (October 1996). "The prosequence of procaricain forms an alpha-helical domain that prevents access to the substrate-binding cleft". Structure. 4 (10): 1193–203. doi:10.1016/s0969-2126(96)00127-x. PMID 8939744.
- ^ Olonen A, Kalkkinen N, Paulin L (July 2003). "A new type of cysteine proteinase inhibitor--the salarin gene from Atlantic salmon (Salmo salar L.) and Arctic charr (Salvelinus alpinus)". Biochimie. 85 (7): 677–81. doi:10.1016/S0300-9084(03)00128-7. PMID 14505823.
- ^ Green TB, Ganesh O, Perry K, Smith L, Phylip LH, Logan TM, Hagen SJ, Dunn BM, Edison AS (April 2004). "IA3, an aspartic proteinase inhibitor from Saccharomyces cerevisiae, is intrinsically unstructured in solution". Biochemistry. 43 (14): 4071–81. doi:10.1021/bi034823n. PMID 15065849.
- ^ Tanaka K, Aoki H, Oda K, Murao S, Saito H, Takahashi H (November 1990). "Nucleotide sequence of the gene for a metalloproteinase inhibitor of Streptomyces nigrescens (SMPI)". Nucleic Acids Res. 18 (21): 6433. doi:10.1093/nar/18.21.6433. PMC 332545. PMID 2243793.
- ^ Murai H, Hara S, Ikenaka T, Oda K, Murao S (January 1985). "Amino acid sequence of Streptomyces metallo-proteinase inhibitor from Streptomyces nigrescens TK-23". J. Biochem. 97 (1): 173–80. doi:10.1093/oxfordjournals.jbchem.a135041. PMID 3888972.
- ^ Ohno A, Tate S, Seeram SS, Hiraga K, Swindells MB, Oda K, Kainosho M (September 1998). "NMR structure of the Streptomyces metalloproteinase inhibitor, SMPI, isolated from Streptomyces nigrescens TK-23: another example of an ancestral beta gamma-crystallin precursor structure". J. Mol. Biol. 282 (2): 421–33. doi:10.1006/jmbi.1998.2022. PMID 9735297.
- ^ Monteiro AC, Abrahamson M, Lima AP, Vannier-Santos MA, Scharfstein J (November 2001). "Identification, characterization and localization of chagasin, a tight-binding cysteine protease inhibitor in Trypanosoma cruzi". J. Cell Sci. 114 (Pt 21): 3933–42. doi:10.1242/jcs.114.21.3933. PMID 11719560.
- ^ Figueiredo da Silva AA; de Carvalho Vieira L; Krieger MA; Goldenberg S; Zanchin NI; Guimarães BG (February 2007). "Crystal structure of chagasin, the endogenous cysteine-protease inhibitor from Trypanosoma cruzi". J. Struct. Biol. 157 (2): 416–23. doi:10.1016/j.jsb.2006.07.017. PMID 17011790.
- ^ Wang SX, Pandey KC, Scharfstein J, Whisstock J, Huang RK, Jacobelli J, Fletterick RJ, Rosenthal PJ, Abrahamson M, Brinen LS, Rossi A, Sali A, McKerrow JH (May 2007). "The structure of chagasin in complex with a cysteine protease clarifies the binding mode and evolution of an inhibitor family". Structure. 15 (5): 535–43. doi:10.1016/j.str.2007.03.012. PMID 17502099.
- ^ Brzin J, Rogelj B, Popovic T, Strukelj B, Ritonja A (June 2000). "Clitocypin, a new type of cysteine proteinase inhibitor from fruit bodies of mushroom clitocybe nebularis". J. Biol. Chem. 275 (26): 20104–9. doi:10.1074/jbc.M001392200. PMID 10748021.
- ^ Iwanaga S, Okada M, Isawa H, Morita A, Yuda M, Chinzei Y (May 2003). "Identification and characterization of novel salivary thrombin inhibitors from the ixodidae tick, Haemaphysalis longicornis". Eur. J. Biochem. 270 (9): 1926–34. doi:10.1046/j.1432-1033.2003.03560.x. PMID 12709051.


