V(D)J 재결합
V(D)J recombinationV(D)J 재조합은 T세포와 B세포의 성숙 초기 단계에서 림프구 발달에서만 발생하는 체세포 재조합 메커니즘이다.그것은 각각 B세포와 T세포에서 발견되는 항체/면역글로불린과 T세포 수용체(TCR)의 매우 다양한 레퍼토리를 낳는다.그 과정은 적응 면역 시스템의 결정적인 특징입니다.
포유류의 V(D)J 재조합은 1차 림프기관(B세포의 골수, T세포의 흉선)과 거의 랜덤한 방식으로 변수(V), 결합(J), 그리고 경우에 따라 다양성(D) 유전자 세그먼트에서 일어난다.이 과정은 궁극적으로 면역글로불린과 TCR의 항원결합 영역에 새로운 아미노산 서열을 만들어 박테리아, 바이러스, 기생충, 웜을 포함한 거의 모든 병원체로부터 항원을 인식할 수 있게 하고 암에서 보이는 "변화된 자기 세포"를 인식하게 한다.또한 인식은 자연에서 알레르기(예: 꽃가루 또는 기타 알레르겐)가 있을 수 있으며 숙주 조직과 일치하여 자가 면역으로 이어질 수 있습니다.
1987년, 도네가와 스스무는 "항체 [1]다양성의 발생을 위한 유전적 원리를 발견한" 공로로 노벨 생리의학상을 수상했다.
배경
인간 항체 분자(B세포 수용체 포함)는 무거운 사슬과 가벼운 사슬로 구성되며, 각각은 유전적으로 세 개의 위치에 부호화된 상수(C) 영역과 가변(V) 영역을 모두 포함한다.
- 면역글로불린 중쇄의 유전자 세그먼트를 포함한 14번 염색체의 면역글로불린 중궤적(IGH@).
- 1종류의 면역글로불린 경쇄에 대한 유전자 세그먼트를 포함한 염색체 2상의 면역글로불린 카파(α) 궤적(IGK@).
- 염색체 22의 면역글로불린 람다()) 궤적(IGL@)은 면역글로불린 경쇄의 다른 유형())에 대한 유전자 세그먼트를 포함한다.
각 중쇄 또는 경쇄 유전자는 항체 단백질의 가변 영역에 대해 세 가지 다른 유형의 유전자 세그먼트의 여러 복사본을 포함한다.예를 들어 인간 면역글로불린 중쇄 영역은 2개의 상수(Cμ 및 C c) 유전자 세그먼트와 44개의 가변(V) 유전자 세그먼트와 27개의 다양성(D) 유전자 세그먼트 및 6개의 결합(J) 유전자 [2]세그먼트를 포함한다.경쇄 유전자는 다수의 V 및 J 유전자 세그먼트를 가진 1개의 (C)) 또는 4개의 (C)) 상수 유전자 세그먼트를 가지지만 D 유전자 [3]세그먼트를 가지지 않는다.DNA 재배열은 주어진 림프구에 각 유형의 유전자 세그먼트의 복사본이 하나씩 들어가도록 하며, 거대한 항체 레퍼토리를 생성한다; 자기 반응성 때문에 일부 제거되기는 하지만 대략 3×1011 조합이 가능하다.
대부분의 T세포 수용체는 가변 알파 사슬과 베타 사슬로 구성되어 있다.T세포 수용체 유전자는 면역 글로불린 유전자와 유사합니다.이는 림프구가 발달하는 동안 세포에 독특한 항원 수용체를 제공하기 위해 재배열된 베타 사슬에 여러 개의 V, D, J 유전자 세그먼트를 포함하고 있습니다.이러한 의미에서 T세포 수용체는 항체의 항원 결합 단편과 동일한 위상이며, 둘 다 면역글로불린 슈퍼패밀리의 일부이다.
자가반응하는 세포를 제거함으로써 자가면역반응을 방지한다.이는 자가면역조절기(AIRE)의 기능을 통해 발현되는 자가항원 배열에 대해 세포를 테스트함으로써 흉선에서 발생합니다.면역글로불린 람다 경쇄 궤적에는 단백질 코드 유전자가 포함되어 있어 재배열과 함께 손실될 수 있습니다.이것은 생리학적 메커니즘에 기초하며 백혈병이나 림프종의 경우 병원성이 아니다.셀은 자가 반응하지 않는 성공적인 제품을 생성하면 유지되며 그렇지 않으면 아포토시스를 통해 플루닝됩니다.
면역글로불린
헤비 체인
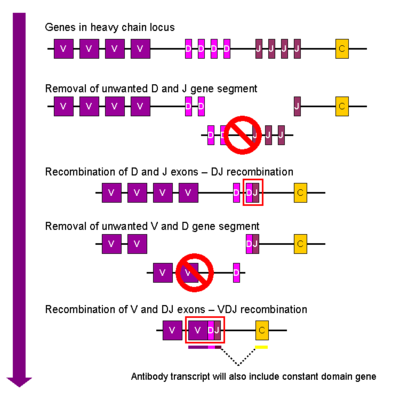
현상 중인 B세포에서 최초로 발생하는 재조합 이벤트는 중쇄 궤적의 1개의 D와 1개의 J 유전자 세그먼트이다.이 두 유전자 세그먼트 사이의 DNA는 모두 삭제됩니다.이 D-J 재조합은 새롭게 형성된 디제이 복합체의 상류에서 하나의 V 유전자 세그먼트가 결합되어 재배열된 VDJ 유전자 세그먼트를 형성한다.V와 D 세그먼트 사이의 다른 모든 유전자 세그먼트는 이제 세포의 게놈에서 삭제되었다.중쇄의 VDJ 영역과 상수 mu 및 델타 체인(Cμ 및 Cδ)을 모두 포함한 1차 전사체(즉, 1차 전사체에는 세그먼트 V-D-J-Cμδ)가 생성된다.1차 RNA는 C 사슬μ 뒤에 폴리아데닐화(poly-A) 꼬리를 추가하고 VDJ 세그먼트와 이 일정한 유전자 세그먼트 사이의 배열을 제거하기 위해 처리된다.이 mRNA의 번역은 IgM 중쇄 단백질의 생산으로 이어진다.
라이트 체인
면역글로불린 경쇄 궤적의 카파())와 람다()) 사슬은 D 세그먼트가 없다는 점을 제외하고는 매우 유사한 방식으로 재배열된다.즉, 경쇄의 재조합의 제1단계는 1차 전사 중에 일정한 사슬 유전자를 부가하기 전에 V와 J사슬의 결합을 통해 VJ복합체를 형성한다.카파사슬 또는 람다사슬 중 하나에 대한 스플라이스 mRNA의 번역은 Ig δ 또는 Ig δ 경쇄단백질을 생성한다.
Igμ중쇄와 그 중 하나의 경쇄가 조립되면 미성숙 B세포 표면에 발현되는 면역글로불린 IgM의 막결합 형태가 형성된다.
T세포수용체
흉선세포가 발달하는 동안 T세포수용체(TCR) 사슬은 기본적으로 면역글로불린에 대해 기술된 것과 같은 순서 있는 재조합 이벤트를 겪는다.D-to-J 재조합은 TCR의 β-사슬에서 먼저 발생한다.이 과정은 6개의β J1β 세그먼트 중 하나에 D1 유전자 세그먼트를 결합하거나 6개의β J2 세그먼트 [3]중 하나에 D2β 유전자 세그먼트를 결합하는 것을 포함할 수 있다.위와 같이 DJ 재조합은 V-DJ 간 재배열로 이루어집니다.새롭게 형성된 복합체 내의 V-D-Jβββ 유전자 세그먼트 간의 모든 유전자 세그먼트는 삭제되고 상수 도메인 유전자(V-D-J-Cββββ)를 포함하는 1차 전사물이 합성된다.mRNA 전사는 모든 간섭 배열을 분리하여 TCR β-사슬을 위한 전장 단백질의 번역을 가능하게 한다.
TCR의 알파(α) 사슬의 재배열은 β 사슬 재배열을 따르며, Ig 경쇄에 대해 설명된 V-to-J 정렬과 유사하다(위 참조).β-사슬과 α-사슬의 결합은 대다수의 T세포에서 발현되는 αβ-TCR을 형성한다.
메커니즘
주요 효소 및 성분
V(D)J 재조합 과정은 다양한 효소 집합인 VDJ 재조합 효소에 의해 매개된다.관련된 주요 효소는 재조합 활성화 유전자 1 및 2(RAG), 말단 디옥시뉴클레오티딜 전이효소(TdT) 및 DNA [4]수복을 위한 유비쿼터스 비호몰로지 엔드 결합(NHEJ) 경로의 구성원인 아르테미스 뉴클레아제이다.이 과정에 관여하는 몇몇 다른 효소는 DNA 의존성 단백질인산화효소(DNA-PK), X선 수복 교차 보완 단백질 4(XRCC4), DNA 리가아제 IV, 비호몰로지 말단 결합인자 1(NHEJ1), Cernunos 또는 XRCC 유사인자 [X-Like]를 포함하는 것으로 알려져 있다.ases µ 및 [5]μ.관련된 일부 효소는 림프구(예: RAG, TdT)에 특유한 반면, 다른 세포 유형에서 발견되고 심지어 보편적으로 발견되기도 한다(예: NHEJ 성분).
재조합의 특이성을 유지하기 위해 V(D)J 재조합효소는 가변(V), 다양성(D) 및 결합(J) 유전자 세그먼트에 인접한 재조합 신호 배열(RSS)을 인식하고 결합한다.RSS는 7개의 보존된 뉴클레오티드의 헵타머, 길이 12 또는 23개의 염기쌍의 스페이서 영역 및 9개의 보존된 뉴클레오티드의 노나머의 세 가지 요소로 구성됩니다.RSS의 대부분은 시퀀스에 따라 다르지만 컨센서스 헵타머와 노나머 시퀀스는 각각 CACAGTG와 ACAAACC입니다.스페이서 영역의 시퀀스는 잘 보존되지 않지만 길이는 매우 [6][7]보존됩니다.스페이서 영역의 길이는 DNA 나선의 약 1개(12개 염기쌍) 또는 2개 회전(23개 염기쌍)에 해당합니다.12/23 규칙으로 알려진 대로, 재조합되는 유전자 세그먼트는 보통 다른 스페이서 길이의 RSS에 인접한다(즉, 하나는 "12RSS"이고 하나는 "23RSS"[8]이다).이는 V(D)J [9]재조합 조절에서 중요한 특징이다.
과정
V(D)J 재조합은 (RAG1의 활성을 통해) V(D)J 재조합이 부호화 유전자 세그먼트(V, D 또는 J) 옆에 있는 RSS를 결합하고 RSS의 첫 번째 염기(헵타머 직전)와 부호화 세그먼트 사이에 DNA에 단일 가닥 흠집을 만들 때 시작된다.이는 본질적으로 에너지적으로 중립적이며(ATP 가수분해 필요 없음), 동일한 가닥에서 3' 하이드록실기와 5' 인산기가 형성된다.반응성 히드록실기는 반대쪽 가닥의 포스포디에스테르 결합을 공격하기 위해 재조합 효소에 의해 위치하여 코딩 세그먼트의 헤어핀(줄기 루프)과 신호 [10]세그먼트의 뭉툭한 끝의 두 개의 DNA 끝을 형성한다.현재 모델은 DNA 절단 및 헤어핀 형성이 재조합 [11][12][13][14]중심이라고 알려진 복합체에서 두 가닥에서 동시에(또는 거의) 일어나는 것입니다.
무딘 신호 끝은 신호 접합으로 알려진 코드 세그먼트 사이의 모든 간섭 서열을 포함하는 DNA의 원형 조각을 형성하기 위해 서로 수평으로 결합됩니다(본질적으로는 원형이지만, 이것은 플라스미드와 혼동해서는 안 됩니다).원래 연속적인 세포 분열 동안 상실되는 것으로 생각되었지만, 신호 관절이 게놈으로 다시 들어가 종양 유전자를 활성화하거나 종양 억제 유전자 기능을 방해함으로써 병리학으로 이어질 수 있다는 증거가 있다[Ref].
부호화 끝은 궁극적으로 접합 [15]다양성으로 이어지는 여러 사건에 의해 결속되기 전에 더 처리된다.DNA-PK가 각각의 망가진 DNA 끝에 결합하고 Artemis, XRCC4, DNA 연결효소 IV, Cernunnos, 그리고 여러 DNA [16]중합효소를 포함한 몇 가지 다른 단백질을 모집할 때 처리가 시작됩니다.DNA-PK는 복합체를 형성하여 아르테미스의 활성화를 초래한다.코드 끝의 머리핀은 [17]Artemis의 활동에 의해 개방됩니다.중앙에서 열리면 무딘 DNA 말단이 생깁니다. 그러나 대부분의 경우, 개구부는 "중앙에서 벗어난" 상태이고 한 가닥에 여분의 염기가 남아 있게 됩니다(오버행).이것들은 DNA 복구 효소가 돌출부를 [18]해결할 때 생성되는 순서의 회문 특성 때문에 회문 뉴클레오티드로 알려져 있습니다.Artemis에 의한 헤어핀 개방 과정은 V(D)J 재조합의 중요한 단계로 심각한 복합면역결핍(scid) 마우스 모델에 결함이 있습니다.
다음으로 XRCC4, Cernunnos 및 DNA-PK는 DNA 말단을 정렬하고 코드 말단에 비템플레이트(N) 뉴클레오티드를 첨가하는 템플릿 비의존성 DNA 중합효소인 말단 디옥시뉴클레오티딜전달효소(TdT)를 채용한다.첨가물은 대부분 무작위이지만, TdT는 G/C 뉴클레오티드에 [19]대한 선호도를 보인다.알려진 모든 DNA 중합효소들과 마찬가지로 TdT는 뉴클레오티드를 5'에서 3' 방향으로 [20]한 가닥에 첨가한다.
마지막으로, 엑소핵산가수분해효소는 코드 말단에서 염기를 제거할 수 있습니다.이어서 DNA 중합효소 δ 및 μ를 필요에 따라 추가 뉴클레오티드를 삽입하여 양끝이 결합에 적합하도록 한다.이것은 확률적 과정이기 때문에, P와 N 뉴클레오티드의 첨가와 외부 핵 분해 제거의 어떤 조합도 일어날 수 있다(또는 전혀 일어나지 않는다).마지막으로 처리한 코드 끝은 DNA 연결효소 [21]IV에 의해 결합된다.
이러한 모든 처리 사건은 같은 유전자 세그먼트가 재결합되어도 매우 가변적인 파라토프를 초래한다.V(D)J 재조합은 유기체나 그 조상(들)이 이전에 접하지 않아도 되는 항원에 대한 면역 글로불린과 T세포 수용체의 생성을 허용하고, 발병하는 새로운 병원체나 자주 변화하는 병원체(예: 계절 인플루엔자)에 대한 적응 면역 반응을 가능하게 한다.그러나 이 과정의 주요 경고는 최종 단백질 생성물에서 올바른 아미노산 배열을 유지하기 위해 DNA 배열을 프레임 안에 유지해야 한다는 것입니다.결과 배열이 프레임 외일 경우, 세포의 발육이 중단되고 세포는 성숙할 때까지 생존하지 못할 것이다.따라서 V(D)J 재조합은 엄격한 규제와 관리가 필요한 매우 비용이 많이 드는 프로세스입니다.
「 」를 참조해 주세요.
레퍼런스
- ^ "The Nobel Prize in Physiology or Medicine 1987". nobelprize.org. Archived from the original on 13 February 2021. Retrieved 26 December 2014.
- ^ Li A, Rue M, Zhou J, et al. (June 2004). "Utilization of Ig heavy chain variable, diversity, and joining gene segments in children with B-lineage acute lymphoblastic leukemia: implications for the mechanisms of VDJ recombination and for pathogenesis". Blood. 103 (12): 4602–9. doi:10.1182/blood-2003-11-3857. PMID 15010366.
- ^ a b Abbas, Abul K. (2018). "Lymphocyte Development and Antigen Receptor Gene Rearrangement". Cellular and Molecular Immunology (9th ed.). Philadelphia, PA: Elsevier. ISBN 978-0-323-47978-3.
- ^ Ma, Yunmei; Lu, Haihui; Schwarz, Klaus; Lieber, Michael (September 2005). "Repair of Double-Strand DNA Breaks by the Human Nonhomologous DNA End Joining Pathway: the Iterative Processing Model". Cell Cycle. 4 (9): 1193–1200. doi:10.4161/cc.4.9.1977. PMID 16082219.
- ^ Malu, Shruti; Malshetty, Vidyasagar; Francis, Dailia; Cortes, Patricia (2012). "Role of non-homologous end joining in V(D)J recombination". Immunologic Research. 54 (1–3): 233–246. doi:10.1007/s12026-012-8329-z. PMID 22569912. S2CID 45771818.
- ^ Ramsden, Dale; Baetz, Kristin; Wu, Gillian (1994). "Conservation of Sequence in Recombination Signal Sequence Spacers". Nucleic Acids Research. 22 (10): 1785–1796. doi:10.1093/nar/22.10.1785. PMC 308075. PMID 8208601.
- ^ Cowell, Lindsay; Davila, Marco; Ramsden, Dale; Kelsoe, Garnett (2004). "Computational tools for understanding sequence variability in recombination signals". Immunological Reviews. 200: 57–69. doi:10.1111/j.0105-2896.2004.00171.x. PMID 15242396. S2CID 40771963.
- ^ van Gent, Dik; Ramsden, Dale; Gellert, Martin (1996). "The RAG1 and RAG2 Proteins Establish the 12/23 Rule in V(D)J Recombination". Cell. 85 (1): 107–13. doi:10.1016/s0092-8674(00)81086-7. PMID 8620529.
- ^ Hiom, Kevin; Gellert, Martin (1998). "Assembly of a 12/23 Paired Signal Complex: a Critical Control Point in V(D)J Recombination". Molecular Cell. 1 (7): 1011–1019. doi:10.1016/s1097-2765(00)80101-x. PMID 9651584.
- ^ Schatz, David; Swanson, Patrick (2011). "V(D)J Recombination: Mechanisms of Initiation". Annual Review of Genetics. 45: 167–202. doi:10.1146/annurev-genet-110410-132552. PMID 21854230.
- ^ Schatz, David; Ji, Yanhong (2011). "Recombination Centres and the Orchestration of V(D)J Recombination". Nature Reviews Immunology. 11 (4): 251–263. doi:10.1038/nri2941. PMID 21394103. S2CID 33489235.
- ^ Curry, John; Geier, Jamie; Schlissel, Mark (2005). "Single-Strand Recombination Signal Sequence Nicks in vivo: Evidence for a Capture Model of Synapsis". Nature Immunology. 6 (12): 1272–1279. doi:10.1038/ni1270. PMID 16286921. S2CID 10975251.
- ^ Agrawal, Alka; Schatz, David (1997). "RAG1 and RAG2 Form a Stable Postcleavage Synaptic Complex with DNA Containing Signal Ends in V(D)J Recombination". Cell. 89 (1): 43–53. doi:10.1016/s0092-8674(00)80181-6. PMID 9094713.
- ^ Fugmann, Sebastian; Lee, AIfred; Shockett, Penny; Villey, Isabelle; Schatz, David (2000). "The RAG Proteins and V(D)J Recombination: Complexes, Ends, and Transposition". Annual Review of Immunology. 18: 495–527. doi:10.1146/annurev.immunol.18.1.495. PMID 10837067.
- ^ Lewis, Susanna (1994). "The Mechanism of V(D)J Joining: Lessons from Molecular, Immunological, and Comparative Analyses". Advances in Immunology Volume 56. Advances in Immunology. Vol. 56. pp. 27–150. doi:10.1016/s0065-2776(08)60450-2. ISBN 9780120224562. PMID 8073949.
- ^ Helmink, Beth; Sleckman, Barry (2012). "The response to and repair of RAG-mediated DNA double-strand breaks". Annual Review of Immunology. 30: 175–202. doi:10.1146/annurev-immunol-030409-101320. PMC 4038028. PMID 22224778.
- ^ Ma, Yunmei; Schwarz, Klaus; Lieber, Michael (2005). "The Artemis:DNA-PKcs Endonuclease Cleaves DNA Loops, Flaps, and Gaps". DNA Repair. 4 (7): 845–851. doi:10.1016/j.dnarep.2005.04.013. PMID 15936993.
- ^ Lu, Haihui; Schwarz, Klaus; Lieber, Michael (2007). "Extent to Which Hairpin Opening by the Artemis:DNA-PKcs Complex can Contribute to Junctional Diversity in V(D)J Recombination". Nucleic Acids Research. 35 (20): 6917–6923. doi:10.1093/nar/gkm823. PMC 2175297. PMID 17932067.
- ^ Gauss, George; Lieber, Michael (1996). "Mechanistic Constraints on Diversity in Human V(D)J Recombination". Molecular and Cellular Biology. 16 (1): 258–269. doi:10.1128/MCB.16.1.258. PMC 230999. PMID 8524303.
- ^ Benedict, Cindy; Gilfillan, Susan; Thai, To-Ha; Kearney, John (2000). "Terminal Deoxynucleotidyl Transferase and Repertoire Development". Immunological Reviews. 175: 150–157. doi:10.1111/j.1600-065x.2000.imr017518.x. PMID 10933600. S2CID 20634390.
- ^ van Gent, DC; van der Burg, M (10 December 2007). "Non-Homologous End-Joining, a Sticky Affair". Oncogene. 26 (56): 7731–40. doi:10.1038/sj.onc.1210871. PMID 18066085.
추가 정보
- Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC (2000). Chapter 24, Evolution at the molecular level. In: Genetics. New York: McGraw-Hill. pp. 805–807. ISBN 978-0-07-299587-9.
- V(D)J 재결합시리즈:실험의학과 생물학의 진보, 제650권 Ferrier, Pierre (Ed.) Landes Bioscience 2009, XI, 199 p.ISBN 978-1-4419-0295-5