녹색 형광 단백질
Green fluorescent protein| 녹색 형광 단백질 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
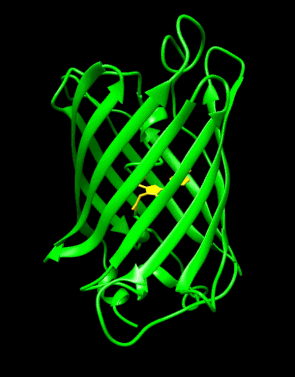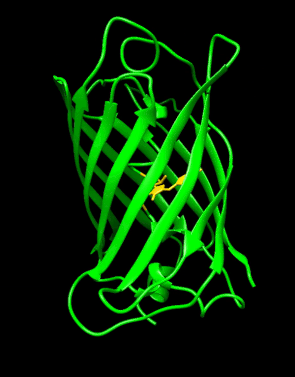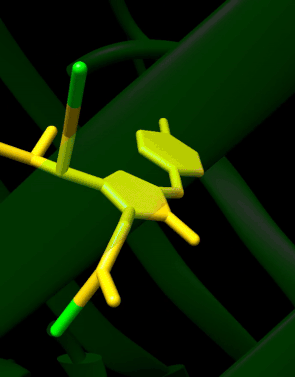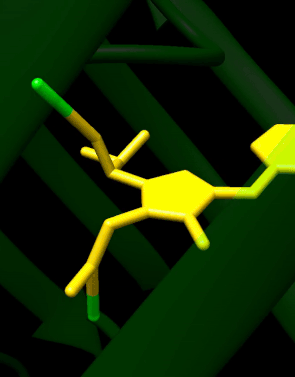
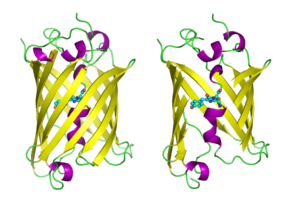
 Aequorea victoria green 형광단백질 [1]구조. | |||||||||
| 식별자 | |||||||||
| 기호. | GFP | ||||||||
| 팜 | PF01353 | ||||||||
| 빠맘 클랜 | CL0069 | ||||||||
| 인터프로 | IPR011584 | ||||||||
| 캐스 | 1ema | ||||||||
| SCOP2 | 1ema/SCOPe/SUPFAM | ||||||||
| |||||||||
| 녹색 형광 단백질 | |||||||
|---|---|---|---|---|---|---|---|
| 식별자 | |||||||
| 유기체 | |||||||
| 기호. | GFP | ||||||
| 유니프로트 | P42212 | ||||||
| |||||||
녹색형광단백질(GFP)은 청색에서 자외선에 [2][3]노출되면 밝은 녹색 형광을 나타내는 단백질이다.GFP라는 라벨은 전통적으로 해파리 Aequorea victoria에서 처음 분리된 단백질을 가리키며 avGFP라고 불리기도 한다.하지만, GFP는 산호, 말미잘, 조석류, 요각류, [4]랜슬렛을 포함한 다른 유기체에서 발견되었다.
A. victoria의 GFP는 파장 395 nm에서 메이저 들뜸 피크, 475 nm에서 마이너 들뜸 피크입니다.방출 피크는 가시 스펙트럼의 녹색 하단 부분에 있는 509 nm이다.GFP의 형광 양자 수율(QY)은 0.79입니다.바다 팬시(Renilla reniformis)의 GFP는 498 nm에서 단일 주요 들뜸 피크를 가진다.GFP는 분자 [5]산소 이외의 보조 요소, 유전자 생성물 또는 효소/기질을 필요로 하지 않고 내부 색소를 형성할 수 있는 능력 때문에 많은 형태의 생물학에서 훌륭한 도구를 만듭니다.
세포 및 분자생물학에서 GFP 유전자는 [6]발현 리포터로 자주 사용된다.그것은 바이오센서를 만들기 위해 변형된 형태로 사용되어 왔고, GFP를 발현하는 많은 동물들이 만들어졌어요. GFP는 주어진 유기체, 선택된 장기, 또는 관심 세포에서 유전자가 발현될 수 있다는 개념의 증거를 보여줍니다.GFP는 트랜스제닉 기술을 통해 동물이나 다른 종에 도입될 수 있으며, 그들의 게놈과 그들의 자손의 게놈에서 유지될 수 있다.지금까지 GFP는 박테리아, 효모, 곰팡이, 물고기, 포유류를 포함한 많은 종에서 발현되어 왔습니다.과학자 Roger Y. 2008년 10월 10일 녹색 형광 단백질을 발견하고 개발한 공로로 첸, 시모무라 오사무, 마틴 찰피는 2008년 노벨 화학상을 수상했다.
GFP와 유사한 형광 단백질에 대해 상업적으로 이용 가능한 대부분의 유전자는 약 730개의 염기쌍이다.자연 단백질은 238개의 아미노산을 가지고 있습니다.그것의 분자 질량은 27kD이다.[7]따라서 GFP 유전자를 해당 단백질의 유전자에 융합시키면 단백질의 크기와 분자량이 크게 증가하고 단백질의 자연기능을 손상시키거나 세포 [8]내 이동 위치나 이동 궤적을 바꿀 수 있다.
배경
와일드 타입 GFP(wtGFP)
GFP는 1960~1970년대 시모무라 [9]오사무가 연구한 해파리 에쿠오레아 빅토리아에서 분리된 발광 단백질 에쿠오린(luminent protein aequorin, 루시페린의 분해를 촉진하고 빛을 방출하는 효소)과 함께 정제됐다.A. victoria에서 GFP 형광은 aequorin이 Ca2+ 이온과 상호작용할 때 발생하며 파란색 빛을 유발한다.이 발광 에너지 중 일부는 GFP로 전달되어 전체 색상이 [10]녹색으로 바뀝니다.하지만, 분자생물학자들을 위한 도구로서의 그것의 유용성은 1992년 더글라스 프라셔가 [11]진에서 wtGFP의 복제와 뉴클레오티드 배열에 대해 보고했을 때 비로소 실현되기 시작했다.이 프로젝트에 대한 자금이 바닥났기 때문에 Prasher는 여러 연구소에 cDNA 샘플을 보냈습니다.마틴 찰피의 연구실은 대장균과 엘레건의 이종 세포에서 처음 몇 개의 아미노산이 제거된 상태에서 wtGFP의 코드 배열을 표현하여 1994년 [12]사이언스에 그 결과를 발표했다.Frederick Tsuji의 연구실은 한 달 후에 [13]이 재조합 단백질의 발현을 독립적으로 보고했다.놀랍게도, GFP 분자는 접혀져 상온에서 형광을 띠었고, 해파리 특유의 외인성 보조 인자는 필요하지 않았다.이 근중량 GFP는 형광성이었지만 이중 피크 들뜸 스펙트럼, pH 민감도, 염화물 민감도, 낮은 형광 양자 수율, 낮은 광안정성, 37°C에서의 낮은 접힘 등 몇 가지 단점이 있었다.
GFP의 첫 번째 결정 구조는 1996년 [14]사이언스의 레밍턴 그룹에 의해 S65T 돌연변이였다.한 달 뒤 필립스 그룹은 네이처 바이오테크놀로지([15]Nature Biotechnology)의 야생형 GFP 구조를 독자적으로 보고했다.이러한 결정 구조는 발색단 형성과 인접 잔류물 상호작용에 대한 중요한 배경을 제공했다.연구진은 유도 및 무작위 돌연변이를 통해 이러한 잔류물을 수정하여 오늘날 사용되는 다양한 GFP 유도체를 생산했습니다.GFP에 대한 추가 연구는 GFP가 세제, 단백질 분해효소, 염화구아니듐(GdmCl) 처리 및 급격한 온도 [16]변화에 내성이 있는 것으로 나타났습니다.
GFP 유도체
광범위한 사용의 가능성과 연구자들의 진화하는 요구로 인해, GFP의 많은 다른 돌연변이들이 [17][18]개발되었습니다.첫 번째 주요 개선은 1995년 Roger Tsien이 [19]Nature에서 보고한 단일점 돌연변이(S65T)였다.이 돌연변이는 GFP의 스펙트럼 특성을 극적으로 개선하여 형광, 광안정성을 증가시키고 주요 들뜸 피크를 488 nm로 이동시켰으며, 최대 방출량은 509 nm로 유지되었다.이는 일반적으로 이용 가능한 FITC 필터 세트의 스펙트럼 특성과 일치하여 일반 연구원의 사용 실용성을 높였다.Thastrup과[20] Falkow의 [21]연구소에서 1995년에 이 발판에 37°C 폴딩 효율성(F64L) 점 변이를 발견하여 향상된 GFP(EGFP)를 산출하였다.EGFP는 포유동물 세포에서 GFP를 실용적으로 사용할 수 있게 했다.EGFP의 소멸계수(')[22]는 55,000Mcm입니다−1−1.EGFP의 형광 양자 수율(QY)은 0.60입니다.상대 휘도는 µ−1−1·QY로 33,000Mcm입니다.
GFP가 제대로 접히지 않은 펩타이드에 융합되어도 빠르게 접히고 성숙할 수 있는 일련의 돌연변이인 Superfolder GFP(sfGFP)는 [23]2006년에 보고되었다.
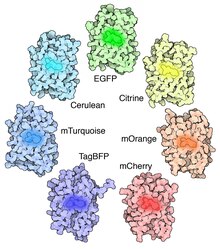
특히 청색 형광 단백질(EBFP, EBFP2, Azurite, mKalama1)과 시안 형광 단백질(ECFP, Cerulean, CyPet, mTurquoise2, 황색 형광 단백질 유도체(YFP, 시트린, 비너스, YET)를 포함한 많은 돌연변이가 이루어졌다.BFP 도함수(mKalama1 제외)는 Y66H 치환을 포함합니다.이들은 자외선 중심에서 380나노미터에 가까운 넓은 흡수대역을 보이며 최대 방출량은 448나노미터이다.Zn(II)과 Cu(II)를 우선적으로 결합하는 녹색 형광 단백질 돌연변이체(BFPms1)가 개발됐다.BFPms1은 및 BFP 발색단(Y66H), 양자수율 향상을 위한 Y145F, 베타배럴에 구멍을 만들기 위한 H148G 및 용해성을 높이는 몇 가지 다른 돌연변이를 포함한다.Zn(II) 결합은 형광 강도를 높이는 반면 Cu(II) 결합은 형광을 억제하고 최대 흡광도를 379nm에서 444nm로 이동시킨다.따라서 Zn biosensor로 [24]사용할 수 있습니다.
색소 결합.시안 유도체의 중요한 돌연변이는 Y66W 치환이며, 이로 인해 발색단이 페놀 성분이 아닌 인돌로 형성됩니다.인돌 그룹의 부피가 증가하기 때문에 이 변형된 색단에 대한 밝기를 복원하려면 주변 배럴에 몇 가지 추가 보상 돌연변이가 필요합니다.ECFP 및 Cerulean에서 7번째 가닥의 N단자 절반은 두 가지 형태를 나타낸다.이러한 배치는 모두 발색단과의 복잡한 일련의 반데르발스 상호작용을 가지고 있다.Cerulean의 Y145A 및 H148D 돌연변이는 이러한 상호작용을 안정화시키고 색소체가 더 평면적이고, 더 잘 채워지고, 충돌 [25]담금질 가능성이 낮아지도록 합니다.
형광 수명 기반 스크리닝과 조합하여 추가적인 부위 지향 랜덤 돌연변이 유발은 7번째 β-스트랜드를 더욱 안정화시켰고, 결과적으로 [26]0.93의 양자 수율(QY)을 가진 밝은 변종 mTurquoise2를 생성했다.YFP 유도체의 적색 편이 파장은 T203Y 돌연변이에 의해 달성되며, 치환된 티로신 잔기와 [3]색소단 사이의 δ-전자 적층 상호작용에 기인한다.이 두 가지 등급의 스펙트럼 변이는 종종 Förster 공명 에너지 전달(FRET) 실험에 사용된다.칼슘 또는 글루탐산염, 단백질 인산화 상태, 단백질 보완, 수용체 이합체 및 기타 과정과 같은 세포 신호 분자에 민감한 유전자 코드 FLET 리포터는 실시간으로 세포 활성의 매우 특정한 광학 판독치를 제공합니다.
다수의 잔류물의 반국가적 돌연변이 유발은 pH 감수성 돌연변이로 알려진 pH 감수성 돌연변이와 이후 초황화 pHluorins로 이어졌다.시냅스 소포 융합 시 pH의 빠른 변화를 이용하여 시냅토브레빈에 태그된 pHluorins는 [27]뉴런의 시냅스 활동을 시각화하는 데 사용되어 왔다.
레독스 감응 GFP(roGFP)는 베타 배럴 구조에 시스테인을 도입하여 설계되었습니다.시스테인의 산화환원 상태는 roGFP의 [28]형광 특성을 결정합니다.
명명법
변경된 GFP의 명명법은 여러 GFP 버전을 하나의 이름으로 매핑하기 때문에 혼동되는 경우가 많습니다.예를 들어 mGFP는 종종 GFP가 세포막에 결합하는 N 말단 팔미토일화를 가진 GFP를 말합니다.단, 같은 용어가 단량체 GFP를 가리키는 경우에도 사용됩니다.단량체 GFP는 대부분의 경우 A206K의 [29]다이머인터페이스가 변환하는 것에 의해서 실현됩니다.야생형 GFP는 5 mg/mL 이상의 농도에서 이합체화 경향이 약하며 mGFP는 식물세포에서 안정적으로 발현하기 위해 아미노산 교환을 통해 최적화한 '변성 GFP'를 의미한다.
자연에서
(1차) 생물발광(aequorin이 루시페린에 작용하여)과 (2차) 해파리에서 GFP의 형광의 목적은 알려져 있지 않다.GFP는 해파리 종 가장자리 주변의 작은 과립에서 에이코린과 함께 발현됩니다.GFP의 2차 들뜸 피크(480 nm)는 에쿼린의 파란색 방출 중 일부를 흡수하여 생물 발광의 녹색을 더 많이 발한다.GFP 발색단의 세린 65 잔기는 야생형 GFP의 이중 피크 들뜸 스펙트럼을 담당한다.원래 Prasher에 의해 복제된 3개의 GFP Isoforms 모두에 보존됩니다.이 잔류물의 거의 모든 돌연변이는 395nm 또는 480nm에서 들뜸 스펙트럼을 단일 피크로 통합한다.이 감도의 정확한 메커니즘은 복잡하지만, 색소 이온화에 [3]영향을 미치는 세린 65에서 글루탐산 222로의 수소 기증을 수반하는 것으로 보인다.단일 돌연변이가 480 nm 들뜸 피크를 극적으로 향상시켜 GFP를 훨씬 더 효율적인 aequorin 파트너로 만들 수 있기 때문에 A. victoria는 진화적으로 덜 효율적인 이중 피크 들뜸 스펙트럼을 선호하는 것으로 보인다.Roger Tsien은 깊이와 함께 변화하는 정수압은 색단에 수소를 공급하고 두 들뜸의 비율을 이동하는 세린 65의 능력에 영향을 미칠 수 있다고 추측했다.따라서, 해파리는 깊이에 따라 생물 발광의 색을 바꿀 수 있다.그러나 GFP가 처음 발견된 금요일 항구 해파리 개체수의 감소는 해파리의 자연환경에서 GFP의 역할에 대한 추가 연구를 방해하고 있다.
대부분의 랜슬렛 종은 [30]몸의 다양한 부분에서 GFP를 생성하는 것으로 알려져 있다.A. victoria와 달리, 랜슬렛은 그들만의 푸른 빛을 내지 않으며, 그들의 내생적인 GFP의 기원은 아직 알려지지 않았다.어떤 사람들은 이것이 플랑크톤을 랜슬렛의 입 쪽으로 끌어들여 수동적인 사냥 메커니즘으로 작용한다고 추측한다.또한 유충의 광보호제 역할을 하여 저강도 청색광으로 변환하여 고강도 청색광에 의한 손상을 방지할 수 있다.그러나 이 이론들은 검증되지 않았다.
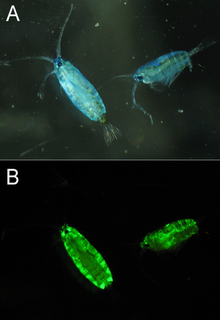
GFP와 유사한 단백질은 여러 종류의 해양 요각류,[31] 특히 폰텔리과와 아이티데과에서 발견되었다.폰텔라 미모세라미에서 분리된 GFP는 0.92의 양자 수율로 높은 밝기를 보여 A.[32] victoria에서 분리된 일반적으로 사용되는 EGFP보다 거의 두 배 밝습니다.
기타 형광단백질
GFP와 같은 단백질군에 속함에도 불구하고 Aequorea victoria에서 직접 유래하지 않는 많은 GFP 유사 단백질이 있습니다.여기에는 dsRed, eqFP611, Dronpa, TagRFP, KFP, EosFP/IrisFP, Dendra 등이 포함됩니다.다른 유기체의 단백질로부터 개발된 이 단백질들은 때때로 발색단 형성에 대한 예상치 못한 접근을 보일 수 있다.이들 중 KFP와 같은 일부는 돌연변이 [33]유발에 의해 크게 개선되는 자연 비 또는 약 형광 단백질로부터 개발된다.스펙트럼 특성이 다른 GFP와 같은 배럴을 사용할 경우, 한 발색단의 들뜸 스펙트럼을 사용하여 다른 발색단(FRET)에 전력을 공급할 수 있어 [34]빛의 파장 간에 변환이 가능하다.
FMN 결합 형광 단백질(FbFPs)은 2007년에 개발되었으며, 청색광 수용체에서 파생된 산소 비의존형광 단백질의 한 종류이다.GFP 발색단 [35]합성과 마찬가지로 플라빈 발색단 형성 및 결합에 분자 산소가 필요하지 않으므로 혐기성 또는 저산소 조건에서 특히 사용하도록 고안되었다.
빌리루빈의 UnaG와 같은 다른 색소체를 가진 형광 단백질은 600nm 이상의 적색 편이 방출 또는 녹색 발광 상태에서 적색 발광 상태로의 광변환과 같은 독특한 특성을 나타낼 수 있다.그들은 붉은 빛과 녹색 빛 사이의 변환을 달성하기에 충분히 멀리 떨어진 들뜸과 방출 파장을 가질 수 있다.
새로운 형광 단백질 클래스는 시아노박테리아(Trichodesmium erithraeum) 피코빌리프로틴, α-allophycocyanin에서 진화하여 2016년에 소형 초적색 형광 단백질(smURFP)로 명명되었다. smURFP는 외부 단백질의 필요 없이 자기 인크루게 된다.[36]Jellyfish-과 coral-derivedGFP-like 단백질과 과산화 수소의 발색단 형성에 화학 량적인 생성된다는 산소를 필요로 하기 때문.[37]smURFP나 과산화 수소를 생산하고 발색단, biliverdin을 사용한다. smURFP고 못은 수수한 양자 수량(0.20), 아주 큰 흡광 계수(18만 M−1 cm−1)이 산소 요구하지 않는다.kes 그것은 생물물리학적 밝기를 eGFP와 비교하고 산호로부터 파생된 대부분의 적색 또는 원적색 형광 단백질보다 약 2배 밝습니다. smURFP 스펙트럼 특성은 유기 염료 Cy5와 [36]유사합니다.
형광 단백질의 새로운 등급과 적용에 대한 리뷰는 인용된 [38][39]리뷰에서 찾을 수 있다.
구조.
GFP는 주름 시트 배열의 11개의 β-스트랜드로 이루어진 베타배럴 구조를 가지며,[3][14][15] 중앙을 관통하는 공유결합 발색단 4-(p-히드록시벤질리덴) 이미다졸리딘-5-온(HBI)을 포함한 알파나선을 가진다.5개의 짧은 알파 나선은 구조의 끝에 캡을 형성합니다.베타 배럴 구조는 거의 완벽한 실린더로, 길이 42Ω, 직경 24Ω([14]일부 연구에서는 직경 30Ω으로[16] 보고됨)으로, GFP [15]계열만의 독특한 "β-can" 형성이 형성되었습니다.트리펩타이드 Ser65–Tyr66–Gly67의 자발적 변형 형태인 HBI는 적절히 접힌 GFP 스켈렛이 없을 때 비형광성이며 주로 wtGFP에서 [40]비이온화 페놀 형태로 존재한다.배럴의 안쪽을 향한 측쇄는 Ser65–Tyr66–Gly67에서 특정 고리화 반응을 유도하여 HBI의 페놀레이트 형태 및 발색단 형성을 유도한다.번역 후 수정 과정을 [41]성숙이라고 합니다.이러한 사이드체인과의 수소 결합 네트워크 및 전자 스택 상호작용은 GFP와 그 수많은 [42]파생물의 색상, 강도 및 광안정성에 영향을 미칩니다.배럴의 촘촘하게 포장된 성질은 용제 분자를 배제하여 물에 의한 발색단 형광을 보호합니다.Ser65-Tyr66-Gly67의 자동 순환에 더해 [16]Tyr66 잔기에서 1,2-탈수소 반응이 일어난다.색소를 형성하는 3개의 잔류물 외에 Gln94, Arg96, His148, Thr203, Glu222 등의 잔류물이 모두 안정제 역할을 한다.Gln94, Arg96, His148의 잔류물은 색소 전하를 디로컬화함으로써 안정시킬 수 있다.Arg96은 HBI 링에서 필요한 구조적 재조정을 촉진하기 때문에 가장 중요한 안정화 잔류물이다.Arg96 잔류물에 대한 돌연변이는 적절한 정전 및 입체 상호작용이 손실되기 때문에 발색단의 발달 속도를 감소시킬 것이다.Tyr66은 수소 결합의 수용체이며 양호한 정전기를 [43]생성하기 위해 이온화되지 않습니다.
wtGFP에서의 발색단 자기촉매 형성
기계적으로, 이 공정은 염기 매개 순환화를 거쳐 탈수 및 산화를 수반한다.7a~8의 반응에서는 이미인으로부터의 에나민의 형성을 수반하는 반면, 7b~9의 반응에서는 양성자가 [44]추상화된다.형성된 HBI 형광체는 녹색으로 강조 표시됩니다.
반응은 잔류물인 Glu222와 Arg96에 [44][45]의해 촉매된다.Ser65 대신 트레오닌을 사용하는 경우에도 유사한 메커니즘이 가능하다.
적용들
리포터 어세스
녹색 형광 단백질을 리포터 [46][47]유전자로 사용해도 좋다.
예를 들어 GFP는 환경독성 수준의 리포터로 사용할 수 있다.이 단백질은 에탄올, p-포름알데히드, 페놀, 트리클로산, 파라벤을 포함한 다양한 화학물질의 독성 수준을 측정하는 효과적인 방법인 것으로 나타났다.GFP는 호스트의 세포 환경에 도입되었을 때 호스트에 영향을 주지 않기 때문에 리포터 단백질로서 매우 좋습니다.이 능력으로 인해 외부 시각화 염색체, ATP 또는 보조 인자가 필요하지 않습니다.오염물질의 농도는 오염물질이 숙주세포에 미치는 영향을 측정하기 위해 형광을 측정하였다.숙주 세포의 세포 밀도도 측정되었다.송·김·서(2016) 연구결과 오염물질 수치가 높아지면서 형광과 세포 밀도가 모두 감소한 것으로 나타났다.이는 세포활동이 감소했음을 나타낸다.GFP가 오염물질 [48]표지자 역할을 하는 메커니즘을 결정하기 위해 이 특정 용도에 대해 더 자세히 조사합니다.GFP를 주입한 제브라피쉬가 GFP를 [49]주입하지 않은 제브라피쉬보다 세포 스트레스를 인지하기 약 20배 더 쉽기 때문에 제브라피쉬에서도 비슷한 결과가 관찰되었습니다.
이점
GFP의 가장 큰 장점은 어떻게 도입되었는지에 따라 유전될 수 있으며, 그것이 발현되는 세포와 조직에 대한 지속적인 연구가 가능하다는 것이다.GFP를 시각화하는 것은 비침습적이어서 파란색 빛으로 조명만 비추면 됩니다.GFP만으로는 생물학적 과정을 방해하지 않지만, 관심 단백질과 융합할 경우 관심 단백질의 기능을 유지하기 위해 링커의 신중한 설계가 필요하다.게다가 단량체와 함께 사용하면 [50]세포 전체에 쉽게 확산될 수 있다.
형광 현미경법
GFP와 그 유도체의 가용성은 형광 현미경과 세포 생물학 및 다른 생물학적 [51]분야에서 사용되는 방법을 완전히 재정의했다.FITC와 같은 대부분의 작은 형광 분자는 살아있는 세포에서 사용될 때 강한 광독성을 가지지만, GFP와 같은 형광 단백질은 살아있는 세포에서 빛나면 훨씬 덜 해롭다.이것은 형광 단백질로 태그된 하나 이상의 단백질을 발현하는 세포를 시간이 지남에 따라 관찰하는데 사용될 수 있는 고도로 자동화된 살아있는 세포 형광 현미경 시스템의 개발을 촉발시켰다.
실시간 세포 이미징 실험에서 GFP를 활용하는 많은 기술이 있습니다.GFP를 이용하는 가장 직접적인 방법은 해당 단백질에 직접 부착하는 것입니다.예를 들어 GFP는 다른 유전자를 발현하는 플라스미드에 포함되어 관심 유전자의 성공적인 전달을 나타낼 수 있다.또 다른 방법은 형광이 시간이 지남에 따라 녹색에서 노란색으로 변하는 변이를 포함하는 GFP를 사용하는 것입니다.이것은 형광 타이머라고 불립니다.형광 타이머를 사용하여, 연구원들은 형광 [52]단백질이 보고한 색을 바탕으로 최근에 활성화, 연속 활성화, 또는 최근에 비활성화된 것과 같은 단백질 생산 상태를 연구할 수 있다.또 다른 예에서 과학자들은 조사 노출 후에야 GFP가 활성화되도록 수정하여 연구자들에게 세포의 특정 부분을 선택적으로 활성화하고 GFP로 태그된 단백질이 시작 [53]위치에서 어디로 이동하는지 관찰할 수 있는 도구를 제공한다.이것들은 형광 마이크로카피의 급증하는 분야에서 단 두 가지 예에 불과하며 GFP와 다른 형광 단백질을 사용하는 바이오센서의 보다 완전한 리뷰는 여기에서 찾을 수 있다.
예를 들어 GFP는 특정 특성의 지표로서 GFP의 발현을 사용할 수 있는 Drosophila melanogaster에서와 같이 식별 목적으로 다양한 생물의 정자 동물에 라벨을 붙이는 데 널리 사용되어 왔다.GFP는 형태학적 구별을 가능하게 하는 다른 구조에서도 발현될 수 있다.이 경우 GFP 생성용 유전자는 표적단백질을 코드하고 동일한 조절배열에 의해 제어되는 DNA 영역의 유기체 게놈에 통합된다.즉, 이 유전자의 조절배열은 태그 부착 단백질 외에 GFP 생성을 제어한다.유전자가 발현되어 태그 부착 단백질이 생성되는 세포에서는 동시에 GFP가 생성된다.따라서, 형광 현미경으로 관찰될 때, 태그가 달린 유전자가 발현되는 세포 또는 표적 단백질이 생성되는 세포만이 형광을 발생시킬 것이다.그러한 시간 경과 영화의 분석은 과거에 고정된(즉, 죽은) 물질을 사용하여 연구되었던 단백질 접힘, 단백질 운반 및 RNA 역학을 포함한 많은 생물학적 과정의 이해를 재정의했다.얻어진 데이터는 또한 세포 내 시스템의 수학적 모델을 교정하고 [55]유전자 발현 속도를 추정하기 위해 사용된다.마찬가지로, GFP는 이종 시스템에서 단백질 발현 지표로 사용될 수 있다.이 시나리오에서 GFP를 포함하는 융합단백질은 간접적으로 또는 태그가 부착된 단백질 자체와 함께 직접 도입된다.이 방법은 형광 현미경으로 고분자 또는 단분자 척도로 태그 부착 단백질의 구조 및 기능적 특성을 연구하는데 유용하다.
SPDM Phymod 기술을 사용하는 Vericoto SMI 현미경은 GFP 및 그 유도체와 같은 형광 염료의 소위 "역광 표백" 효과를 사용하여 10 nm의 광학 분해능에서 단일 분자로 국부화시킵니다.이것은, 2개의 GFP 파생 모델(2CLM)[56]의 공동 현지화로서도 실행할 수 있습니다.
GFP의 또 다른 강력한 사용은 단백질을 특정 세포의 작은 집합에서 발현시키는 것입니다.이것은 연구자들이 특정한 종류의 세포를 광학적으로 시험관내 또는 심지어 생체내에서도 [57]검출할 수 있게 해준다.GFP의 여러 스펙트럼 변형을 유전적으로 결합하는 것은 뇌 회로 분석에 유용한 기술이다.[58]문헌에서 형광 단백질의 다른 흥미로운 사용으로는 FP를 뉴런막 [59]전위의 센서로 사용하는 것,[60] 세포막의 AMPA 수용체 추적, 바이러스 진입 및 개별 인플루엔자 바이러스 및 렌티바이러스 바이러스의 [61][62]감염 등이 있다.
또한 유전자 변형 GFP 쥐의 새로운 라인이 유전자 치료뿐만 아니라 재생 [63]의학에도 관련이 있는 것으로 밝혀졌다.트랜스제닉 래트는 "고발현형" GFP를 사용함으로써 대부분의 조직에서 높은 발현을 보이고, 이전의 GFP 트랜스제닉 래트에서 특징지어지지 않았거나 특징지어지지 않았던 많은 세포들이 나타난다.
GFP는 생존가능성 분석으로서 저온생물학에서 유용한 것으로 나타났다.트리판 블루 어세이에서 측정한 생존성의 상관관계는 0.97이었다.[64]또 다른 적용은 포유동물 [65]세포의 전달 효율을 위한 내부 제어로서 GFP 공감염을 사용하는 것이다.
GFP의 새로운 가능한 사용은 인간 배아 신장 세포주로 이루어진 eGFP 레이저 시스템을 통해 세포 내 프로세스의 민감한 모니터로서 GFP를 사용하는 것을 포함한다.최초의 살아있는 공학적 레이저는 반사광 공동 내부의 세포를 표현하고 푸른 빛의 펄스로 그것을 때리는 eGFP에 의해 만들어집니다.특정 펄스 임계값에서는 eGFP의 광출력이 밝아지고 파장 516nm의 순수 녹색으로 완전히 균일해집니다.이 빛은 레이저광으로 방출되기 전에 공진기 캐비티 내에서 앞뒤로 튀어 나와 셀을 여러 번 통과한다.광학 활동의 변화를 연구함으로써, 연구원들은 세포 [66][67]과정을 더 잘 이해할 수 있을 것이다.
GFP는 암세포에 라벨을 붙이고 추적하기 위해 암 연구에 널리 사용된다.GFP라는 라벨이 붙은 암세포는 암세포가 먼 [68]장기로 전이되는 과정을 모형화하는 데 사용되어 왔다.
GFP 분할
GFP는 단백질의 혈소판정 분석을 위해 사용될 수 있다.이것은 단백질을 자가 조립할 수 있는 두 조각으로 "분할"한 다음, 각각의 단백질을 관심 있는 두 단백질에 결합함으로써 달성된다.이러한 불완전한 GFP fragment만으로는 형광을 발생시킬 수 없습니다.단, 관심 있는 두 개의 단백질이 열원소화되면 두 개의 GFP 조각이 함께 모여 형광을 발생시킬 수 있는 GFP와 같은 구조를 형성한다.따라서 형광 수준을 측정함으로써 관심 있는 두 단백질이 결장하는지 여부를 [69]결정할 수 있다.
매크로 포토그래피
GFP [70]라벨을 사용하여 바이러스 감염 확산과 같은 거시적 생물학적 과정을 따를 수 있습니다.과거에는 돌연변이 유발 자외선(UV)이 GFP 발현을 검출하고 촬영하기 위해 생물(예: 보기[71])을 비추는 데 사용되었습니다.최근에는 비변형 LED[72] 조명을 이용한 매크로 [73]사진 기술이 개발되고 있다.이 기술은 에피 형광 현미경의 구성에 사용된 것과 동일한 원리에 기초한 에피 형광 카메라[74] 부속품을 사용합니다.
트랜스제닉 애완동물
녹색 형광 토끼 알바는 에두아르도 카크가 예술과 사회 [75]해설을 목적으로 GFP를 이용해 프랑스 연구소에 의뢰해 만들었다.미국 Yorktown Technologies사는 처음에는 수로의 오염을 감지하기 위해 개발된 녹색 형광 얼룩말 물고기(GloFish)를 수족관 상점에 판매하고 있습니다.미국에 본사를 둔 NeonPets는 녹색 형광 생쥐를 NeonMice로 [76]애완동물 업계에 판매했다.노엘로 알려진 녹색 형광 돼지는 대만 [77]국립대학교 동물과학기술과의 우신치 박사가 이끄는 연구진에 의해 사육되었다.일본계 미국인 팀은 녹색 형광 고양이를 개념의 증거로 만들어 질병, 특히 HIV의 [78]모델 유기체로 사용할 수 있도록 했다.2009년, 서울대학교의 한국팀은 말미잘에서 나온 섬유아세포로 최초의 유전자 변형 비글을 번식시켰다.이 개들은 붉은 형광등을 발산하며 과학자들이 기면증과 [79]실명과 같은 인간의 질병을 일으키는 유전자를 연구할 수 있도록 하기 위한 것이다.
예체능
독일 태생의 단백질 조각 전문 화가인 [80]줄리안 보스-안드레에는 GFP의 구조를 바탕으로 녹색 형광 단백질(2004년)[81]과 1.40m(2006년)의 강철 해파리(2006년) 등 조형물을 만들었다.후자의 조각상은 1962년 시모무라 워싱턴대 금요항 [82]연구소에 의해 GFP가 발견된 장소에 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Ormö M, Cubitt AB, Kallio K, Gross LA, Tsien RY, Remington SJ (September 1996). "Crystal structure of the Aequorea victoria green fluorescent protein". Science. 273 (5280): 1392–5. Bibcode:1996Sci...273.1392O. doi:10.1126/science.273.5280.1392. PMID 8703075. S2CID 43030290.
- ^ Prendergast FG, Mann KG (Aug 1978). "Chemical and physical properties of aequorin and the green fluorescent protein isolated from Aequorea forskålea". Biochemistry. 17 (17): 3448–53. doi:10.1021/bi00610a004. PMID 28749.
- ^ a b c d Tsien RY (1998). "The green fluorescent protein" (PDF). Annual Review of Biochemistry. 67: 509–44. doi:10.1146/annurev.biochem.67.1.509. PMID 9759496.
- ^ Salih A (2019). "Fluorescent Proteins". In Cox G (ed.). Fundamentals of Fluorescence Imaging. Boca Raton: Jenny Stanford Publishing. p. 122. doi:10.1201/9781351129404. ISBN 9781351129404.
- ^ Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (Aug 2008). "Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes". Current Protein & Peptide Science. 9 (4): 338–69. doi:10.2174/138920308785132668. PMC 2904242. PMID 18691124.
- ^ Phillips GJ (Oct 2001). "Green fluorescent protein--a bright idea for the study of bacterial protein localization". FEMS Microbiology Letters. 204 (1): 9–18. doi:10.1016/S0378-1097(01)00358-5. PMID 11682170.
- ^ Uckert W, Pedersen L, Günzburg W (2000). "Green fluorescent protein retroviral vector: generation of high-titer producer cells and virus supernatant". Methods in Molecular Medicine. 35: 275–85. doi:10.1385/1-59259-086-1:275. ISBN 1-59259-086-1. PMID 21390811.
- ^ Goodman SR, ed. (2008). "Chapter 1 - Tools of the Cell Biologist: Green Fluorescent Protein". Medical Cell Biology (3rd ed.). Amsterdam: Elsevier/Academic Press. pp. 14–25. doi:10.1016/B978-0-12-370458-0.50006-2. ISBN 978-0-12-370458-0.
- ^ Shimomura O, Johnson FH, Saiga Y (Jun 1962). "Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea". Journal of Cellular and Comparative Physiology. 59 (3): 223–39. doi:10.1002/jcp.1030590302. PMID 13911999.
- ^ Morise H, Shimomura O, Johnson FH, Winant J (Jun 1974). "Intermolecular energy transfer in the bioluminescent system of Aequorea". Biochemistry. 13 (12): 2656–62. doi:10.1021/bi00709a028. PMID 4151620.
- ^ Prasher DC, Eckenrode VK, Ward WW, Prendergast FG, Cormier MJ (Feb 1992). "Primary structure of the Aequorea victoria green-fluorescent protein". Gene. 111 (2): 229–33. doi:10.1016/0378-1119(92)90691-H. PMID 1347277.
- ^ Chalfie M, Tu Y, Euskirchen G, Ward WW, Prasher DC (Feb 1994). "Green fluorescent protein as a marker for gene expression". Science. 263 (5148): 802–5. Bibcode:1994Sci...263..802C. doi:10.1126/science.8303295. PMID 8303295. S2CID 9043327.
- ^ Inouye S, Tsuji FI (Mar 1994). "Aequorea green fluorescent protein. Expression of the gene and fluorescence characteristics of the recombinant protein". FEBS Letters. 341 (2–3): 277–80. doi:10.1016/0014-5793(94)80472-9. PMID 8137953.
- ^ a b c Ormö M, Cubitt AB, Kallio K, Gross LA, Tsien RY, Remington SJ (Sep 1996). "Crystal structure of the Aequorea victoria green fluorescent protein". Science. 273 (5280): 1392–5. Bibcode:1996Sci...273.1392O. doi:10.1126/science.273.5280.1392. PMID 8703075. S2CID 43030290.
- ^ a b c Yang F, Moss LG, Phillips GN (Oct 1996). "The molecular structure of green fluorescent protein" (PDF). Nature Biotechnology. 14 (10): 1246–51. doi:10.1038/nbt1096-1246. hdl:1911/19233. PMID 9631087. S2CID 34713931.
- ^ a b c Brejc, K.; Sixma, T. K.; Kits, P. A.; Kain, S. R. Chain, R. Tsien, Ormö, M.; 레밍턴, S. J. 형광색 빅토리아 단백질의 이중 들뜸 및 광이성화를 위한 구조적 기초.검사님, Natl. 학술원, 과학부, 1997, 94(6), 2306-2311.
- ^ Shaner NC, Steinbach PA, Tsien RY (Dec 2005). "A guide to choosing fluorescent proteins" (PDF). Nature Methods. 2 (12): 905–9. doi:10.1038/nmeth819. PMID 16299475. S2CID 10024284.
- ^ Wilhelmsson M, Tor Y (2016). Fluorescent Analogs of Biomolecular Building Blocks: Design and Applications. New Jersey: Wiley. ISBN 978-1-118-17586-6.
- ^ Heim R, Cubitt AB, Tsien RY (Feb 1995). "Improved green fluorescence" (PDF). Nature. 373 (6516): 663–4. Bibcode:1995Natur.373..663H. doi:10.1038/373663b0. PMID 7854443. S2CID 40179694.
- ^ 미국 특허 6172188, Thastrup O, Tullin S, Kongsbak Poulsen L, Björn S, "형광단백질", 2001-01-09 발행
- ^ Cormack BP, Valdivia RH, Falkow S (1996). "FACS-optimized mutants of the green fluorescent protein (GFP)". Gene. 173 (1 Spec No): 33–38. doi:10.1016/0378-1119(95)00685-0. PMID 8707053.
- ^ McRae SR, Brown CL, Bushell GR (May 2005). "Rapid purification of EGFP, EYFP, and ECFP with high yield and purity". Protein Expression and Purification. 41 (1): 121–127. doi:10.1016/j.pep.2004.12.030. PMID 15802229.
- ^ Pédelacq JD, Cabantous S, Tran T, Terwilliger TC, Waldo GS (Jan 2006). "Engineering and characterization of a superfolder green fluorescent protein". Nature Biotechnology. 24 (1): 79–88. doi:10.1038/nbt1172. PMID 16369541. S2CID 2966399.
- ^ Barondeau DP, Kassmann CJ, Tainer JA, Getzoff ED (Apr 2002). "Structural chemistry of a green fluorescent protein Zn biosensor". Journal of the American Chemical Society. 124 (14): 3522–3524. doi:10.1021/ja0176954. PMID 11929238.
- ^ Lelimousin M, Noirclerc-Savoye M, Lazareno-Saez C, Paetzold B, Le Vot S, Chazal R, Macheboeuf P, Field MJ, Bourgeois D, Royant A (Oct 2009). "Intrinsic dynamics in ECFP and Cerulean control fluorescence quantum yield". Biochemistry. 48 (42): 10038–10046. doi:10.1021/bi901093w. PMID 19754158.
- ^ Goedhart J, von Stetten D, Noirclerc-Savoye M, Lelimousin M, Joosen L, Hink MA, van Weeren L, Gadella TW, Royant A (2012). "Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%". Nature Communications. 3: 751. Bibcode:2012NatCo...3..751G. doi:10.1038/ncomms1738. PMC 3316892. PMID 22434194.
- ^ Miesenböck G, De Angelis DA, Rothman JE (Jul 1998). "Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins". Nature. 394 (6689): 192–5. Bibcode:1998Natur.394..192M. doi:10.1038/28190. PMID 9671304. S2CID 4320849.
- ^ Hanson GT, Aggeler R, Oglesbee D, Cannon M, Capaldi RA, Tsien RY, Remington SJ (Mar 2004). "Investigating mitochondrial redox potential with redox-sensitive green fluorescent protein indicators". The Journal of Biological Chemistry. 279 (13): 13044–53. doi:10.1074/jbc.M312846200. PMID 14722062.
- ^ Zacharias DA, Violin JD, Newton AC, Tsien RY (May 2002). "Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells". Science. 296 (5569): 913–16. Bibcode:2002Sci...296..913Z. doi:10.1126/science.1068539. PMID 11988576. S2CID 14957077.
- ^ Yue JX, Holland ND, Holland LZ, Deheyn DD (June 2016). "The evolution of genes encoding for green fluorescent proteins: insights from cephalochordates (amphioxus)". Scientific Reports. 6 (1): 28350. Bibcode:2016NatSR...628350Y. doi:10.1038/srep28350. PMC 4911609. PMID 27311567.
- ^ Hunt ME, Scherrer MP, Ferrari FD, Matz MV (July 2010). "Very bright green fluorescent proteins from the Pontellid copepod Pontella mimocerami". PLOS ONE. 5 (7): e11517. Bibcode:2010PLoSO...511517H. doi:10.1371/journal.pone.0011517. PMC 2904364. PMID 20644720.
- ^ "eGFP". FPbase.
- ^ Chudakov DM, Belousov VV, Zaraisky AG, Novoselov VV, Staroverov DB, Zorov DB, Lukyanov S, Lukyanov KA (February 2003). "Kindling fluorescent proteins for precise in vivo photolabeling". Nature Biotechnology. 21 (2): 191–4. doi:10.1038/nbt778. PMID 12524551. S2CID 52887792.
- ^ Wiens MD, Shen Y, Li X, Salem MA, Smisdom N, Zhang W, Brown A, Campbell RE (December 2016). "A Tandem Green-Red Heterodimeric Fluorescent Protein with High FRET Efficiency". ChemBioChem. 17 (24): 2361–2367. doi:10.1002/cbic.201600492. PMID 27781394. S2CID 4301322.
- ^ Drepper, T., Eggert, T., Circolone, F., Heck, A., Krauss, U., Guterl, J. K., Wendorff, M., Losi, A., Gärtner, W., Jaeger, K. E. (2007). "Reporter proteins for in vivo fluorescence without oxygen". Nat Biotechnol. 25 (4): 443–445. doi:10.1038/nbt1293. PMID 17351616. S2CID 7335755.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ a b Rodriguez EA, Tran GN, Gross LA, Crisp JL, Shu X, Lin JY, Tsien RY (September 2016). "A far-red fluorescent protein evolved from a cyanobacterial phycobiliprotein". Nature Methods. 13 (9): 763–9. doi:10.1038/nmeth.3935. PMC 5007177. PMID 27479328.
- ^ Tsien RY (1998-01-01). "The green fluorescent protein". Annual Review of Biochemistry. 67 (1): 509–44. doi:10.1146/annurev.biochem.67.1.509. PMID 9759496. S2CID 8138960.
- ^ Rodriguez EA, Campbell RE, Lin JY, Lin MZ, Miyawaki A, Palmer AE, Shu X, Zhang J, Tsien RY (February 2017). "The Growing and Glowing Toolbox of Fluorescent and Photoactive Proteins". Trends in Biochemical Sciences. 42 (2): 111–129. doi:10.1016/j.tibs.2016.09.010. PMC 5272834. PMID 27814948.
- ^ Montecinos-Franjola F, Lin JY, Rodriguez EA (2020-11-16). "Fluorescent proteins for in vivo imaging, where's the biliverdin?". Biochemical Society Transactions. 48 (6): 2657–2667. doi:10.1042/BST20200444. PMID 33196077.
- ^ Bokman SH, Ward WW (1982). "Reversible denaturation of Aequorea green-fluorescent protein: physical separation and characterization of the renatured protein". Biochemistry. 21 (19): 4535–4540. doi:10.1021/bi00262a003. PMID 6128025.
- ^ Pouwels LJ, Zhang L, Chan NH, Dorrestein PC, Wachter RM (Sep 2008). "Kinetic isotope effect studies on the de novo rate of chromophore formation in fast- and slow-maturing GFP variants". Biochemistry. 47 (38): 10111–22. doi:10.1021/bi8007164. PMC 2643082. PMID 18759496.
- ^ Chudakov DM, Matz MV, Lukyanov S, Lukyanov KA (Jul 2010). "Fluorescent proteins and their applications in imaging living cells and tissues". Physiological Reviews. 90 (3): 1103–63. doi:10.1152/physrev.00038.2009. PMID 20664080. S2CID 10767597.
- ^ 스테파네코, 버지니아 주, 베르쿠샤, 버지니아 주, 샤블로프스키, 일리노이 주, 쿠즈네초바, 버지니아 주, 우베르스키, 영국 투로베로프녹색 형광 단백질의 구조와 안정성에서 Arg96의 역할을 이해한다.단백질: 구조, 펑트, 바이오인피1999, 73(3), 539-551.
- ^ a b Rosenow, Matthew A.; Huffman, Holly A.; Phail, Marlene E.; Wachter, Rebekka M. (2004-04-01). "The Crystal Structure of the Y66L Variant of Green Fluorescent Protein Supports a Cyclization−Oxidation−Dehydration Mechanism for Chromophore Maturation ,". Biochemistry. 43 (15): 4464–4472. doi:10.1021/bi0361315. ISSN 0006-2960.
- ^ Ma, Yingying; Yu, Jian-Guo; Sun, Qiao; Li, Zhen; Smith, Sean C. (2015-07-01). "The mechanism of dehydration in chromophore maturation of wild-type green fluorescent protein: A theoretical study". Chemical Physics Letters. 631–632: 42–46. doi:10.1016/j.cplett.2015.04.061. ISSN 0009-2614.
- ^ Jugder BE, Welch J, Braidy N, Marquis CP (2016-07-26). "Construction and use of a Cupriavidus necator H16 soluble hydrogenase promoter (PSH) fusion to gfp (green fluorescent protein)". PeerJ. 4: e2269. doi:10.7717/peerj.2269. PMC 4974937. PMID 27547572.
- ^ Arun KH, Kaul CL, Ramarao P (2005). "Green fluorescent proteins in receptor research: an emerging tool for drug discovery". Journal of Pharmacological and Toxicological Methods. 51 (1): 1–23. doi:10.1016/j.vascn.2004.07.006. PMID 15596111.
- ^ Song YH, Kim CS, Seo JH (April 2016). "Noninvasive monitoring of environmental toxicity through green fluorescent protein expressing Escherichia coli". Korean Journal of Chemical Engineering. 33 (4): 1331–6. doi:10.1007/s11814-015-0253-1. S2CID 62828580.
- ^ Pan Y, Leifert A, Graf M, Schiefer F, Thoröe-Boveleth S, Broda J, Halloran MC, Hollert H, Laaf D, Simon U, Jahnen-Dechent W (March 2013). "High-sensitivity real-time analysis of nanoparticle toxicity in green fluorescent protein-expressing zebrafish". Small. Weinheim an Der Bergstrasse, Germany. 9 (6): 863–9. doi:10.1002/smll.201201173. PMID 23143852.
- ^ Chalfie M (Jun 2009). "GFP: Lighting up life". Proceedings of the National Academy of Sciences of the United States of America. 106 (25): 10073–10080. Bibcode:2009PNAS..10610073C. doi:10.1073/pnas.0904061106. PMC 2700921. PMID 19553219.
- ^ Yuste R (Dec 2005). "Fluorescence microscopy today". Nature Methods. 2 (12): 902–4. doi:10.1038/nmeth1205-902. PMID 16299474. S2CID 205418407.
- ^ Terskikh A, Fradkov A, Ermakova G, Zaraisky A, Tan P, Kajava AV, et al. (November 2000). ""Fluorescent timer": protein that changes color with time". Science. 290 (5496): 1585–8. Bibcode:2000Sci...290.1585T. doi:10.1126/science.290.5496.1585. PMID 11090358.
- ^ Patterson GH, Lippincott-Schwartz J (September 2002). "A photoactivatable GFP for selective photolabeling of proteins and cells". Science. 297 (5588): 1873–7. Bibcode:2002Sci...297.1873P. doi:10.1126/science.1074952. PMID 12228718. S2CID 45058411.
- ^ Lin W, Mehta S, Zhang J (October 2019). "Genetically encoded fluorescent biosensors illuminate kinase signaling in cancer". The Journal of Biological Chemistry. 294 (40): 14814–14822. doi:10.1074/jbc.REV119.006177. PMC 6779441. PMID 31434714.
- ^ Komorowski M, Finkenstädt B, Rand D (Jun 2010). "Using a single fluorescent reporter gene to infer half-life of extrinsic noise and other parameters of gene expression". Biophysical Journal. 98 (12): 2759–2769. Bibcode:2010BpJ....98.2759K. doi:10.1016/j.bpj.2010.03.032. PMC 2884236. PMID 20550887.
- ^ Gunkel M, Erdel F, Rippe K, Lemmer P, Kaufmann R, Hörmann C, Amberger R, Cremer C (Jun 2009). "Dual color localization microscopy of cellular nanostructures". Biotechnology Journal. 4 (6): 927–38. doi:10.1002/biot.200900005. PMID 19548231. S2CID 18162278.
- ^ Chudakov DM, Lukyanov S, Lukyanov KA (Dec 2005). "Fluorescent proteins as a toolkit for in vivo imaging". Trends in Biotechnology. 23 (12): 605–13. doi:10.1016/j.tibtech.2005.10.005. PMID 16269193.
- ^ Livet J, Weissman TA, Kang H, Draft RW, Lu J, Bennis RA, Sanes JR, Lichtman JW (Nov 2007). "Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system". Nature. 450 (7166): 56–62. Bibcode:2007Natur.450...56L. doi:10.1038/nature06293. PMID 17972876. S2CID 4402093.
- ^ Baker BJ, Mutoh H, Dimitrov D, Akemann W, Perron A, Iwamoto Y, Jin L, Cohen LB, Isacoff EY, Pieribone VA, Hughes T, Knöpfel T (Aug 2008). "Genetically encoded fluorescent sensors of membrane potential". Brain Cell Biology. 36 (1–4): 53–67. doi:10.1007/s11068-008-9026-7. PMC 2775812. PMID 18679801.
- ^ Adesnik H, Nicoll RA, England PM (Dec 2005). "Photoinactivation of native AMPA receptors reveals their real-time trafficking". Neuron. 48 (6): 977–85. doi:10.1016/j.neuron.2005.11.030. PMID 16364901.
- ^ Lakadamyali M, Rust MJ, Babcock HP, Zhuang X (Aug 2003). "Visualizing infection of individual influenza viruses". Proceedings of the National Academy of Sciences of the United States of America. 100 (16): 9280–5. Bibcode:2003PNAS..100.9280L. doi:10.1073/pnas.0832269100. PMC 170909. PMID 12883000.
- ^ Joo KI, Wang P (Oct 2008). "Visualization of targeted transduction by engineered lentiviral vectors". Gene Therapy. 15 (20): 1384–96. doi:10.1038/gt.2008.87. PMC 2575058. PMID 18480844.
- ^ Remy S, Tesson L, Usal C, Menoret S, Bonnamain V, Nerriere-Daguin V, Rossignol J, Boyer C, Nguyen TH, Naveilhan P, Lescaudron L, Anegon I (Oct 2010). "New lines of GFP transgenic rats relevant for regenerative medicine and gene therapy". Transgenic Research. 19 (5): 745–63. doi:10.1007/s11248-009-9352-2. PMID 20094912. S2CID 42499768.
- ^ Elliott G, McGrath J, Crockett-Torabi E (Jun 2000). "Green fluorescent protein: A novel viability assay for cryobiological applications". Cryobiology. 40 (4): 360–369. doi:10.1006/cryo.2000.2258. PMID 10924267.
- ^ Fakhrudin N, Ladurner A, Atanasov AG, Heiss EH, Baumgartner L, Markt P, Schuster D, Ellmerer EP, Wolber G, Rollinger JM, Stuppner H, Dirsch VM (Apr 2010). "Computer-aided discovery, validation, and mechanistic characterization of novel neolignan activators of peroxisome proliferator-activated receptor gamma". Molecular Pharmacology. 77 (4): 559–66. doi:10.1124/mol.109.062141. PMC 3523390. PMID 20064974.
- ^ Gather MC, Yun SH (2011). "Single-cell biological lasers". Nature Photonics. 5 (7): 406–410. Bibcode:2011NaPho...5..406G. doi:10.1038/nphoton.2011.99. S2CID 54971962.
- ^ Matson J (2011). "Green Fluorescent Protein Makes for Living Lasers". Scientific American. Retrieved 2011-06-13.
- ^ Kouros-Mehr H, Bechis SK, Slorach EM, Littlepage LE, Egeblad M, Ewald AJ, Pai SY, Ho IC, Werb Z (Feb 2008). "GATA-3 links tumor differentiation and dissemination in a luminal breast cancer model". Cancer Cell. 13 (2): 141–52. doi:10.1016/j.ccr.2008.01.011. PMC 2262951. PMID 18242514.
- ^ Cabantous S, Terwilliger TC, Waldo GS (January 2005). "Protein tagging and detection with engineered self-assembling fragments of green fluorescent protein". Nature Biotechnology. 23 (1): 102–7. doi:10.1038/nbt1044. PMID 15580262. S2CID 25833063.
- ^ Rodman MK, Yadav NS, Artus NN (2002-09-01). "Progression of geminivirus-induced transgene silencing is associated with transgene methylation". New Phytologist. 155 (3): 461–468. doi:10.1046/j.1469-8137.2002.00467.x. PMID 33873315.
- ^ Zhu YJ, Agbayani R, Moore PH (Apr 2004). "Green fluorescent protein as a visual selection marker for papaya (Carica papaya L.) transformation". Plant Cell Reports. 22 (9): 660–7. doi:10.1007/s00299-004-0755-5. PMID 14749892. S2CID 23198182.
- ^ Niwa Y, Hirano T, Yoshimoto K, Shimizu M, Kobayashi H (1999). "Non-invasive quantitative detection and applications of non-toxic, S65T-type green fluorescent protein in living plants". The Plant Journal. 18 (4): 455–63. doi:10.1046/j.1365-313X.1999.00464.x. PMID 10406127. S2CID 292648.
- ^ Baker SS, Vidican CB, Cameron DS, Greib HG, Jarocki CC, Setaputri AW, Spicuzza CH, Burr AA, Waqas MA, Tolbert DA (2012-01-01). "An epifluorescent attachment improves whole-plant digital photography of Arabidopsis thaliana expressing red-shifted green fluorescent protein". AoB PLANTS. 2012: pls003. doi:10.1093/aobpla/pls003. PMC 3296078. PMID 22479674.
- ^ "PlantEdDL - Using SRL digital cameras in quantitative investigations of plants expressing green fluorescent protein (GFP)". planted.botany.org. Retrieved 2016-03-23.
- ^ Eduardo Kac. "GFP Bunny".
- ^ "Glow-In-The Dark NeonMice". Archived from the original on February 14, 2009. Retrieved August 30, 2016.
- ^ 대만 과학자들은 형광 녹색 돼지를 기른다.
- ^ Wongsrikeao P, Saenz D, Rinkoski T, Otoi T, Poeschla E (2011). "Antiviral restriction factor transgenesis in the domestic cat". Nature Methods. 8 (10): 853–9. doi:10.1038/nmeth.1703. PMC 4006694. PMID 21909101.
- ^ "Fluorescent puppy is world's first transgenic dog".
- ^ Voss-Andreae J (2005). "Protein Sculptures: Life's Building Blocks Inspire Art". Leonardo. 38: 41–45. doi:10.1162/leon.2005.38.1.41. S2CID 57558522.
- ^ Pawlak A (2005). "Inspirierende Proteine". Physik Journal. 4: 12.
- ^ "Julian Voss-Andreae Sculpture". Retrieved 2007-06-14.
추가 정보
- Pieribone V, Gruber D (2006). Aglow in the Dark: The Revolutionary Science of Biofluorescence. Cambridge: Belknap Press. ISBN 978-0-674-01921-8. OCLC 60321612. GFP의 역사와 발견을 기술한 인기 과학 서적
- Zimmer M (2005). Glowing Genes: A Revolution In Biotechnology. Buffalo, NY: Prometheus Books. ISBN 978-1-59102-253-4. OCLC 56614624.
외부 링크
| 라이브러리 리소스 정보 녹색 형광 단백질 |
- 스콜라피디아 형광단백질 종합기사
- 랜드마크 GFP 논문 요약
- GFP 발색단 형성의 배후에 있는 화학작용을 보여주는 인터랙티브 자바 애플릿
- 2008년 Roger Tsien 형광단백질 강연 동영상
- 다양한 형광 단백질에 대한 들뜸 및 방출 스펙트럼
- 2008년 노벨 화학상 수상자인 시모무라 오사무 교수, 마틴 찰피 교수, 로저 Y 교수에게 바치는 녹색 형광 단백질 화학 Soc Rev 테마호. 첸
- Molecular of the Month, 2003년 6월: David Goodsell의 GFP 개요 그림.
- Molecular of the Month, 2014년 6월: David Goodsell의 GFP 유사 변종 개요 그림.
- 형광단백질 데이터베이스인 FPBase의 녹색 형광단백질
- PDBe-KB의 UniProt: P42212(녹색 형광 단백질)에 대한 PDB의 모든 구조 정보 개요.