LIG1
LIG1| LIG1 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||
| 에일리어스 | LIG1, DNA 리가아제1, LIGI, hLig1, IMD96 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 126391 MGI: 101789 HomoloGene: 197 GeneCard: LIG1 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 맞춤법 | |||||||||||||||||||||||||
| 종. | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레즈 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) | |||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 장소(UCSC) | Chr 19: 48.12 ~48.17 Mb | Chr 7: 13.01 ~13.05 Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키데이터 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
DNA 리가아제 1은 LIG1 유전자에 의해 인간에게 코드되는 효소이다.DNA 연결효소 I은 DNA 복제와 수복에 모두 관여하는 유일한 진핵생물 DNA 연결효소로서, 연결효소 중 가장 많이 연구되었다.
검출
DNA 복제는 이중 DNA 가닥의 파괴를 통해 일어난 것으로 알려졌으나 가닥을 다시 묶는 효소와 작용 메커니즘은 1967년 [5]리먼, 겔러트, 리처드슨, 후르비츠 연구소가 DNA 연결효소를 발견하는 데 큰 기여를 할 때까지 알려지지 않았다.
채용 및 규제
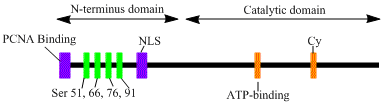
LIG1 유전자는 DNA 연결효소 I로 알려진 120kDa 효소, 길이 919개의 잔류물을 암호화합니다.DNA 리가아제 I 폴리펩타이드는 N-말단복제공장표적배열(RFTS)에 이어 핵국재배열(NLS) 및 3개의 기능 [6]도메인을 포함한다.3개의 도메인은 N 말단 DNA 결합 도메인(DBD)과 촉매 뉴클레오티딜전달효소(NTAase) 및 C 말단 올리고뉴클레오티드/올리고당 결합(OB) 도메인으로 구성된다.펩타이드의 N 말단은 촉매 활성이 없지만 세포 내 활성을 위해 필요하다.단백질의 N 말단에는 복제 팩토리라고 알려진 DNA 복제 사이트에 그것을 모집하는 데 사용되는 복제 팩토리 타깃팅 시퀀스가 포함되어 있습니다.
DNA 연결효소 I의 활성화와 획득은 번역 후 변형과 관련이 있는 것으로 보인다.N 말단 도메인은 이 도메인에서 4개의 세린 잔기 Ser51, Ser76 및 Ser91의 인산화 과정을 통해 사이클린 의존성 키나제(CDK)에 의해, Ser66은 카제인 키나제 II(CKII)에 의해 완성된다.이들 잔류물의 인산화(특히 Ser66)는 S상 동안 [6][7]리게아제 I이 복제 공장으로 모집될 때 RFTS와 증식 세포핵항원(PCNA) 사이의 상호작용을 조절하는 것으로 나타났다.Rossi 등은 Ser66이 탈인산화되면 연결효소 I의 RFTS가 PCNA와 상호작용하며, 이는 Tom 등에 의해 체외에서 확인되었다.두 데이터 세트 모두 결합효소 I의 N 말단 영역이 [7][8]핵에서 생체 내 효소 기능에 조절 역할을 한다는 그럴듯한 증거를 제공한다.또한 촉매 C 말단 도메인에서 사이클린 결합(Cy) 모티브의 식별은 돌연변이 분석을 통해 세린 91, 76의 인산화에서 역할을 하는 것으로 나타났다.N 말단 세린은 CDK와 CKII의 기질로서 세포주기의 [6][9]S상 동안 복제공장으로의 중요한 조절역할 DNA 연결효소 I 신병을 수행하는 것으로 보인다.
기능 및 메커니즘
LIG1은 DNA 복제 [10]및 염기 절제 복구 과정에서 기능하는 DNA 연결효소 I을 암호화합니다.
진핵생물 DNA 연결효소 1은 모든 연결효소에 화학적으로 보편적인 반응을 촉매한다.DNA 연결효소 1은 아데노신3인산(ATP)을 이용하여 DNA 복제 및 복구 모두에서 에너지적으로 유리한 결찰 이벤트를 촉매한다.진핵세포주기의 합성단계(S상)에서 DNA복제가 일어난다.DNA리가아제1은 DNA중합효소γ가 RNA프라이머뉴클레오티드를 DNA뉴클레오티드로 치환한 후 DNA의 후행성 가닥에 불연속 DNA합성 중에 형성된 오카자키 단편을 결합하는 역할을 한다.만약 오카자키 조각들이 제대로 결합되지 않는다면, 결합되지 않은 DNA ('닉'을 포함한)는 유전자 변이를 일으키는 것으로 알려진 현상인 이중 가닥 파괴로 쉽게 분해될 수 있다.이러한 조각들을 함께 결합하기 위해 연결효소는 세 가지 단계를 거칩니다.
아데닐화 중 촉매 리신으로부터 ATP의 알파인산에 대한 친핵성 공격이 있어 DNA 연결효소 1의 활성 부위에서 무기 피로인산(PPi)과 공유 결합 리신-AMP 중간체가 생성된다.
AMP 전달 단계에서 DNA 연결효소는 DNA와 관련지어 니크를 찾아 DNA 니크의 5' 인산염 부위에서 반응을 촉매한다.DNA 닉의 5'인산 위의 음이온산소는 친핵체로서 기능하며, AMP가 공유결합 중간체(DNA-AMP 중간체)로 결합되는 원인이 되는 공유결합 AMP의 알파인산을 공격한다.
포스포디에스테르 결합을 형성하기 위해서는 DNA-AMP 중간체를 절단해야 한다.이 과제를 달성하기 위해 업스트림 3'-히드록실로부터 5'-인산에 대한 친핵성 공격이 있으며, 이는 포스포디에스터 결합을 형성한다.이 친핵성 공격 중에 AMP기를 5'인산염에서 밀어내 Nick을 봉합하고 AMP를 방출함으로써 1사이클의 DNA 결찰을 완료한다.
최적 상태가 아닌 조건에서는 완전한 반응이 완료되기 전에 연결효소가 DNA에서 분리될 수 있다.마그네슘 수치는 닉 봉합 과정을 늦출 수 있고, 연결효소가 DNA에서 분리되도록 하여, 중단된 아데닐화 중간체는 포스포디에스테라아제의 도움 없이 고정될 수 없게 만들 수 있는 것으로 나타났다.아파탁신(포스포디에스테라아제)은 AMP-인산 결합의 가수분해를 통해 중단된 DNA 중간체에 작용하여 연결효소가 [12][13]반응하기 전의 초기 상태로 DNA를 복원하는 것으로 나타났다.
파손된 베이스 수리의 역할
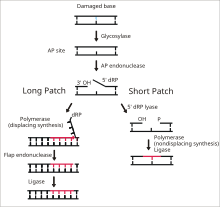
DNA 연결효소 I은 염기절제 복구([14]BER) 경로의 마지막 단계에서 단일 가닥 DNA 파괴를 결합하는 기능을 한다.DNA의 질소 염기는 일반적으로 활성 산소종, 독소, 이온화 방사선과 같은 환경적 위험에 의해 손상됩니다.BER은 손상된 염기를 제거하고 교체하는 주요 수리 경로입니다.리가아제 I은 LP-BER 경로에 관여하는 반면, 리가아제 III는 주요 SN-BER 경로(2)[15]에 관여한다.LP-BER는 4개의 촉매 단계로 진행됩니다.첫째, DNA 글리코실라아제는 N-글리코시드 결합을 분해하여 손상된 염기를 방출하고 퓨린 또는 피리미딘 염기가 없는 부위인 AP 부위를 만듭니다.다음 단계에서 AP 엔도핵산가수분해효소는 AP 부위의 5' 말단에 니크를 생성하여 AP 부위 대신 매달린 디옥시리보스 인산(dRP) 잔기를 생성한다.DNA 중합효소는 5'에서 3' 방향으로 여러 개의 새로운 염기를 합성하여 dRP를 5' 끝에 두고 늘어뜨린 DNA를 생성한다.이 단계에서 SN-BER와 LP-BER는 메커니즘에서 분기합니다. SNBER에서는 단일 뉴클레오티드만 첨가되고 DNA 중합효소는 AP 부위를 제거하기 위한 리아제 역할을 합니다.LP-BER에서는 여러 염기가 합성되어 플랩 엔도핵산가수분해효소에 의해 절단되는 DNA의 리프팅 플랩을 생성한다.이것은 DNA 연결효소에 [14][15][16]의해 감지되고 연결된 절단된 DNA 가닥을 남긴다.리가아제 I의 작용은 다른 LP-BER 효소, 특히 AP-엔도핵산가수분해효소 [16]및 DNA 중합효소에 의해 자극된다.
임상적 의의
DNA 연결효소 I 결핍을 초래하는 LIG1의 돌연변이는 면역결핍을 초래하고 DNA 손상제에 [10]대한 민감도를 증가시킨다.
유전된 돌연변이 대립 유전자에 기인한 리가아제 I 결핍증을 보이는 환자에 대한 드문 보고가 있다.첫 번째 사례는 성장과 발육의 저하와 면역 결핍으로 나타난다.환자로부터 파생된 세포주를 기반으로 마우스 모델이 만들어졌으며, 돌연변이 연결 효소가 게놈 불안정성을 초래하는 복제 오류를 유발한다는 것을 확인했습니다.특히 돌연변이 생쥐들은 종양 [8]발생의 증가를 보였다.바이알릴 돌연변이를 가진 3개 혈연종 환자 5명의 분자, 세포 및 임상 특징이 보고되었다.환자는 저감마글로불린혈증, 림프감소증, 순환하는 δT세포의 비율 증가, 매우 큰 적혈구(대형세포증)를 보였다.임상적 심각도는 경미한 항체 결핍에서 조혈모세포 이식이 필요한 복합 면역 결핍까지 다양했다.화학 및 방사선 결함은 DNA 복구 경로를 손상시키는 것으로 입증되었다.따라서 DNA 연결효소 1의 결함은 상염색체 열성, 부분 DNA 연결효소 1 결핍의 다른 형태를 초래할 수 있으며,[17] 이는 다양한 심각도의 면역 결핍을 초래할 수 있다.
리가아제 I은 또한 양성 종양 세포주나 정상적인 인간 세포와는 달리 종양 세포 증식에서 높은 조절을 받는 것으로 밝혀졌다.또한 이들 세포에서 리가아제I 발현을 억제하는 것이 세포독성 효과를 가져올 수 있다는 것이 밝혀져 리가아제I 억제제가 실행 가능한 화학요법제일 [18]수 있음을 시사한다.
(DNA 연결효소 I이 아데닐화 DNA 중간생성물을 중단한 후) DNA 재컨디셔닝에 관여하는 포스포디에스테라아제인 아파탁신의 결핍은 신경변성과 관련이 있다.이것은 DNA가 연결효소 [13]오류를 교정하기 위한 추가적인 백업 기계 없이는 복구 경로로 재진입할 수 없음을 시사한다.
DNA의 구조가 잘 알려져 있고 그것의 조작, 수리, 사용에 필요한 많은 성분들이 확인되고 특성화 되면서, 연구원들은 질병을 치료하고 암을 퇴치할 능력을 가진 살아있는 유기체에 통합될 나노스코프 기계의 개발을 조사하기 시작했다.그리고 유기체가 나노소픽 기계에 제공하는 생물학적 자극에 기초한 약물을 방출한다.DNA 연결효소는 아마도 그러한 [19]기계에 통합되어야 할 것이다.
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG00000105486 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG000056394 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Kresge N, Simoni RD, Hill RL (January 2007). "Insights into DNA Joining: I. Robert Lehman's work on DNA Ligase". Journal of Biological Chemistry. 282 (2): e1–e3. doi:10.1016/S0021-9258(20)73504-0.
- ^ a b c Ferrari G, Rossi R, Arosio D, Vindigni A, Biamonti G, Montecucco A (September 2003). "Cell cycle-dependent phosphorylation of human DNA ligase I at the cyclin-dependent kinase sites". The Journal of Biological Chemistry. 278 (39): 37761–7. doi:10.1074/jbc.M304462200. PMID 12851383.
- ^ a b Rossi R, Villa A, Negri C, Scovassi I, Ciarrocchi G, Biamonti G, Montecucco A (October 1999). "The replication factory targeting sequence/PCNA-binding site is required in G(1) to control the phosphorylation status of DNA ligase I". The EMBO Journal. 18 (20): 5745–54. doi:10.1093/emboj/18.20.5745. PMC 1171641. PMID 10523317.
- ^ a b c Ellenberger T, Tomkinson AE (2008). "Eukaryotic DNA ligases: structural and functional insights". Annual Review of Biochemistry. 77: 313–38. doi:10.1146/annurev.biochem.77.061306.123941. PMC 2933818. PMID 18518823.
- ^ Prigent C, Lasko DD, Kodama K, Woodgett JR, Lindahl T (August 1992). "Activation of mammalian DNA ligase I through phosphorylation by casein kinase II". The EMBO Journal. 11 (8): 2925–33. doi:10.1002/j.1460-2075.1992.tb05362.x. PMC 556774. PMID 1639065.
- ^ a b "Entrez Gene: LIG1 ligase I, DNA, ATP-dependent".
- ^ Sriskanda V, Shuman S (January 1998). "Chlorella virus DNA ligase: nick recognition and mutational analysis". Nucleic Acids Research. 26 (2): 525–31. doi:10.1093/nar/26.2.525. PMC 147278. PMID 9421510.
- ^ Taylor MR, Conrad JA, Wahl D, O'Brien PJ (July 2011). "Kinetic mechanism of human DNA ligase I reveals magnesium-dependent changes in the rate-limiting step that compromise ligation efficiency". The Journal of Biological Chemistry. 286 (26): 23054–62. doi:10.1074/jbc.M111.248831. PMC 3123073. PMID 21561855.
- ^ a b Rass U, Ahel I, West SC (March 2007). "Actions of aprataxin in multiple DNA repair pathways". The Journal of Biological Chemistry. 282 (13): 9469–74. doi:10.1074/jbc.M611489200. PMID 17276982.
- ^ a b Sattler U, Frit P, Salles B, Calsou P (April 2003). "Long-patch DNA repair synthesis during base excision repair in mammalian cells". EMBO Reports. 4 (4): 363–7. doi:10.1038/sj.embor.embor796. PMC 1319152. PMID 12671676.
- ^ a b Hegde ML, Hazra TK, Mitra S (January 2008). "Early steps in the DNA base excision/single-strand interruption repair pathway in mammalian cells". Cell Research. 18 (1): 27–47. doi:10.1038/cr.2008.8. PMC 2692221. PMID 18166975.
- ^ a b Balakrishnan L, Brandt PD, Lindsey-Boltz LA, Sancar A, Bambara RA (May 2009). "Long patch base excision repair proceeds via coordinated stimulation of the multienzyme DNA repair complex". The Journal of Biological Chemistry. 284 (22): 15158–72. doi:10.1074/jbc.M109.000505. PMC 2685697. PMID 19329425.
- ^ Maffucci P, Chavez J, Jurkiw TJ, O'Brien PJ, Abbott JK, Reynolds PR, et al. (December 2018). "Biallelic mutations in DNA ligase 1 underlie a spectrum of immune deficiencies". The Journal of Clinical Investigation. 128 (12): 5489–5504. doi:10.1172/JCI99629. PMC 6264644. PMID 30395541.
- ^ Sun D, Urrabaz R, Nguyen M, Marty J, Stringer S, Cruz E, et al. (December 2001). "Elevated expression of DNA ligase I in human cancers". Clinical Cancer Research. 7 (12): 4143–8. PMID 11751514.
- ^ Macdonald J. "Smart DNA: Programming the Molecule of Life for Work and Play [Preview]". scientificamerican. Retrieved 2013-02-22.
추가 정보
- Leonhardt H, Cardoso MC (1996). "Targeting and association of proteins with functional domains in the nucleus: the insoluble solution". Structural and Functional Organization of the Nuclear Matrix. International Review of Cytology. Vol. 162B. pp. 303–35. doi:10.1016/S0074-7696(08)62620-0. ISBN 9780123645661. PMID 8557490.
- Tomkinson AE, Mackey ZB (February 1998). "Structure and function of mammalian DNA ligases". Mutation Research. 407 (1): 1–9. doi:10.1016/s0921-8777(97)00050-5. PMID 9539976.
- Perrigot M, Pierrot-Deseilligny E, Bussel B, Held JP (April 1976). "[Paralysis following Dimer X radiculography]". La Nouvelle Presse Médicale. 5 (17): 1120–2. PMID 934827.
- Webster AD, Barnes DE, Arlett CF, Lehmann AR, Lindahl T (June 1992). "Growth retardation and immunodeficiency in a patient with mutations in the DNA ligase I gene". Lancet. 339 (8808): 1508–9. doi:10.1016/0140-6736(92)91266-B. PMID 1351188. S2CID 11874717.
- Barnes DE, Tomkinson AE, Lehmann AR, Webster AD, Lindahl T (May 1992). "Mutations in the DNA ligase I gene of an individual with immunodeficiencies and cellular hypersensitivity to DNA-damaging agents". Cell. 69 (3): 495–503. doi:10.1016/0092-8674(92)90450-Q. PMID 1581963. S2CID 11736507.
- Barnes DE, Kodama K, Tynan K, Trask BJ, Christensen M, De Jong PJ, et al. (January 1992). "Assignment of the gene encoding DNA ligase I to human chromosome 19q13.2-13.3". Genomics. 12 (1): 164–6. doi:10.1016/0888-7543(92)90422-O. PMID 1733856.
- Petrini JH, Huwiler KG, Weaver DT (September 1991). "A wild-type DNA ligase I gene is expressed in Bloom's syndrome cells". Proceedings of the National Academy of Sciences of the United States of America. 88 (17): 7615–9. Bibcode:1991PNAS...88.7615P. doi:10.1073/pnas.88.17.7615. PMC 52352. PMID 1881902.
- Lasko DD, Tomkinson AE, Lindahl T (July 1990). "Mammalian DNA ligases. Biosynthesis and intracellular localization of DNA ligase I". The Journal of Biological Chemistry. 265 (21): 12618–22. doi:10.1016/S0021-9258(19)38388-7. PMID 2197279.
- Barnes DE, Johnston LH, Kodama K, Tomkinson AE, Lasko DD, Lindahl T (September 1990). "Human DNA ligase I cDNA: cloning and functional expression in Saccharomyces cerevisiae". Proceedings of the National Academy of Sciences of the United States of America. 87 (17): 6679–83. Bibcode:1990PNAS...87.6679B. doi:10.1073/pnas.87.17.6679. PMC 54600. PMID 2204063.
- Montecucco A, Savini E, Weighardt F, Rossi R, Ciarrocchi G, Villa A, Biamonti G (November 1995). "The N-terminal domain of human DNA ligase I contains the nuclear localization signal and directs the enzyme to sites of DNA replication". The EMBO Journal. 14 (21): 5379–86. doi:10.1002/j.1460-2075.1995.tb00222.x. PMC 394647. PMID 7489727.
- Maruyama K, Sugano S (January 1994). "Oligo-capping: a simple method to replace the cap structure of eukaryotic mRNAs with oligoribonucleotides". Gene. 138 (1–2): 171–4. doi:10.1016/0378-1119(94)90802-8. PMID 8125298.
- Trask B, Fertitta A, Christensen M, Youngblom J, Bergmann A, Copeland A, et al. (January 1993). "Fluorescence in situ hybridization mapping of human chromosome 19: cytogenetic band location of 540 cosmids and 70 genes or DNA markers". Genomics. 15 (1): 133–45. doi:10.1006/geno.1993.1021. PMID 8432525.
- Petrini JH, Walsh ME, DiMare C, Chen XN, Korenberg JR, Weaver DT (September 1995). "Isolation and characterization of the human MRE11 homologue". Genomics. 29 (1): 80–6. doi:10.1006/geno.1995.1217. PMID 8530104.
- Bentley D, Selfridge J, Millar JK, Samuel K, Hole N, Ansell JD, Melton DW (August 1996). "DNA ligase I is required for fetal liver erythropoiesis but is not essential for mammalian cell viability". Nature Genetics. 13 (4): 489–91. doi:10.1038/ng0896-489. PMID 8696349. S2CID 20264173.
- Suzuki Y, Yoshitomo-Nakagawa K, Maruyama K, Suyama A, Sugano S (October 1997). "Construction and characterization of a full length-enriched and a 5'-end-enriched cDNA library". Gene. 200 (1–2): 149–56. doi:10.1016/S0378-1119(97)00411-3. PMID 9373149.
- Rossi R, Villa A, Negri C, Scovassi I, Ciarrocchi G, Biamonti G, Montecucco A (October 1999). "The replication factory targeting sequence/PCNA-binding site is required in G(1) to control the phosphorylation status of DNA ligase I". The EMBO Journal. 18 (20): 5745–54. doi:10.1093/emboj/18.20.5745. PMC 1171641. PMID 10523317.
- Matsumoto Y, Kim K, Hurwitz J, Gary R, Levin DS, Tomkinson AE, Park MS (November 1999). "Reconstitution of proliferating cell nuclear antigen-dependent repair of apurinic/apyrimidinic sites with purified human proteins". The Journal of Biological Chemistry. 274 (47): 33703–8. doi:10.1074/jbc.274.47.33703. PMID 10559261.
- Vispé S, Satoh MS (September 2000). "DNA repair patch-mediated double strand DNA break formation in human cells". The Journal of Biological Chemistry. 275 (35): 27386–92. doi:10.1074/jbc.M003126200. PMID 10827190.