뉴클레오솜
Nucleosome
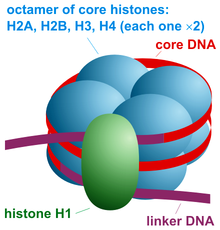
뉴클레오솜은 진핵생물에서 DNA 패키징의 기본 구조 단위이다.뉴클레오좀의 구조는 8개의 히스톤[1] 단백질에 감긴 DNA의 부분으로 구성되며 스풀에 감긴 실과 유사합니다.뉴클레오솜은 염색질의 기본 서브 유닛이다.각각의 뉴클레오솜은 히스톤이라고 불리는 여덟 개의 단백질 세트를 감싸고 있는 두 바퀴 미만의 DNA로 구성되어 있는데, 이것은 히스톤 옥타머로 알려져 있다.각 히스톤 옥타머는 히스톤 단백질 H2A, H2B, H3, H4의 2개의 복사본으로 구성된다.
DNA는 세포핵에 [2]맞도록 뉴클레오솜으로 압축되어야 한다.뉴클레오솜 포장 외에 진핵생물 크로마틴은 일련의 보다 복잡한 구조로 접혀져 최종적으로 염색체를 형성함으로써 더욱 압축된다.각각의 인간 세포는 약 3천만 개의 뉴클레오솜을 [3]가지고 있다.
뉴클레오솜은 핵심 히스톤의 공유 변형의 형태로 후생적으로 물려받은 정보를 가지고 있는 것으로 생각된다.게놈의 뉴클레오솜 위치는 무작위가 아니며, 이것이 조절 [4]단백질에 대한 DNA의 접근성을 결정하기 때문에 각 뉴클레오솜이 어디에 위치하는지 아는 것이 중요하다.
뉴클레오솜은 1974년 [5]돈 올린스와 에이다 올린스에 의해 전자 현미경의 입자로 처음 관찰되었고, 그들의 존재와 구조(약 200개의 염기쌍의 DNA로 둘러싸인 히스톤 옥타머)는 로저 콘버그에 [6][7]의해 제안되었다.전사의 조절제로서의 뉴클레오솜의 역할은 1987년 Lorch 등에 의해 시험관내[8], 1988년 Han과[9] Grunstein, Clark-Adams [10]등에 의해 입증되었다.
뉴클레오솜 코어 입자는 히스톤 옥타머를 중심으로 약 146개의 염기쌍(bp)의 DNA로[11] 구성되어 있으며, 각각 2개의 코어 히스톤 H2A, H2B, H3, [12]H4로 구성됩니다.핵심 입자는 링커 DNA의 연장선에 의해 연결되며, 길이는 약 80bp까지 될 수 있습니다.기술적으로 뉴클레오솜은 코어 입자와 이러한 링커 영역 중 하나로 정의됩니다. 그러나 이 단어는 종종 코어 [13]입자와 동의어입니다.게놈 전체 뉴클레오솜 위치 지도는 현재 쥐의 간과 [14]뇌를 포함한 많은 모델 유기체에 사용할 수 있습니다.
H1과 같은 링커히스톤과 그 등소형은 염색질 압축에 관여하며 DNA의 [15]링커 영역에 대한 DNA 입출구 결합 부근에 있는 뉴클레오좀의 밑부분에 위치한다.링커히스톤이 없는 비응축뉴클레오솜은 전자현미경 [16]하에서 "DNA 줄 위의 비드"와 유사하다.
대부분의 진핵 세포와 대조적으로, 성숙한 정자 세포는 그들의 게놈 DNA를 포장하기 위해 프로타민을 주로 사용하며, 훨씬 더 높은 포장 [17]비율을 달성할 가능성이 높다.히스톤 당량과 단순화된 염색질 구조 또한 고고학에서 [18]발견되었는데, 이는 진핵생물만이 뉴클레오솜을 사용하는 유기체가 아니라는 것을 암시한다.
구조.
코어 입자의 구조
개요
1980년대 Aaron Klug의 그룹에 의한 선구적인 구조 연구는 히스톤 단백질의 옥타머가 왼손 [19]초나선의 약 1.7 회전으로 DNA를 감싼다는 첫 번째 증거를 제공했다.1997년에 뉴클레오좀의 원자 분해능에 가까운 첫 결정 구조가 리치몬드 그룹에 의해 해결되어 입자의 가장 중요한 세부 사항을 보여주었다.1997년 뉴클레오솜 결정 구조를 달성하는 데 중요한 인간 알파 위성 회문 DNA는 [20][21][22][23][24]테네시 오크리지 국립 연구소의 버닉 그룹에 의해 개발되었다.20개 이상의 다른 핵소체 핵심 입자의 구조는 지금까지 [25]해결되었으며, 여기에는 다양한 종의 히스톤 변종과 히스톤이 포함되어 있습니다.뉴클레오솜 코어 입자의 구조는 현저하게 보존되어 있으며 개구리와 효모 히스톤 사이의 잔류물이 100개 이상 변화해도 전체 루트 평균 제곱 편차가 1.6Ω에 [26]불과한 전자 밀도 맵이 생성된다.
뉴클레오솜 코어 입자(NCP)
뉴클레오솜 코어 입자(그림 참조)는 히스톤 옥타머를 중심으로 약 1.67개의 초나선 회전에 싸인 약 146개의 염기쌍의[11] DNA로 구성되어 있으며, 코어 히스톤 H2A, H2B, H3, H4 각각 2개의 복사본으로 구성되어 있다.인접한 뉴클레오솜은 링커 DNA라고 불리는 자유 DNA의 연장선에 의해 결합된다. (종과 조직 유형에[18] 따라 길이가 10 - 80 bp이다.)전체 구조는 직경 11nm, 높이 5.5nm의 실린더를 생성합니다.
중간상 염색질을 처리하여 염색질을 부분적으로 펼쳤을 때 뉴클레오솜 코어 입자가 관찰된다.전자 현미경을 통해 생성된 이미지는 "줄에 박힌 구슬"입니다.실은 DNA이고, 뉴클레오솜의 각 구슬은 핵심 입자입니다.뉴클레오솜 핵심 입자는 DNA와 히스톤 [29]단백질로 구성되어 있다.
염색질의 부분적인 DNAse 소화는 염색질의 뉴클레오솜 구조를 드러낸다.뉴클레오솜 코어 입자의 DNA 부분은 연결 부분보다 DNAe에 접근하기 어렵기 때문에, DNA는 뉴클레오솜 사이의 거리의 다수와 같은 길이의 조각으로 소화된다(180, 360, 540 염기쌍 등).따라서 [27]그 DNA의 겔 전기영동 중에 사다리와 유사한 매우 특징적인 패턴을 볼 수 있다.이러한 소화는 또한 아포토시스("세포 자살" 또는 프로그램된 세포사망") 동안 자연 조건에서도 일어날 수 있는데, 이는 일반적으로 DNA의 자동 파괴가 그 역할이기 때문이다.
뉴클레오솜 내 단백질 상호작용
핵심 히스톤 단백질은 두 개의 루프(L1-2)로 분리된 세 개의 알파 헬리(α1-3)로 구성된 "히스톤 폴드"라고 불리는 특징적인 구조적 모티브를 포함한다.히스톤은 H2A-H2B 헤테로디머 및 H3-H4 헤테로테트라미터를 형성한다.히스톤은 긴 α2나선을 반평행 방향으로 이합화하며, H3 및 H4의 경우, 그러한 2개의 이합체가 광범위한 H3-H3' 상호작용에 의해 안정화 된 4-나선다발을 형성한다.H2A/H2B 이합체는 소수성 클러스터의 형성을 포함한 [12]H4와 H2B 사이의 상호작용으로 인해 H3/H4 사량체에 결합합니다.히스톤 옥타머는 두 개의 H2A/H2B 이합체 사이에 끼인 중앙 H3/H4 4중합체에 의해 형성됩니다.히스톤 옥타머는 4개의 코어 히스톤의 높은 염기성 전하로 인해 DNA 또는 매우 높은 염분 농도가 존재하는 경우에만 안정적입니다.
히스톤 - DNA 상호작용
뉴클레오솜은 120개 이상의 직접 단백질-DNA 상호작용과 수백 개의 물 매개 상호작용을 포함합니다.[30]직접 단백질 - DNA 상호작용은 옥타머 표면 주위에 고르게 분포되지 않고 개별 부위에 위치한다.이는 옥타머 내에서 두 가지 유형의 DNA 결합 부위가 형성되기 때문이다. 즉, 인접한 두 히스톤의 α1 나선을 사용하는 α1 부위와 L1 및 L2 루프로 형성된 L1L2 부위가 그것이다.측쇄 염기기와 수산기 사이의 염결합과 수소결합 그리고 DNA 백본 인산염과의 주쇄 아미드 사이의 염결합은 DNA와의 상호작용의 대부분을 형성한다.게놈을 따라 뉴클레오좀의 유비쿼터스 분포가 비배열 특이적 DNA 결합 인자를 필요로 한다는 것을 고려하면 이것은 중요하다.비록 뉴클레오솜이 다른 [31]DNA 배열보다 몇몇 DNA 배열들을 선호하는 경향이 있지만, 그것들은 실질적으로 어떤 배열에도 결합할 수 있고, 이것은 이러한 물 매개 상호작용의 형성에서의 유연성 때문인 것으로 생각된다.또한 단백질 측쇄와 디옥시리보스기 사이에 무극성 상호작용이 이루어지며, 옥타머 표면에 면하는 14개소 모두에서 아르기닌 측쇄가 DNA 소구내에 인터클레이션된다.옥타머 표면 주변의 DNA 결합 부위의 분포와 강도는 뉴클레오솜 코어 내의 DNA를 왜곡시킨다.DNA는 균일하지 않게 구부러져 있으며 비틀림 결함도 포함되어 있습니다.용액에서 자유 B형 DNA의 꼬임은 1회당 10.5bp이다.그러나 뉴클레오솜 DNA의 전체 꼬임은 턴당 10.2bp에 불과하며, 턴당 9.4~10.9bp의 값에서 변화한다.
히스톤 테일 도메인
히스톤 꼬리 확장은 히스톤 질량에 의해 최대 30%를 구성하지만, 높은 고유 유연성 때문에 뉴클레오솜의 결정 구조에서는 보이지 않으며,[32] 대부분 구조화되지 않은 것으로 여겨져 왔다.히스톤 H3와 H2B의 N 말단 꼬리는 20bp마다 DNA에서 돌출된 두 DNA 가닥의 작은 홈에 의해 형성된 채널을 통과합니다.한편 히스톤 H4의 N말단 꼬리는 결정 구조에서 다른 뉴클레오솜의 H2A-H2B 이합체의 고산성 표면 영역과 상호작용을 형성하여 뉴클레오솜의 고차 구조와 잠재적으로 관련이 있는 고염기 아미노산 영역(16~25)을 가지고 있다.이 상호작용은 생리적인 조건에서도 일어나는 것으로 생각되며, H4 꼬리의 아세틸화가 염색질의 고차 구조를 왜곡시킨다는 것을 시사한다.
고차 구조
뉴클레오솜에 의해 달성된 DNA의 구성은 세포핵에서 관찰된 DNA의 포장을 완전히 설명할 수 없다.세포핵으로의 염색질의 추가 압축이 필요하지만, 아직 잘 이해되지 않았다.현재의 이해는[25] 개입된 "링커" DNA와 함께 뉴클레오솜을 반복하는 것이 "줄 위의 구슬"로 묘사되는 10nm 섬유를 형성하고 약 5에서 [18]10의 패킹 비율을 갖는다는 것이다.뉴클레오솜 사슬은 30nm 섬유 내에 배치할 수 있으며, 패킹비는 50[18] 이하이며, 형성은 H1 히스톤의 존재에 따라 결정된다.
테트라뉴클레오좀의 결정구조를 제시하여 30nm 섬유의 제안된 구조를 2-start 나선으로 [33]구축하기 위해 사용한다.이 모델은 최근의 전자 현미경 검사 [34]데이터와 호환되지 않기 때문에 여전히 어느 정도 논란이 있습니다.이 밖에도 염색질의 구조는 잘 알려져 있지 않지만, 30 nm 섬유는 중심 단백질 골격을 따라 루프로 배열되어 전사 활성 에크로마틴을 형성하는 것이 고전적으로 제안되고 있다.추가적인 압박은 전사적으로 비활성 헤테로크로마틴으로 이어진다.
다이내믹스
뉴클레오솜은 매우 안정적인 단백질-DNA 복합체이지만 정적이지 않고 뉴클레오솜 슬라이딩 및 DNA 부위 노출을 포함한 여러 가지 다른 구조적 재배열을 거치는 것으로 나타났다.상황에 따라 뉴클레오솜은 전사인자 결합을 억제하거나 촉진할 수 있다.뉴클레오솜 위치는 세 가지 주요 기여에 의해 제어됩니다.첫째, 히스톤 옥타머의 내인성 결합 친화성은 DNA 배열에 따라 달라진다.둘째, 뉴클레오솜은 다른 단백질 인자의 경쟁적 또는 협동적 결합에 의해 치환되거나 획득될 수 있다.셋째, 뉴클레오좀은 ATP의존성 리모델링 [35]복합체에 의해 능동적으로 전이될 수 있다.
뉴클레오솜 슬라이딩
브래드베리 실험실에서 수행된 연구는 5S DNA 위치 결정 배열에 재구성된 뉴클레오솜이 [36]열적으로 배양되었을 때 인접한 배열에 번역적으로 재배치될 수 있다는 것을 보여주었다.이후 연구 결과, 이 위치 변경은 히스톤 옥타머의 교란을 필요로 하지 않았지만 시스에서 DNA를 따라 "슬라이드"할 수 있는 뉴클레오솜과 일치했다.2008년에는 CTCF 결합 부위가 뉴클레오좀 위치 결정 앵커로서 작용하여 다양한 게놈 신호를 정렬할 때 다중 측면 뉴클레오솜을 쉽게 [37]식별할 수 있다는 것이 추가로 밝혀졌다.뉴클레오솜은 본질적으로 이동성이지만, 진핵생물은 염색질 구조를 바꾸기 위해 ATP 의존적인 염색질 재모델링 효소의 큰 패밀리를 진화시켰으며, 그들 중 다수는 뉴클레오솜 슬라이딩을 통해 그렇게 한다.2012년, 비나 필라이의 실험실은 뉴클레오솜 슬라이딩이 유전자의 대규모 조직 특이적 발현을 위한 가능한 메커니즘 중 하나라는 것을 증명했다.이 연구는 특정 조직에서 발현되는 유전자의 전사 시작 부위가 뉴클레오솜이 고갈된 반면, 발현되지 않는 다른 조직에서는 동일한 세트의 유전자가 뉴클레오솜과 [14]결합되어 있다는 것을 보여준다.
DNA부위노출
위돔 연구소의 연구는 핵염색체 DNA가 포장된 상태와 포장되지 않은 상태 사이의 균형을 이루고 있다는 것을 보여주었다.시간 분해 FLET를 사용하여 이러한 속도를 측정한 결과, 뉴클레오솜 내의 DNA는 10-50ms 동안 포장을 풀었다가 빠르게 다시 [38]포장을 하기 전에 250ms 동안만 완전히 포장을 유지한다는 것이 밝혀졌다.이것은 DNA가 뉴클레오솜에서 능동적으로 분리될 필요는 없지만 완전히 접근할 수 있는 상당한 시간이 있다는 것을 암시한다.실제로, 이것은 뉴클레오솜 내에 DNA 결합 배열을 도입하는 것이 [39]결합되었을 때 DNA의 인접 영역의 접근성을 증가시킨다는 관찰로 확장될 수 있다.이 뉴클레오솜 내의 DNA가 "호흡"하는 경향은 염색질 [38]환경에서 작동하는 모든 DNA 결합 단백질에 중요한 기능적 결과를 가져온다.특히 뉴클레오좀의 동적 호흡은 전사 [40]신장 중에 RNA 중합효소 II의 진행을 제한하는 데 중요한 역할을 한다.
뉴클레오솜프리영역
활성 유전자의 프로모터는 뉴클레오좀 프리 영역(NFR)을 가지고 있다.이것은 전사인자와 같은 다양한 단백질에 대한 프로모터 DNA 접근을 가능하게 한다.뉴클레오솜 자유 영역은 일반적으로 세레비새에서[41] 200개의 뉴클레오티드에 걸쳐 있다. 잘 배치된 뉴클레오솜은 NFR의 경계를 형성한다.이러한 뉴클레오솜은 +1-뉴클레오솜과 -1-뉴클레오솜이라고 불리며, 전사가 시작되는 [42]부위에서 각각 하류 및 상류로 표준 거리에 위치한다. +1-뉴클레오솜과 몇몇 하류 뉴클레오솜도 H2A를 포함하는 경향이 있다.Z히스톤 [42]변종
변조 뉴클레오솜 구조
진핵생물 게놈은 어디에나 크로마틴과 연관되어 있지만, 세포는 부피 크로마틴과는 독립적으로 공간적이고 시간적으로 특정한 위치를 조절해야 한다.DNA 복제, 복구, 전사 등 핵 과정을 조정하는 데 필요한 높은 수준의 제어를 달성하기 위해 세포는 국소적이고 구체적으로 크로마틴 구조와 기능을 조절하는 다양한 수단을 개발했다.이것은 히스톤의 공유성 변형, 히스톤 변이체의 통합, ATP 의존성 리모델링 효소에 의한 비공유성 재모델링을 포함할 수 있다.
히스톤 번역 후 수정
1960년대 중반에 발견된 이래 히스톤의 변형은 [43]전사에 영향을 미칠 것으로 예측되어 왔다.발견된 대부분의 초기 번역 후 수정이 뉴클레오솜 코어에서 돌출된 꼬리 연장선 내에 집중되었다는 사실은 히스톤 수정 메커니즘에 관한 두 가지 주요 이론을 이끈다.첫 번째 이론은 그것들이 염색질 구조를 느슨하게 하기 위해 히스톤 꼬리와 DNA 사이의 정전적 상호작용에 영향을 미칠 수 있다는 것을 시사했다.나중에 이러한 변형들의 조합이 다른 단백질을 [44]모집하는 결합 에피토프를 생성할 수 있다는 것이 제안되었다.최근 히스톤의 구조화된 영역에서 더 많은 수정이 발견됨에 따라, 이러한 수정이 뉴클레오솜 코어 내의 히스톤-DNA 및 히스톤-히스톤[46] 상호작용에 영향을 미칠 수 있다는 주장이 제기되었다.구상 히스톤 코어의 전하를 낮추는 변형(아세틸화 또는 인산화 등)은 코어-DNA 결합을 "느슨하게" 할 것으로 예측된다. 효과의 강도는 [47]코어 내의 변형 위치에 따라 달라진다.몇몇 변형들은 유전자 소음과 관련이 있는 것으로 보여졌고, 다른 변형들은 유전자 활성화와 관련이 있는 것으로 보인다.일반적인 변형으로는 아세틸화, 메틸화 또는 리신의 유비퀴티화, 아르기닌의 메틸화, 세린의 인산화 등이 있다.이렇게 저장된 정보는 DNA에 암호화되지 않지만 여전히 딸 세포에 유전되기 때문에 후생유전적인 것으로 간주됩니다.억제되거나 활성화된 유전자 상태를 유지하는 것은 세포 [18]분화를 위해 종종 필요하다.
히스톤 변종
히스톤은 진화 과정에서 현저하게 보존되지만, 몇 가지 변형 형태가 확인되었다.히스톤 기능의 다양화는 H2A와 H3로 제한되며, H2B와 H4는 대부분 불변이다.H2A는 H2AZ(뉴클레오솜 안정성 저하로 이어짐) 또는 H2AX(DNA 복구 및 T세포 분화와 관련됨)로 대체될 수 있는 반면, 포유류의 비활성 X 염색체는 매크로H2A로 농축된다.H3는 H3.3(활성유전자 및 조절요소와의 상관관계)으로 대체될 수 있으며, 동원체 H3는 [18]CENPA로 대체된다.
ATP 의존성 뉴클레오솜 리모델링
ATP 의존성 크로마틴 리모델링이라는 용어와 많은 뚜렷한 반응들이 연관되어 있다.리모델링 효소는 DNA를 [48]따라 뉴클레오솜을 슬라이드시키고, H2A/H2B 이합체를[49][50] 불안정하게 만들 정도로 히스톤-DNA 접촉을 방해하며, DNA와 염색질에 [51]음의 초 나선적 비틀림을 발생시키는 것으로 나타났다.최근에는 Swr1 리모델링 효소가 변형 히스톤 H2A를 도입하는 것으로 나타났다.Z를 뉴클레오솜으로 [52]만들어요현재, 이 모든 것이 별개의 반응을 나타내는 것인지, 아니면 단지 공통 메커니즘의 대체 결과를 나타내는 것인지 명확하지 않다.ATP 의존성 염색질 리모델링의 특징인 공통점은 모두 DNA 접근성이 변화시킨다는 것입니다.
생체 내 유전자[53] 활성화와 더 놀랍게도, 체외[54] 리모델링에 대한 연구는 염색질 리모델링 이벤트와 전사 인자 결합이 본질적으로 주기적이고 주기적이라는 것을 밝혀냈다.염색질 리모델링의 반응 메커니즘에 대한 이것의 결과는 알려져 있지 않지만, 시스템의 동적 특성은 외부 자극에 더 빨리 반응할 수 있게 할 수 있다.최근의 연구는 뉴클레오솜 위치가 마우스 배아줄기세포가 발달하는 동안 크게 변화하며,[55] 이러한 변화는 발달전사인자의 결합과 관련이 있다는 것을 보여준다.
효모 게놈 전체의 동적 뉴클레오솜 재모델링
2007년 연구는 효모의 뉴클레오솜 위치를 목록화했고, 프로모터 영역과 [56][57][58]복제의 기원에서 뉴클레오솜이 고갈된다는 것을 보여주었다.효모 게놈의 약 80%는 뉴클레오솜에[59] 의해 덮여 있는 것으로 보이며 뉴클레오솜의 위치 결정 패턴은 전사를 조절하는 DNA 영역, 전사되는 영역 및 DNA [60]복제를 시작하는 영역과 명확하게 관련되어 있습니다.가장 최근에, 새로운 연구는 효모의 게놈 전체 전사 변화 동안 뉴클레오솜 치환에 [61]대한 영향을 설명하기 위해 글로벌 전사 재프로그래밍 이벤트 동안 뉴클레오솜 위치 변경의 동적 변화를 조사했다.그 결과 프로모터 영역에 국소화된 뉴클레오솜이 스트레스(열충격 등)에 반응하여 치환되는 것으로 나타났다.또한, 뉴클레오좀의 제거는 보통 전사 활성화에 대응하고 뉴클레오좀의 치환은 보통 전사 억제에 대응하며, 아마도 전사인자 결합 부위가 각각 어느 정도 접근 가능하게 되었기 때문일 것이다.일반적으로 이러한 전사 변화를 일으키기 위해 프로모터에서 하나 또는 두 개의 뉴클레오솜만 재배치되었다.그러나, 전사 변화와 관련이 없는 염색체 영역에서도 뉴클레오좀의 위치가 관찰되어 전사 DNA의 피복과 발견이 반드시 전사 이벤트를 발생시키는 것은 아님을 시사했다.전사 후, rDNA 영역은 어떠한 손상으로부터도 보호되어야 하며, HMGB 단백질은 뉴클레오솜 [62][63]프리 영역을 보호하는 데 중요한 역할을 한다고 제안했다.
DNA 트위스트 결함
DNA 트위스트 결손은 한 DNA 세그먼트에서 하나 또는 몇 개의 염기쌍의 추가가 다음 세그먼트로 전달되어 DNA 트위스트의 변화를 일으키는 경우이다.이것은 DNA의 뒤틀림을 바꿀 뿐만 아니라 [64]길이도 바꿀 것이다.이 꼬임 결함은 결국 염기쌍의 전달을 통해 뉴클레오솜 주위를 이동하는데, 이것은 DNA 꼬임이 [65]뉴클레오솜의 미끄러짐을 야기할 수 있다는 것을 의미합니다.뉴클레오솜 결정 구조는 뉴클레오솜 위의 초나선 위치 2와 5가 일반적인 리모델링 결합 [66]부위이기 때문에 일반적으로 DNA 트위스트 결함이 발생하는 것으로 밝혀졌다.다양한 염색질 리모델링제가 있지만 모두 ATP의 [67]결합과 가수분해를 통해 DNA에서 염색질이 미끄러지는 것을 촉진하는 ATPase 모터의 존재를 공유합니다.ATP 분해효소는 개방과 폐쇄 상태를 가지고 있다.ATPase 모터가 개방 및 폐쇄 상태에서 변화할 때, DNA 듀플렉스는 형상을 바꾸고 염기쌍 기울기를 보입니다.[66]ATPase 모터를 통해 트위스트 결함이 시작되면 리모델링 부위 주변에 장력이 축적됩니다.DNA의 슬라이딩이 반대 방향으로 두 개의 트위스트 결점([67]각 가닥에 하나씩)의 확산을 통해 뉴클레오솜 전체에 걸쳐 완료되면 장력이 방출됩니다.
시험관내 뉴클레오솜 조립체
뉴클레오솜은 정제된 천연 히스톤 [68][69]또는 재조합 히스톤을 사용하여 시험관내에서 조립할 수 있다.히스톤 주위에 DNA를 로드하는 한 가지 표준 기술은 소금 투석을 사용하는 것이다.히스톤 옥타머와 벌거벗은 DNA 템플릿으로 이루어진 반응을 2M의 염농도로 배양할 수 있으며 염분 농도를 꾸준히 감소시킴으로써 히스톤 옥타머를 감싸는 위치까지 평형화되어 뉴클레오솜을 형성한다.적절한 조건에서 이 재구성 과정은 주어진 배열의 뉴클레오솜 위치결정 친화성을 [70]실험적으로 매핑할 수 있게 한다.
디술피드 가교 뉴클레오솜 코어 입자
안정성이 향상된 뉴클레오솜 코어 입자의 생산의 최근 발전은 부위 특이적 디술피드 [71]가교와 관련이 있다.두 개의 다른 가교 링크는 뉴클레오솜 코어 입자에 도입될 수 있다.첫 번째 것은 도입 시스테인(N38C)을 통해 H2A의 두 복사본을 가교함으로써 뉴클레오솜 재구성 중 H2A/H2B 이합체 손실에 대해 안정된 히스톤 옥타머를 생성한다.H3 N 말단 히스톤 꼬리와 뉴클레오좀 DNA 말단 사이에 내장된 컨버터블 뉴클레오티드를 [72]통해 제2의 가교 링크를 도입할 수 있다.DNA-히스톤 옥타머 교차 링크는 매우 낮은 입자 농도와 높은 소금 농도로 뉴클레오솜 코어 입자를 DNA 해리에 대해 안정화시킵니다.
생체내 뉴클레오솜 조립체
뉴클레오솜은 DNA가 감겨있는 히스톤 단백질로 만들어진 DNA의 기본 포장 단위이다.그것들은 유전자 발현 조절 층뿐만 아니라 고차 염색질 구조의 형성을 위한 발판 역할을 한다.뉴클레오솜은 복제 포크 뒤에 있는 새로 합성된 DNA에 빠르게 조립됩니다.
H3 및 H4
분해된 오래된 뉴클레오솜에서 나온 히스톤 H3, H4가 근방에 유지되어 새롭게 합성된 [73]DNA 상에 랜덤하게 분포한다.이들은 3개의 서브유닛(p150, p60 및 p48)[74]으로 구성된 크로마틴 조립률-1(CAF-1) 복합체에 의해 조립됩니다.새롭게 합성된 H3, H4는 Replication Coupling Assembly Factor(RCAF)에 의해 조립된다.RCAF는 새롭게 합성된 H3 [75]및 H4 단백질에 결합하는 서브유닛 Af1을 포함한다.오래된 H3 및 H4 단백질은 후생유전적 특징의 전승에 기여하는 화학적 변형을 유지한다.새로 합성된 H3 및 H4 단백질은 염색질 성숙 [76]과정의 일부로 서로 다른 리신 잔기에서 점차 아세틸화된다.또한 새로운 뉴클레오솜의 오래된 H3 및 H4 단백질은 새로운 히스톤을 표시하는 히스톤 수정 효소를 모집하여 후생유전기억에 기여하는 것으로 생각된다.
H2A 및 H2B
구식 H3, H4와 달리 구식 H2A 및 H2B 히스톤 단백질이 방출 및 분해되므로 신규 조립 H2A 및 H2B 단백질이 신규 뉴클레오솜에 [77]혼입된다.H2A 및 H2B를 이합체로 조립한 후 뉴클레오솜 조립단백질-1(NAP-1)에 의해 뉴클레오솜에 적재하여 뉴클레오솜 [78]슬라이딩을 돕는다.뉴클레오솜은 또한 Isw1 Ino80, Chd1과 같은 효소를 포함하는 ATP 의존성 뉴클레오솜-리모델링 복합체에 의해 간격을 두고 있으며, 이후 고차 [79][80]구조로 조립된다.
갤러리
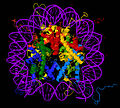
뉴클레오솜 코어 입자(PDB: 1EQZ[28])의 결정 구조 - 히스톤 접힘 및 구성에 대한 세부사항을 보여주는 다른 관점.히스톤 H2A, H2B, H3, H4 및 DNA를 착색한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Reece J, Campbell N (2006). Biology. San Francisco: Benjamin Cummings. ISBN 978-0-8053-6624-2.
- ^ Alberts B (2002). Molecular biology of the cell (4th ed.). New York: Garland Science. p. 207. ISBN 978-0-8153-4072-0.
- ^ lberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2002). Chromosomal DNA and Its Packaging in the Chromatin Fiber.
- ^ Teif VB, Clarkson CT (2019). "Nucleosome Positioning". Encyclopedia of Bioinformatics and Computational Biology. 2: 308–317. doi:10.1016/B978-0-12-809633-8.20242-2. ISBN 9780128114322. S2CID 43929234.
- ^ Olins AL, Olins DE (January 1974). "Spheroid chromatin units (v bodies)". Science. 183 (4122): 330–332. Bibcode:1974Sci...183..330O. doi:10.1126/science.183.4122.330. PMID 4128918. S2CID 83480762.
- ^ McDonald D (December 2005). "Milestone 9, (1973-1974) The nucleosome hypothesis: An alternative string theory". Nature Milestones: Gene Expression. doi:10.1038/nrm1798.
- ^ Kornberg RD (May 1974). "Chromatin structure: a repeating unit of histones and DNA". Science. 184 (4139): 868–871. Bibcode:1974Sci...184..868K. doi:10.1126/science.184.4139.868. PMID 4825889.
- ^ Lorch Y, LaPointe JW, Kornberg RD (April 1987). "Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones". Cell. 49 (2): 203–210. doi:10.1016/0092-8674(87)90561-7. PMID 3568125. S2CID 21270171.
- ^ Han M, Grunstein M (December 1988). "Nucleosome loss activates yeast downstream promoters in vivo". Cell. 55 (6): 1137–1145. doi:10.1016/0092-8674(88)90258-9. PMID 2849508. S2CID 41520634.
- ^ Clark-Adams CD, Norris D, Osley MA, Fassler JS, Winston F (February 1988). "Changes in histone gene dosage alter transcription in yeast". Genes & Development. 2 (2): 150–159. doi:10.1101/gad.2.2.150. PMID 2834270.
- ^ a b 다른 결정에서 146과 147의 염기쌍 값이 관찰되었다.
- ^ a b Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (September 1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648): 251–260. Bibcode:1997Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ Alberts B (2007). Molecular Biology of the Cell (5th ed.). New York: Garland Science. p. 211. ISBN 978-0-8153-4106-2.
- ^ a b Bargaje R, Alam MP, Patowary A, Sarkar M, Ali T, Gupta S, et al. (October 2012). "Proximity of H2A.Z containing nucleosome to the transcription start site influences gene expression levels in the mammalian liver and brain". Nucleic Acids Research. 40 (18): 8965–8978. doi:10.1093/nar/gks665. PMC 3467062. PMID 22821566.
- ^ Zhou YB, Gerchman SE, Ramakrishnan V, Travers A, Muyldermans S (September 1998). "Position and orientation of the globular domain of linker histone H5 on the nucleosome". Nature. 395 (6700): 402–405. Bibcode:1998Natur.395..402Z. doi:10.1038/26521. PMID 9759733. S2CID 204997317.
- ^ Thoma F, Koller T, Klug A (November 1979). "Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin". The Journal of Cell Biology. 83 (2 Pt 1): 403–427. doi:10.1083/jcb.83.2.403. PMC 2111545. PMID 387806.
- ^ Clarke HJ (1992). "Nuclear and chromatin composition of mammalian gametes and early embryos". Biochemistry and Cell Biology. 70 (10–11): 856–866. doi:10.1139/o92-134. PMID 1297351.
- ^ a b c d e f Felsenfeld G, Groudine M (January 2003). "Controlling the double helix". Nature. 421 (6921): 448–453. Bibcode:2003Natur.421..448F. doi:10.1038/nature01411. PMID 12540921.
- ^ Richmond TJ, Finch JT, Rushton B, Rhodes D, Klug A (1984). "Structure of the nucleosome core particle at 7 A resolution". Nature. 311 (5986): 532–7. Bibcode:1984Natur.311..532R. doi:10.1038/311532a0. PMID 6482966. S2CID 4355982.
- ^ Harp JM, Palmer EL, York MH, Gewiess A, Davis M, Bunick GJ (October 1995). "Preparative separation of nucleosome core particles containing defined-sequence DNA in multiple translational phases". Electrophoresis. 16 (10): 1861–1864. doi:10.1002/elps.11501601305. PMID 8586054. S2CID 20178479.
- ^ Palmer EL, Gewiess A, Harp JM, York MH, Bunick GJ (October 1995). "Large-scale production of palindrome DNA fragments". Analytical Biochemistry. 231 (1): 109–114. doi:10.1006/abio.1995.1509. PMID 8678288.
- ^ Harp JM, Uberbacher EC, Roberson AE, Palmer EL, Gewiess A, Bunick GJ (March 1996). "X-ray diffraction analysis of crystals containing twofold symmetric nucleosome core particles". Acta Crystallographica. Section D, Biological Crystallography. 52 (Pt 2): 283–288. doi:10.1107/S0907444995009139. PMID 15299701.
- ^ Harp JM, Hanson BL, Timm DE, Bunick GJ (December 2000). "Asymmetries in the nucleosome core particle at 2.5 A resolution". Acta Crystallographica. Section D, Biological Crystallography. 56 (Pt 12): 1513–1534. doi:10.1107/s0907444900011847. PMID 11092917.
- ^ Hanson BL, Alexander C, Harp JM, Bunick GJ (2004). "Preparation and crystallization of nucleosome core particle". Methods in Enzymology. 375: 44–62. doi:10.1016/s0076-6879(03)75003-4. ISBN 9780121827793. PMID 14870658.
- ^ a b Chakravarthy S, Park YJ, Chodaparambil J, Edayathumangalam RS, Luger K (February 2005). "Structure and dynamic properties of nucleosome core particles". FEBS Letters. 579 (4): 895–898. doi:10.1016/j.febslet.2004.11.030. PMID 15680970. S2CID 41706403.
- ^ White CL, Suto RK, Luger K (September 2001). "Structure of the yeast nucleosome core particle reveals fundamental changes in internucleosome interactions". The EMBO Journal. 20 (18): 5207–5218. doi:10.1093/emboj/20.18.5207. PMC 125637. PMID 11566884.
- ^ a b Stryer L (1995). Biochemistry (fourth ed.). New York - Basingstoke: W. H. Freeman and Company. ISBN 978-0716720096.
- ^ a b Harp JM, Hanson BL, Timm DE, Bunick GJ (December 2000). "Asymmetries in the nucleosome core particle at 2.5 A resolution". Acta Crystallographica. Section D, Biological Crystallography. 56 (Pt 12): 1513–1534. doi:10.1107/S0907444900011847. PMID 11092917. PDB ID: 1EQZ.
- ^ Alberts B (2009). Essential Cell Biology (2nd ed.). New York: Garland Science.
- ^ Davey CA, Sargent DF, Luger K, Maeder AW, Richmond TJ (June 2002). "Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 a resolution". Journal of Molecular Biology. 319 (5): 1097–1113. doi:10.1016/S0022-2836(02)00386-8. PMID 12079350.
- ^ Segal E, Fondufe-Mittendorf Y, Chen L, Thåström A, Field Y, Moore IK, et al. (August 2006). "A genomic code for nucleosome positioning". Nature. 442 (7104): 772–778. Bibcode:2006Natur.442..772S. doi:10.1038/nature04979. PMC 2623244. PMID 16862119.
- ^ Zheng C, Hayes JJ (April 2003). "Structures and interactions of the core histone tail domains". Biopolymers. 68 (4): 539–546. doi:10.1002/bip.10303. PMID 12666178.
- ^ Schalch T, Duda S, Sargent DF, Richmond TJ (July 2005). "X-ray structure of a tetranucleosome and its implications for the chromatin fibre". Nature. 436 (7047): 138–141. Bibcode:2005Natur.436..138S. doi:10.1038/nature03686. PMID 16001076. S2CID 4387396.
- ^ Robinson PJ, Fairall L, Huynh VA, Rhodes D (April 2006). "EM measurements define the dimensions of the "30-nm" chromatin fiber: evidence for a compact, interdigitated structure". Proceedings of the National Academy of Sciences of the United States of America. 103 (17): 6506–6511. Bibcode:2006PNAS..103.6506R. doi:10.1073/pnas.0601212103. PMC 1436021. PMID 16617109.
- ^ Teif VB, Rippe K (September 2009). "Predicting nucleosome positions on the DNA: combining intrinsic sequence preferences and remodeler activities". Nucleic Acids Research. 37 (17): 5641–5655. doi:10.1093/nar/gkp610. PMC 2761276. PMID 19625488.
- ^ Pennings S, Muyldermans S, Meersseman G, Wyns L (May 1989). "Formation, stability and core histone positioning of nucleosomes reassembled on bent and other nucleosome-derived DNA". Journal of Molecular Biology. 207 (1): 183–192. doi:10.1016/0022-2836(89)90449-X. PMID 2738923.
- ^ Fu Y, Sinha M, Peterson CL, Weng Z (July 2008). Van Steensel B (ed.). "The insulator binding protein CTCF positions 20 nucleosomes around its binding sites across the human genome". PLOS Genetics. 4 (7): e1000138. doi:10.1371/journal.pgen.1000138. PMC 2453330. PMID 18654629.
- ^ a b Li G, Levitus M, Bustamante C, Widom J (January 2005). "Rapid spontaneous accessibility of nucleosomal DNA". Nature Structural & Molecular Biology. 12 (1): 46–53. doi:10.1038/nsmb869. PMID 15580276. S2CID 14540078.
- ^ Li G, Widom J (August 2004). "Nucleosomes facilitate their own invasion". Nature Structural & Molecular Biology. 11 (8): 763–769. doi:10.1038/nsmb801. PMID 15258568. S2CID 11299024.
- ^ Hodges C, Bintu L, Lubkowska L, Kashlev M, Bustamante C (July 2009). "Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II". Science. 325 (5940): 626–628. Bibcode:2009Sci...325..626H. doi:10.1126/science.1172926. PMC 2775800. PMID 19644123.
- ^ Yuan GC, Liu YJ, Dion MF, Slack MD, Wu LF, Altschuler SJ, Rando OJ (July 2005). "Genome-scale identification of nucleosome positions in S. cerevisiae". Science. 309 (5734): 626–630. Bibcode:2005Sci...309..626Y. doi:10.1126/science.1112178. PMID 15961632. S2CID 43625066.
- ^ a b Lai WK, Pugh BF (September 2017). "Understanding nucleosome dynamics and their links to gene expression and DNA replication". Nature Reviews. Molecular Cell Biology. 18 (9): 548–562. doi:10.1038/nrm.2017.47. PMC 5831138. PMID 28537572.
- ^ Allfrey VG, Faulkner R, Mirsky AE (May 1964). "Acetylation and Methylation of Histones and Their Possible Role in the Regulation of RNA Synthesis". Proceedings of the National Academy of Sciences of the United States of America. 51 (5): 786–794. Bibcode:1964PNAS...51..786A. doi:10.1073/pnas.51.5.786. PMC 300163. PMID 14172992.
- ^ Strahl BD, Allis CD (January 2000). "The language of covalent histone modifications". Nature. 403 (6765): 41–45. Bibcode:2000Natur.403...41S. doi:10.1038/47412. PMID 10638745. S2CID 4418993.
- ^ Cosgrove MS, Boeke JD, Wolberger C (November 2004). "Regulated nucleosome mobility and the histone code". Nature Structural & Molecular Biology. 11 (11): 1037–1043. doi:10.1038/nsmb851. PMID 15523479. S2CID 34704745.
- ^ Ye J, Ai X, Eugeni EE, Zhang L, Carpenter LR, Jelinek MA, et al. (April 2005). "Histone H4 lysine 91 acetylation a core domain modification associated with chromatin assembly". Molecular Cell. 18 (1): 123–130. doi:10.1016/j.molcel.2005.02.031. PMC 2855496. PMID 15808514.
- ^ Fenley AT, Adams DA, Onufriev AV (September 2010). "Charge state of the globular histone core controls stability of the nucleosome". Biophysical Journal. 99 (5): 1577–1585. Bibcode:2010BpJ....99.1577F. doi:10.1016/j.bpj.2010.06.046. PMC 2931741. PMID 20816070.
- ^ Whitehouse I, Flaus A, Cairns BR, White MF, Workman JL, Owen-Hughes T (August 1999). "Nucleosome mobilization catalysed by the yeast SWI/SNF complex". Nature. 400 (6746): 784–787. Bibcode:1999Natur.400..784W. doi:10.1038/23506. PMID 10466730. S2CID 2841873.
- ^ Kassabov SR, Zhang B, Persinger J, Bartholomew B (February 2003). "SWI/SNF unwraps, slides, and rewraps the nucleosome". Molecular Cell. 11 (2): 391–403. doi:10.1016/S1097-2765(03)00039-X. PMID 12620227.
- ^ Bruno M, Flaus A, Stockdale C, Rencurel C, Ferreira H, Owen-Hughes T (December 2003). "Histone H2A/H2B dimer exchange by ATP-dependent chromatin remodeling activities". Molecular Cell. 12 (6): 1599–1606. doi:10.1016/S1097-2765(03)00499-4. PMC 3428624. PMID 14690611.
- ^ Havas K, Flaus A, Phelan M, Kingston R, Wade PA, Lilley DM, Owen-Hughes T (December 2000). "Generation of superhelical torsion by ATP-dependent chromatin remodeling activities". Cell. 103 (7): 1133–1142. doi:10.1016/S0092-8674(00)00215-4. PMID 11163188. S2CID 7911590.
- ^ Mizuguchi G, Shen X, Landry J, Wu WH, Sen S, Wu C (January 2004). "ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex". Science. 303 (5656): 343–348. Bibcode:2004Sci...303..343M. doi:10.1126/science.1090701. PMID 14645854. S2CID 9881829.
- ^ Métivier R, Penot G, Hübner MR, Reid G, Brand H, Kos M, Gannon F (December 2003). "Estrogen receptor-alpha directs ordered, cyclical, and combinatorial recruitment of cofactors on a natural target promoter". Cell. 115 (6): 751–763. doi:10.1016/S0092-8674(03)00934-6. PMID 14675539. S2CID 145525.
- ^ Nagaich AK, Walker DA, Wolford R, Hager GL (April 2004). "Rapid periodic binding and displacement of the glucocorticoid receptor during chromatin remodeling". Molecular Cell. 14 (2): 163–174. doi:10.1016/S1097-2765(04)00178-9. PMID 15099516.
- ^ Teif VB, Vainshtein Y, Caudron-Herger M, Mallm JP, Marth C, Höfer T, Rippe K (November 2012). "Genome-wide nucleosome positioning during embryonic stem cell development". Nature Structural & Molecular Biology. 19 (11): 1185–1192. doi:10.1038/nsmb.2419. PMID 23085715. S2CID 34509771.
- ^ Albert I, Mavrich TN, Tomsho LP, Qi J, Zanton SJ, Schuster SC, Pugh BF (March 2007). "Translational and rotational settings of H2A.Z nucleosomes across the Saccharomyces cerevisiae genome". Nature. 446 (7135): 572–576. Bibcode:2007Natur.446..572A. doi:10.1038/nature05632. PMID 17392789. S2CID 4416890.
- ^ Li B, Carey M, Workman JL (February 2007). "The role of chromatin during transcription". Cell. 128 (4): 707–719. doi:10.1016/j.cell.2007.01.015. PMID 17320508. S2CID 1773333.
- ^ Whitehouse I, Rando OJ, Delrow J, Tsukiyama T (December 2007). "Chromatin remodelling at promoters suppresses antisense transcription". Nature. 450 (7172): 1031–1035. Bibcode:2007Natur.450.1031W. doi:10.1038/nature06391. PMID 18075583. S2CID 4305576.
- ^ Lee W, Tillo D, Bray N, Morse RH, Davis RW, Hughes TR, Nislow C (October 2007). "A high-resolution atlas of nucleosome occupancy in yeast". Nature Genetics. 39 (10): 1235–1244. doi:10.1038/ng2117. PMID 17873876. S2CID 12816925.
- ^ Eaton ML, Galani K, Kang S, Bell SP, MacAlpine DM (April 2010). "Conserved nucleosome positioning defines replication origins". Genes & Development. 24 (8): 748–753. doi:10.1101/gad.1913210. PMC 2854390. PMID 20351051.
- ^ Shivaswamy S, Bhinge A, Zhao Y, Jones S, Hirst M, Iyer VR (March 2008). "Dynamic remodeling of individual nucleosomes across a eukaryotic genome in response to transcriptional perturbation". PLOS Biology. 6 (3): e65. doi:10.1371/journal.pbio.0060065. PMC 2267817. PMID 18351804.
- ^ Murugesapillai D, McCauley MJ, Huo R, Nelson Holte MH, Stepanyants A, Maher LJ, et al. (August 2014). "DNA bridging and looping by HMO1 provides a mechanism for stabilizing nucleosome-free chromatin". Nucleic Acids Research. 42 (14): 8996–9004. doi:10.1093/nar/gku635. PMC 4132745. PMID 25063301.
- ^ Murugesapillai D, McCauley MJ, Maher LJ, Williams MC (February 2017). "Single-molecule studies of high-mobility group B architectural DNA bending proteins". Biophysical Reviews. 9 (1): 17–40. doi:10.1007/s12551-016-0236-4. PMC 5331113. PMID 28303166.
- ^ Winger J, Nodelman IM, Levendosky RF, Bowman GD (May 2018). "A twist defect mechanism for ATP-dependent translocation of nucleosomal DNA". eLife. 7: e34100. doi:10.7554/eLife.34100. PMC 6031429. PMID 29809147.
- ^ Bowman GD (August 2019). "Uncovering a New Step in Sliding Nucleosomes". Trends in Biochemical Sciences. 44 (8): 643–645. doi:10.1016/j.tibs.2019.05.001. PMC 7092708. PMID 31171402.
- ^ a b Nodelman IM, Bowman GD (May 2021). "Biophysics of Chromatin Remodeling". Annual Review of Biophysics. 50 (1): 73–93. doi:10.1146/annurev-biophys-082520-080201. PMC 8428145. PMID 33395550.
- ^ a b Brandani GB, Takada S (November 2018). Onufriev A (ed.). "Chromatin remodelers couple inchworm motion with twist-defect formation to slide nucleosomal DNA". PLOS Computational Biology. 14 (11): e1006512. Bibcode:2018PLSCB..14E6512B. doi:10.1371/journal.pcbi.1006512. PMC 6237416. PMID 30395604.
- ^ Hayes JJ, Lee KM (May 1997). "In vitro reconstitution and analysis of mononucleosomes containing defined DNAs and proteins". Methods. 12 (1): 2–9. doi:10.1006/meth.1997.0441. PMID 9169189.
- ^ Dyer PN, Edayathumangalam RS, White CL, Bao Y, Chakravarthy S, Muthurajan UM, Luger K (2004). "Reconstitution of nucleosome core particles from recombinant histones and DNA". Methods in Enzymology. 375: 23–44. doi:10.1016/s0076-6879(03)75002-2. ISBN 9780121827793. PMID 14870657.
- ^ Yenidunya A, Davey C, Clark D, Felsenfeld G, Allan J (April 1994). "Nucleosome positioning on chicken and human globin gene promoters in vitro. Novel mapping techniques". Journal of Molecular Biology. 237 (4): 401–414. doi:10.1006/jmbi.1994.1243. PMID 8151701.
- ^ Frouws TD, Barth PD, Richmond TJ (January 2018). "Site-Specific Disulfide Crosslinked Nucleosomes with Enhanced Stability". Journal of Molecular Biology. 430 (1): 45–57. doi:10.1016/j.jmb.2017.10.029. PMC 5757783. PMID 29113904.
- ^ Ferentz AE, Verdine GL (1994). "The Convertible Nucleoside Approach: Structural Engineering of Nucleic Acids by Disulfide Cross-Linking". In Eckstein F, Lilley DM (eds.). Nucleic Acids and Molecular Biology. Nucleic Acids and Molecular Biology. Vol. 8. pp. 14–40. doi:10.1007/978-3-642-78666-2_2. ISBN 978-3-642-78668-6.
- ^ Yamasu K, Senshu T (January 1990). "Conservative segregation of tetrameric units of H3 and H4 histones during nucleosome replication". Journal of Biochemistry. 107 (1): 15–20. doi:10.1093/oxfordjournals.jbchem.a122999. PMID 2332416.
- ^ Kaufman PD, Kobayashi R, Kessler N, Stillman B (June 1995). "The p150 and p60 subunits of chromatin assembly factor I: a molecular link between newly synthesized histones and DNA replication". Cell. 81 (7): 1105–1114. doi:10.1016/S0092-8674(05)80015-7. PMID 7600578. S2CID 13502921.
- ^ Tyler JK, Adams CR, Chen SR, Kobayashi R, Kamakaka RT, Kadonaga JT (December 1999). "The RCAF complex mediates chromatin assembly during DNA replication and repair". Nature. 402 (6761): 555–560. Bibcode:1999Natur.402..555T. doi:10.1038/990147. PMID 10591219. S2CID 205097512.
- ^ Benson LJ, Gu Y, Yakovleva T, Tong K, Barrows C, Strack CL, et al. (April 2006). "Modifications of H3 and H4 during chromatin replication, nucleosome assembly, and histone exchange". The Journal of Biological Chemistry. 281 (14): 9287–9296. doi:10.1074/jbc.M512956200. PMID 16464854.
- ^ Louters L, Chalkley R (June 1985). "Exchange of histones H1, H2A, and H2B in vivo". Biochemistry. 24 (13): 3080–3085. doi:10.1021/bi00334a002. PMID 4027229.
- ^ Park YJ, Chodaparambil JV, Bao Y, McBryant SJ, Luger K (January 2005). "Nucleosome assembly protein 1 exchanges histone H2A-H2B dimers and assists nucleosome sliding". The Journal of Biological Chemistry. 280 (3): 1817–1825. doi:10.1074/jbc.M411347200. PMID 15516689.
- ^ Vincent JA, Kwong TJ, Tsukiyama T (May 2008). "ATP-dependent chromatin remodeling shapes the DNA replication landscape". Nature Structural & Molecular Biology. 15 (5): 477–484. doi:10.1038/nsmb.1419. PMC 2678716. PMID 18408730.
- ^ Yadav T, Whitehouse I (April 2016). "Replication-Coupled Nucleosome Assembly and Positioning by ATP-Dependent Chromatin-Remodeling Enzymes". Cell Reports. 15 (4): 715–723. doi:10.1016/J.CELREP.2016.03.059. PMC 5063657. PMID 27149855.