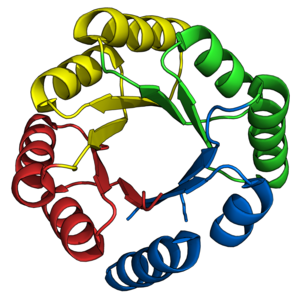
TIM 배럴
TIM barrel| 알돌라아제형 TIM 배럴 | |
|---|---|
 | |
| 식별자 | |
| 기호. | 알돌라아제_팀 |
| 빠맘 클랜 | CL0036 |
| 인터프로 | IPR013785 |
| 캐스 | 8박자 |
| SCOP2 | 8 tim / SCOPe / SUPFAM |
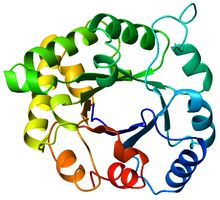
알파/베타 [1]: 252 배럴이라고도 하는 TIM 배럴(트리오스-인산 이성질화효소)은 펩타이드 [2]골격을 따라 교대로 움직이는 8개의 알파 헬리클(α-헬리체)과 8개의 평행 베타 스트랜드(β-스트랜드)로 구성된 보존된 단백질 접힘이다.이 구조는 보존된 대사 [3]효소인 트리오스-인산 이성질화효소의 이름을 따서 명명되었다.TIM 배럴은 어디에나 존재하며, 모든 효소의 약 10%가 이 [4]접힘을 채택합니다.또한 7종류의 효소 커미션(EC) 효소 클래스 중 5종은 TIM 배럴 [5][6]단백질을 포함한다.TIM 배럴 폴드는 진화적으로 오래된 것으로,[7] 오늘날 많은 구성원들이 거의 유사성을 가지고 있지 않고, 대신 순서 [8][9]유사성의 황혼 영역에 속합니다.
내부 베타 배럴(β-barrel)은 많은 경우 복잡한 소금 [10]브릿지 네트워크에 의해 안정화됩니다.β-배럴의 C-말단 단부에 있는 루프는 촉매[11][12] 활성을 담당하며, N-말단 단부 루프는 TIM-배럴의 안정성에 중요합니다.확장 루프에서 독립 단백질 도메인에 이르는 구조적 삽입물은 이러한 루프 대신 또는 N-말단/C-말단에 삽입될 수 있다.TIM 배럴은 유전자 복제와 절반 배럴 [13]단백질의 도메인 융합 이벤트를 통해 진화한 것으로 보이며, 대부분의 TIM 배럴은 공통의 조상으로부터 유래했다.이로 인해 많은 TIM 배럴이 내부 대칭을 [14]갖게 되었습니다.이 조상 TIM 배럴의 추가적인 유전자 복제 사건은 오늘날 관찰된 기능적 다양성을 가진 발산 효소로 이어졌다.TIM 배럴은 또한 단백질 설계자들의 오랜 목표물이었다.성공적인 TIM 배럴 설계에는 기존 단백질의 도메인 퓨전 및 de novo 설계가 모두 포함됩니다.도메인 퓨전 실험은 많은 성공적인 설계를 [15][16][17][18][19][20][21]낳았지만, de novo 설계는 28년간의 증분 [22]개발 후에야 성공을 거두었다.
구조.


토폴로지
TIM 배럴의 이름은 트리오스-인산 이성질화효소(TIM)에서 유래했는데, TIM은 이 성분은 결정화된 [3]첫 번째 단백질이었다.TIM 배럴은 8개의 알파 헬리클(α-헬리체)과 8개의 베타 스트랜드(β-스트랜드)로 접힌 200-250개의 아미노산 [2]잔류물을 포함합니다.β 가닥은 평행 베타 배럴(β-barrel)로 배열되어 있으며, 8개의 α-헬리에 둘러싸여 있다.TIM β-배럴의 정의 특성은 항상 [2]8의 전단 수를 갖는다는 것이다.전단 수는 β-스트랜드-1의 잔류물 x를 선택하고 원래 β-스트랜드-1의 잔류물 y에 도달할 때까지 β-배럴을 따라 가닥 방향에 수직 방향으로 이동함으로써 결정된다.시작 위치와 끝 위치 사이의 잔류물 수(y-x )는 전단 [24]수입니다.스트랜드 수가 전단 수와 같기 때문에 사이드 체인은 모공과 코어를 번갈아 가리키며 4배의 대칭을 이룬다.α-나선은 내부 β-배럴을 둘러싸고 완전히 감싸고 있다.짧은 루프는 일반적으로 α 및 β 2차 구조를 연결하여 (βα)8 반복 토폴로지를 형성합니다.경우에 따라서는 확장 루프에서 독립 도메인에 이르는 구조가 이러한 루프 대신 삽입되거나 N/C 터미널에 연결될 수 있습니다.모든 TIM 배럴 효소는 β [25]배럴의 C 말단 단부에 촉매 부위가 있으며, 이 단부에 가까운 구조적 삽입물이 촉매 활성에 도움이 될 수 있습니다.
코어 및 모공 영역
TIM 배럴은 아미노산 잔류물이 이웃에 완전히 둘러싸여 용매에 접근할 수 없는 두 개의 뚜렷한 매립 영역을 포함합니다.이 영역 내에 용제 채널이 없기 때문에 '포어'라는 용어는 잘못된 명칭입니다.코어 영역은 α-β 계면을 구성하는 모든 잔류물로 구성되며 중심 β-배럴 외부에 있다.모공 영역은 모든 내부 β-배럴 잔기로 구성되며, 이들은 β-배럴 골격으로 둘러싸여 있다.
β-스트랜드의 주름 특성으로 인해 스트랜드를 따른 대체 잔류물은 모공(53%)과 코어(47%) 사이에서 거의 균등하게 분할된다.β-배럴의 경우 코어 잔기의 95%가 매립되어 있다.코어 잔기의 11%만이 극성이며, 물에 대한 친화력을 가지고 있으며, 수소 결합이나 염교 [10]형성 능력을 가지고 있다.마찬가지로 β-스트랜드 모공 잔기의 84%가 매립되어 있다.그러나 모공 잔류물의 42%는 극성이다.이러한 잔류물은 용제의 접근성 부족을 보완하기 위해 복잡한 염교 네트워크를 형성합니다.
TIM 배럴 안정화 요소
TIM 배럴 모공 내의 소금 브릿지는 접힘의 전반적인 안정성에 기여하는 것으로 생각됩니다.2-디옥시리보스-5-인산알돌라아제에서 대형 염교망의 예를 찾을 수 있다.이 네트워크는 클래스 I 알돌라아제 패밀리에 걸쳐 보존되어 있는 것으로 판명되었습니다.
극성 잔류물과 모공 내 소금 브릿지가 과도하게 나타나는 정확한 이유는 여전히 불분명하다.한 연구는 TIM 배럴의 열역학적 안정성보다는 접힘성을 개선한다고 제안합니다.접는 과정에서 β-스트랜드에 있는 내부 모공 잔류물은 물에 노출된다.폴드온이라고 불리는 부분 접힌 βαβα 모듈은 이 폴딩 단계에서 극성 모공 잔류물에 의해 에너지적으로 안정화된다.
S. solfataric indole-3-glycerol phosphate synthase TIM barrel 단백질과 관련된 또 다른 연구에서 보존된 βαβ 모듈은 다른 2차 구조의 접힘을 안내하는 필수 접힘 템플릿인 것으로 밝혀졌다.β-피복 폐쇄는 폴딩 과정의 마지막에만 발생했다.그러나 이 경우, 저자들은 분기 지방족 아미노산(발린, 류신, 이소류신)이 폴드온의 안정성을 인정하였다.
TIM 배럴의 또 다른 안정화 요소는 베타 헤어핀 클램프입니다.짝수 β-스트랜드의 N-termini 측쇄 H-결합 공여자는 종종 앞의 홀수 β-스트랜드에서 주쇄 아미드 수소와 H-결합을 형성한다.이러한 클램프(또는 소수성 측쇄교 아날로그)는 박테리아와 고고학계의 3개의 인돌-3-글리세롤인산합성효소 TIM 배럴 오솔로그에 보존되어 있으며, 이는 그들이 마지막 공통 조상에서 발생했으며 10억 년 이상 보존되었음을 암시한다.
구조 인서트

TIM 배럴 단백질의 N/C 말단 및 루프 영역은 단순한 2차 구조 모티브에서 완전한 도메인에 이르는 구조적 삽입물을 호스팅할 수 있다.이러한 영역은 기판 인식 및 촉매 활성에 도움이 됩니다.추가 모티브와 도메인을 포함하는 TIM 배럴의 4가지 다양한 예가 아래에 설명되어 있습니다.
Bacillus subtilis Orotidine 5'-인산탈카르복실화효소(PDB: 1DBT)는 β-barrel(잔류 35-42, 89-91, 126-133, 215-219)의 C 말단에 전형적으로 존재하는 βα 루프 대신 4개의 α-헬리스를 나타내는 TIM 배럴 단백질이다.이들 나선형(R215→K219) 중 하나는 오로티딘5γ-일인산염의 인산염 부분과의 상호작용에 필요한 보존 아르기닌레시듀(R215)를 포함한다.다른 나선은 촉매 활성에 중요한 잔류물을 수용하는 것으로 발견되지 않았으며 구조적 역할을 할 수 있습니다.
마이코박테륨 결핵 2관능성 히스티딘/트립토판 생합성 이성질효소(PriA)(PDB: 2Y85)는 (i) HisA 반응: N-[(5-포스포리보실) 포르미노]-5-아미노이미다졸-4-뉴클레오박스의 2가지 반응을 촉매하는 능력을 가진다.우클레오티드(PRFAR) 및 (ii) TrpF 반응: N-(5'-포스포리보실)-안트라닐산(PRA)~1-(O-카르복시페닐아미노)-1'-디옥시리불로스-5'-인산(CdRP)PriA는 활성 부위 루프(루프 1, 5, 6, β-배럴의 C 말단 단부에서 확장된 βα 루프)를 사용하여 두 기질을 수용하는 TIM 배럴 효소입니다.루프 1은 ProFAR이 존재하는 경우에만 활성화사이트를 덮어씁니다Loop5는 활성화사이트를 감싸고 CdRP가 있는 경우 β시트 컨피규레이션 또는 ProFAR가 있는 경우 매듭 모양의 컨피규레이션을 채택합니다.루프 6은 모든 반응물질의 활성 부위를 덮습니다.
락토코커스 락티스 디히드로트로테이트탈수소효소A(DHODA) (PDB: 2DOR)는 β-시트와 β-배럴의 C-말단에 걸쳐 확장된 루프를 가진 TIM 배럴의 예이다.DODA는 디히드로오로테이트의 산화를 오로테이트로 촉매하며, 이는 데노보우리딘 5'-일인산(UMP) 합성 경로의 일부이다.이 산화는 플라빈 모노뉴클레오티드(FMN)에 의해 매개된다.여기서, β-시트와 확장 루프는 공동을 형성하는 활성 부위를 둘러싸는 동시에 여러 촉매 잔류물을 수용한다.
메틸로필루스메틸로프로푸스트리메틸아민탈수소효소(PDB:2TMD) TIM 배럴은 완전한 도메인 삽입의 한 예이다.여기서 TIM-barrel의 C 말단부에 로스만 폴드 도메인을 삽입한다.트리메틸아민탈수소효소는 트리메틸아민의 포름알데히드 전환을 촉매한다.이 반응에는 환원된 6-S-시스테이닐 플라빈 모노뉴클레오티드(FMN) 보조인자와 환원된 철-설푸르([4Fe-4S])+ 중심부가 모두 필요합니다.FMN은 β배럴의 C말단 영역 내에 공유 결합되어 있다.[4Fe-4S]+ 중앙은 TIM 배럴 내에 수용하기에는 너무 크고, TIM 배럴과 Rossmann 폴드 도메인 사이의 인터페이스에 7º 떨어진 가까운 곳에 배치됩니다.
접이식 메커니즘
TIM 배럴 폴드의 보존은 계통학적으로 다른 혈통을 가진 세균성 패럴로그의 평형 및 운동 폴딩 메커니즘 보존에 의해 반영된다.여러 자연[27][28] 및 2개의 설계된 TIM 배럴[28] 변형의 화학적 변성에는 반드시 고밀도 평형 중간체가 포함된다.고도로 변성된 용액에서 희석된 후에 나타나는 운동 중간체는 생산적인 접힘 [27][28]경로에 접근하기 위해 적어도 부분적으로 펼쳐져야 하는 초기 잘못 접힌 종을 포함한다.접힘의 속도 제한 단계는 평형 [29]중간체에 해당하는 앞의 개방 배럴 형태로 8가닥 β 배럴을 닫는 것이다.원어민 중심 분자 역학 시뮬레이션은 실험 결과를 재점검하고 복잡한 접이식 [30]메커니즘에 대한 테스트 가능한 계산 모델을 제시합니다.
피트니스 환경 보호
TIM 배럴 단백질은 비정상적으로 높은 염기서열 가소성을 가지고 있으며, 널리 분산된 유기체에서 직교 및 병렬 효소의 큰 패밀리를 형성합니다.이러한 가소성은 기능을 유지하면서 주로 계통 발생 이력과 무관한 다양한 환경 조건에 대한 단백질 적응을 가능하게 하는 배열 풍경을 시사합니다.IGPS가 결여된 효모 숙주의 성장을 지원하는 3개의 고열성 인돌-3-글리세롤인산합성효소(IGPS) TIM 배럴 효소의 위치에 걸쳐 가능한 모든 아미노산 돌연변이의 적합성을 결정하기 위해 심층 돌연변이[31] 스캔 접근법과 경쟁 분석을[32] 사용했다.2개의 박테리아 효소와 1개의 고고 IGPS 효소는 순서상 30-40%만 동일했지만, 이들의 적합성 환경은 강한 상관관계가 있었다. 즉, 세 개의 다른 단백질에서 동일한 위치에 있는 동일한 아미노산은 매우 유사한 적합성을 보였다.상관관계는 진화시간에 걸쳐 TIM 배럴 효소에 대한 적합성 환경의 보존으로 생각할 수 있다.
루프 영역
TIM 배럴을 완전히 형성하기 위해 필요한 약 200개의 잔류물 중 약 160개는 이 접힘을 공유하는 다른 단백질 간에 구조적으로 동등한 것으로 간주된다.나머지 잔류물은 나선과 가닥을 연결하는 루프 영역에 위치합니다; 가닥의 C-말단 끝의 루프는 활성 부위를 포함하는 경향이 있습니다, 이것이 이 접힘이 매우 일반적인 이유 중 하나입니다: 구조를 유지하는 데 필요한 잔류물과 효소 촉매 작용에 효과적인 잔류물은 대부분의 개별 서브셋에 대한 것입니다.: 링크 루프는 사실 너무 길어서 다른 단백질 도메인을 포함할 수 있습니다.[33]최근에는 촉매 루프가 다른 [34]TIM 배럴 효소 간에 작용기의 반자율 단위로 교환될 수 있다는 것이 입증되었다.
진화와 기원


TIM 배럴 진화에 대한 지배적인 이론은 결국 완전한 TIM 배럴을 형성한 반 배럴에서 시작하여 유전자 복제와 융합을 포함한다.여러 연구가 단일 조상으로부터의 발산 진화 이론을 뒷받침하며, 아래에서 논의된다.
공통 조상으로부터의 진화
1990년대 초, 당시 해결된 모든 TIM 배럴 구조는 효소로 공통 [11][12]조상으로부터의 차이를 나타냈다.또한 모든 TIM 배럴은 β-배럴의 C 말단부에 활성 부위를 가지고 있었다. 작은 α-나선 및 TIM 배럴 루프-7/8에 의해 형성된 공통 인산염 결합 부위는 분화 [35]진화를 강하게 나타낸다.23개의 SCOP TIM 배럴 패밀리 중 12개가 공통 [36]조상에서 분리되었다는 결론을 내린 인산염 그룹에 대한 추가 연구.마찬가지로 21개의 CATH TIM 배럴 패밀리 [7]중 17개의 공통 조상에 대한 힌트가 있었다.이러한 보고에 근거해, 대부분의 TIM 배럴 단백질이 공통의 조상으로부터 진화한 것이 타당하다고 생각된다.
유전자 복제와 도메인 융합을 통한 기원

많은 TIM 배럴 단백질은 2배, 4배 또는 8배 내부 대칭을 가지며, 이는 TIM 배럴이 유전자 복제와 도메인 융합을 통해 조상(βα),4 (βα)2 또는 βα 모티브에서 진화했음을 시사한다.Thermotoga maritima 히스티딘 생합성 [13]경로의 ProFAR 이성질화효소(HisA) 및 이미다졸 글리세롤 인산합성효소(HisF)에서 2배 내부 대칭의 좋은 예가 관찰된다.이들은 경로에서 2개의 연속 반응을 촉매하고, 25%의 염기서열 상동성을 가지며, 1.5~2Ω 사이의 RMSD(root-mean-square deviations)를 가지며, 공통 조상으로부터의 분리를 시사한다.더 흥미로운 것은 HisA와 HisF의 C 말단 루프가 2배 반복 패턴을 보여 공통 조상이 2배 내부 대칭을 가졌음을 시사한다.이러한 관찰을 이용하여, TIM [13]배럴의 진화를 위한 모델이 구성되었습니다.조상의 반배럴은 유전자 복제와 융합 현상을 겪었고, 그 결과 두 개의 반배럴 도메인을 포함하는 단일 단백질이 생성되었다.구조적 적응이 발생하여 이러한 도메인이 결합되어 폐쇄된 β 배럴을 형성하고 조상 TIM 배럴을 형성하게 된다.기능적 적응도 일어나 β-배럴의 C 말단부에서 새로운 촉매 활성의 진화를 초래했을 것이다.이 시점에서 HisA와 HisF의 공통 조상은 두 번째 유전자 복제 이벤트를 겪었을 것이다.조상 TIM 배럴의 중복 유전자가 서로 다르게 진화했다면 HisA와 HisF가 형성되었을 것이다.
흥미롭게도, 이 진화 모델은 합리적인 단백질 설계와 유도된 진화를 사용하여 실험적으로 검증되었습니다.Höcker 등은 먼저 HisF의 두 개의 C 말단 반쪽을 융합하여 HisF-CC를 생성했다.그런 다음 내부 염교를 삽입하여 HisF-C*[17]C를 생성함으로써 이 구조가 안정화되었습니다.HisF-C*C는 하프배럴 인터페이스를 최적화하여 [15][16]HisF-C**C와 HisF-C**C를 생성함으로써 더욱 단계적인 안정화 및 가용화를 달성하였다.HisF-C***C의 결정구조는 2배의 대칭 TIM 배럴을 나타내 자연영역 융접 가능성을 입증했다.게다가, 회커는 최초의 키메라 히스를 창조했다.HisA와 HisF 하프 [17]배럴을 사용하는 AF와 HisFA TIM 배럴.이러한 실험은 기존의 TIM 배럴 사이에서 (βα)4의 반배럴 도메인의 교환을 통해 TIM 배럴 효소의 다양화와 진화를 위한 새로운 방법의 제안으로 이어졌다.이 아이디어에 따라 HIS의 촉매 활성도가 높아집니다.AF구축이 [18]확립되었습니다.마찬가지로 키메라βα-플라보독신5 유사주름(CheY)/HisF TIM [19][20]배럴 및 완벽하게 2배 대칭 HisF 기반의 TIM[21][28] 배럴도 생성되었다.
TIM 배럴 [14]시퀀스의 계산 분석에 기초하여 4/8배 내부 대칭의 존재가 제안되었다.예를 들어 대장균 KDPG 알돌라아제[37](PDB: 1FQ0)는 구별 가능한 8배 대칭과 뚜렷한 4배 대칭을 갖는 것으로 제안되었다.4배 대칭 TIM[22] 배럴의 설계로 자연 TIM 배럴의 내부 대칭 차수가 높아질 가능성이 확인되었으며, 다음 섹션에서 자세히 설명하겠습니다.8배 대칭 TIM 배럴의 존재에 대한 실험적인 증거는 현재까지 보고되지 않았다.
De novo TIM 배럴 설계
TIM 배럴 폴드는 de novo 단백질 디자이너들에게 오랜 목표였습니다.앞서 설명한 바와 같이, 다수의 TIM 배럴은 기존의 천연 하프 배럴을 기반으로 성공적으로 설계되었습니다.반면 TIM 배럴의 de novo 설계는 28년에 [38]걸쳐 점진적으로 이루어졌다.
단백질의 옥타렐린 시리즈[39][40][41][42][43](Octarelin I→VI)는 de novo TIM 배럴을 생성하기 위한 첫 번째 시도였다.단백질 설계 분야는 아직 초기 단계였기 때문에 이러한 설계 시도는 제한적인 성공만을 거뒀습니다.이들은 αβ 단백질 및 일부 협력적 접힘 특성과 일치하는 원형 이색성 스펙트럼을 보였지만, 모든 옥타렐린 계열 펩타이드는 불용성이었으며, 추가적인 특성화를 위해 포접체에서 재규화되어야 했다.흥미롭게도, 옥타렐린 V[44].1은 공동 결정 조건에서 로스만과 같은 접힘을 보였다.
Symmetrin 시리즈 단백질(Symmetrin-1→4)은 보다 양호한 생물물리학적 특성을 보였다.Symmetrin-1은 쉽게 용해되며, αβ 단백질과 일치하는 원형 이색성 스펙트럼을 보였으며, 뛰어난 협동 전개 및 리폴딩 특성을 보였다.이러한 발전에도 불구하고, 이 패밀리의 모든 단백질은 NMR(핵자기공명)을 이용하여 분석했을 때 녹은 특성을 보였으며, 그 구조를 해결하기 위한 추가 작업을 추구할 수 없었다.
sTIM[22] 시리즈의 단백질은 최초의 성공적인 de novo TIM 배럴 [45][38]설계를 나타낸다. sTIM-11(PDB: 5BVL)은 Rosetta 소프트웨어 제품군을 [46]사용하여 계산 설계의 복잡성을 줄이기 위해 내부 4중 대칭으로 설계되었다.이전에 도출된 첫 번째[47] 원칙은 이차 구조 토폴로지 및 길이를 기술하기 위해 사용되었다. sTIM-11은 의도된 구조를 채택한 높은 내열성과 협동적으로 접히는 설계임이 입증되었다.
「 」를 참조해 주세요.
레퍼런스
![]() 이 문서는 CC BY 4.0, § 4.0 라이선스(2020)(리뷰어 리포트)에 따라 다음 소스로부터 수정되었습니다. Deepesh Nagarajan; Neha Nanajkar (2020). "The TIM barrel fold" (PDF). WikiJournal of Science. 3 (1): 4. doi:10.15347/WJS/2020.004. ISSN 2470-6345. Wikidata Q87400003.
이 문서는 CC BY 4.0, § 4.0 라이선스(2020)(리뷰어 리포트)에 따라 다음 소스로부터 수정되었습니다. Deepesh Nagarajan; Neha Nanajkar (2020). "The TIM barrel fold" (PDF). WikiJournal of Science. 3 (1): 4. doi:10.15347/WJS/2020.004. ISSN 2470-6345. Wikidata Q87400003.
- ^ Voet D, Voet JG (2011). "Chapter 8. Three-Dimensional Structures of Proteins". Biochemistry (4th ed.). John Wiley & Sons, Inc. ISBN 978-0470-91745-9.
- ^ a b c Wierenga RK (March 2001). "The TIM-barrel fold: a versatile framework for efficient enzymes". FEBS Letters. 492 (3): 193–8. doi:10.1016/s0014-5793(01)02236-0. PMID 11257493. S2CID 42044123.
- ^ Jansen R, Gerstein M (March 2000). "Analysis of the yeast transcriptome with structural and functional categories: characterizing highly expressed proteins". Nucleic Acids Research. 28 (6): 1481–8. doi:10.1093/nar/28.6.1481. PMC 111042. PMID 10684945.
- ^ Nagano N, Hutchinson EG, Thornton JM (October 1999). "Barrel structures in proteins: automatic identification and classification including a sequence analysis of TIM barrels". Protein Science. 8 (10): 2072–84. doi:10.1110/ps.8.10.2072. PMC 2144152. PMID 10548053.
- ^ Webb EC (1992). Enzyme nomenclature: Recommendations of the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology on the Nomenclature and Classification of Enzymes. Academic Press. ISBN 978-0-12-227164-9.
- ^ a b Nagano N, Orengo CA, Thornton JM (August 2002). "One fold with many functions: the evolutionary relationships between TIM barrel families based on their sequences, structures and functions". Journal of Molecular Biology. 321 (5): 741–65. doi:10.1016/s0022-2836(02)00649-6. PMID 12206759.
- ^ Livesay DR, La D (May 2005). "The evolutionary origins and catalytic importance of conserved electrostatic networks within TIM-barrel proteins". Protein Science. 14 (5): 1158–70. doi:10.1110/ps.041221105. PMC 2253277. PMID 15840824.
- ^ Chung SY, Subbiah S (October 1996). "A structural explanation for the twilight zone of protein sequence homology". Structure. 4 (10): 1123–7. doi:10.1016/s0969-2126(96)00119-0. PMID 8939745.
- ^ a b Vijayabaskar MS, Vishveshwara S (2012). "Insights into the fold organization of TIM barrel from interaction energy based structure networks". PLOS Computational Biology. 8 (5): e1002505. Bibcode:2012PLSCB...8E2505V. doi:10.1371/journal.pcbi.1002505. PMC 3355060. PMID 22615547.
- ^ a b Farber GK, Petsko GA (June 1990). "The evolution of alpha/beta barrel enzymes". Trends in Biochemical Sciences. 15 (6): 228–34. doi:10.1016/0968-0004(90)90035-A. PMID 2200166.
- ^ a b Reardon D, Farber GK (April 1995). "The structure and evolution of alpha/beta barrel proteins". FASEB Journal. 9 (7): 497–503. doi:10.1096/fasebj.9.7.7737457. PMID 7737457. S2CID 23208817.
- ^ a b c d Lang D, Thoma R, Henn-Sax M, Sterner R, Wilmanns M (September 2000). "Structural evidence for evolution of the beta/alpha barrel scaffold by gene duplication and fusion". Science. 289 (5484): 1546–50. Bibcode:2000Sci...289.1546L. doi:10.1126/science.289.5484.1546. PMID 10968789.
- ^ a b Söding J, Remmert M, Biegert A (July 2006). "HHrep: de novo protein repeat detection and the origin of TIM barrels". Nucleic Acids Research. 34 (Web Server issue): W137-42. doi:10.1093/nar/gkl130. PMC 1538828. PMID 16844977.
- ^ a b Seitz T, Bocola M, Claren J, Sterner R (September 2007). "Stabilisation of a (betaalpha)8-barrel protein designed from identical half barrels". Journal of Molecular Biology. 372 (1): 114–29. doi:10.1016/j.jmb.2007.06.036. PMID 17631894.
- ^ a b Höcker B, Lochner A, Seitz T, Claren J, Sterner R (February 2009). "High-resolution crystal structure of an artificial (betaalpha)(8)-barrel protein designed from identical half-barrels". Biochemistry. 48 (6): 1145–7. doi:10.1021/bi802125b. PMID 19166324.
- ^ a b c Höcker B, Claren J, Sterner R, Makar AB, McMartin KE, Palese M, Tephly TR (June 1975). "Formate assay in body fluids: application in methanol poisoning". Biochemical Medicine. 13 (2): 117–26. doi:10.1016/0006-2944(75)90147-7. PMC 534502. PMID 15539462.
- ^ a b Claren J, Malisi C, Höcker B, Sterner R (March 2009). "Establishing wild-type levels of catalytic activity on natural and artificial (beta alpha)8-barrel protein scaffolds". Proceedings of the National Academy of Sciences of the United States of America. 106 (10): 3704–9. Bibcode:2009PNAS..106.3704C. doi:10.1073/pnas.0810342106. PMC 2656144. PMID 19237570.
- ^ a b Bharat TA, Eisenbeis S, Zeth K, Höcker B (July 2008). "A beta alpha-barrel built by the combination of fragments from different folds". Proceedings of the National Academy of Sciences of the United States of America. 105 (29): 9942–7. Bibcode:2008PNAS..105.9942B. doi:10.1073/pnas.0802202105. PMC 2481348. PMID 18632584.
- ^ a b Eisenbeis S, Proffitt W, Coles M, Truffault V, Shanmugaratnam S, Meiler J, Höcker B (March 2012). "Potential of fragment recombination for rational design of proteins". Journal of the American Chemical Society. 134 (9): 4019–22. doi:10.1021/ja211657k. PMID 22329686.
- ^ a b Fortenberry C, Bowman EA, Proffitt W, Dorr B, Combs S, Harp J, et al. (November 2011). "Exploring symmetry as an avenue to the computational design of large protein domains". Journal of the American Chemical Society. 133 (45): 18026–9. doi:10.1021/ja210593m. PMC 3781211. PMID 21978247.
- ^ a b c Huang PS, Feldmeier K, Parmeggiani F, Velasco DA, Höcker B, Baker D (January 2016). "De novo design of a four-fold symmetric TIM-barrel protein with atomic-level accuracy". Nature Chemical Biology. 12 (1): 29–34. doi:10.1038/nchembio.1966. PMC 4684731. PMID 26595462.
- ^ Nagarajan D, Deka G, Rao M (August 2015). "Design of symmetric TIM barrel proteins from first principles". BMC Biochemistry. 16 (1): 18. doi:10.1186/s12858-015-0047-4. PMC 4531894. PMID 26264284.
- ^ Murzin AG, Lesk AM, Chothia C (March 1994). "Principles determining the structure of beta-sheet barrels in proteins. I. A theoretical analysis". Journal of Molecular Biology. 236 (5): 1369–81. doi:10.1016/0022-2836(94)90064-7. PMID 8126726.
- ^ Brändén C (1991). "The TIM barrel—the most frequently occurring folding motif in proteins". Current Opinion in Structural Biology. 1 (6): 978–983. doi:10.1016/0959-440x(91)90094-a.
- ^ Barber MJ, Neame PJ, Lim LW, White S, Matthews FS (April 1992). "Correlation of x-ray deduced and experimental amino acid sequences of trimethylamine dehydrogenase". The Journal of Biological Chemistry. 267 (10): 6611–9. doi:10.1016/S0021-9258(19)50471-9. PMID 1551870.
- ^ a b Forsyth WR, Bilsel O, Gu Z, Matthews CR (September 2007). "Topology and sequence in the folding of a TIM barrel protein: global analysis highlights partitioning between transient off-pathway and stable on-pathway folding intermediates in the complex folding mechanism of a (betaalpha)8 barrel of unknown function from B. subtilis". Journal of Molecular Biology. 372 (1): 236–53. doi:10.1016/j.jmb.2007.06.018. PMID 17619021.
- ^ a b c d Carstensen L, Sperl JM, Bocola M, List F, Schmid FX, Sterner R (August 2012). "Conservation of the folding mechanism between designed primordial (βα)8-barrel proteins and their modern descendant". Journal of the American Chemical Society. 134 (30): 12786–91. doi:10.1021/ja304951v. PMID 22758610.
- ^ Gu Z, Rao MK, Forsyth WR, Finke JM, Matthews CR (November 2007). "Structural analysis of kinetic folding intermediates for a TIM barrel protein, indole-3-glycerol phosphate synthase, by hydrogen exchange mass spectrometry and Gō model simulation". Journal of Molecular Biology. 374 (2): 528–46. doi:10.1016/j.jmb.2007.09.024. PMC 2735044. PMID 17942114.
- ^ Halloran KT, Wang Y, Arora K, Chakravarthy S, Irving TC, Bilsel O, et al. (August 2019). "Frustration and folding of a TIM barrel protein". Proceedings of the National Academy of Sciences of the United States of America. 116 (33): 16378–16383. doi:10.1073/pnas.1900880116. PMC 6697809. PMID 31346089.
- ^ Chan YH, Venev SV, Zeldovich KB, Matthews CR (March 2017). "Correlation of fitness landscapes from three orthologous TIM barrels originates from sequence and structure constraints". Nature Communications. 8: 14614. Bibcode:2017NatCo...814614C. doi:10.1038/ncomms14614. PMC 5343507. PMID 28262665.
- ^ Hietpas RT, Jensen JD, Bolon DN (May 2011). "Experimental illumination of a fitness landscape". Proceedings of the National Academy of Sciences of the United States of America. 108 (19): 7896–901. doi:10.1073/pnas.1016024108. PMC 3093508. PMID 21464309.
- ^ Ochoa-Leyva A, Soberón X, Sánchez F, Argüello M, Montero-Morán G, Saab-Rincón G (April 2009). "Protein design through systematic catalytic loop exchange in the (beta/alpha)8 fold". Journal of Molecular Biology. 387 (4): 949–64. doi:10.1016/j.jmb.2009.02.022. PMID 19233201.
- ^ Ochoa-Leyva A, Barona-Gómez F, Saab-Rincón G, Verdel-Aranda K, Sánchez F, Soberón X (August 2011). "Exploring the Structure-Function Loop Adaptability of a (β/α)(8)-Barrel Enzyme through Loop Swapping and Hinge Variability". Journal of Molecular Biology. 411 (1): 143–57. doi:10.1016/j.jmb.2011.05.027. PMID 21635898.
- ^ Brändén CI (1991). "The TIM barrel—the most frequently occurring folding motif in proteins". Current Opinion in Structural Biology. 1 (6): 978–983. doi:10.1016/0959-440x(91)90094-a.
- ^ Copley RR, Bork P (November 2000). "Homology among (betaalpha)(8) barrels: implications for the evolution of metabolic pathways". Journal of Molecular Biology. 303 (4): 627–41. doi:10.1006/jmbi.2000.4152. PMID 11054297.
- ^ Wymer N, Buchanan LV, Henderson D, Mehta N, Botting CH, Pocivavsek L, et al. (January 2001). "Directed evolution of a new catalytic site in 2-keto-3-deoxy-6-phosphogluconate aldolase from Escherichia coli". Structure. 9 (1): 1–9. doi:10.1016/S0969-2126(00)00555-4. PMID 11342129.
- ^ a b Borman S (2015). "Protein designers roll out a barrel". Chemical & Engineering News. Vol. 93, no. 47. p. 6.
- ^ Goraj K, Renard A, Martial JA (March 1990). "Synthesis, purification and initial structural characterization of octarellin, a de novo polypeptide modelled on the alpha/beta-barrel proteins". Protein Engineering. 3 (4): 259–66. doi:10.1093/protein/3.4.259. PMID 2188263.
- ^ Beauregard M, Goraj K, Goffin V, Heremans K, Goormaghtigh E, Ruysschaert JM, Martial JA (October 1991). "Spectroscopic investigation of structure in octarellin (a de novo protein designed to adopt the alpha/beta-barrel packing)". Protein Engineering. 4 (7): 745–9. doi:10.1093/protein/4.7.745. PMID 1798699.
- ^ Houbrechts A, Moreau B, Abagyan R, Mainfroid V, Préaux G, Lamproye A, et al. (March 1995). "Second-generation octarellins: two new de novo (beta/alpha)8 polypeptides designed for investigating the influence of beta-residue packing on the alpha/beta-barrel structure stability". Protein Engineering. 8 (3): 249–59. doi:10.1093/protein/8.3.249. PMID 7479687.
- ^ Offredi F, Dubail F, Kischel P, Sarinski K, Stern AS, Van de Weerdt C, et al. (January 2003). "De novo backbone and sequence design of an idealized alpha/beta-barrel protein: evidence of stable tertiary structure" (PDF). Journal of Molecular Biology. 325 (1): 163–74. doi:10.1016/S0022-2836(02)01206-8. PMID 12473459.
- ^ Figueroa M, Oliveira N, Lejeune A, Kaufmann KW, Dorr BM, Matagne A, et al. (2013). "Octarellin VI: using rosetta to design a putative artificial (β/α)8 protein". PLOS ONE. 8 (8): e71858. Bibcode:2013PLoSO...871858F. doi:10.1371/journal.pone.0071858. PMC 3747059. PMID 23977165.
- ^ Figueroa M, Sleutel M, Vandevenne M, Parvizi G, Attout S, Jacquin O, et al. (July 2016). "The unexpected structure of the designed protein Octarellin V.1 forms a challenge for protein structure prediction tools". Journal of Structural Biology. 195 (1): 19–30. doi:10.1016/j.jsb.2016.05.004. hdl:2268/199167. PMID 27181418.
- ^ Nanda V (January 2016). "Protein Design: Getting to the bottom of the TIM barrel". Nature Chemical Biology. 12 (1): 2–3. doi:10.1038/nchembio.1987. PMID 26678608.
- ^ Kaufmann KW, Lemmon GH, Deluca SL, Sheehan JH, Meiler J (April 2010). "Practically useful: what the Rosetta protein modeling suite can do for you". Biochemistry. 49 (14): 2987–98. doi:10.1021/bi902153g. PMC 2850155. PMID 20235548.
- ^ Koga N, Tatsumi-Koga R, Liu G, Xiao R, Acton TB, Montelione GT, Baker D (November 2012). "Principles for designing ideal protein structures". Nature. 491 (7423): 222–7. Bibcode:2012Natur.491..222K. doi:10.1038/nature11600. PMC 3705962. PMID 23135467.
외부 링크
- TIM 배럴 폴드를 채택한 단백질의 SCOP 리스트
- Babu MM (1998). "TIM Barrel Analysis". Center for Biotechnology, Anna University.