사이트별 재조합 기술
Site-specific recombinase technology이 글은 대부분의 독자들이 이해하기에는 너무 기술적인 것일 수도 있다.(2019년 5월)(이를 과 시기 |
사이트별 재조합효소 기술은 DNA의 표적 부분을 대체하기 위해 재조합효소에 의존하는 게놈공학 도구다.
역사
1980년대 후반에 머린 배아줄기세포(ESC)를 대상으로 한 유전자가 돌연변이를 생쥐 세균선으로 전이시킬 수 있게 했고, 게놈에 존재하는 규제 네트워크의 유전적 근거를 연구하는 새로운 선택지로 떠올랐다.그러나, 재조합 ESC를 선택하기 위해 도입되어야 했던 마커 유전자에 의해 유전자 기능이 불가역적으로 파괴됨에 따라, 고전적인 유전자 타겟팅은 여러 가지 면에서 제한적이라는 것이 증명되었다.이러한 초기 단계들은 동물들로 이어졌고, 처음부터 몸의 모든 세포에 돌연변이가 존재하여 복잡한 표현형이나 초기 치사성으로 이어졌다.이러한 돌연변이를 개발의 특정 지점과 특정 세포 유형으로 제한하는 방법이 분명히 필요했다.이 꿈은 미국의 집단이 박테리오파지와 효모에서 유래한 사이트별 재조합(SSR-) 시스템을 쥐뿐만 아니라 포유류 세포에도 도입할 수 있게 되면서 현실이 되었다.[1][2][3]
분류, 속성 및 전용 애플리케이션
일반적인 유전공학 전략에는 대상 게놈의 영구적인 수정이 필요하다.이를 위해 유전자변형물의 전달에 적용되는 경로설계에 큰 정교함이 투자되어야 한다.생명공학적인 목적에서 무작위 통합은 여전히 일반적이지만, 가변 트랜스젠 복사 번호, 통합 사이트에 대한 제어 능력 부족 및 관련 돌연변이로 인해 예측할 수 없는 유전자 발현이 발생할 수 있다.줄기세포 분야의 분자 요구사항은 훨씬 더 엄격하다.여기서, 동질 재조합(HR)은 원칙적으로 통합 프로세스에 특수성을 제공할 수 있지만, 진핵생물의 경우 극히 낮은 효율에 의해 타협된다.메가뉴클레아제, 아연-손가락 및 전사 활성제(ZFNs, TALENs)는 HR을 지원하는 실제 도구지만 예측 가능한 성질을 가진 셀 라인의 합리적 구축을 촉발한 것은 현장 고유 재조합제(SSR)의 가용성이었다.오늘날 HR과 SSR은 매우 효율적인 "태그 앤 교환 기술"[4]로 결합될 수 있다.
많은 현장 고유 재조합 시스템은 다양한 목적으로 이러한 DNA 재조정을 수행하기 위해 확인되었지만, 이들 중 거의 모든 것이 메커니즘에 따라 Tyrosine 재조합(YR)과 세린 재조합(SR) 두 제품군 중 하나에 속한다.이 두 가문은 그 기원과 구조에 따라 서로 다른 반응 경로를 따라 최대 3가지 종류의 DNA 재배열(통합, 절연/해결, 반전)을 중재할 수 있다.[5]
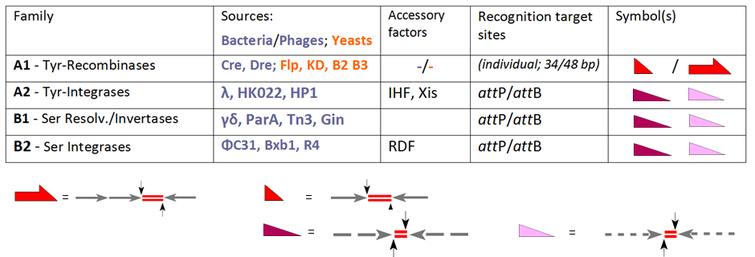
YR 계열의 창립 멤버는 박테리오파지 λ으로 인코딩된 람다 통합효소로서, 페이지 DNA를 박테리아 게놈에 통합할 수 있다.이 세분류의 공통적인 특징은 보존된 티로신 핵종이 가위 DNA-인산염을 공격하여 3'-인산염 연계를 형성한다는 것이다.SR 계열의 초기 구성원은 박테리아 트랜스포존 Tn3 및 Δ로부터 나온 분해효소/DNA 인버타아제로서, 가위 인산염 공격을 담당하는 촉매 세린에 의존하여 5'-인산염 연계를 형성한다.그러나 이러한 논쟁의 여지가 없는 사실들은, 예를 들어 YR 재조합은 "통합가족"의 새로운 구성원으로서 환영받았던 Cre와 Flp(통합, 분리/해결은 물론 반전도 가능)를 현장으로 재조합하는 등, 다른 구성원들이 현장에 진입할 때 상당한 혼란에 의해 타협되었다.역류 예로는 PhiC31과 관련 SR이 있는데, 이 SR은 원래 분해효소/반전효소로 도입되었지만, 보조 요인이 없는 경우에는 통합이 유일한 기능이다.오늘날 각 효소의 표준 활성은 가족 구성원을 위한 일반적인 용어 "recombinase"를 예약하는 그것의 분류를 결정하는데, 이는 INT, RES, INV의 세 가지 경로로 구성된다.
본 표는 기존의 SSR 시스템의 선택을 확장하고, 이러한 시스템을 성능에 따라 그룹화한다.이 모든 효소는 동일한 (하위 제품군 A1) 또는 구별되는 (A2, B1, B2의 피지 유도 효소) 두 개의 표적 부위를 재결합시킨다.[6]A1의 경우 이러한 사이트는 개별 지정("FRT")을 가지고 있는 반면(Flp-recombinase의 경우, 크레-recombinase의 경우 loxP), "attP" 및 "attB"(각각각 페이지와 박테리아 부분의 첨부 사이트)라는 용어는 유효하다.하위 패밀리 A1의 경우 우리는 8bp 스페이서(빨간색 선 이중으로 표시된 교차 영역) 옆에 있는 2개의 (근접) 13bp 암(화살표)으로 구성된 짧은 (일반적으로 34bp-) 사이트를 처리해야 한다.[7]Flp의 경우 세 개의 팔을 가진 48 bp 사이트가 있으며 각각 Flp 유닛(일명 "프로토머")을 수용한다는 점에 유의하십시오.attP-와 attB-s는 유사한 아키텍처 규칙을 따르지만, 여기서 암은 부분적인 정체성(파손된 선으로 표시됨)만 보이며 두 경우 모두 다르다.이러한 특징은 관련 차이를 설명한다.
- 두 개의 동일한 연결 부지의 재결합은 동일한 구성의 제품 부지로 이어진다. 비록 두 기판에서 무기를 포함하고 있지만, 이러한 변환은 되돌릴 수 있다.
- attP x attB 재결합 교차점은 되돌릴 수 없는 방식으로 두 개의 다른 제품(attP x attB → attR + attL)을 유도하는 프로세스에서 이 보완적 파트너들 사이에서만 발생할 수 있다.
본 장의 효율화를 위해 현재 주로 지시된 게놈 수정 시 사용되는 도구를 주파수가 포함하기 때문에 다음의 구현은 두 개의 재조합(Flp 및 Cre)과 단 하나의 통합체(PhiC31)에 초점을 맞출 것이다.이것은 다음의 개요의 틀에서 이루어질 것이다.
반응 경로
모드 통합/해상도 및 반전(INT/RES 및 INV)은 이러한 attP와 attB 쌍 중에서 대상 사이트(RTS) 재결합 방향에 따라 달라진다.섹션 C는 간소화된 방식으로 (통합이 아닌, 분해능이 뒤따르는) 동기식 이중 레시프로칼 크로스를 통해 재조합형 카세트 교환(RMCE)에 도달할 수 있는 방법을 나타낸다.[8][9]
Tyr-Recombinases는 되돌릴 수 있는 반면 Ser-Integratease는 단방향이다.물론, 되돌릴 수 있는 Flp(34bp 최소값 대신) 통합/해상도를 48bp(34bp) FRT 버전으로 변조하는 방법이 있다. 13bp 암은 Flp-INT와 Flp-RMCE 기능의 맥락에서 시냅스 복합체의 형성에 기여하는 Flp "착륙 경로" 역할을 한다(각 평형 상황 참조).크레의 경우 섹션 A의 통합(엔트로피 구동)의 역전을 거의 막을 수 없고, Flp의 경우 달성하기 어려운 반면, 전방과 역반작용의 바이몰리드 특성 때문에 공여 플라스미드가 과도하게 제공되면 RMCE를 완료할 수 있다.두 FRT 사이트를 역방향으로 배치하면 삽입(녹색 화살표)에 대한 두 방향 모두 평형이 된다.Flp와 대조적으로, Ser integrase PhiC31(하단 표현)은 적어도 재조합-방향성(RDF-) 인자가 없는 경우 단방향 통합으로 이어진다.[10]서로 다른 두 가지("Heterospecific") FRT-spacer 돌연변이("Heterospecific")가 필요한 Flp-RMCE에 비해, 기증자 카세트가 대상(cf. 대체 제품)으로 들어가는 방향에 대한 통제가 없을 정도로 첫 번째 반응 atTP 사이트의 반응 파트너(attB)가 임의로 타격된다.또한 Flp-RMCE와 달리, 이질성(비교차적) attP/attB 조합의 부족으로 인해 몇 개의 뚜렷한 RMCE 표적을 병렬로 장착할 수 없다.
크레이 재조합효소
크레 재조합효소(Cre)는 공동 인자 없이도 DNA의 특정 시퀀스를 재조합할 수 있다.이 효소는 loxP라고 불리는 34개의 염기쌍 DNA 염기서열("Page P1의 교차점")을 인식한다.서로에 대한 대상 사이트의 방향에 따라, 크레는 DNA 시퀀스를 통합/배출 또는 반전시킬 것이다.특정 DNA 부위의 절연(원형 기질의 경우 "해상도"라고 함)에 따라 정상적인 유전자 발현이 상당히 손상되거나 종료된다.[11]
크레의 두드러진 분해능 활동으로 인해, 크레가 관심 조직에서 발현된 후 그러한 플록스 유전자의 세포 고유 유전자 결핍으로 이어지는 roxP-flanked("floxed") 유전자의 제거가 최초 적용의 하나였다.현재의 기술에는 크레 활동의 공간적 제어와 시간적 제어를 모두 허용하는 방법이 통합되어 있다.유전적 변화의 공간적 제어를 용이하게 하는 일반적인 방법은 크레 표현을 추진하기 위한 조직 특유의 촉진자를 선택하는 것이다.그러한 프로모터의 통제 하에 크레를 배치하면 지역적이고 조직적인 표현이 된다.예를 들어, Leone 등.미엘린 프로테롤리피드 단백질(PLP) 유전자의 규제 순서에 따라 전사 장치를 제어하여 과두 유전자와 슈완 세포에서 표적 유전자 순서의 제거를 유도한다.[12]크레가 인정한 특정 DNA 파편은 세포에 그대로 남아 PLP 유전자를 표현하지 않고 있다. 이는 중추신경계(CNS)와 말초신경계(PNS)의 신경섬유를 둘러싸고 있는 미엘린 피복의 게놈 변형의 국부적 효과에 대한 경험적 관찰을 용이하게 한다.[13]선택적 크레 표현은 다른 많은 세포 유형과 조직에서도 달성되었다.
절제 반응의 시간적 활동을 제어하기 위해 다양한 리간드 결합 도메인을 이용하는 크레 형태가 개발되었다.특정한 일시적 Cre 활동을 유도하기 위한 하나의 성공적인 전략은 인간 에스트로겐 수용체(ERT)를 위한 돌연변이 리간드 바인딩 도메인과 효소를 융합하는 것을 포함한다.타목시펜(에스트로겐 수용체 길항제)의 도입에 따라 크레-ERT 구조물은 핵에 침투해 표적 돌연변이를 유도할 수 있다.ERT는 타목시펜을 내생성 에스트로겐보다 더 높은 친화력으로 묶어, Cre-ERT가 타목시펜으로 처리되지 않은 동물에서 세포질성 상태를 유지할 수 있게 한다.타목시펜에 의한 SSR 활동의 시간적 제어는 나중에 발생 및/또는 성인 조직에서 유전적 변화를 유도할 수 있도록 한다.[12]이를 통해 연구자들은 대상 유전자의 기능을 조사하는 동시에 배아적 치사율을 우회할 수 있다.
이러한 일반적인 개념의 최근 확장은 "Cre-zoo" 즉, 표적형 Cre 표현에 의해 정의된 유전자를 삭제할 수 있는 수백 개의 마우스 변종들을 발생시켰다.[3]
FLp 재조합효소
FLP/FRT 시스템은 그것의 자연적 호스트(S. cerebisiae)에서, 동일하지만 반대 방향의 FRT 사이트 두 개("flippase" activity)에 의해 측면에 있는 세그먼트를 뒤집어서 "2μ 플라스미드"의 복제를 가능하게 한다.이러한 반대로 "롤링 서클"을 가능하게 하는 플라스미드 내에서 복제 포크의 상대적 방향은 변경된다. 즉, 다중 중간자체가 복수의 모노메릭 제품을 방출하기로 결정되기 전에 원형 2μ 실체를 확대한다.34 bp 최소 FRT 사이트는 Cre의 아날로그 loxP 사이트와 유사한 범위에서 분리/해결을 선호하지만, 보다 확장된 48 bp FRT 변형은 Cre-와 PhiC31과 같은 페이징 효소에 대해 설명된 특정 비규칙적 상호작용을 극복하면서 더 높은 수준의 통합을 가능하게 한다.[6]또 다른 장점은 동일한 파트너와 교차하지만 와일드 타입 FRT와는 비교가 되지 않는 이질적인 FRT 사이트를 생성하기 위해 간단한 규칙을 적용할 수 있다는 점이다.이러한 사실은 1994년 이후 재조합형 카세트 교환(RMCE-) 전략의 개발과 지속적인 개선을 가능하게 하여 들어오는 기증 카세트에 대한 표적 카세트의 깨끗한 교환을 가능하게 했다.[6]
그 RMCE 기술을 기반으로,pre-characterized ES-strains의 더 이상의 상술에 적합 특정 리소스는 EUCOMM(조건화 마우스 Mutagenesis 유럽)프로그램의 틀 속에, 지금은 설립되 Cre- 및/또는Flp-based"FlExing"(Flp-mediated excision/inversion)setups,[6]은 절제 관련한 연구에 기반하고 진화해 왔다.에서버전 활동2005년에 시작된 이 프로젝트는 우선 마우스 게놈(IKMC, International Knockout-Mouse Consortium)의 완전한 기능 주석을 가능하게 하기 위해 포화성 돌연변이 유발에 초점을 맞췄으며, 모든 단백질 유전자를 뮤린 ES 세포에서 유전자 트래핑과 표적을 통해 변이시키는 궁극적인 목표를 가지고 있다.[14]이러한 노력들은 "태그"가 새로운 (또는 기존의) 유전 정보를 도입하는 주소 역할을 할 수 있도록 뚜렷한 유전적 사이트에 태그를 붙이는 데 전념하는 다양한 "태그 앤 교환" 전략의 상위를 나타낸다.태그 지정 단계는 기본적으로 단방향으로 작용하는 역방향 통합 또는 PhiC31과 같은 사이트별 통합 선호도를 이용하여 특정 종류의 통합 사이트를 다룰 수 있다.
전통적인 "태그 앤 교환" 절차는 선택 표식 유전자로 구성된 태그를 도입하는 첫 번째 단계인 두 개의 연속적인 동질 재조합(HR-) 단계에 의존했다.그 후 "HR2"는 "GOI"로 마커를 대체하기 위해 사용되었다.첫 번째("knock-out"-) 반응에서 유전자는 선택 가능한 마커(일반적으로 G418 저항을 제공하는 hygtk([+/-]) 카세트를 삽입하여 태그가 지정되었다.다음 "노크인" 단계에서 태그가 붙은 게놈 시퀀스는 특정 돌연변이가 있는 동질 게놈 시퀀스로 대체되었다.그러면 세포 클론은 HSV-tk 유전자("부정 선택")의 상실로 인해 간시클로비에 대한 저항으로 격리될 수 있다.이러한 기존의 2단계 태그 및 교환 절차는 RMCE의 등장 이후 간소화될 수 있으며, 이는 Knock-in 단계에 효율성을 더할 수 있다.
PhiC31 집적효소
의심할 여지 없이, 세르 통합은 대부분 (항상 그렇지는 않지만) 페이지는 AttB가 포함된 기증자 벡터를 유인한다는 점에서 페이지는 ATP 사이트를 모방하는, 잘 이해되는 유전체 수용자 사이트의 제한된 숫자에 트랜스젠다를 통합하기 위한 현재의 선택 도구다.이때 가장 두드러진 구성원은 인간과 쥐 게놈의 맥락에서 잠재력이 입증된 PhiC31-INT이다.
위의 Tyr 재조합과는 반대로 PhiC31-INT는 단방향으로 작용하여 유전적으로 고정된 표적에서 공여 벡터를 단단히 잠근다.이 시스템의 분명한 장점은 수정되지 않은 네이티브 attP(수용자) 및 attB 기증 사이트에 의존할 수 있다는 것이다.(특정 합병증과 함께) 추가적인 이득은 쥐와 인간 게놈의 세포당 제한된 수의 내생성 표적(일명 "attP-pseudosites"라고 함)을 포함하고 있다는 사실에서 발생할 수 있다.이용 가능한 정보에 따르면 상당한 DNA 시퀀스 요구사항으로 인해 통합업체는 레트로바이러스나 심지어 잘 확립된 다수의 유전체 사이트에서 운송 및 삽입을 위한 우수한 운반 차량으로서의 경력을 여는 트랜스포세 기반 통합 시스템보다 적은 수의 사이트를 인식할 수 있으며, 그 중 일부는 소위 "세이프-하버(safe-harbor)"라고 한다.로퍼티[10]
특정 (attP x attB) 재조합 경로의 사실을 이용하여 RMCE는 합성, 이질적 attits에 대한 요구사항 없이 가능해진다.그러나 이러한 분명한 이점은 카세트의 종류 또는 방향성에 대한 통제력 부족과 같은 특정 단점을 희생하여 발생한다.[6]불가역성이 "시리얼 RMCE" 반응, 즉 주어진 유전체 위치에서의 반복적인 카세트 교환을 포함한 표준 멀티플렉싱-RMCE 설정을 허용하지 않는다는 사실에 의해 추가 제한이 부과된다.
전망 및 전망
인간과 쥐 게놈의 주석을 통해 >20,000의 단백질 코딩 유전자와 >3,000의 비코딩 RNA 유전자를 확인하게 되어 유기체의 발달을 수정에서 발생생성까지 안내하고 있다.극적인 진전이 눈에 띄지만, 희귀 유전자 변형의 관련성은 여전히 연구의 중심 주제로 남아 있다.
척추동물 유전자 기능을 대규모로 다루기 위한 가장 중요한 플랫폼 중 하나로 돌연변이 뮤린 ES세포의 게놈 폭의 유전자원들이 확립되었다.이를 위해 유럽과 북미에서 마우스 게놈의 포화 돌연변이 발생을 목표로 하는 4개의 국제 프로그램이 설립되었다(EUCOMM, KOMP, NorCOMM, TIGM). 국제 녹아웃 마우스 컨소시엄(IKSC)이 공동 주관하는 이 ES-세포 저장소는 국제 연구단위 간 교환이 가능하다.현재의 자원은 11개의 539개의 독특한 유전자에 돌연변이를 구성하는데, 이 중 414개는 조건부 유전자다.[14]
관련 기술은 현재 iPS(유인 전능) 지위를 가진 다른 포유류 종과 인간 줄기 세포에 대한 확장을 허용하는 수준에 도달했다.
참고 항목
참조
- ^ Sauer, Brian; Henderson, Nancy (1988). "Site-Specific DNA Recombination in Mammalian Cells by the Cre Recombinase of Bacteriophage P1". Proceedings of the National Academy of Sciences of the United States of America. 85 (14): 5166–70. Bibcode:1988PNAS...85.5166S. doi:10.1073/pnas.85.14.5166. JSTOR 32380. PMC 281709. PMID 2839833.
- ^ O'Gorman, Stephen; Fox, Daniel T.; Wahl, Geoffrey M. (1991). "Recombinase-mediated gene activation and site-specific integration in mammalian cells". Science. 251 (4999): 1351–5. Bibcode:1991Sci...251.1351O. doi:10.1126/science.1900642. JSTOR 2875533. PMID 1900642.
- ^ a b Rajewsky, Klaus (2007). "From a Dream to Reality". European Journal of Immunology. 37: S134–7. doi:10.1002/eji.200737819. PMID 17972357.
- ^ Branda, Catherine S.; Dymecki, Susan M. (2004). "Talking about a RevolutionThe Impact of Site-Specific Recombinases on Genetic Analyses in Mice". Developmental Cell. 6 (1): 7–28. doi:10.1016/S1534-5807(03)00399-X. PMID 14723844.
- ^ a b Nern, A.; Pfeiffer, B. D.; Svoboda, K.; Rubin, G. M. (2011). "Multiple new site-specific recombinases for use in manipulating animal genomes". Proceedings of the National Academy of Sciences. 108 (34): 14198–203. Bibcode:2011PNAS..10814198N. doi:10.1073/pnas.1111704108. PMC 3161616. PMID 21831835.
- ^ a b c d e f Turan, S.; Bode, J. (2011). "Site-specific recombinases: From tag-and-target- to tag-and-exchange-based genomic modifications". The FASEB Journal. 25 (12): 4088–107. doi:10.1096/fj.11-186940. PMID 21891781. S2CID 7075677.
- ^ Bode, Jürgen; Schlake, Thomas; Iber, Michaela; Schübeler, Dirk; Seibler, Jost; Snezhkov, Evgeney; Nikolaev, Lev (2000). "The Transgeneticists Toolbox: Novel Methods for the Targeted Modification of Eukaryotic Genomes". Biological Chemistry. 381 (9–10): 801–13. doi:10.1515/BC.2000.103. PMID 11076013. S2CID 36479502.
- ^ Lyznik, Leszek A.; Mitchell, Jon C.; Hirayama, Lynne; Hodges, Thomas K. (1993). "Activity of yeast FLP recombinase in maize and rice protoplasts". Nucleic Acids Research. 21 (4): 969–75. doi:10.1093/nar/21.4.969. PMC 309231. PMID 8451196.
- ^ Lauth, M.; Spreafico, F; Dethleffsen, K; Meyer, M (2002). "Stable and efficient cassette exchange under non-selectable conditions by combined use of two site-specific recombinases". Nucleic Acids Research. 30 (21): 115e. doi:10.1093/nar/gnf114. PMC 135837. PMID 12409474.
- ^ a b Karow, Marisa; Calos, Michele P (2011). "The therapeutic potential of phiC31 integrase as a gene therapy system". Expert Opinion on Biological Therapy. 11 (10): 1287–96. doi:10.1517/14712598.2011.601293. PMID 21736536. S2CID 2674915.
- ^ Oumard, André; Qiao, Junhua; Jostock, Thomas; Li, Jiandong; Bode, Juergen (2006). "Recommended Method for Chromosome Exploitation: RMCE-based Cassette-exchange Systems in Animal Cell Biotechnology". Cytotechnology. 50 (1–3): 93–108. doi:10.1007/s10616-006-6550-0. PMC 3476001. PMID 19003073.
- ^ a b Leone, Dino P; Genoud, S.Téphane; Atanasoski, Suzana; Grausenburger, Reinhard; Berger, Philipp; Metzger, Daniel; MacKlin, Wendy B; Chambon, Pierre; Suter, Ueli (2003). "Tamoxifen-inducible glia-specific Cre mice for somatic mutagenesis in oligodendrocytes and Schwann cells". Molecular and Cellular Neuroscience. 22 (4): 430–40. doi:10.1016/S1044-7431(03)00029-0. PMID 12727441. S2CID 624620.
- ^ Koenning, M.; Jackson, S.; Hay, C. M.; Faux, C.; Kilpatrick, T. J.; Willingham, M.; Emery, B. (2012). "Myelin Gene Regulatory Factor is Required for Maintenance of Myelin and Mature Oligodendrocyte Identity in the Adult CNS". Journal of Neuroscience. 32 (36): 12528–42. doi:10.1523/JNEUROSCI.1069-12.2012. PMC 3752083. PMID 22956843.
- ^ a b Bradley, Allan; Anastassiadis, Konstantinos; Ayadi, Abdelkader; Battey, James F.; Bell, Cindy; Birling, Marie-Christine; Bottomley, Joanna; Brown, Steve D.; et al. (2012). "The mammalian gene function resource: The international knockout mouse consortium". Mammalian Genome. 23 (9–10): 580–6. doi:10.1007/s00335-012-9422-2. PMC 3463800. PMID 22968824.
- ^ Askew, G. Roger; Doetschman, Thomas; Lingrel, Jerry B. (1993). "Site-directed point mutations in embryonic stem cells: A gene-targeting tag-and-exchange strategy". Molecular and Cellular Biology. 13 (7): 4115–24. doi:10.1128/MCB.13.7.4115. PMC 359961. PMID 8391633.
외부 링크
- http://www.knockoutmouse.org/
- Emes, RD; Goodstadt, L; Winter, EE; Ponting, CP (2003). "Comparison of the genomes of human and mouse lays the foundation of genome zoology". Hum Mol Genet. 12 (7): 701–9. doi:10.1093/hmg/ddg078. PMID 12651866.




