마이크로 어레이 분석 기술
Microarray analysis techniques마이크로어레이 분석기법은 DNA(유전자칩 분석), RNA, 단백질 마이크로어레이 실험으로 생성된 데이터를 해석하는 데 사용되며, 이를 통해 연구자들은 한 번의 [citation needed]실험으로 다수의 유전자(많은 경우 유기체의 전체 게놈)의 발현 상태를 조사할 수 있다.그러한 실험은 연구자들이 세포나 유기체의 전반적인 상태를 평가할 수 있도록 하면서 매우 많은 양의 데이터를 생성할 수 있다.이렇게 많은 양의 데이터는 컴퓨터 프로그램의 도움 없이 분석하는 것은 불가능하지는 않더라도 어렵습니다.
서론
마이크로 어레이 데이터 분석은 마이크로 어레이 칩에 의해 생성된 데이터를 읽고 처리하는 마지막 단계입니다.샘플은 마이크로칩을 이용한 정제, 스캔 등 다양한 과정을 거쳐 컴퓨터 소프트웨어를 통해 처리해야 하는 대량의 데이터를 생성한다.아래 그림과 같이 몇 가지 단계를 수행합니다.스텝 중 하나를 변경하면 분석 결과가 바뀌기 때문에 MAQC 프로젝트는[1] 일련의 표준 전략을 식별하기 위해 작성되었습니다.MAQC 프로토콜을 사용하여 완전한 [2]분석을 수행하는 회사가 존재합니다.
기술
Affymetrix 및 [3]Agilent와 같은 대부분의 마이크로 어레이 제조업체는 마이크로 어레이 제품과 함께 상용 데이터 분석 소프트웨어를 제공합니다.마이크로 어레이 데이터를 분석하기 위해 다양한 방법을 활용하는 오픈 소스 옵션도 있습니다.
집약과 정규화
두 개의 다른 어레이 또는 동일한 어레이에 혼성된 두 개의 다른 샘플을 비교하는 것은 일반적으로 절차와 염료 강도 효과의 차이로 인한 시스템 오류에 대한 조정을 포함합니다.두 가지 색상 배열에 대한 염료 정규화는 종종 국소 회귀를 통해 이루어집니다.LIMMA는 배경 보정 및 스케일링을 위한 도구 세트를 제공하며, 슬라이드 상의 중복 스팟을 [4]평균화하는 옵션도 제공합니다.배열이 얼마나 잘 정규화되어 있는지를 평가하는 일반적인 방법은 데이터의 MA 그림을 표시하는 것입니다.MA 그림은 R, MATLAB 및 Excel과 같은 프로그램 및 언어를 사용하여 생성할 수 있습니다.
Raw Affy 데이터에는 동일한 RNA 표적에 대한 프로브가 약 20개 포함되어 있습니다.이 중 절반은 목표 시퀀스와 정확하게 일치하지 않는 "불일치 지점"입니다.이들은 이론적으로 특정 대상에 대한 비특이적 결합의 양을 측정할 수 있습니다.RMA(Robust Multi-Array Average)는 이러한 미스매치 스팟을 이용하지 않는 정규화 어프로치이지만, 중간값의 [6]광택을 통해 완전한 일치를 요약해야 합니다.중위수 다듬기 알고리즘은 강력하지만 [7]분석된 표본 수에 따라 다르게 작동합니다.또한 RMA의 일부이기도 한 정량 정규화는 더 많은 비교를 위해 배지를 정규화하는 합리적인 접근법 중 하나입니다.
현재 Affymetrix MAS5 알고리즘은 완전일치 프로브와 미스매치 프로브를 모두 사용하고 있어 계속 인기를 끌고 있으며 헤드 투 헤드 [8]테스트에서도 좋은 결과를 얻고 있습니다.
FARMS([9]Factor Analysis for Robust Microarray Summaryization)는 어레이 데이터를 완전 일치 프로브 수준에서 요약하기 위한 모델 기반 기술입니다.이는 베이지안 최대 사후 방법이 가우스 측정 노이즈를 가정하여 모델 매개변수를 최적화하는 인자 분석 모델에 기초한다.Affycomp[10] 벤치마크에 따르면 FARMS는 민감도 및 특수성 측면에서 다른 모든 요약 방법을 능가합니다.
유의미분식의 식별
비정상적인 수준의 과잉 표현 또는 과소 표현을 나타내는 어레이 프로브를 식별하기 위한 많은 전략이 있습니다.가장 간단한 방법은 치료 그룹 간에 평균 2배 이상 차이가 나는 모든 프로브를 "중대"라고 부르는 것입니다.보다 정교한 접근법은 종종 효과 크기와 가변성을 모두 고려하는 t-테스트 또는 기타 메커니즘과 관련이 있다.특이하게도, 특정 유전자와 관련된 p-값은 반복 실험 사이에 잘 재생되지 않으며, 직선 접힘 변화에 의해 생성된 리스트는 훨씬 더 [11][12]잘 수행된다.실험을 수행하는 지점이 일반적인 동작을 예측하는 것과 관련이 있기 때문에 이것은 매우 중요한 관찰을 나타냅니다.MAQC 그룹은 폴드 변화 평가와 비스트링 p-값 컷오프를 사용할 것을 권고하며, 배경 보정 및 스케일링 프로세스의 변화는 폴드 변화 차이의 순위 순서에는 최소한의 영향만 미치지만 p-값에는 상당한 영향을 미친다는 점을 추가로 지적한다.
클러스터링
클러스터링은 유사한 발현 패턴을 가진 유전자를 그룹화하기 위해 사용되는 데이터 마이닝 기술이다.계층적 군집화 및 k-평균 군집화는 마이크로 어레이 분석에서 널리 사용되는 기법입니다.
계층 클러스터링
계층적 군집화는 비교적 균일한 군집을 찾기 위한 통계적 방법입니다.계층형 클러스터링은 두 개의 개별 단계로 구성됩니다.우선 유전자 간의 모든 쌍방향 거리를 포함하는 거리행렬을 계산한다.피어슨의 상관관계와 스피어맨의 상관관계는 종종 차이점 추정치로 사용되지만 맨해튼 거리나 유클리드 거리 같은 다른 방법도 적용할 수 있습니다.사용 가능한 거리 측정의 수와 클러스터링 알고리즘 결과에 미치는 영향을 고려할 때, 여러 연구는 마이크로 어레이 데이터의 고유한 특성과 [13][14][15]소음에 대한 견고성을 고려하여 마이크로 어레이 데이터의 클러스터링을 위한 다른 거리 측정을 비교하고 평가하였다.초기 거리 매트릭스 계산 후 계층형 클러스터링 알고리즘은 (A) 단일 데이터 포인트에서 시작하는 가장 가까운 두 개의 클러스터를 반복적으로 결합하거나 (B) 전체 집합에서 시작하는 파티션 클러스터(분할, 하향식 접근)를 반복 결합한다.각 단계가 끝나면 새로 형성된 클러스터와 다른 클러스터 간의 새로운 거리 행렬이 다시 계산됩니다.계층 클러스터 분석 방법에는 다음이 포함됩니다.
- 단일 링크(최소 방식, 가장 가까운 네이버)
- 평균 링크(UPGMA).
- 완전한 링크(최대 방식, 가장 먼 네이버)
다른 연구들은 이미 단일 연결 클러스터링 알고리즘이 유전자 발현 마이크로 어레이 데이터에 사용될 때 좋지 않은 결과를 생성하므로 [15][16]피해야 한다는 것을 경험적으로 보여주었다.
K-평균 군집화
K-평균 군집화는 패턴을 기반으로 유전자 또는 샘플을 K 그룹으로 그룹화하는 알고리즘입니다.그룹화는 데이터와 해당 군집 중심 사이의 거리 제곱의 합을 최소화하여 수행됩니다.따라서 K-평균 군집화의 목적은 유사한 [17]식을 기반으로 데이터를 분류하는 것입니다.K-평균 클러스터링 알고리즘과 그 변형체(k-medoids 포함) 중 일부는 유전자 발현 데이터에 대해 좋은 결과를 생성하는 것으로 나타났다(적어도 계층적 클러스터링 방법보다 더 좋다).k-평균, k-중위수, 계층적 방법 및 다른 거리 측정의 경험적 비교는 [15][16]문헌에서 확인할 수 있다.
패턴 인식
Inneguity[18] and Pathway[19] 스튜디오와 같은 유전자 네트워크 분석을 위한 상업적인 시스템은 현재의 과학 문헌에 기초하여 다르게 발현되는 유전자의 시각적 표현을 만듭니다.FunRich,[20] GenMAPP 및 Moksiskaan과 같은 비상업적 도구는 또한 하나 또는 여러 개의 마이크로 어레이 실험에서 얻은 유전자 네트워크 데이터를 정리하고 시각화하는 데 도움을 준다.R 프로그래밍 언어로 작성된 바이오컨덕터를 통해 다양한 마이크로 어레이 분석 도구를 사용할 수 있습니다.자주 인용되는 SAM 모듈 및 기타 마이크로 어레이[21] 툴은 스탠포드 대학에서 이용할 수 있습니다.또 다른 세트는 하버드와 [22]MIT에서 구할 수 있습니다.
특정 표현형과 관련된 유전자 또는 유전자 세트를 식별하는 데 도움을 주기 위해 기준 상태에 상대적인 마이크로 어레이 실험에서 유전자의 과잉 또는 과소 발현 정도를 결정하기 위한 통계 분석을 위한 특수 소프트웨어 도구도 개발되었다.유전자 세트 농축 분석(GSEA)으로 알려진 그러한 분석 방법 중 하나는 함께 [23]조절되는 유전자 그룹을 식별하기 위해 콜모고로프-스미르노프 스타일의 통계를 사용한다.이 서드파티 통계 패키지는 NCBI의 GenBank와 같은 데이터베이스 및 Biocarta[24] 및 Gene Ontology와 같은 큐레이티드 데이터베이스의 엔트리에 대한 링크를 포함하여 유전자 또는 유전자 세트에 대한 사용자 정보를 제공합니다.단백질 복합체 농축 분석 도구(COMPLEAT)는 단백질 [25]복합체 수준에서 유사한 농축 분석을 제공합니다.이 도구는 다양한 조건 또는 시점에서의 동적 단백질 복합체 조절을 식별할 수 있습니다.관련[26] 시스템, PAINT 및[27] SCOPE는 유전자 프로모터 영역에 대한 통계 분석을 수행하여 이전에 확인된 전사 인자 응답 요소의 과잉 및 과소 표현을 식별한다.또 다른 통계 분석 도구는 유전자 세트 수집을 위한 RsGsc(Rank Sum Statistics for Gene Set Collections)로, 실험 [28]데이터를 설명하는 유전자 세트를 찾기 위해 순위 합 확률 분포 함수를 사용합니다.또 다른 접근법은 맥락 메타 분석, 즉 유전자 클러스터가 다양한 실험 맥락에 어떻게 반응하는지 알아내는 것이다.Genevestigator는 해부학적 부분, 발달 단계 및 질병, 화학 물질, 스트레스 및 신생물 반응과 같은 맥락에 걸쳐 맥락 메타 분석을 수행하는 공용 도구입니다.
마이크로어레이의 유의성 분석(SAM)
미세 배열의 유의성 분석(SAM)은 유전자 발현 변화가 통계적으로 유의한지 여부를 판단하기 위해 버지니아 터셔, 로버트 티비라니, 길버트 추에 의해 2001년에 확립된 통계 기법이다.DNA 마이크로어레이의 등장으로, 이제 단일 교배 실험에서 수천 개의 유전자의 발현을 측정할 수 있게 되었다.생성되는 데이터는 상당하며, 무엇이 중요하고 무엇이 중요하지 않은지를 선별하는 방법이 필수적이다.SAM은 Stanford University에 의해 R패키지 형태로 배포됩니다.
SAM은 유전자 특이 t-테스트를 실시함으로써 통계적으로 유의한 유전자를 식별하고 유전자 발현과 반응 [29][30][31]변수 사이의 관계의 강도를 측정하는 각 유전자 j에 대한 통계j d를 계산한다.데이터가 정규 분포를 따르지 않을 수 있으므로 이 분석에서는 비모수 통계량을 사용합니다.반응 변수는 실험 조건을 기반으로 데이터를 설명하고 그룹화합니다.이 방법에서는 데이터의 반복된 배열을 사용하여 어떤 유전자의 발현도 반응에 관련되어 있는지 여부를 판단한다.치환 기반 분석의 사용은 유전자의 상관관계를 설명하고 개별 유전자의 분포에 대한 파라메트릭 가정을 회피한다.이는 유전자의 [32]등분산 및/또는 독립성을 가정하는 다른 기술(예: 분산 분석 및 본페로니)에 비해 유리하다.
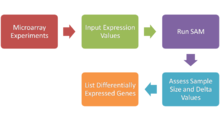
기본 프로토콜
- 마이크로 어레이 실험 실행(올리고 및 cDNA 프라이머를 사용한 DNA 마이크로 어레이, SNP 어레이, 단백질 어레이 등)
- Microsoft Excel에서의 입력식 분석 - 아래 참조
- Microsoft Excel 애드인으로 SAM 실행
- SAM Plot Controller(SAM 플롯 컨트롤러)에서 평균 발현 차이를 계산하여 상당한 수의 유전자와 FDR(허용 가능한 거짓 발견 속도) 및 Assess Sample Size(샘플 크기 평가)를 얻도록 Delta 조정 매개 변수를 조정합니다.
- 차분 발현 유전자 목록(양성 및 음성 발현 유전자)
SAM 실행
- SAM은 등록 절차 완료 후 http://www-stat.stanford.edu/~tibs/SAM/에서 온라인으로 다운로드할 수 있습니다.
- SAM은 Excel Add-In으로 실행되며 SAM 플롯컨트롤러에서는 False Discovery Rate와 Delta를 커스터마이즈할 수 있습니다.SAM 플롯과 SAM 출력 기능에서는 중요한 유전자 목록, 델타 테이블 및 샘플크기 평가가 생성됩니다.
- 순열은 표본 수를 기반으로 계산됩니다.
- 블록 순열
SAM을 실행하기 위해 데이터 세트의 올바른 값을 지정할 때 사용자가 순열 수를 설정합니다.
응답 형식
종류:[29]
- 정량적 - 실제 값(심박수 등)
- 한 클래스 - 평균 유전자 발현과 0이 다른지 여부를 테스트합니다.
- 2개의 클래스 - 2개의 측정 세트
- 비쌍성 — 측정 단위는 두 그룹에서 다르다. 예를 들어, 서로 다른 환자의 샘플을 가진 대조군과 치료군
- 쌍체 - 동일한 실험 단위를 두 그룹으로 측정한다. 예를 들어, 동일한 환자의 치료 전후의 샘플
- 멀티클래스 - 각각 다른 실험 단위를 포함하는 두 개 이상의 그룹, 쌍이 없는 두 개의 클래스 유형 일반화
- 생존 - 이벤트(사망 또는 재발 등)까지의 시간 데이터
- 시간 과정 - 각 실험 단위는 여러 시점에서 측정됩니다.실험 단위는 1개 또는 2개의 클래스 설계로 분류됩니다.
- 패턴 검출 - 명시적 응답 파라미터는 지정되지 않습니다.사용자는 식 데이터의 eigengene(주요 컴포넌트)을 지정하여 정량적 응답으로 취급합니다.
알고리즘.
SAM은 발현 데이터의 치환 해석에 근거해 유전자 발현의 상대적 차이에 관한 테스트 통계량을 산출해 오발견률을 산출한다.프로그램의 주요 계산은 다음과 같습니다.[29][30][31]
so 상수는 d.r의i 변동i 계수를 최소화하기 위해 선택된다. r은 y 실험 조건에서 유전자 i의 발현 수준(x)과 같다.
적어도 소정의 양 이상의 유의한 변화라고 불리는 유전자를 보증하기 위해 접힘 변화(t)를 특정한다.즉, 두 가지 조건 각각에서 유전자의 평균 발현 수준의 절대값이 폴드 변화(t)보다 커야 양성이며 폴드 변화(t)의 역치보다 작아야 음성이 된다.
SAM 알고리즘은 다음과 같이 기술할 수 있습니다.
- 크기에 따라 검정 통계의 순서 지정
- 각 치환에 대해 순서가 매겨진 null(해당되지 않음) 점수를 계산합니다.
- 순서 있는 검정 통계량을 기대 Null 점수에 대해 그림으로 표시
- 해당 유전자에 대한 테스트 통계량에서 해당 유전자에 대한 평균 테스트 통계량을 뺀 절대값이 명시된 임계값보다 클 경우 각 유전자를 유의하다고 부른다.
- 예측값과 관측값을 바탕으로 오검출률 추정
산출량
- 유의한 유전자 세트
- 양성 유전자 세트 - 유전자 세트 내 대부분의 유전자의 높은 발현과 표현형의 높은 값과의 상관관계y
- 음성 유전자 세트 - 유전자 세트 내 대부분의 유전자의 낮은 발현과 높은 표현형 값의 상관관계y
SAM 기능
- SAM에서는[30][31] Oligo 또는 cDNA 어레이, SNP 어레이, 단백질 어레이 등의 데이터를 이용할 수 있습니다.
- 발현 데이터를 임상 매개[32] 변수와 연관시킵니다.
- 식 데이터와 시간[29] 상관 관계
- 데이터 치환을 사용하여 여러[30][31][32][35] 테스트의 오검출률을 추정합니다.
- 로컬 오검출률(유전자와 [29]유사한i d를 가진 유전자의 FDR) 및 누락률을 보고합니다.
- 처리를 어레이의[29] 다른 배치 내에서 적용할 경우 블록 설계로 작업할 수 있습니다.
- 유의라고[29] 불리는 유전자 수를 결정하는 임계값을 조정할 수 있습니다.
오류 수정 및 품질 관리
품질 관리
전체 어레이는 육안 검사, 동일한 실험 그룹의 어레이와의 쌍별 비교 또는 RNA 열화 [36]분석을 통해 탐지할 수 있는 명백한 결함이 있을 수 있습니다.이러한 어레이를 분석에서 완전히 삭제함으로써 결과가 개선될 수 있습니다.
배경 보정
어레이의 종류에 따라 형광체의 비특이적 결합에 관련된 신호를 감산하여 보다 나은 결과를 얻을 수 있다.한 가지 방법은 지점 간 영역의 평균 신호 강도를 빼는 것입니다.배경 보정 및 추가 분석을 위한 다양한 도구는 [38]TIGR,[37] 애질런트(GeneSpring) 및 Ocimum Bio Solutions(Genowiz)[39]에서 제공합니다.
스폿 필터링
인쇄 또는 세척 결함과 같은 국소 아티팩트의 시각적 식별도 마찬가지로 개별 스폿 제거를 제안할 수 있습니다.이 작업은 어레이 제조 품질에 따라 상당한 시간이 걸릴 수 있습니다.또, 일부의 순서에서는, 특정의 강도 역치를 밑도는 표현치를 가지는 모든 스팟을 삭제할 필요가 있습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Dr. Leming Shi, National Center for Toxicological Research. "MicroArray Quality Control (MAQC) Project". U.S. Food and Drug Administration. Retrieved 2007-12-26.
- ^ "GenUs BioSystems - Services - Data Analysis". Retrieved 2008-01-02.
- ^ "Agilent DNA Microarrays". Archived from the original on December 22, 2007. Retrieved 2008-01-02.
- ^ "LIMMA Library: Linear Models for Microarray Data". Retrieved 2008-01-01.
- ^ Irizarry, RA; Hobbs, B; Collin, F; Beazer-Barclay, YD; Antonellis, KJ; Scherf, U; Speed, TP (2003). "Exploration, normalization, and summaries of high density oligonucleotide array probe level data". Biostatistics. 4 (2): 249–64. doi:10.1093/biostatistics/4.2.249. PMID 12925520.
- ^ Bolstad BM, Irizarry RA, Astrand M, Speed TP (2003). "A comparison of normalization methods for high density oligonucleotide array data based on variance and bias". Bioinformatics. 19 (2): 185–93. doi:10.1093/bioinformatics/19.2.185. PMID 12538238.
- ^ Giorgi FM, Bolger AM, Lohse M, Usadel B (2010). "Algorithm-driven Artifacts in median polish summarization of Microarray data". BMC Bioinformatics. 11: 553. doi:10.1186/1471-2105-11-553. PMC 2998528. PMID 21070630.
- ^ Lim WK, Wang K, Lefebvre C, Califano A (2007). "Comparative analysis of microarray normalization procedures: effects on reverse engineering gene networks". Bioinformatics. 23 (13): i282–8. doi:10.1093/bioinformatics/btm201. PMID 17646307.
- ^ Hochreiter S, Clevert DA, Obermayer K (2006). "A new summarization method for affymetrix probe level data". Bioinformatics. 22 (8): 943–949. doi:10.1093/bioinformatics/btl033. PMID 16473874.
- ^ "Affycomp III: A Benchmark for Affymetrix GeneChip Expression Measures".
- ^ Shi L, Reid LH, Jones WD, et al. (2006). "The MicroArray Quality Control (MAQC) project shows inter- and intraplatform reproducibility of gene expression measurements". Nat. Biotechnol. 24 (9): 1151–61. doi:10.1038/nbt1239. PMC 3272078. PMID 16964229.
- ^ Guo L, Lobenhofer EK, Wang C, et al. (2006). "Rat toxicogenomic study reveals analytical consistency across microarray platforms". Nat. Biotechnol. 24 (9): 1162–9. doi:10.1038/nbt1238. PMID 17061323. S2CID 8192240.
- ^ Gentleman, Robert; et al. (2005). Bioinformatics and computational biology solutions using R and Bioconductor. New York: Springer Science+Business Media. ISBN 978-0-387-29362-2.
- ^ Jaskowiak, Pablo A.; Campello, Ricardo J.G.B.; Costa, Ivan G. (2013). "Proximity Measures for Clustering Gene Expression Microarray Data: A Validation Methodology and a Comparative Analysis". IEEE/ACM Transactions on Computational Biology and Bioinformatics. 10 (4): 845–857. doi:10.1109/TCBB.2013.9. PMID 24334380. S2CID 760277.
- ^ a b c Jaskowiak, Pablo A; Campello, Ricardo JGB; Costa, Ivan G (2014). "On the selection of appropriate distances for gene expression data clustering". BMC Bioinformatics. 15 (Suppl 2): S2. doi:10.1186/1471-2105-15-S2-S2. PMC 4072854. PMID 24564555.
- ^ a b de Souto, Marcilio C. P.; Costa, Ivan G.; de Araujo, Daniel S. A.; Ludermir, Teresa B.; Schliep, Alexander (2008). "Clustering cancer gene expression data: a comparative study". BMC Bioinformatics. 9 (1): 497. doi:10.1186/1471-2105-9-497. PMC 2632677. PMID 19038021.
- ^ http://www.biostat.ucsf.edu
- ^ "Ingenuity Systems". Retrieved 2007-12-31.
- ^ "Ariadne Genomics: Pathway Studio". Archived from the original on 2007-12-30. Retrieved 2007-12-31.
- ^ "FunRich: Functional Enrichment Analysis". Retrieved 2014-09-09.
- ^ ["Significance Analysis of Microarrays". Retrieved 2007-12-31. ]
- ^ "Software - Broad". Retrieved 2007-12-31.
- ^ Subramanian A, Tamayo P, Mootha VK, et al. (2005). "Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles". Proc. Natl. Acad. Sci. U.S.A. 102 (43): 15545–50. doi:10.1073/pnas.0506580102. PMC 1239896. PMID 16199517.
- ^ "BioCarta - Charting Pathways of Life". Retrieved 2007-12-31.
- ^ Vinayagam A, Hu Y, Kulkarni M, Roesel C, et al. (2013). "Protein Complex-Based Analysis Framework for High-Throughput Data Sets. 6, rs5 (2013)". Sci. Signal. 6 (r5): rs5. doi:10.1126/scisignal.2003629. PMC 3756668. PMID 23443684.
- ^ "DBI Web". Archived from the original on 2007-07-05. Retrieved 2007-12-31.
- ^ "SCOPE". Archived from the original on 2011-08-17. Retrieved 2007-12-31.
- ^ "RssGsc". Retrieved 2008-10-15.
- ^ a b c d e f g h i Chu, G., Narasimhan, B, Tibshirani, R, Tusher, V. "SAM "Significantance Analysis of Microarays" 사용자 가이드 및 기술 문서 [1]
- ^ a b c d e f g h i Zang, S.; Guo, R.; et al. (2007). "Integration of statistical inference methods and a novel control measure to improve sensitivity and specificity of data analysis in expression profiling studies". Journal of Biomedical Informatics. 40 (5): 552–560. doi:10.1016/j.jbi.2007.01.002. PMID 17317331.
- ^ a b c d e f g h i <Zhang, S. (2007년)"SAM, SAM R 패키지에 대한 포괄적인 평가 및 성능 향상을 위한 간단한 수정." BMC BioInformatics 8: 230.
- ^ a b c Tusher, V. G.; Tibshirani, R.; et al. (2001). "Significance analysis of microarrays applied to the ionizing radiation response" (PDF). Proceedings of the National Academy of Sciences. 98 (9): 5116–5121. Bibcode:2001PNAS...98.5116G. doi:10.1073/pnas.091062498. PMC 33173. PMID 11309499.
- ^ Dinu, I. P.; JD; Mueller, T; Liu, Q; Adewale, AJ; Jhangri, GS; Einecke, G; Famulski, KS; Halloran, P; Yasui, Y. (2007). "Improving gene set analysis of microarray data by SAM-GS". BMC Bioinformatics. 8: 242. doi:10.1186/1471-2105-8-242. PMC 1931607. PMID 17612399.
- ^ Jeffery, I. H.; DG; Culhane, AC. (2006). "Comparison and evaluation of methods for generating differentially expressed gene lists from microarray data". BMC Bioinformatics. 7: 359. doi:10.1186/1471-2105-7-359. PMC 1544358. PMID 16872483.
- ^ Larsson, O. W. C; Timmons, JA. (2005). "Considerations when using the significance analysis of microarrays (SAM) algorithm". BMC Bioinformatics. 6: 129. doi:10.1186/1471-2105-6-129. PMC 1173086. PMID 15921534.
- ^ Wilson CL, Miller CJ (2005). "Simpleaffy: a BioConductor package for Affymetrix Quality Control and data analysis". Bioinformatics. 21 (18): 3683–5. doi:10.1093/bioinformatics/bti605. PMID 16076888.
- ^ "J. Craig Venter Institute -- Software". Retrieved 2008-01-01.
- ^ "Agilent GeneSpring GX". Retrieved 2008-01-02.
- ^ "Ocimum Biosolutions Genowiz". Archived from the original on 2009-11-24. Retrieved 2009-04-02.
외부 링크
- ArrayExplector - 마이크로 어레이를 나란히 비교하여 연구 요구에 가장 적합한 어레이를 찾을 수 있습니다.
- FARMS - 견고한 마이크로 어레이 집약에 대한 인자 분석, R 패키지: 소프트웨어
- StatsArray - 온라인 마이크로 어레이 분석 서비스 - 소프트웨어
- ArrayMining.net - 마이크로 어레이 데이터 온라인 분석용 웹 애플리케이션 - 소프트웨어
- FunRich - 유전자 세트 농축 분석 실행 - 소프트웨어
- 생명과학 레퍼런스 모듈의 전사체 비교 분석
- SAM 다운로드 순서
- GeneChip® 발현 분석-데이터 분석 기초(Affymetrix 기준)
- Duke data_analysis_fundamentals_manual