게놈 전체 연관성 연구
Genome-wide association study유전체학에서 전체 게놈 연관 연구(WGA 연구, 또는 WGAS)라고도 알려진 게놈 전체 연관 연구(GWA 연구, 또는 GWAS)는 어떤 변종이 어떤 특성과 연관되어 있는지를 확인하기 위해 다른 개인들의 게놈 전체 유전자 변이들의 집합체를 관찰한 연구다.GWA 연구는 일반적으로 단일 뉴클레오티드 다형성(SNP)과 주요 인간 질병과 같은 특징들 사이의 연관성에 초점을 맞추고 있지만, 다른 유전적 변종과 다른 유기체에도 동일하게 적용될 수 있다.
인간 데이터에 적용할 때, GWA 연구는 특정 특성이나 질병에 대해 다양한 표현형을 가진 참가자들의 DNA를 비교한다.이러한 참가자는 질병(사례)이 있는 사람일 수도 있고, 질병(통제)이 없는 유사한 사람일 수도 있고, 혈압과 같은 특정 성질에 대해 다른 표현형을 가진 사람일 수도 있다.이 접근방식은 유전자형 우선과는 반대로 참가자를 임상적 발현에 의해 먼저 분류하는 표현형 우선이라고 알려져 있다.각 사람은 DNA 샘플을 제공하는데, 이 샘플에서 수백만 개의 유전자 변형이 SNP 배열을 사용하여 판독된다.한 종류의 변종(한 개의 알레르)이 질병에 걸린 사람들에게 더 자주 발생한다면, 그 변종은 질병과 관련이 있다고 한다.관련 SNP는 질병의 위험에 영향을 미칠 수 있는 인간 게놈의 영역을 표시하는 것으로 간주된다.
GWA 연구는 소수의 사전 지정된 유전 영역을 구체적으로 테스트하는 방법과 대조적으로 전체 게놈을 조사한다.따라서 GWAS는 유전자 특정 후보 주도 연구와는 대조적으로 비후보 주도 접근방식이다.GWA 연구는 질병과 관련된 DNA에서 SNP와 다른 변종을 확인하지만, 그들은 어떤 유전자가 인과인지 그들 스스로 특정할 수는 없다.[1][2][3]
2002년에 발표된 첫 번째 성공적인 GWAS는 심근경색을 연구했다.[4]이후 이 연구 설계는 연령 관련 황반변성 환자를 조사한 랜드마크 GWA 2005 연구에서 구현되었으며, 건강한 대조군에 비해 알레르기가 상당히 변경된 두 개의 SNP를 발견했다.[5]2017년[update] 현재 3,000개 이상의 인간 GWA 연구가 1,800개 이상의 질병과 특성을 조사했으며, 수천 개의 SNP 연관성이 발견되었다.[6]희귀한 유전질환의 경우를 제외하고는 이러한 연관성은 매우 약하지만, 위험의 상당 부분을 설명하지는 못하지만, 중요한 유전자와 경로에 대한 통찰력을 제공한다.
배경

어떤 두 개의 인간 게놈도 수백만 가지의 다른 방식으로 다르다.게놈(SNP)의 개별 뉴클레오티드는 물론 삭제, 삽입, 복사 번호 변동 등 더 큰 변형이 많다.이들 중 어떤 것이든 개인의 특성, 즉 표현형에 변화를 일으킬 수 있는데, 이것은 질병 위험에서부터 키와 같은 물리적 특성에 이르는 모든 것이 될 수 있다.[8]GWA 연구가 도입되기 전인 2000년을 전후해 1차적인 조사방법은 가족 내 유전적 연계에 대한 상속 연구를 통한 것이었다.이 접근법은 단일 유전자 장애에 매우 유용하다는 것이 입증되었다.[9][8][10]그러나 일반적이고 복잡한 질병의 경우 유전적 연계 연구의 결과는 번식하기 어려운 것으로 판명되었다.[8][10]연계 연구의 제안된 대안은 유전적 연관성 연구였다.이 연구 유형은 관심의 표현형(예를 들어 질병이 연구되고 있는 경우)을 가진 개인에서 유전자 변종의 대립이 예상보다 더 자주 발견되는지를 묻는다.통계적 힘에 대한 초기 계산은 이 접근법이 약한 유전적 영향을 탐지하는 연계 연구보다 나을 수 있다는 것을 보여주었다.[11]
개념 체계 외에도 몇 가지 추가 요소들이 GWA 연구를 가능하게 했다.하나는 연구를 위해 충분한 수의 생물학적 표본을 수집하는 비용과 어려움을 크게 줄인 인간 유전 물질의 보고인 바이오뱅크의 출현이었다.[12]또 다른 하나는 국제합동지도프로젝트였는데, 2003년부터 GWA 연구에서 조사된 일반 SNP의 대다수를 확인했다.[13]HapMap 프로젝트에 의해 식별된 HapLock 구조는 또한 대부분의 변동을 설명하는 SNP의 하위 집합에 초점을 맞출 수 있었다.또한 유전자형 어레이를 사용하여 이러한 모든 SNP를 유전자형으로 만드는 방법의 개발은 중요한 전제조건이었다.[14]
방법들
GWA 연구의 가장 일반적인 접근방식은 환자-대조군 설정이다. 환자-대조군 두 개, 건강한 대조군 한 개, 질병의 영향을 받는 환자군 한 개를 비교한다.각 그룹의 모든 개인은 일반적으로 알려진 대다수의 SNP에 대해 유전자형이다.정확한 SNP의 수는 유전자형 기술에 따라 다르지만, 일반적으로 백만 개 이상이다.[7]이러한 각 SNP에 대해 사례와 대조군 그룹 간에 알레르기가 유의하게 변화하는지 여부를 조사한다.[16]이러한 설정에서 효과 크기를 보고하는 기본 단위는 승산비다.승산비는 두 개의 오즈의 비율인데, GWA 연구의 맥락에서 특정 알레르기가 있는 개인의 경우 사례의 확률과 동일한 알레르기가 없는 개인의 경우 사례의 확률이다.
예제: 두 개의 알레르기가 있다고 가정합시다. T와 C.사례집단의 개인수는 'A'로, 대조집단의 개인수는 'B'로 나타낸다.마찬가지로, 사례군에서 C가 있는 개인의 수는 'X'로, C가 있는 대조군에서 개인 수는 'Y'로 표시된다.이 경우 알레르 T의 승산비는 A:B(표준 오즈 용어로 'A~B'를 의미함)로 X:Y로 나눈 값이며, 수학 표기법으로는 단순히 (A/B)/(X/Y)이다.
사례군에서 발생 빈도가 대조군보다 훨씬 높은 경우, 승산비는 1보다 높으며, 낮은 발생 빈도의 경우 그 반대의 경우도 마찬가지다.또한 승산비의 유의성에 대한 P-값은 일반적으로 단순 카이-제곱 검정을 사용하여 계산된다.1과 현저히 다른 승산비를 찾는 것은 GWA 연구의 목적이다. 왜냐하면 이것은 SNP가 질병과 연관되어 있다는 것을 보여주기 때문이다.[16]매우 많은 변형이 시험되기 때문에 변형을 유의미하게 고려하기 위해 p-값이 5×10보다−8 낮도록 요구하는 것이 표준 관행이다.
사례-제어 접근방식의 변화.환자-대조군 GWA 연구의 일반적인 대안은 높이, 바이오마커 농도 또는 유전자 발현과 같은 정량적 표현형 데이터의 분석이다.마찬가지로 우위 또는 열성 침투 패턴을 위해 설계된 대체 통계를 사용할 수 있다.[16]계산은 일반적으로 SNPTEST와 PLINK와 같은 생물정보학 소프트웨어를 사용하여 수행되며, 이 소프트웨어에는 이러한 대체 통계 중 많은 부분에 대한 지원도 포함된다.[15][17]GWAS는 개별 SNP의 효과에 초점을 맞춘다.그러나, 둘 이상의 SNP들 사이의 복잡한 상호작용, 즉 인식은 복잡한 질병의 원인이 될 수도 있다.잠재적으로 지수적인 교호작용 수로 인해 GWAS 데이터에서 통계적으로 유의한 교호작용을 검출하는 것은 계산적으로나 통계적으로 어려운 일이다.이 작업은 데이터 마이닝에서 영감을 받은 알고리즘을 사용하는 기존 출판물에서 다루어졌다.[18]게다가, 연구원들은 GWA 데이터를 단백질-단백질 상호작용 네트워크와 같은 다른 생물학적 데이터와 통합하여 더 유익한 결과를 도출하려고 노력한다.[19][20]
대부분의 GWA 연구의 주요 단계는 연구에 사용된 유전자형 칩이 아닌 SNP에서 유전자형의 귀속이다.[21]이 과정은 연관성을 시험할 수 있는 SNP의 수를 크게 증가시키고, 연구의 힘을 증가시키며, 구별되는 코호트에 걸쳐 GWAS의 메타분석을 용이하게 한다.유전자형 귀납은 GWAS 데이터와 하플라타입의 기준 패널을 결합한 통계적 방법에 의해 수행된다.이 방법들은 알레르기를 유발하기 위해 짧은 순서에 걸쳐 개인들 간에 happlotype을 공유하는 것을 이용한다.유전자형 귀신을 위한 기존 소프트웨어 패키지에는 IMMUTUT2,[22] Minimac, Beagle[23], MaCH 등이 있다.[24]
연관성 계산 외에도 결과를 잠재적으로 혼동할 수 있는 변수를 고려하는 것이 일반적이다.성별과 나이는 교란 변수의 일반적인 예다.게다가 많은 유전적 변이가 돌연변이가 처음 발생한 지리적, 역사적 인구와 연관되어 있다는 것도 알려져 있다.[25]이러한 연관성 때문에, 연구는 인구 계층화라고 불리는 것을 통제함으로써 참가자들의 지리적, 민족적 배경을 고려해야 한다.만약 그들이 그렇게 하지 못한다면, 이러한 연구들은 잘못된 양성 결과를 낳을 수 있다.[26]
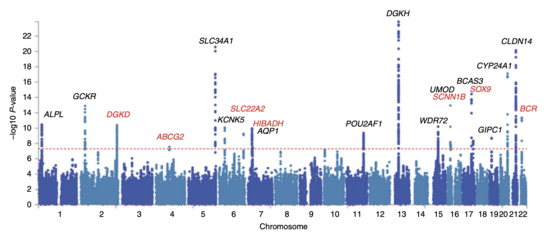
모든 SNP에 대해 오즈 비와 P 값이 계산된 후, 공통 접근법은 맨해튼 플롯을 만드는 것이다.GWA 연구의 맥락에서 이 그래프는 게놈 위치의 함수로서 P-값의 음수 로그를 보여준다.따라서 가장 유의한 연관성을 갖는 SNP가 그림에서 두드러지게 나타나며, 대개 하플로블록 구조 때문에 점의 스택으로 나타난다.중요한 것은 유의성에 대한 P-값 임계값이 여러 시험 문제에 대해 수정된다는 것이다.정확한 임계값은 연구에 따라 다르지만,[27] 기존의 게놈 전체 중요도 임계값은 5×10으로−8 수십만에서 수백만 개의 SNP 테스트에 직면했을 때 유의할 것이다.[7][16][28]GWA 연구는 일반적으로 발견 코호트에서 첫 번째 분석을 수행한 후 독립 유효성 검사 코호트에서 가장 중요한 SNP의 유효성 검사를 수행한다.[29]
결과.
GWA 연구에서 확인된 SNP의 포괄적인 카탈로그를 만들기 위한 시도가 있었다.[31]2009년 현재 질병과 관련된 SNP는 수천 개에 달한다.[32]
2005년에 시행된 첫 번째 GWA 연구는 연령 관련 황반변성(ARMD) 환자 96명과 50명의 건강한 대조군을 비교했다.[33]그것은 두 그룹 사이에 알레르기가 상당히 변경된 두 개의 SNP를 식별했다.이러한 SNP는 유전자 부호화 보완 인자 H에 위치했는데, ARMD의 연구에서 뜻밖의 발견이었다.이러한 첫 번째 GWA 연구에서 나온 결과는 이후 ARMD의 보완 시스템을 치료적으로 조작하기 위한 추가적인 기능적 연구를 촉발시켰다.[34]
GWA 연구 역사상 또 다른 획기적인 출판물은 2007년 발행 당시 실시된 GWA 연구 중 역대 최대 규모인 WTCCC(Wellcome Trust Case Control Consortium) 연구였다.WTCCC에는 7가지 흔한 질병(관상동맥 심장병, 제1형 당뇨, 제2형 당뇨, 류마티스 관절염, 크론병, 조울증, 고혈압 각각 약 2천 명) 1만 4천 건과 3천 건의 공동관제 대상자가 포함됐다.[15]이 연구는 이러한 질병의 기저에 있는 많은 새로운 질병 유전자를 밝혀내는 데 성공했다.[15][35]
이러한 최초의 획기적인 GWA 연구 이후, 두 가지 일반적인 추세가 있었다.[36]하나는 점점 더 큰 표본 크기에 대한 것이었다.2018년에는 교육성취에 대한[37] 게놈전역 연구 110만 명, 130만 명의 개인이 포함된 불면증 연구 등 여러 게놈전역 연구들이 총 100만 명 이상의 참가자들의 표본 크기에 도달하고 있다.[38]그 이유는 오즈비가 작고 알레르기가 적은 SNP를 신뢰성 있게 검출하기 위한 추진력 때문이다.또 다른 경향은 혈중 지질, 프로이슬린 또는 유사한 바이오마커와 같이 보다 좁게 정의된 표현형을 사용하는 것이었다.[39][40]이것들은 중간 표현형이라고 불리며, 그들의 분석은 바이오마커에 대한 기능적 연구에 가치가 있을 수 있다.[41]
GWAS의 변형은 질병에 걸린 사람들의 1급 친족인 참가자들을 이용한다.이러한 유형의 연구는 GWAX(Genome-wide association study)로 명명되었다.[42]
GWA 연구에 대한 논쟁의 중심점은 GWA 연구에 의해 발견되는 SNP 변형의 대부분은 질병의 위험 증가와 관련이 있을 뿐이고, 예측 가치도 작다는 것이다.중위 오즈비는 위험-SNP당 1.33이며, 소수의 오즈비만이 3.0 이상을 보여준다.[1][43]이러한 크기는 유전적 변동의 많은 부분을 설명하지 않기 때문에 작은 것으로 간주된다.이 유전적 변화는 일란성 쌍둥이에 기초한 유전성 연구로부터 추정되었다.[44]예를 들어, 키의 분산의 80-90%는 유전적 차이로 설명할 수 있다고 알려져 있지만, GWA 연구는 이러한 분산의 소수만을 차지한다.[44]
임상 애플리케이션 및 예
향후 성공적인 GWA 연구의 과제는 신약 및 진단 설계의 청사진으로서 건강을 유지하는 데 있어 유전적 연구의 통합과 유전적 변동의 역할에 초점을 맞추는 등 약물 및 진단 개발을 가속화하는 방향으로 연구 결과를 적용하는 것이다.[45]여러 연구에서는 예후의 정확성을 직접적으로 향상시키는 수단으로서 위험-SNP 마커의 사용을 조사했다.일부는 예후의 정확성이 향상된다는 것을 발견했고,[46] 다른 일부는 이 사용으로 인한 사소한 이익만을 보고한다.[47]일반적으로 이러한 직접적인 접근법의 문제는 관찰된 효과의 작은 규모다.작은 효과는 궁극적으로 사례와 통제의 분리가 제대로 이루어지지 않아 예후 정확도가 약간 향상되는 것으로 해석된다.따라서 대안적 적용은 GWA 연구가 병리학을 해명할 수 있는 가능성이다.[48]
C형 간염 치료
그러한 성공 중 하나는 항혈당염 C 바이러스 치료와 관련된 유전적 변종을 식별하는 것과 관련이 있다.GWA 연구에서는[49] 리바비린과 결합된 Pegylated interferon-alpha-2a 또는 Pegylated interferon-alpha-2b로 치료된 유전자형 1형 간염에 대해, 인터페론 람다 3을 인코딩하는 인간 IL28B 유전자 근처의 SNP가 치료에 대한 반응에서 유의미한 차이와 관련이 있다는 것을 보여주었다.나중에 발표된 보고서는 동일한 유전적 변형이 유전자형 1형 간염 C형 간염 바이러스의 자연적인 간극과도 관련이 있다는 것을 증명했다.[50]이러한 주요 발견은 개인화된 의학의 개발을 촉진시켰고 의사는 환자의 유전자형에 기초하여 의학적 결정을 맞춤화할 수 있게 했다.[51]
eQTL, LDL 및 심혈관 질환
병태생리학의 해명목표는 또한 위험-SNPs와 인근 유전자의 유전자 발현 사이의 연관성에 대한 관심을 증가시켰는데, 이른바 표현 정량적 특성 loci(eQTL) 연구들이다.[52]그 이유는 GWAS 연구가 위험-SNP를 식별하지만 위험 유전자는 식별하지 못하며 유전자의 사양은 실행 가능한 약물 표적에 한 걸음 더 가까워졌기 때문이다.그 결과, 2011년까지의 주요 GWA 연구는 일반적으로 광범위한 eQTL 분석을 포함했다.[53][54][55]GWA 식별 위험 SNP에 대해 관측된 가장 강력한 eQTL 효과 중 하나는 SOCR1 로케우스다.[39]작은 간섭 RNA와 유전자 녹아웃 생쥐를 이용한 이 위치의 기능적 추적 연구는 심혈관 질환에 중요한 임상적 영향을 미치는 저밀도 지단백질의 신진대사를 밝혀냈다.[39][56][57]
심방세동
예를 들어 2018년에 이루어진 메타 분석 결과 심방세동과 관련된 70개의 새로운 로키가 발견되었다.심장전도 조절에 관여하는 TBX3 및 TBX5, NKX2-5 o PITX2와 같이 이온 채널 변조 및 심장발달에 있어 전사 인자 코딩겐과 관련된 다양한 변형이 확인되었다.또한 빈맥(CASQ2)이나 심장근육세포통신(PKP2)의 변화와 관련된 새로운 유전자도 확인되었다.[58]
정신분열증
어떤 연구는 504명 새로운 단백질-단백질 상호 작용(PPIs)을 발견했다 컴퓨터 모델(HiPPIP) High-Precision 단백질 상호 작용 예측을 사용하여는 유전자 증거를 정신 분열증의 유전적 근거 지원 schizophrenia,[59][60]와 연결된 사실과 limita에 시달릴 수 있은 논란과 관련된입니다.tion의이 연구 [61]방법
농업적용
플랜트 성장 단계 및 수율 구성 요소
GWA 연구는 식물 번식에 중요한 도구로 작용한다.GWAS는 대형 유전자형·표현형 데이터로 스파이크 당 곡물 수, 곡물 무게, 식물 구조 등 중요한 산출 성분인 형질의 복잡한 유전 모드를 분석하는 데 강력하다.GWAS는 봄밀의 GWAS에 관한 연구에서 부팅 데이터, 바이오매스, 스파이크 당 곡물 수 등과 곡물 생산의 강한 상관관계를 밝혀냈다.[62] GWA 연구는 쌀의 복잡한 형질을 가진 유전자 구조를 연구하는 데도 성공적이다.[63]
식물 병원균
식물 병원균의 출현으로 식물 건강과 생물 다양성에 심각한 위협이 되고 있다.이러한 고려사항 하에서 특정 병원균에 대한 자연적인 저항성을 갖는 야생 유형의 식별이 매우 중요할 수 있다.더 나아가 어떤 알레르기가 저항과 연관되어 있는지 예측할 필요가 있다.GWA 연구는 특정 변종의 관계와 식물 병원체에 대한 저항성을 탐지하는 강력한 도구로, 새로운 병원체 재배 품종을 개발하는 데 이롭다.[64]
치킨
첫 번째 GWA 연구는 2007년에 아바쉬트와 라몬트에 의해 수행되었다.이 새로운 도구는 이전에 발견된 F2 모집단의 비만 특성을 연구하기 위해 사용되었다.이들이 발견한 SNP는 10개 염색체(1, 2, 3, 4, 7, 8, 10, 12, 15, 27)에서 검출됐다.
제한 사항
GWA 연구는 적절한 품질 관리와 연구 설정을 통해 해결할 수 있는 몇 가지 문제와 한계를 가지고 있다.잘 정의된 사례와 대조군의 부족, 불충분한 표본 크기, 다중 검사에 대한 대조군 및 모집단 층화에 대한 대조군이 일반적인 문제들이다.[2]특히 다중 시험의 통계 문제에서 "대량의 통계 시험이 시행되면 거짓 양성 결과가 발생할 수 있는 전례가 없기 때문에 GWA 접근방식이 문제가 될 수 있다"[2]고 언급했다.이러한 수정 가능한 문제를 무시하는 것은 GWA 방법론에 대한 일반적인 문제의식에 기여하는 것으로 언급되어 왔다.[66]이러한 문제들과 같이 쉽게 고칠 수 있는 문제들 외에도, 좀 더 미묘하지만 중요한 문제들이 표면화되었다.장수와 관련된 SNP를 식별하기 위해 수명이 매우 긴 개인을 조사한 유명한 GWA 연구가 그 예다.[67]이 출판물은 케이스의 유전자형 배열의 유형과 대조군 사이의 불일치로 인해 여러 SNP가 장수와 관련된 것으로 잘못 부각되었다.[68]그 연구는 이후 철회되었지만,[69] 수정된 원고가 나중에 출판되었다.[70]
이러한 예방 가능한 문제 외에도, GWA 연구는 보다 근본적인 비판을 이끌어냈는데, 주로 공통 유전적 변화가 일반 질병의 유전적 변이를 설명하는 데 큰 역할을 한다는 가정 때문이었다.[71]실제로 대부분의 조건에서 일반 SNP에 귀속되는 SNP 유전성은 <0.05>라고 추정되었다.[72]GWA 연구의 이런 측면은 비록 전진적으로 알 수는 없었지만, GWA 연구는 궁극적으로 지출할 가치가 없다는 비판을 받아왔다.[48]GWA 연구는 또한 질병 상태에 대한 개인의 반응이나 보상 메커니즘의 광범위한 변화는 질병과 관련된 잠재적 유전자나 인과 변형을 감추고 취소한다는 비판에 직면해 있다.[73]또한 GWA 연구는 분석을 수행하는 모집단의 후보 위험 변형을 식별하며, 유럽 데이터베이스에서 비롯된 대부분의 GWA 연구에서는 확인된 위험 변형을 다른 비유럽 모집단으로 변환하는 것이 부족하다.[74]대안 전략에는 연계 분석이 포함된다.[75][76]보다 최근에는 완전한 게놈 염기서열 분석의 가격이 급격히 하락하면서 배열 기반의 GWA 연구에 대한 현실적인 대안이 제공되기도 했다.이 새로운 기법의 사용을 여전히 GWA 연구라고 언급하고 있는지는 논의될 수 있지만, 고투과 시퀀싱은 비시퀀싱 GWA의 일부 단점을 보조할 수 있는 잠재력을 가지고 있다.[77]
미세 맵핑
GWAS를 위해 고안된 유전자형 배열은 일부 변종의 유전자형을 만들어 전체 게놈의 커버리지를 제공하기 위해 비정형 연결에 의존한다.이 때문에 보고된 관련 변형은 실제 인과 변형이 아닐 가능성이 높다.관련 영역은 넓은 지역에 걸쳐 있고 다양한 유전자를 포괄하는 수백 가지의 변종을 포함할 수 있어 GWAS loci의 생물학적 해석을 더욱 어렵게 한다.정밀 지도는 이러한 관련 변종 리스트를 인과 변종을 포함할 가능성이 가장 높은 세트로 세분화하는 과정이다.
정밀 매핑을 위해서는 관련 영역의 모든 변형이 유전자형 또는 귀납형(감지 범위), 고품질 유전자형을 초래하는 매우 엄격한 품질 관리 및 고도로 상관된 신호를 분리하기에 충분한 큰 표본 크기가 필요하다.미세 맵핑을 수행하는 방법에는 여러 가지가 있으며, 모든 방법은 그 위치의 변종이 인과적일 수 있는 후확률을 산출한다.요구사항이 충족되기 어려운 경우가 많기 때문에, 이러한 방법이 더 일반적으로 적용되는 예는 여전히 제한적이다.
참고 항목
참조
- ^ a b Manolio TA (July 2010). "Genomewide association studies and assessment of the risk of disease". The New England Journal of Medicine. 363 (2): 166–76. doi:10.1056/NEJMra0905980. PMID 20647212.
- ^ a b c Pearson TA, Manolio TA (March 2008). "How to interpret a genome-wide association study". JAMA. 299 (11): 1335–44. doi:10.1001/jama.299.11.1335. PMID 18349094.
- ^ "Genome-Wide Association Studies". National Human Genome Research Institute.
- ^ Ozaki K, Ohnishi Y, Iida A, Sekine A, Yamada R, Tsunoda T, et al. (December 2002). "Functional SNPs in the lymphotoxin-alpha gene that are associated with susceptibility to myocardial infarction". Nature Genetics. 32 (4): 650–4. doi:10.1038/ng1047. PMID 12426569. S2CID 21414260.
- ^ Klein RJ, Zeiss C, Chew EY, Tsai JY, Sackler RS, Haynes C, et al. (April 2005). "Complement factor H polymorphism in age-related macular degeneration". Science. 308 (5720): 385–9. Bibcode:2005Sci...308..385K. doi:10.1126/science.1109557. PMC 1512523. PMID 15761122.
- ^ "GWAS Catalog: The NHGRI-EBI Catalog of published genome-wide association studies". European Molecular Biology Laboratory. European Molecular Biology Laboratory. Retrieved 18 April 2017.
- ^ a b c Bush WS, Moore JH (2012). Lewitter F, Kann M (eds.). "Chapter 11: Genome-wide association studies". PLOS Computational Biology. 8 (12): e1002822. Bibcode:2012PLSCB...8E2822B. doi:10.1371/journal.pcbi.1002822. PMC 3531285. PMID 23300413.
- ^ a b c Strachan T, Read A (2011). Human Molecular Genetics (4th ed.). Garland Science. pp. 467–495. ISBN 978-0-8153-4149-9.
- ^ "Online Mendelian Inheritance in Man". Archived from the original on 5 December 2011. Retrieved 6 December 2011.
- ^ a b Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M (November 2001). "Genomewide scans of complex human diseases: true linkage is hard to find". American Journal of Human Genetics. 69 (5): 936–50. doi:10.1086/324069. PMC 1274370. PMID 11565063.
- ^ Risch N, Merikangas K (September 1996). "The future of genetic studies of complex human diseases". Science. 273 (5281): 1516–7. Bibcode:1996Sci...273.1516R. doi:10.1126/science.273.5281.1516. PMID 8801636. S2CID 5228523.
- ^ Greely HT (2007). "The uneasy ethical and legal underpinnings of large-scale genomic biobanks". Annual Review of Genomics and Human Genetics. 8: 343–64. doi:10.1146/annurev.genom.7.080505.115721. PMID 17550341.
- ^ The International HapMap Project, Gibbs RA, Belmont JW, Hardenbol P, Willis TD, Yu F, Yang H, Ch'Ang LY, Huang W (December 2003). "The International HapMap Project" (PDF). Nature. 426 (6968): 789–96. Bibcode:2003Natur.426..789G. doi:10.1038/nature02168. hdl:2027.42/62838. PMID 14685227. S2CID 4387110.
- ^ Schena M, Shalon D, Davis RW, Brown PO (October 1995). "Quantitative monitoring of gene expression patterns with a complementary DNA microarray". Science. 270 (5235): 467–70. Bibcode:1995Sci...270..467S. doi:10.1126/science.270.5235.467. PMID 7569999. S2CID 6720459.
- ^ a b c d Wellcome Trust Case Control Consortium, Burton PR (June 2007). "Genome-wide association study of 14,000 cases of seven common diseases and 3,000 shared controls". Nature. 447 (7145): 661–78. Bibcode:2007Natur.447..661B. doi:10.1038/nature05911. PMC 2719288. PMID 17554300.
- ^ a b c d Clarke GM, Anderson CA, Pettersson FH, Cardon LR, Morris AP, Zondervan KT (February 2011). "Basic statistical analysis in genetic case-control studies". Nature Protocols. 6 (2): 121–33. doi:10.1038/nprot.2010.182. PMC 3154648. PMID 21293453.
- ^ Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, et al. (September 2007). "PLINK: a tool set for whole-genome association and population-based linkage analyses". American Journal of Human Genetics. 81 (3): 559–75. doi:10.1086/519795. PMC 1950838. PMID 17701901.
- ^ Llinares-López F, Grimm DG, Bodenham DA, Gieraths U, Sugiyama M, Rowan B, Borgwardt K (June 2015). "Genome-wide detection of intervals of genetic heterogeneity associated with complex traits". Bioinformatics. 31 (12): i240-9. doi:10.1093/bioinformatics/btv263. PMC 4559912. PMID 26072488.
- ^ Ayati M, Erten S, Chance MR, Koyutürk M (December 2015). "MOBAS: identification of disease-associated protein subnetworks using modularity-based scoring". EURASIP Journal on Bioinformatics & Systems Biology. 2015 (1): 7. doi:10.1186/s13637-015-0025-6. PMC 5270451. PMID 28194175.
- ^ Ayati M, Koyutürk M (1 January 2015). "Assessing the Collective Disease Association of Multiple Genomic Loci". Proceedings of the 6th ACM Conference on Bioinformatics, Computational Biology and Health Informatics. BCB '15. New York, NY, USA: ACM. pp. 376–385. doi:10.1145/2808719.2808758. ISBN 978-1-4503-3853-0. S2CID 5942777.
- ^ Marchini J, Howie B (July 2010). "Genotype imputation for genome-wide association studies". Nature Reviews Genetics. 11 (7): 499–511. doi:10.1038/nrg2796. PMID 20517342. S2CID 1465707.
- ^ Howie B, Marchini J, Stephens M (November 2011). "Genotype imputation with thousands of genomes". G3. 1 (6): 457–70. doi:10.1534/g3.111.001198. PMC 3276165. PMID 22384356.
- ^ Browning BL, Browning SR (February 2009). "A unified approach to genotype imputation and haplotype-phase inference for large data sets of trios and unrelated individuals". American Journal of Human Genetics. 84 (2): 210–23. doi:10.1016/j.ajhg.2009.01.005. PMC 2668004. PMID 19200528.
- ^ Li Y, Willer CJ, Ding J, Scheet P, Abecasis GR (December 2010). "MaCH: using sequence and genotype data to estimate haplotypes and unobserved genotypes". Genetic Epidemiology. 34 (8): 816–34. doi:10.1002/gepi.20533. PMC 3175618. PMID 21058334.
- ^ Novembre J, Johnson T, Bryc K, Kutalik Z, Boyko AR, Auton A, Indap A, King KS, Bergmann S, Nelson MR, Stephens M, Bustamante CD (November 2008). "Genes mirror geography within Europe". Nature. 456 (7218): 98–101. Bibcode:2008Natur.456...98N. doi:10.1038/nature07331. PMC 2735096. PMID 18758442.
- ^ Charney E (January 2017). "Genes, behavior, and behavior genetics". Wiley Interdisciplinary Reviews. Cognitive Science. 8 (1–2): e1405. doi:10.1002/wcs.1405. hdl:10161/13337. PMID 27906529.
- ^ Wittkowski KM, Sonakya V, Bigio B, Tonn MK, Shic F, Ascano M, Nasca C, Gold-Von Simson G (January 2014). "A novel computational biostatistics approach implies impaired dephosphorylation of growth factor receptors as associated with severity of autism". Translational Psychiatry. 4 (1): e354. doi:10.1038/tp.2013.124. PMC 3905234. PMID 24473445.
- ^ Barsh GS, Copenhaver GP, Gibson G, Williams SM (July 2012). "Guidelines for genome-wide association studies". PLOS Genetics. 8 (7): e1002812. doi:10.1371/journal.pgen.1002812. PMC 3390399. PMID 22792080.
- ^ Smith SM, Douaud G, Chen W, Hanayik T, Alfaro-Almagro F, Sharp K, Elliott LT (2021). "An expanded set of genome-wide association studies of brain imaging phenotypes in UK Biobank". Nat Neurosci. 24 (5): 737–745. doi:10.1038/s41593-021-00826-4. PMC 7610742. PMID 33875891.
- ^ Sanna S, Li B, Mulas A, Sidore C, Kang HM, Jackson AU, et al. (July 2011). Gibson G (ed.). "Fine mapping of five loci associated with low-density lipoprotein cholesterol detects variants that double the explained heritability". PLOS Genetics. 7 (7): e1002198. doi:10.1371/journal.pgen.1002198. PMC 3145627. PMID 21829380.
- ^ Hindorff LA, Sethupathy P, Junkins HA, Ramos EM, Mehta JP, Collins FS, Manolio TA (June 2009). "Potential etiologic and functional implications of genome-wide association loci for human diseases and traits". Proceedings of the National Academy of Sciences of the United States of America. 106 (23): 9362–7. Bibcode:2009PNAS..106.9362H. doi:10.1073/pnas.0903103106. PMC 2687147. PMID 19474294.
- ^ Johnson AD, O'Donnell CJ (January 2009). "An open access database of genome-wide association results". BMC Medical Genetics. 10: 6. doi:10.1186/1471-2350-10-6. PMC 2639349. PMID 19161620.
- ^ Haines JL, Hauser MA, Schmidt S, Scott WK, Olson LM, Gallins P, Spencer KL, Kwan SY, Noureddine M, Gilbert JR, Schnetz-Boutaud N, Agarwal A, Postel EA, Pericak-Vance MA (April 2005). "Complement factor H variant increases the risk of age-related macular degeneration". Science. 308 (5720): 419–21. Bibcode:2005Sci...308..419H. doi:10.1126/science.1110359. PMID 15761120. S2CID 32716116.
- ^ Fridkis-Hareli M, Storek M, Mazsaroff I, Risitano AM, Lundberg AS, Horvath CJ, Holers VM (October 2011). "Design and development of TT30, a novel C3d-targeted C3/C5 convertase inhibitor for treatment of human complement alternative pathway-mediated diseases". Blood. 118 (17): 4705–13. doi:10.1182/blood-2011-06-359646. PMC 3208285. PMID 21860027.
- ^ "Largest ever study of genetics of common diseases published today" (Press release). Wellcome Trust Case Control Consortium. 6 June 2007. Retrieved 19 June 2008.
- ^ Ioannidis JP, Thomas G, Daly MJ (May 2009). "Validating, augmenting and refining genome-wide association signals". Nature Reviews Genetics. 10 (5): 318–29. doi:10.1038/nrg2544. PMC 7877552. PMID 19373277. S2CID 6463743.
- ^ Lee JJ, Wedow R, Okbay A, Kong E, Maghzian O, Zacher M, Nguyen-Viet TA, Bowers P, Sidorenko J, Karlsson Linnér R, et al. (July 2018). "Gene discovery and polygenic prediction from a genome-wide association study of educational attainment in 1.1 million individuals". Nature Genetics. 50 (8): 1112–1121. doi:10.1038/s41588-018-0147-3. PMC 6393768. PMID 30038396.
- ^ Jansen PR, Watanabe K, Stringer S, Skene N, Bryois J, Hammerschlag AR, et al. (January 2018). "Genome-wide Analysis of Insomnia (N=1,331,010) Identifies Novel Loci and Functional Pathways". doi:10.1101/214973.
{{cite journal}}:Cite 저널은 필요로 한다.journal=(도움말) - ^ a b c Kathiresan S, Willer CJ, Peloso GM, Demissie S, Musunuru K, Schadt EE, et al. (January 2009). "Common variants at 30 loci contribute to polygenic dyslipidemia". Nature Genetics. 41 (1): 56–65. doi:10.1038/ng.291. PMC 2881676. PMID 19060906.
- ^ Strawbridge RJ, Dupuis J, Prokopenko I, Barker A, Ahlqvist E, Rybin D, et al. (October 2011). "Genome-wide association identifies nine common variants associated with fasting proinsulin levels and provides new insights into the pathophysiology of type 2 diabetes". Diabetes. 60 (10): 2624–34. doi:10.2337/db11-0415. PMC 3178302. PMID 21873549.
- ^ Danesh J, Pepys MB (November 2009). "C-reactive protein and coronary disease: is there a causal link?". Circulation. 120 (21): 2036–9. doi:10.1161/CIRCULATIONAHA.109.907212. PMID 19901186.
- ^ Liu JZ, Erlich Y, Pickrell JK (March 2017). "Case-control association mapping by proxy using family history of disease". Nature Genetics. 49 (3): 325–331. doi:10.1038/ng.3766. PMID 28092683. S2CID 5598845.
- ^ Ku CS, Loy EY, Pawitan Y, Chia KS (April 2010). "The pursuit of genome-wide association studies: where are we now?". Journal of Human Genetics. 55 (4): 195–206. doi:10.1038/jhg.2010.19. PMID 20300123.
- ^ a b Maher B (November 2008). "Personal genomes: The case of the missing heritability". Nature. 456 (7218): 18–21. doi:10.1038/456018a. PMID 18987709.
- ^ Iadonato SP, Katze MG (September 2009). "Genomics: Hepatitis C virus gets personal". Nature. 461 (7262): 357–8. Bibcode:2009Natur.461..357I. doi:10.1038/461357a. PMID 19759611. S2CID 7602652.

- ^ Muehlschlegel JD, Liu KY, Perry TE, Fox AA, Collard CD, Shernan SK, Body SC (September 2010). "Chromosome 9p21 variant predicts mortality after coronary artery bypass graft surgery". Circulation. 122 (11 Suppl): S60–5. doi:10.1161/CIRCULATIONAHA.109.924233. PMC 2943860. PMID 20837927.
- ^ Paynter NP, Chasman DI, Paré G, Buring JE, Cook NR, Miletich JP, Ridker PM (February 2010). "Association between a literature-based genetic risk score and cardiovascular events in women". JAMA. 303 (7): 631–7. doi:10.1001/jama.2010.119. PMC 2845522. PMID 20159871.
- ^ a b Couzin-Frankel J (June 2010). "Major heart disease genes prove elusive". Science. 328 (5983): 1220–1. Bibcode:2010Sci...328.1220C. doi:10.1126/science.328.5983.1220. PMID 20522751.

- ^ Ge D, Fellay J, Thompson AJ, Simon JS, Shianna KV, Urban TJ, Heinzen EL, Qiu P, Bertelsen AH, Muir AJ, Sulkowski M, McHutchison JG, Goldstein DB (September 2009). "Genetic variation in IL28B predicts hepatitis C treatment-induced viral clearance". Nature. 461 (7262): 399–401. Bibcode:2009Natur.461..399G. doi:10.1038/nature08309. PMID 19684573. S2CID 1707096.
- ^ Thomas DL, Thio CL, Martin MP, Qi Y, Ge D, O'Huigin C, Kidd J, Kidd K, Khakoo SI, Alexander G, Goedert JJ, Kirk GD, Donfield SM, Rosen HR, Tobler LH, Busch MP, McHutchison JG, Goldstein DB, Carrington M (October 2009). "Genetic variation in IL28B and spontaneous clearance of hepatitis C virus". Nature. 461 (7265): 798–801. Bibcode:2009Natur.461..798T. doi:10.1038/nature08463. PMC 3172006. PMID 19759533.
- ^ Lu YF, Goldstein DB, Angrist M, Cavalleri G (July 2014). "Personalized medicine and human genetic diversity". Cold Spring Harbor Perspectives in Medicine. 4 (9): a008581. doi:10.1101/cshperspect.a008581. PMC 4143101. PMID 25059740.
- ^ Folkersen L, van't Hooft F, Chernogubova E, Agardh HE, Hansson GK, Hedin U, Liska J, Syvänen AC, Paulsson-Berne G, Paulssson-Berne G, Franco-Cereceda A, Hamsten A, Gabrielsen A, Eriksson P (August 2010). "Association of genetic risk variants with expression of proximal genes identifies novel susceptibility genes for cardiovascular disease". Circulation: Cardiovascular Genetics. 3 (4): 365–73. doi:10.1161/CIRCGENETICS.110.948935. PMID 20562444.
- ^ Bown MJ, Jones GT, Harrison SC, Wright BJ, Bumpstead S, Baas AF, et al. (November 2011). "Abdominal aortic aneurysm is associated with a variant in low-density lipoprotein receptor-related protein 1". American Journal of Human Genetics. 89 (5): 619–27. doi:10.1016/j.ajhg.2011.10.002. PMC 3213391. PMID 22055160.
- ^ Coronary Artery Disease (C4D) Genetics Consortium (March 2011). "A genome-wide association study in Europeans and South Asians identifies five new loci for coronary artery disease". Nature Genetics. 43 (4): 339–44. doi:10.1038/ng.782. PMID 21378988. S2CID 39712343.

- ^ Johnson T, Gaunt TR, Newhouse SJ, Padmanabhan S, Tomaszewski M, Kumari M, et al. (December 2011). "Blood pressure loci identified with a gene-centric array". American Journal of Human Genetics. 89 (6): 688–700. doi:10.1016/j.ajhg.2011.10.013. PMC 3234370. PMID 22100073.
- ^ Dubé JB, Johansen CT, Hegele RA (June 2011). "Sortilin: an unusual suspect in cholesterol metabolism: from GWAS identification to in vivo biochemical analyses, sortilin has been identified as a novel mediator of human lipoprotein metabolism". BioEssays. 33 (6): 430–7. doi:10.1002/bies.201100003. PMID 21462369.

- ^ Bauer RC, Stylianou IM, Rader DJ (April 2011). "Functional validation of new pathways in lipoprotein metabolism identified by human genetics". Current Opinion in Lipidology. 22 (2): 123–8. doi:10.1097/MOL.0b013e32834469b3. PMID 21311327. S2CID 24020035.

- ^ Roselli C, Chafin M, Weng L (2018). "Multi-ethnic genome-wide association study for atrial fibrillation". Nature Genetics. 50 (9): 1225–1233. doi:10.1038/s41588-018-0133-9. PMC 6136836. PMID 29892015.
- ^ Ganapathiraju MK, Thahir M, Handen A, Sarkar SN, Sweet RA, Nimgaonkar VL, Loscher CE, Bauer EM, Chaparala S (27 April 2016). "Schizophrenia interactome with 504 novel protein-protein interactions". NPJ Schizophrenia. 2: 16012. doi:10.1038/npjschz.2016.12. PMC 4898894. PMID 27336055. Lay summary – psychcentral.com.
{{cite journal}}:Cite는 사용되지 않는 매개 변수를 사용한다.lay-url=(도움말) - ^ Ganapathiraju M, Chaparala S, Lo C (April 2018). "F200. Elucidating The Role of Cilia in Neuropsychiatric Diseases Through Interactome Analysis". Schizophrenia Bulletin. 44 (suppl_1): S298-9. doi:10.1093/schbul/sby017.731. PMC 5887623.
- ^ Johnson EC, Border R, Melroy-Greif WE, de Leeuw CA, Ehringer MA, Keller MC (November 2017). "No Evidence That Schizophrenia Candidate Genes Are More Associated With Schizophrenia Than Noncandidate Genes". Biological Psychiatry. 82 (10): 702–708. doi:10.1016/j.biopsych.2017.06.033. PMC 5643230. PMID 28823710.
- ^ Turuspekov Y, Baibulatova A, Yermekbayev K, Tokhetova L, Chudinov V, Sereda G, et al. (November 2017). "GWAS for plant growth stages and yield components in spring wheat (Triticum aestivum L.) harvested in three regions of Kazakhstan". BMC Plant Biology. 17 (Suppl 1): 190. doi:10.1186/s12870-017-1131-2. PMC 5688510. PMID 29143598.
- ^ Zhao, Keyan; Tung, Chih-Wei; Eizenga, Georgia C.; Wright, Mark H.; Ali, M. Liakat; Price, Adam H.; Norton, Gareth J.; Islam, M. Rafiqul; Reynolds, Andy; Mezey, Jason; McClung, Anna M. (13 September 2011). "Genome-wide association mapping reveals a rich genetic architecture of complex traits in Oryza sativa". Nature Communications. 2 (1): 467. doi:10.1038/ncomms1467. ISSN 2041-1723.
- ^ Bartoli C, Roux F (2017). "Genome-Wide Association Studies In Plant Pathosystems: Toward an Ecological Genomics Approach". Frontiers in Plant Science. 8: 763. doi:10.3389/fpls.2017.00763. PMC 5441063. PMID 28588588.
- ^ Abasht, B.; Lamont, S. J. (9 August 2007). "Genome-wide association analysis reveals cryptic alleles as an important factor in heterosis for fatness in chicken F2 population: Genome-wide association analysis". Animal Genetics. 38 (5): 491–498. doi:10.1111/j.1365-2052.2007.01642.x.
- ^ Pickrell J, Barrett J, MacArthur D, Jostins L (23 November 2011). "Size matters, and other lessons from medical genetics". Genomes Unzipped. Retrieved 7 December 2011.
- ^ Sebastiani P, Solovieff N, Puca A, Hartley SW, Melista E, Andersen S, Dworkis DA, Wilk JB, Myers RH, Steinberg MH, Montano M, Baldwin CT, Perls TT (July 2010). "Genetic signatures of exceptional longevity in humans". Science. 2010. doi:10.1126/science.1190532. PMID 20595579.(회수됨)

- ^ MacArthur D (8 July 2010). "Serious flaws revealed in "longevity genes" study". Wired. Retrieved 7 December 2011.
- ^ Sebastiani P, Solovieff N, Puca A, Hartley SW, Melista E, Andersen S, Dworkis DA, Wilk JB, Myers RH, Steinberg MH, Montano M, Baldwin CT, Perls TT (July 2011). "Retraction". Science. 333 (6041): 404. doi:10.1126/science.333.6041.404-a. PMID 21778381.

- ^ Sebastiani P, Solovieff N, Dewan AT, Walsh KM, Puca A, Hartley SW, Melista E, Andersen S, Dworkis DA, Wilk JB, Myers RH, Steinberg MH, Montano M, Baldwin CT, Hoh J, Perls TT (18 January 2012). "Genetic signatures of exceptional longevity in humans". PLOS ONE. 7 (1): e29848. Bibcode:2012PLoSO...729848S. doi:10.1371/journal.pone.0029848. PMC 3261167. PMID 22279548.
- ^ Visscher PM, Brown MA, McCarthy MI, Yang J (January 2012). "Five years of GWAS discovery". American Journal of Human Genetics. 90 (1): 7–24. doi:10.1016/j.ajhg.2011.11.029. PMC 3257326. PMID 22243964.
- ^ Patron J, Serra-Cayuela A, Han B, Li C, Wishart D (July 2019). "Assessing the performance of genome-wide association studies for predicting disease risk". bioRxiv 10.1101/701086.
- ^ Santolini M, Romay MC, Yukhtman CL, Rau CD, Ren S, Saucerman JJ, Wang JJ, Weiss JN, Wang Y, Lusis AJ, Karma A (24 February 2018). "A personalized, multiomics approach identifies genes involved in cardiac hypertrophy and heart failure". NPJ Systems Biology and Applications. 4 (1): 12. doi:10.1038/s41540-018-0046-3. PMC 5825397. PMID 29507758.
- ^ Rosenberg NA, Huang L, Jewett EM, Szpiech ZA, Jankovic I, Boehnke M (May 2010). "Genome-wide association studies in diverse populations". Nature Reviews Genetics. 11 (5): 356–66. doi:10.1038/nrg2760. PMC 3079573. PMID 20395969.
- ^ Sham PC, Cherny SS, Purcell S, Hewitt JK (May 2000). "Power of linkage versus association analysis of quantitative traits, by use of variance-components models, for sibship data". American Journal of Human Genetics. 66 (5): 1616–30. doi:10.1086/302891. PMC 1378020. PMID 10762547.
- ^ Borecki IB (2006). "Linkage and Association Studies". Encyclopedia of Life Sciences. eLS. John Wiley & Sons, Ltd. doi:10.1038/npg.els.0005483. ISBN 9780470015902.
- ^ Visscher PM, Goddard ME, Derks EM, Wray NR (May 2012). "Evidence-based psychiatric genetics, AKA the false dichotomy between common and rare variant hypotheses". Molecular Psychiatry. 17 (5): 474–85. doi:10.1038/mp.2011.65. PMID 21670730.

외부 링크
| 위키미디어 커먼스는 게놈 전반에 걸친 연관 연구와 관련된 미디어를 보유하고 있다. |
- OmicX 상의 Genotype-photype 상호 작용 소프트웨어 도구 및 데이터베이스
- 게놈전역 연관성 연구 분석을 위한 통계적 방법 [비디오 강의 시리즈]
- 전체 게놈 연관 연구 - 국립 인간 게놈 연구소의 연구
- GWAS Central — 요약 수준 유전자 관련 소견의 중앙 데이터베이스
- Barrett J (18 July 2010). "How to read a genome-wide association study". Genomes Unzipped.
- GWAS(Genome-wide association studies) - Bennett SN, Caporaso, NE, et al.
- PLINK — 전체 게놈 연관 분석 도구 세트
- ENCODE 스레드 탐색기 기능 정보가 변동 이해에 미치는 영향.자연(저널)