미코플라스마 산부인과
Mycoplasma laboratorium이 문서에서는 JCVI-syn3.0의 분할 형태학 문제와 JCVI-syn3A의 수정에 대한 정보가 누락되어 있습니다.은 에 될 수 . (2021년 3월) |
| 미코플라스마 산부인과 | |
|---|---|
| 과학적 분류 | |
| 도메인: | 박테리아 |
| 문: | 마이코플라즈마토타 |
| 클래스: | 연체동물 |
| 주문: | 미코플라즈마목 |
| 패밀리: | 미코플라스마과 |
| 속: | 미코플라스마 |
| 종류: | |
| 아종: | M. m. JCVI-syn1.0 |
| 삼항명 | |
| 마이코플라스마 마이코이데스 JCVI-syn1.0 깁슨 외, 2010년[a 1] | |
| 동의어[a2] | |
| 미코플라스마 산부인과 라이히, 2000 | |
마이코플라스마 라보토리엄 또는[b 1] 신시아는 합성 균주를 말한다.새로운 박테리아를 만드는 프로젝트는 시작부터 발전해 왔다.처음에 목표는 마이코플라스마 제니탈리움 게놈에서 생명을 유지하는데 필요한 최소한의 유전자 세트를 확인하고, 이러한 유전자를 합성하여 "새로운" 유기체를 만드는 것이었다.마이코플라스마 제니탈리움이 원래 이 프로젝트의 기초로 선택된 이유는 당시 분석된 모든 유기체 중 가장 적은 수의 유전자를 가지고 있었기 때문이다.이후 마이코플라스마 마이코이데스로 초점을 바꿔 시행착오를 거듭했다.[b 2]
생명에 필요한 최소한의 유전자를 식별하기 위해 M. 생식기의 482개의 유전자를 각각 개별적으로 삭제하고 그 결과 생성된 돌연변이의 생존성을 테스트했다.이것은 이론적으로 최소한의 [a 3]게놈을 나타내야 하는 382개의 최소 유전자 세트를 확인하는 결과를 낳았다.2008년 M. 생식기 유전자의 완전한 세트가 [b 3][a 4]합성 유전자를 식별하기 위해 비침이 추가되어 실험실에서 구성되었다.그러나 M. 생식기는 매우 느리게 성장하며 [b 4]M. mycoides는 성장에 실제로 필요한 유전자 세트를 결정하기 위한 실험을 가속화하기 위한 새로운 초점으로 선택되었습니다.
2010년, 컴퓨터 기록에서 M. mycoides의 완전한 게놈이 성공적으로 합성되어 DNA가 [b 5]제거된 마이코플라스마 카프리콜룸의 기존 세포에 이식되었다.이 프로젝트에 사용된 합성 게놈은 [b 4]제작하는 데 4천만 달러, 200명의 인력이 소요된 것으로 추정됩니다.이 새로운 박테리아는 자랄 수 있었고 이름은 JCVI-syn1.0, 즉 Synthia였다.기능성 유기체를 생산할 수 있는 더 작은 유전자 세트를 식별하기 위한 추가 실험 후, 473개의 [b 2]유전자를 포함하는 JCVI-syn3.0이 생성되었다.이 유전자들 중 149개는 알려지지 않은 [b 2]기능이다.JCVI-syn3.0의 게놈은 참신하기 때문에 최초의 진정한 합성 유기체로 여겨진다.
최소한의 게놈 프로젝트
신시아의 생산은 노벨상 수상자인 해밀턴 스미스가 이끄는 약 20명의 과학자 팀과 DNA 연구원 크레이그 벤터와 미생물학자 클라이드 A에 의한 J. 크레이그 벤터 연구소의 합성 생물학에 대한 노력이다. 허치슨 3세전체적인 목표는 살아있는 유기체를 필수 요소로 줄이고,[a 3] 따라서 새로운 유기체를 만드는데 무엇이 필요한지 처음부터 이해하는 것이다.초기 초점은 582,970개의 염기쌍으로 구성된 482개의 유전자로 구성된 필수 세포 내 기생충인 M. 제니탈리움 박테리아였다. 이 유전자는 하나의 원형 염색체에 배열되어 있었다. (프로젝트가 시작될 당시, 이것은 자유 배양에서 자랄 수 있는 알려진 자연 유기체 중 가장 작은 게놈이었다.)그들은 유기체의 성장에 필수적이지 않은 유전자를 식별하기 위해 트랜스포존 돌연변이 유발을 사용했고, 그 결과 최소 382개의 [a 3]유전자가 생성되었다.이 노력은 최소 게놈 [a 5]프로젝트로 알려져 있다.
유기체의 선택
미코플라스마
마이코플라스마(Mycoplasma)는 마이코플라스마토타(옛 테네리쿠테스)에 속하는 몰리쿠테스류(Mollicutes)의 박테리아로, 기생 또는 공생 생활 방식 때문에 세포벽이 없는 것이 특징이다.분자생물학에서, 이 속은 포유동물 세포 배양에서 악명높은 제거가 어려운 오염물질이며(베타락탐과 다른 [a 6]항생제에 면역이 있음), 그리고 작은 게놈 [a 7]크기 때문에 모델 유기체로 사용될 수 있다는 점에서 많은 관심을 받아왔다.Synthia 프로젝트의 속 선택은 Karl Reich가 Mycoplasma [a 2]laboratorium이라는 문구를 만든 2000년으로 거슬러 올라간다.
게놈이 작은 기타 유기체
2005년 현재, 펠라기박터(리케치아목의 α-단백질박터)는 자유 생물 중 가장 작은 게놈(1,308,759개의 염기쌍)을 가지고 있으며, 알려진 가장 작은 자기 복제 세포 중 하나이다.그것은 아마도 세계에서 가장 많은 박테리아일 것이다(약28 10개의 개별 세포). 그리고 SAR11 분류군의 다른 구성원과 함께,[a 8] 바다에 있는 모든 박테리아 또는 고고세포의 1/4에서 1/2를 구성하는 것으로 추정된다.2002년에 rRNA 시퀀스에 의해 확인되었으며 [a 9]2005년에 완전히 염기서열 분석되었다.연구실 [a 10][a 11]배양에서 높은 성장밀도에 도달하지 않은 종을 배양하는 것은 매우 어렵다.몇몇 새로 발견된 종족입니다, 하지만:Hodgkinia cicadicola에 놓치고 있는 많은 필수 유전자 독립 생활을 하는 것은 아니다 M. 플라즈마 제니탈 리움균보다 적은 유전자, Sulcia muelleri, Baumannia cicadellinicola(cicadas의 symbionts)과 Carsonella ruddi(hackberrypetiole에 뻔뻔스럽게 psyllid의 symbiote, Pachypsylla venusta[12])은 호스트 nucle으로 표현될 수 있다.us.[13]2013년 현재 알려진 가장 작은 유전자 세트를 가진 유기체는 필수 공생 동물인 나수이아 델토케팔리니콜라이다.그것은 단지 137개의 유전자와 112kb의 [a 14][b 6]게놈 크기를 가지고 있다.
| 종명 | 유전자수 | 크기(Mbp) |
|---|---|---|
| 칸디다투스 호지키니아 반달팽이[1] | 169 | 0.14 |
| 칸디다투스 카르소넬라 루디 PV [2] | 182 | 0.16 |
| 칸디다투스 술레리 GWSS [3] | 227 | 0.25 |
| 칸디다투스 술레리 SMDSEM [4] | 242 | 0.28 |
| 부체네라 진딧물떼새.시나라 세드리 [5] | 357 | 0.4261 |
| 마이코플라스마 제니탈리움 G37 [6] | 475 | 0.58 |
| 칸디다투스 피토플라스마 말리 [7] | 479 | 0.6 |
| 부체네라 진딧물떼새.바이종기아 피스타치아에 [8] | 504 | 0.6224 |
| Nanoarchaeum equitans Kin4-M [9] | 540 | 0.49 |
기술
매우 큰 DNA 조각의 합성과 조작이 필요했기 때문에 몇몇 실험실 기술이 프로젝트를 위해 개발되거나 적응되어야 했다.
세균 게놈 이식
2007년, Venter의 팀은 다음과 같은 방법으로 Mycoplasma mycoides 종의 염색체를 Mycoplasma caprolum으로 옮기는 데 성공했다고 보고했다.
- M. mycoides 게놈 분리: 한천에 갇힌 세포의 완만한 용해(세포와 혼합하여 겔을 형성하기 위해 남겨진 용융) 후 펄스장 젤 전기영동과 정확한 크기(원형 1.25Mbp)의 띠 분리;
- M. caprolum의 수용체 세포를 유능하게 만든다: 풍부한 배지의 성장이 DNA 복제의 억제와 형태학의 변화를 초래하는 빈약한 배지의 기아를 뒤따른다.
- 폴리에틸렌 글리콜 매개로 원형 염색체를 DNA가 없는 세포로 변환한 후 선택을 [a 15]한다.
변환이라는 용어는 박테리아 세포에 벡터를 삽입하는 것(전기 충격 또는 열 충격에 의한)을 가리키는 데 사용됩니다.여기서, 이식은 핵이식과 유사하게 사용된다.
세균염색체합성
2008년 Venter의 그룹은 계층적 [a 16]전략을 통해 M. Generalium G37 배열 L43967의 복사본인 합성 게놈의 생산을 설명했다.
- 합성 → 1kbp:게놈 시퀀스는 Blue Heron에 의해 80bp 중복이 있는 1,078개의 1080bp 카세트와 NotI 제한 부위(비효율적이지만 드물게 절단)에서 합성되었습니다.
- 결찰 → 10kbp: 연속된 10개의 카세트 109개 그룹을 플라스미드의 대장균에 묶어서 복제하였으며 염기서열을 통해 올바른 치열을 확인하였다.
- 다중 PCR → 100kbp: 연속 10kbp 어셈블리(효모에서 성장)의 11개 그룹이 각 10kbp 어셈블리에 대한 프라이머 쌍을 사용하여 다중 PCR에 결합되었다.
- 분리 및 재조합 → 2차 어셈블리는 벡터 배열 없이 분리, 결합 및 효모 스페로스트로 변환되었다(조립체 811-900에 있음).
이 2008년 결과의 게놈인 M. 제니탈륨 JCVI-1.0은 GenBank에 CP001621.1로 발표되었다.이는 M.[a 16] mycoides에 기초한 JCVI-syn이라는 라벨이 붙은 최신 합성 유기체와 혼동해서는 안 된다.
합성 게놈
2010년 Venter와 동료들은 합성 [a 1]게놈을 사용하여 Mycoplasma mycoides 균주 JCVI-syn1.0을 만들었습니다.처음에는 합성 구조가 작동하지 않았기 때문에 전체 프로젝트에서[b 4] 3개월의 지연을 초래한 오류를 정확히 파악하기 위해 일련의 반합성 구조가 생성되었습니다.장애의 원인은 복제 개시 [a 1]요인인 DnaA의 단일 프레임 시프트 돌연변이가 원인입니다.
합성 게놈으로 세포를 만드는 목적은 향후 수정된 게놈을 만드는 단계로서 방법론을 테스트하는 것이었다.천연 게놈을 템플릿으로 사용함으로써 잠재적인 실패 원인을 최소화할 수 있었습니다.Mycoplasma mycoides JCVI-syn1.0에는 기준 게놈에 대해 몇 가지 차이가 있으며, 특히 E.coli 트랜스포존 IS1(10kb 단계에서 감염)과 85bp 복제 및 효모에서의 증식에 필요한 요소 및 제한 [a 1]부위의 잔류물 등이 있다.
JCVI-syn1.0이 진짜 합성 유기체인지에 대한 논란이 있었다.게놈은 화학적으로 여러 조각으로 합성되었지만, 부모 게놈과 밀접하게 일치하도록 구성되어 자연 세포의 세포질에 이식되었다.DNA만으로는 생존 가능한 세포를 만들 수 없다: DNA를 읽기 위해서는 단백질과 RNA가 필요하고, DNA와 세포질을 구분하기 위해서는 지질막이 필요하다.JCVI-syn1.0에서는 기증자와 수용자로 사용되는 두 종이 같은 속이기 때문에 숙주 세포질과 새로운 [a 17]게놈의 단백질 불일치 문제가 발생할 수 있다.폴 케이임(플래그스태프 소재 노던 애리조나 대학의 분자 유전학자)은 "유전자 공학자들이 유기체의 게놈을 처음부터 섞고, 일치시키고, 완전히 설계하기까지는 앞으로 큰 도전들이 있을 것"[b 4]이라고 언급했다.
워터마크

JCVI-syn1.0의 널리 알려진 기능은 워터마크시퀀스의 존재입니다4개의 워터마크(논문[a 1] 보충자료의 그림 S1에 표시)는 각각 길이 1246, 1081, 1109 및 1222 염기쌍의 DNA에 기록된 코드화된 메시지이다.이 메시지들은 3개의 DNA 염기서열이 아미노산을 암호화하는 표준 유전자 코드가 아니라 이를 위해 개발된 새로운 코드를 사용해 독자들이 [b 7]해결해야 했다.워터마크의 내용은 다음과 같습니다.
- 워터마크 1: 디코더를 축하하는 텍스트로 브라우저에 읽어주는HTML 스크립트 및 디코딩을 증명하기 위해 작성자에게 이메일을 보내는 방법에 대한 설명입니다.
- 워터마크 2: 작가 목록과 제임스 조이스의 인용구: "살기, 실수하기, 추락하기, 승리하기, 삶 밖으로 삶을 재현하기 위해"
- 워터마크 3: 더 많은 저자와 로버트 오펜하이머의 인용문(비신뢰):"사물을 있는 그대로 보는 것이 아니라 있는 그대로 보는 것"
- 워터마크 4: 더 많은 저자와 리처드 파인만의 인용문: "내가 만들 수 없는 것은 이해할 수 없다."
JCVI-syn3.0
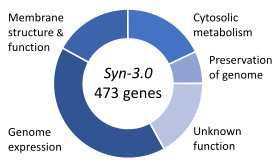
2016년, 벤터 연구소는 531,560개의 염기쌍과 473개의 [b 8]유전자를 포함하는 JCVI-syn3.0이라고 불리는 작은 게놈을 합성하기 위해 JCVI-syn1.0의 유전자를 사용했다.1996년, 다른 작은 박테리아 해모필러스 인플루엔자와 M. 생식기를 비교한 후, Arcady Mushegian과 Eugene Koonin은 [b 9][a 19]생존에 필요한 최소 유전자 세트가 될 수 있는 256개의 공통 유전자 세트가 있을 수 있다고 제안했다.이 새로운 유기체에서는 유전자의 수를 473개로 줄일 수 있는데, 그 중 149개는 전혀 [b 9]알려지지 않은 기능을 가지고 있다.2022년 현재 미지의 집합은 약 [1]100개로 좁혀졌습니다.2019년 Syn3.0 세포에서 모든 경로의 완전한 계산 모델이 발표되었으며, 이는 살아있는 최소 [a 20]유기체에 대한 최초의 완전한 실리콘 모델을 나타낸다.
우려와 논란
접수처
2007년 10월 6일, 크레이그 벤터는 영국의 가디언지와의 인터뷰에서 같은 팀이 화학적으로 마이코플라스마 제니탈리움 단일 염색체의 변형된 버전을 합성했다고 발표했다.합성된 게놈은 아직 작동하는 세포에 이식되지 않았다.다음 날 캐나다 생명윤리단체인 ETC 그룹은 그들의 대표인 팻 무니를 통해 성명을 발표하면서 벤터의 창조는 "거의 모든 것을 만들 수 있는 섀시"라고 말했다.신약과 같은 인류에 대한 기여가 될 수도 있고 생물무기와 같은 인류에 대한 엄청난 위협이 될 수도 있다고 말했다.Venter는 다음과 같이 말했습니다. "우리는 큰 아이디어를 다루고 있습니다.우리는 삶의 새로운 가치 체계를 만들기 위해 노력하고 있습니다.이 정도 규모로는 모두가 [b 10]행복할 수 없습니다.
2010년 5월 21일, 사이언스는 벤터 그룹이 컴퓨터 기록에서 마이코플라스마 마이코이데스 박테리아의 게놈을 성공적으로 합성하고 DNA가 제거된 마이코플라스마 카프리콜룸 박테리아의 기존 세포에 이 합성 게놈을 이식했다고 보도했다."합성" 박테리아는 생존할 수 있었고, 즉,[b 1] 복제할 수 있었다.벤터는 그것을 "첫 번째 종…"이라고 묘사했다.부모가 컴퓨터가 되는 것"이라고 말했다.[b 11]
새로운 합성 박테리아 JCVI-3.0의 생성은 2016년 3월 25일 사이언스에 발표되었습니다.그것은 473개의 유전자만 가지고 있다.벤터는 그것을 "역사상 최초의 디자이너 유기체"라고 불렀고 필요한 유전자 중 149개가 알려지지 않은 기능을 가지고 있다는 것은 "생물학 전체 분야가 생명에 필수적인 것의 3분의 1을 놓치고 있다"[a 21]는 것을 의미한다고 주장했다.
언론 보도
이 프로젝트는 벤터의 쇼맨십으로 인해 언론으로부터 많은 주목을 받았고, "우리가 필요한 유일한 규제는 동료의 입"[b 12]이라고 말할 정도로 선구적인 합성 생물학자이자 에이미리스의 설립자인 제이 키슬링은 말했다.
효용.
벤터는 합성 박테리아가 수소와 바이오 연료를 생산하고 이산화탄소와 다른 온실 가스를 흡수하기 위해 유기체를 만드는 단계라고 주장해왔다.조지 M. 합성 생물학의 또 다른 선구자인 처치는 대장균이 모든 여분의 DNA를 가지고도 M. 생식기보다 더 효율적으로 성장하기 때문에 완전한 합성 게놈을 만들 필요가 없다는 상반된 견해를 표현했다; 그는 합성 유전자가 위의 작업들 [b 13]중 일부를 수행하기 위해 대장균에 통합되었다고 논평했다.
지적 재산.
J. Craig Venter Institute는 2006년 [b 14][b 15][a 22]미국과 국제적으로 마이코플라스마 연구소의 게놈(미니멀 박테리아 게놈) 특허를 출원했다.캐나다 생명윤리단체인 ETC그룹은 특허 [b 16]범위가 너무 넓다는 이유로 반발했다.
유사한 프로젝트
2002년부터 2010년까지 헝가리 과학아카데미의 한 팀은 MDS42라고 불리는 대장균 변종을 만들었고, 이것은 현재 "클린 게놈"이라는 이름으로 미국 매디슨의 스카랍 게노믹스에 의해 판매되고 있다."E.coli"[b 17]는 분자생물학적 효율을 돕기 위해 부모주(E. coli K-12 MG1655)의 게놈의 15%를 제거하여 IS 요소, 의사유전자 및 페이지를 제거함으로써 종종 트랜스포존에 [a 23][a 24][a 25]의해 활성화되지 않는 플라스미드로 인코딩된 독성 유전자의 유지보수를 개선하였다.생화학 및 복제 기계는 변경되지 않았다.
레퍼런스
주요 소스
- ^ a b c d e Gibson, D. G.; Glass, J. I.; Lartigue, C.; Noskov, V. N.; Chuang, R.-Y.; Algire, M. A.; Benders, G. A.; Montague, M. G.; Ma, L.; Moodie, M. M.; Merryman, C.; Vashee, S.; Krishnakumar, R.; Assad-Garcia, N.; Andrews-Pfannkoch, C.; Denisova, E. A.; Young, L.; Qi, Z.-Q.; Segall-Shapiro, T. H.; Calvey, C. H.; Parmar, P. P.; Hutchison, C. A.; Smith, H. O.; Venter, J. C. (20 May 2010). "Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome". Science. 329 (5987): 52–56. Bibcode:2010Sci...329...52G. doi:10.1126/science.1190719. PMID 20488990.
- ^ a b Reich, KA (June 2000). "The search for essential genes". Research in Microbiology. 151 (5): 319–24. doi:10.1016/S0923-2508(00)00153-4. PMID 10919511.
In addition, the difficult genetics in these organisms makes subsequent verification of essentiality by directed knockouts problematic and virtually precludes the possibility of performing a de novo synthesis of ‘M. laboratorium’, the origin of the attention in the popular press.
- ^ a b c Glass, John I.; Nacyra Assad-Garcia; Nina Alperovich; Shibu Yooseph; Matthew R. Lewis; Mahir Maruf; Clyde A. Hutchison; Hamilton O. Smith; J. Craig Venter (2006-01-10). "Essential genes of a minimal bacterium". Proceedings of the National Academy of Sciences. 103 (2): 425–430. Bibcode:2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
- ^ Gibson, D. G.; Benders, G. A.; Andrews-Pfannkoch, C.; Denisova, E. A.; Baden-Tillson, H.; Zaveri, J.; Stockwell, T. B.; Brownley, A.; Thomas, D. W. (2008-02-29). "Complete Chemical Synthesis, Assembly, and Cloning of a Mycoplasma genitalium Genome". Science. 319 (5867): 1215–1220. Bibcode:2008Sci...319.1215G. doi:10.1126/science.1151721. ISSN 0036-8075. PMID 18218864. S2CID 8190996.
- ^ Hutchison CA, Montague MG (2002). "Mycoplasmas and the minimal genome concept". Molecular Biology and Pathogenicity of Mycoplasmas (Razin S, Herrmann R, eds.). New York: Kluwer Academic/Plenum. pp. 221–54. ISBN 978-0-306-47287-9.
- ^ Young L, Sung J, Stacey G, Masters JR. "세포 배양 내 마이코플라스마 검출"NAT Protoc.2010년 5월 5일 : 929 ~34일Epub 2010년 4월 22일
- ^ Fraser CM, Gocayne JD, White O, et al. (October 1995). "The minimal gene complement of Mycoplasma genitalium". Science. 270 (5235): 397–403. Bibcode:1995Sci...270..397F. doi:10.1126/science.270.5235.397. PMID 7569993. S2CID 29825758.
- ^ Morris RM, et al. (2002). "SAR11 clade dominates ocean surface bacterioplankton communities". Nature. 420 (6917): 806–10. Bibcode:2002Natur.420..806M. doi:10.1038/nature01240. PMID 12490947. S2CID 4360530.
- ^ Stephen J. Giovannoni, H. James Tripp, et al. (2005). "Genome Streamlining in a Cosmopolitan Oceanic Bacterium". Science. 309 (5738): 1242–1245. Bibcode:2005Sci...309.1242G. doi:10.1126/science.1114057. PMID 16109880. S2CID 16221415.
- ^ Rappé MS, Connon SA, Vergin KL, Giovannoni SL (2002). "Cultivation of the ubiquitous SAR11 marine bacterioplankton clade". Nature. 418 (6898): 630–33. Bibcode:2002Natur.418..630R. doi:10.1038/nature00917. PMID 12167859. S2CID 4352877.
- ^ Tripp HJ, Kitner JB, Schwalbach MS, Dacey JW, Wilhelm LJ, Giovannoni SJ (Apr 10, 2008). "SAR11 marine bacteria require exogenous reduced sulphur for growth". Nature. 452 (7188): 741–4. Bibcode:2008Natur.452..741T. doi:10.1038/nature06776. PMID 18337719. S2CID 205212536.
- ^ Nakabachi, A.; Yamashita, A.; Toh, H.; Ishikawa, H.; Dunbar, H. E.; Moran, N. A.; Hattori, M. (2006). "The 160-Kilobase Genome of the Bacterial Endosymbiont Carsonella". Science. 314 (5797): 267. doi:10.1126/science.1134196. PMID 17038615. S2CID 44570539.
- ^ McCutcheon, J. P.; McDonald, B. R.; Moran, N. A. (2009). "Convergent evolution of metabolic roles in bacterial co-symbionts of insects". Proceedings of the National Academy of Sciences. 106 (36): 15394–15399. Bibcode:2009PNAS..10615394M. doi:10.1073/pnas.0906424106. PMC 2741262. PMID 19706397.
- ^ Nancy A. Moran and Gordon M. Bennett (2014). "The Tiniest Tiny Genomes". Annual Review of Microbiology. 68: 195–215. doi:10.1146/annurev-micro-091213-112901. PMID 24995872.
{{cite journal}}: CS1 maint: 작성자 파라미터 사용(링크) - ^ Lartigue C, Glass JI, Alperovich N, Pieper R, Parmar PP, Hutchison CA 3rd, Smith HO, Venter JC (Aug 3, 2007). "Genome transplantation in bacteria: changing one species to another". Science. 317 (5838): 632–8. Bibcode:2007Sci...317..632L. CiteSeerX 10.1.1.395.4374. doi:10.1126/science.1144622. PMID 17600181. S2CID 83956478.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ a b Gibson, B; Clyde A. Hutchison; Cynthia Pfannkoch; J. Craig Venter; et al. (2008-01-24). "Complete Chemical Synthesis, Assembly, and Cloning of a Mycoplasma genitalium Genome". Science. 319 (5867): 1215–20. Bibcode:2008Sci...319.1215G. doi:10.1126/science.1151721. PMID 18218864. S2CID 8190996.
- ^ Povolotskaya, IS; Kondrashov, FA (Jun 2010). "Sequence space and the ongoing expansion of the protein universe". Nature. 465 (7300): 922–6. Bibcode:2010Natur.465..922P. doi:10.1038/nature09105. PMID 20485343. S2CID 4431215.
- ^ Hutchison, Clyde A.; Chuang, Ray-Yuan; Noskov, Vladimir N.; Assad-Garcia, Nacyra; Deerinck, Thomas J.; Ellisman, Mark H.; Gill, John; Kannan, Krishna; Karas, Bogumil J. (2016-03-25). "Design and synthesis of a minimal bacterial genome". Science. 351 (6280): aad6253. Bibcode:2016Sci...351.....H. doi:10.1126/science.aad6253. ISSN 0036-8075. PMID 27013737.
- ^ Arcady R. Mushegian and Eugene V. Koonin (September 1996). "A minimal gene set for cellular life derived by comparison of complete bacterial genomes". Proc. Natl. Acad. Sci. USA. 93 (19): 10268–10273. Bibcode:1996PNAS...9310268M. doi:10.1073/pnas.93.19.10268. PMC 38373. PMID 8816789.
{{cite journal}}: CS1 maint: 작성자 파라미터 사용(링크) - ^ Breuer, Marian; Earnest, Tyler M.; Merryman, Chuck; Wise, Kim S.; Sun, Lijie; Lynott, Michaela R.; Hutchison, Clyde A.; Smith, Hamilton O.; Lapek, John D.; Gonzalez, David J.; De Crécy-Lagard, Valérie; Haas, Drago; Hanson, Andrew D.; Labhsetwar, Piyush; Glass, John I.; Luthey-Schulten, Zaida (2019). "Essential metabolism for a minimal cell". eLife. 8. doi:10.7554/eLife.36842. PMC 6609329. PMID 30657448.
- ^ Herper, Matthew. "After 20 Year Quest, Biologists Create Synthetic Bacteria With No Extra Genes". Forbes. Retrieved 2019-07-02.
- ^ 미국 특허출원: 20070122826
- ^ Umenhoffer K, Fehér T, Balikó G, Ayaydin F, Pósfai J, Blattner FR, Pósfai G (2010). "Reduced evolvability of Escherichia coli MDS42, an IS-less cellular chassis for molecular and synthetic biology applications". Microbial Cell Factories. 9: 38. doi:10.1186/1475-2859-9-38. PMC 2891674. PMID 20492662.
- ^ Pósfai G, Plunkett G 3rd, Fehér T, Frisch D, Keil GM, Umenhoffer K, Kolisnychenko V, Stahl B, Sharma SS, de Arruda M, Burland V, Harcum SW, Blattner FR (2006). "Emergent properties of reduced-genome Escherichia coli". Science. 312 (5776): 1044–6. Bibcode:2006Sci...312.1044P. doi:10.1126/science.1126439. PMID 16645050. S2CID 43287314.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Kolisnychenko V, Plunkett G 3rd, Herring CD, Fehér T, Pósfai J, Blattner FR, Pósfai G (April 2002). "Engineering a reduced Escherichia coli genome". Genome Res. 12 (4): 640–7. doi:10.1101/gr.217202. PMC 187512. PMID 11932248.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크)
대중 매체
- ^ a b Roberta Kwok (2010). "Genomics: DNA's master craftsmen". Nature. 468 (7320): 22–5. Bibcode:2010Natur.468...22K. doi:10.1038/468022a. PMID 21048740.
- ^ a b c Callaway, Ewen (2016). "'Minimal' cell raises stakes in race to harness synthetic life". Nature. 531 (7596): 557–558. Bibcode:2016Natur.531..557C. doi:10.1038/531557a. ISSN 0028-0836. PMID 27029256.
- ^ Ball, Philip (2008-01-24). "Genome stitched together by hand". Nature. doi:10.1038/news.2008.522. ISSN 0028-0836.
- ^ a b c d Pennisi E (May 2010). "Genomics. Synthetic genome brings new life to bacterium" (PDF). Science. 328 (5981): 958–9. doi:10.1126/science.328.5981.958. PMID 20488994.
- ^ Katsnelson, Alla (2010-05-20). "Researchers start up cell with synthetic genome". Nature. doi:10.1038/news.2010.253. ISSN 0028-0836.
- ^ Zimmer, Carl (2013-08-23). "And the Genomes Keep Shrinking..." National Geographic.
- ^ Ken Shirriff (2010-06-10). "Using Arc to decode Venter's secret DNA watermark". Ken Shirriff's blog. Retrieved 2010-10-29.
- ^ 최초의 최소 합성 세균 세포.우주생물학 웹2016년 3월 24일
- ^ a b Yong, Ed (March 24, 2016). "The Mysterious Thing About a Marvelous New Synthetic Cell".
- ^ Pilkington, Ed (6 Oct 2009). "I am creating artificial life, declares US gene pioneer". The Guardian. London. Retrieved 23 Nov 2012.
- ^ "How scientists made 'artificial life'". BBC News. 2010-05-20. Retrieved 2010-05-21.
- ^ 앤드류 폴락, 그의 기업 전략:The Scientific Method, NYTimes, 2010년 9월 4일
- ^ 지금까지 가장 긴 합성 DNA 조각, Scientific American News, 2008년 1월 24일
- ^ "인공생명체: 특허출원중", 이코노미스트, 2007년 6월 14일.2007년 10월 7일 취득.
- ^ Roger Highfield, "무제한 바이오 연료를 만드는 인공 미생물", 텔레그래프, 2007년 6월 8일.2007년 10월 7일 취득.
- ^ Ianculovici, Elena (2008-01-15). "The Emerging Field of Synthetic Biology: "Syn" or Salvation?". Science in the News. Retrieved 2019-07-03.
- ^ "Scarab Genomics LLC. Company web site".
- ^ Somers, James (7 March 2022). "The Final Frontier". The New Yorker. New York: Condé Nast.