피위
Piwi| 피위 번 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 피로코쿠스 [1]후리오스 아르고누테 단백질의 구조 | |||||||||||
| 식별자 | |||||||||||
| 기호 | 피위 | ||||||||||
| Pfam | PF02171 | ||||||||||
| 인터프로 | IPR003165 | ||||||||||
| 프로사이트 | PS50822 | ||||||||||
| CDD | cd02826 | ||||||||||
| |||||||||||

피위(또는 PIWI) 유전자는 줄기세포와 세균 세포 분화를 담당하는 규제 단백질로 확인되었다.[4]피위(Piwi)는 드로소필라에서 P-element Induced WImpy testis의 약칭이다.[5]피위 단백질은 RNA 결합 단백질로 식물과 동물 모두에 존재한다.[6]피위 단백질은 아르고누테/피위과에 속하며 핵 단백질로 분류되어 왔다.드로소필라에 대한 연구는 또한 피위 단백질이 피위 영역의 존재에 의해 부여된 더 작은 활동을 가지고 있다는 것을 보여주었다.[7]또한 피위는 후생유전 수식어인 이염색체 단백질 1과 PiRNA-완성순서와 연관된다.이것들은 피위가 후생유전적 규제에서 하는 역할의 표시들이다.피위 단백질은 또한 많은 피위 단백질들이 전구적인 피라나를 성숙한 피라나로 가공할 수 있는 슬라이스 활성을 포함하고 있기 때문에 피라나의 생물 발생을 조절하는 것으로 생각된다.
단백질 구조 및 기능
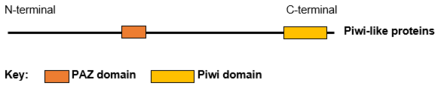
여러 피위(Piwi)와 아르고누테(Argonaute) 단백질(Ago)의 구조가 해결되었다.피위 단백질은 RNA를 결합하는 단백질로 2개 또는 3개의 영역을 가지고 있다.N-단자 PAZ 영역은 가이드 RNA의 3' 끝부분을 결합하고, 중간 MID 영역은 5'-인산 RNA의 인산염을 결합하며, C-단자 PIWI 영역은 RNA를 분쇄할 수 있는 RNase H 내분비체의 역할을 한다.[8][9]아고 단백질의 작은 RNA 파트너는 miRNA이다.이전에 단백질은 miRNA를 이용하여 전이 후 유전자를 침묵시키거나 전사 후와 전승 후 사일화 메커니즘에 siRNA(small-interfering RNA)를 사용한다.피위 단백질은 miRNA와 siRNA보다 긴 piRNA(28~33 뉴클레오티드)와 상호작용하며, 이들의 기능이 아전의 단백질과 구별됨을 시사한다.[8]
인간 피위 단백질
현재 알려진 4개의 인간 피위 단백질이 있다.PIWI 유사 단백질 1, PIWI 유사 단백질 2, PIWI 유사 단백질 3, PIWI 유사 단백질 4인간 피위 단백질은 모두 PAZ와 피위라는 두 개의 RNA 결합 영역을 포함하고 있다.4개의 PIWI와 같은 단백질은 PAZ 영역 내에 넓은 결합 부위가 있어 피위 상호작용 RNA의 3의 끝에서 부피가 큰 2'-OCH3를 결합할 수 있다.[10]
세미노마와 같은 종양의 형성에 관련되는 인간의 주요 호몰로 중 하나는 히위(인간 피위)라고 불린다.[11]
쥐의 동질 단백질은 미위(쥐 피위)라고 불려왔다.[12]
세균선 세포에서의 역할
PIWI 단백질은 동물과 동물에 걸친 생식력과 세균의 발달에 결정적인 역할을 한다.최근 극과립 성분으로 확인된 PIWI 단백질은 세균 세포 형성을 너무 많이 조절하는 것으로 나타나 PIWI 단백질이 없을 때 세균 세포 형성이 현저하게 감소한다.PIWI, MII, MIWI, MIWI2의 마우스 호몰로에서도 유사한 관찰이 이루어졌다.이러한 동음이의어는 정조세포에 존재하는 것으로 알려져 있다.미위는 세르톨리 세포에서 미위2가 표현되는 정조세포 형성과 정자 연장의 다양한 단계로 표현된다.밀리나 미위-2가 부족한 생쥐는 정조세포 감소를 경험했고 미위-2가 부족한 생쥐는 정조세포 감소를 경험했다.[13]인간과 쥐의 세균선에서 피위 단백질의 영향은 피위와 작은 비코딩 RNA인 피위 상호 작용 RNA(piRNA)가 폴리솜을 공동 추출하는 것으로 알려져 있기 때문에 번역 제어에 관여하는 데서 기인하는 것으로 보인다.piwi-piRNA 경로도 중앙에서 이질 색소 침착을 유도하여 전사하는 데 영향을 미친다.[14]Piwi-piRNA 경로도 게놈을 보호하기 위해 나타난다.Dosophila에서 처음 관찰된 돌연변이 Piwi-piRNA 경로로 인해 난소 세균 세포에서 dsDNA 균열이 직접 증가하였다.트랜스폰 소음에서 piwi-piRNA 경로의 역할은 세균 세포에서 dsDNA 파단 감소에 책임이 있을 수 있다.
RNA 간섭에서의 역할
피위 영역은[15] 피위 단백질과 관련 핵산 결합 단백질, 특히 RNA를 결합하고 분쇄하는 단백질에서 발견되는 단백질 영역이다.도메인의 기능은 관련 단백질의 아르고네이트 계열에서 결정된 단일 좌초된 RNA의 이중 좌초-RNA 유도 가수분해다.[1]핵산 결합 단백질의 가장 잘 연구된 계열인 아르고뉴테스는 RNA 유도 음소거 콤플렉스(RISC)의 촉매 기능을 수행하는 RNase H형 효소다.잘 알려진 RNA 간섭 세포 과정에서 RISC 콤플렉스 내 아르고뉴트 단백질은 리보누크레제 디케르가 생산한 내생성 비코딩 RNA에서 생성된 작은 간섭 RNA(siRNA)와 마이크로RNA(miRNA) 모두를 결합시켜 RNA-RISC 콤플렉스를 형성할 수 있다.이 콤플렉스는 메신저 RNA를 결합하고 분리하여 그것을 파괴하고 단백질로의 변환을 막는다.결정화된 피위 영역은 결합 RNA의 5의 끝을 위한 보존된 기본 결합 부지를 가지고 있다. siRNA 가닥을 결합하는 아르곤나이트 단백질의 경우, 또한 염기와 인접한 티로신 잔류물 사이의 염기 쌓기 상호작용으로 siRNA의 마지막 비절연 뉴클레오티드 베이스도 안정화된다.[16]
최근의 증거는 세균선 결정에서 피위 단백질의 기능적 역할이 miRNA와 상호작용하는 능력 때문이라는 것을 시사한다.miRNA 경로의 구성요소는 극 플라스마에 존재하는 것으로 보이며, 세균 라인 유지관리가 광범위하게 연구된 드로필라 멜라노가스터 배아의 초기 개발과 형태생식에 핵심적인 역할을 한다.[17]
PIRNA 및 트랜스폰 소음
피위 상호작용 RNA(PIRNA)로 알려진 평균보다 긴 miRNA의 새로운 등급이 포유류 세포에서 정의되었으며, 약 21개의 뉴클레오티드의 전형적인 miRNA 또는 siRNA에 비해 긴 26~31개의 뉴클레오티드가 있다.이러한 piRNA는 주로 포유류 고환에서 정조세포로 표현된다.[18]하지만 연구는piRNA 표현 무척추 동물의 난소 체세포와 뉴런 세포와 다른 많은 포유류의 체세포에서 찾아볼 수 있다. piRNAs는 생쥐, 쥐, 그리고 사람들의 게놈에 그는 반복적으로 지역에서 비롯될 수 있는 특이한"밀집"게놈 organization[19]으로 밝혀졌다 보도했다.이름e는 일반적으로 이질 색소로 구성된 역트랜스포존이나 영역과 같은 것으로, 일반적으로 이중 가닥 RNA의 항이센스 가닥에서 파생된다.[20] piRNA는 따라서 반복적으로 연관된 작은 간섭 RNA(rasiRNA)로 분류되었다.[21]
비록 그들의 생물 발생이 아직 잘 이해되지는 않았지만, PiRNA와 Piwi 단백질은 역트랜스포존스와 같은 이기적인 유전적 원소의 발현을 잠재우기 위한 내생적 체계를 형성하고, 따라서 그러한 염기서열의 유전자 생산물이 세균 세포 형성에 간섭하는 것을 막는 것으로 생각된다.[20][22]
참조
- ^ a b Rivas FV, Tolia NH, Song JJ, et al. (April 2005). "Purified Argonaute2 and an siRNA form recombinant human RISC". Nat. Struct. Mol. Biol. 12 (4): 340–9. doi:10.1038/nsmb918. PMID 15800637. S2CID 2021813.
- ^ "Uniprot: The Universal knowledge database". Nucleic Acids Research. 45 (D1): D158–D169. 2017. doi:10.1093/nar/gkw1099. PMC 5210571. PMID 27899622.
- ^ Lindse K (2013). "Piwi-RNAs, the Defenders of the Genome".
{{cite journal}}:Cite 저널은 필요로 한다.journal=(도움말) - ^ Cox DN, Chao A, Lin H (2000). "piwi encodes a nucleoplasmic factor whose activity modulates the number and division rate of germline stem cells". Development. 127 (3): 503–14. doi:10.1242/dev.127.3.503. PMID 10631171.
- ^ Lin H, Spradling AC (1997). "A novel group of pumilio mutations affects the asymmetric division of germline stem cells in the Drosophila ovary". Development. 124 (12): 2463–2476. doi:10.1242/dev.124.12.2463. PMID 9199372.
- ^ Cox DN, Chao A, Baker J, Chang L, Qiao D, Lin H (1998). "A novel class of evolutionarily conserved genes defined by piwi are essential for stem cell self-renewal". Genes Dev. 12 (23): 3715–27. doi:10.1101/gad.12.23.3715. PMC 317255. PMID 9851978.
- ^ Darricarrere N, Liu N, Watanabe T, Lin H (2013). "Function of Piwi, a nuclear Piwi/Argonaute protein, is independent of its slicer activity". Proc Natl Acad Sci USA. 110 (6): 1297–1302. Bibcode:2013PNAS..110.1297D. doi:10.1073/pnas.1213283110. PMC 3557079. PMID 23297219.
- ^ a b Zeng, Lei; Zhang, Qiang; Yan, Kelley; Zhou, Ming-Ming (2011-06-01). "Structural insights into piRNA recognition by the human PIWI-like 1 PAZ domain". Proteins: Structure, Function, and Bioinformatics. 79 (6): 2004–2009. doi:10.1002/prot.23003. ISSN 1097-0134. PMC 3092821. PMID 21465557.
- ^ Wei, Kai-Fa; Wu, Ling-Juan; Chen, Juan; Chen, Yan-feng; Xie, Dao-Xin (August 2012). "Structural evolution and functional diversification analyses of argonaute protein". Journal of Cellular Biochemistry. 113 (8): 2576–2585. doi:10.1002/jcb.24133. ISSN 1097-4644. PMID 22415963. S2CID 25990631.
- ^ Tian Y, Simanshu D, Ma J, Patel D (2010). "Structural basis for piRNA 2'-O-methylated 3'-end recognition by Piwi PAZ (Piwi/Argonaute/Zwille) domains". Proc. Natl. Acad. Sci. USA. 108 (3): 903–910. doi:10.1073/pnas.1017762108. PMC 3024652. PMID 21193640.
- ^ Qiao D, Zeeman AM, Deng W, Looijenga LH, Lin H (2002). "Molecular characterization of hiwi, a human member of the piwi gene family whose overexpression is correlated to seminomas". Oncogene. 21 (25): 3988–99. doi:10.1038/sj.onc.1205505. PMID 12037681.
- ^ Deng W, Lin H (2002). "miwi, a murine homolog of piwi, encodes a cytoplasmic protein essential for spermatogenesis". Dev Cell. 2 (6): 819–30. doi:10.1016/s1534-5807(02)00165-x. PMID 12062093.
- ^ Mani S, Juliano C (2013). "Untangling the Web: The Diverse Functions of the PIWI/piRNA Pathway". Mol. Reprod. Dev. 80 (8): 632–664. doi:10.1002/mrd.22195. PMC 4234069. PMID 23712694.
- ^ Thomson T, Lin H (2009). "The Biogenesis and Function PIWI Proteins and piRNAs: Progress and Prospect". Annu. Rev. Cell Dev. Biol. 25: 355–376. doi:10.1146/annurev.cellbio.24.110707.175327. PMC 2780330. PMID 19575643.
- ^ Cerutti L, Mian N, Bateman A (October 2000). "Domains in gene silencing and cell differentiation proteins: the novel PAZ domain and redefinition of the Piwi domain". Trends Biochem. Sci. 25 (10): 481–2. doi:10.1016/S0968-0004(00)01641-8. PMID 11050429.
- ^ Ma J, Yuan Y, Meister G, Pei Y, Tuschl T, Patel D (2005). "Structural basis for 5'-end-specific recognition of guide RNA by the A. fulgidus Piwi protein". Nature. 434 (7033): 666–70. Bibcode:2005Natur.434..666M. doi:10.1038/nature03514. PMC 4694588. PMID 15800629.
- ^ Megosh HB, Cox DN, Campbell C, Lin H (2006). "The role of PIWI and the miRNA machinery in Drosophila germline determination". Curr Biol. 16 (19): 1884–94. doi:10.1016/j.cub.2006.08.051. PMID 16949822. S2CID 6397874.
- ^ Kim VN (2006). "Small RNAs just got bigger: Piwi-interacting RNAs (piRNAs) in mammalian testes". Genes Dev. 20 (15): 1993–7. doi:10.1101/gad.1456106. PMID 16882976.
- ^ Girard A, Sachidanandam R, Hannon GJ, Carmell MA (2006). "A germline-specific class of small RNAs binds mammalian Piwi proteins". Nature. 442 (7099): 199–202. Bibcode:2006Natur.442..199G. doi:10.1038/nature04917. PMID 16751776. S2CID 3185036.
- ^ a b Vagin VV, Sigova A, Li C, Seitz H, Gvozdev V, Zamore PD (2006). "A distinct small RNA pathway silences selfish genetic elements in the germline". Science. 313 (5785): 320–4. Bibcode:2006Sci...313..320V. doi:10.1126/science.1129333. PMID 16809489. S2CID 40471466.
- ^ Saito K, Nishida KM, Mori T, Kawamura Y, Miyoshi K, Nagami T, Siomi H, Siomi MC (2006). "Specific association of Piwi with rasiRNAs derived from retrotransposon and heterochromatic regions in the Drosophila genome". Genes Dev. 20 (16): 2214–22. doi:10.1101/gad.1454806. PMC 1553205. PMID 16882972.
- ^ Ozata DM, Gainetdinov I, Zoch A, Phillip D, Zamore PD (2019). "PIWI-interacting RNAs: small RNAs with big functions" (PDF). Nature Reviews Genetics. 20 (2): 89–108. doi:10.1038/s41576-018-0073-3. PMID 30446728. S2CID 53565676.