장기간섭핵원소
Long interspersed nuclear element| 장기간섭핵원소 | |
|---|---|
 인간 LINE-1 역트랜스포존 표적핵산가수분해효소 결정구조 | |
| 식별자 | |
| 기호. | 선 |
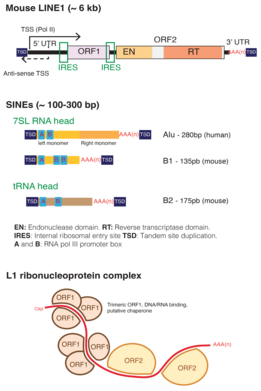
롱 인터스페이드 핵원소(LINEs)[1]는 많은 [4][5]진핵생물의 게놈에 널리 분포하는 비LTR(롱 인터스페이드 핵원소 또는 롱 인터스페이드[3] 원소라고도[2] 함) 역트랜스포존의 그룹이다.그들은 인간 [6][7][8]게놈의 약 21.1%를 차지한다.LINE은 트랜스포존 패밀리를 구성하며, 각 LINE의 길이는 약 7,000개입니다.LINE은 mRNA로 변환되고 단백질로 변환되어 역전사 효소로 작용한다.역전사효소는 새로운 부위의 게놈에 통합될 수 있는 LINE RNA의 DNA 복사본을 만듭니다.
인간에게 풍부한 LINE은 LINE1뿐입니다.인간 게놈에는 약 10만 개의 잘린 원소와 4,000개의 전장 LINE-1 [9]원소가 포함되어 있습니다.랜덤 돌연변이의 축적으로 인해 많은 LINE의 배열은 더 이상 전사되거나 번역되지 않을 정도로 퇴화되었다.LINE DNA 배열의 비교는 게놈에 트랜스포존을 삽입하는 날짜를 측정하기 위해 사용될 수 있다.
발견 이력
약 6.4kb 길이의 LINE 파생 시퀀스에 대한 최초의 설명은 1980년 [10]J. Adams 등에 의해 발표되었다.
종류들
구조적 특징과 핵심 효소인 역전사효소(RT)의 계통 발생에 기초하여 LINEs는 L1, RTE, R2, I 및 Jockey라고 불리는 5개의 주요 그룹으로 분류되며, 최소 28개의 [11]: fig. 1 계층으로 세분될 수 있다.
식물 게놈에서는 지금까지 L1과 RTE의 LINE만 [12][13][14]보고되었다.L1 요소는 여러 하위 계층으로 다양하지만, RTE 유형 LINE은 보존성이 높아 종종 단일 [15][16]계열을 구성한다.
균류에서는 Tad, L1, CRE, Deachiver 및 Inkcap 유사 원소가 [17]확인되었으며, Tad 유사 원소는 균류 [18]게놈에서만 나타난다.
모든 LINE은 RT 도메인과 엔도핵산가수분해효소(EN) 도메인(N-말단 APE 또는 C-말단 RLE 중 하나 또는 거의 둘 다)을 포함하는 최소 1개의 단백질 ORF2를 부호화한다.때때로 리보핵산가수분해효소 H 도메인이 존재한다.진화적 고대 R2 및 RTE 슈퍼패밀리를 제외하고 LINEs는 일반적으로 Gag-knuckle, L1 유사 RRM(InterPro: IPR035300) 및/또는 에스테라아제를 포함할 수 있는 ORF1이라는 또 다른 단백질을 암호화한다.LINE 요소는 식물, 곰팡이 또는 곤충에서 LTR-retro transposon에 비해 상대적으로 드물지만, 특히 게놈의 [11]: fig. 1 약 20%를 차지하는 척추동물과 포유류에서 우세하다.
L1 요소
LINE-1/L1-원소는 오늘날에도 여전히 인간 게놈에서 활성화된 요소 중 하나입니다.그것은 메가비트를 [20]제외한 모든[19] 포유동물에서 발견된다.
기타 요소
L2와 L3 [8]원소의 잔여물은 인간 게놈에서 발견됩니다.L2와 L3 원소는 2억-3억 년 전까지 활동했을 것으로 추정됩니다.L1 요소와 달리 L2 요소는 측면 대상 사이트 중복이 [21]없습니다.L2(및 L3) 요소는 CR1 클래드와 같은 그룹 Jockey에 [22]있습니다.
발생률
인간으로
첫 번째 인간 게놈 초안에서는 인간 게놈의 LINE 요소의 비율이 21%, 복사 번호가 85만이었다.이 중 L1, L2 및 L3 요소는 각각 516,000개, 315,000개 및 37,000개의 복사본을 구성했습니다.증식을 위해 L1 원소에 의존하는 비자율 SINE 원소는 인간 게놈의 13%를 차지하며 약 150만 [8]개의 복사 번호를 가지고 있습니다.LINE의 [23]RTE 패밀리에서 유래했을 가능성이 있습니다.최근의 추정에 따르면 일반적인 인간 게놈은 이동 가능성이 있는 평균 100개의 L1 원소를 포함하고 있지만 상당한 변화가 있고 일부 개인은 더 많은 수의 활성 L1 원소를 포함할 수 있기 때문에 이러한 개인은 L1 유도 돌연변이 [24]유발에 더 잘 걸린다.
또한 정신분열증 환자의 뇌에서 L1 복사본 수가 증가하여 LINE 요소가 일부 신경 질환에서 [25]역할을 할 수 있음을 나타냅니다.
전파
LINE 요소는 소위 Targeted Reverse Transcription Mechanism(TPRT)에 의해 전파됩니다.TPRT는 누에 Bombyx mori에서 R2 요소에 대해 처음 기술되었습니다.
ORF2(존재하는 경우 ORF1) 단백질은 주로 cis에서 mRNA를 코드하는 것과 관련지어 2개의 ORF2와 알려지지 않은 수의 ORF1 삼량체로 [26]구성될 가능성이 있는 리보핵단백질(RNP) 복합체를 형성한다.복합체는 다시 핵으로 운반되어 ORF2 핵산가수분해효소 도메인이 DNA를 연다(포유동물에서[27] TTAAAA 헥사뉴클레오티드 모티브).따라서, 3'OH 그룹은 LINE RNA 전사의 주요 역전사로 역전사 효소에 유리된다.역전사에 따라 대상 가닥이 절단되고 새로 생성된 cDNA가 통합됩니다[28].
새로운 삽입물은 짧은 표적 부위 복제(TSD)를 생성하며, 새로운 삽입물의 대부분은 심각하게 5' 절단되어 있다(사람의 경우 평균 삽입 크기 900bp). 종종 반전된다(Szak 등, 2002).5'가 부족하기 때문입니다.UTR, 대부분의 새 삽입물이 작동하지 않습니다.
LINE 액티비티 규제
숙주 세포는 예를 들어 후생성 소음화를 통해 L1 후전위 활성을 조절하는 것으로 나타났다.예를 들어 L1 배열에서 파생된 작은 간섭 RNA의 RNA 간섭(RNAi) 메커니즘은 L1 역위치를 [29]억제할 수 있다.
식물 게놈에서 LINE의 후생유전학적 수정은 인근 유전자의 발현 변화와 심지어 표현형 변화로 이어질 수 있다.오일 팜 게놈에서 카르마 타입의 LINE의 메틸화는 이 식물의 '[30]망토' 변종인 소마클로널의 기초가 되어 급격한 수율 손실을 일으킨다.
인간 APOBC3C 매개 LINE-1 요소 제한이 보고되었으며, 역전사효소 [31]활성에 영향을 미치는 것은 A3C와 ORF1p 사이의 상호작용 때문이다.
질병과의 관련성
L1-전염병의 역사적 예는 삽입성 돌연변이 [32]유발에 의해 발생하는 혈우병 A이다.암과 신경계 [33]질환을 포함한 소자 삽입에 의해 야기되는 알려진 질병의 거의 100가지 예가 있다.상피세포암(암)[34]에 대해 L1 동원과 종양 발생 사이의 상관관계가 보고되었다.LINES의 저메틸화는 염색체 불안정성과 유전자 발현[35] 변화와 관련이 있으며 다양한 조직 [36][35]유형의 다양한 암세포 유형에서 발견된다.MET onco 유전자에 위치한 특정 L1의 저메틸화는 방광암 종양 [37]형성과 관련이 있으며, Shift work sleep[38] disorder는 밤에 빛을 쬐면 L1에 의해 유발되는 게놈의 [39]불안정성을 감소시키는 것으로 보여지는 호르몬인 멜라토닌을 감소시키기 때문에 암 위험 증가와 관련이 있다.
레퍼런스
- ^ Ewing AD, Kazazian HH (June 2011). "Whole-genome resequencing allows detection of many rare LINE-1 insertion alleles in humans". Genome Research. 21 (6): 985–90. doi:10.1101/gr.114777.110. PMC 3106331. PMID 20980553.
- ^ Huang X, Su G, Wang Z, Shangguan S, Cui X, Zhu J, et al. (March 2014). "Hypomethylation of long interspersed nucleotide element-1 in peripheral mononuclear cells of juvenile systemic lupus erythematosus patients in China". International Journal of Rheumatic Diseases. 17 (3): 280–90. doi:10.1111/1756-185X.12239. PMID 24330152. S2CID 6530689.
- ^ Rodić N, Burns KH (March 2013). "Long interspersed element-1 (LINE-1): passenger or driver in human neoplasms?". PLOS Genetics. 9 (3): e1003402. doi:10.1371/journal.pgen.1003402. PMC 3610623. PMID 23555307.
- ^ Singer MF (March 1982). "SINEs and LINEs: highly repeated short and long interspersed sequences in mammalian genomes". Cell. 28 (3): 433–4. doi:10.1016/0092-8674(82)90194-5. PMID 6280868. S2CID 22129236.
- ^ Jurka, J. (1998). "Repeats in genomic DNA: Mining and meaning". Current Opinion in Structural Biology. 8 (3): 333–337. doi:10.1016/S0959-440X(98)80067-5. PMID 9666329.
- ^ Lindblad-Toh K, Wade CM, Mikkelsen TS, Karlsson EK, Jaffe DB, Kamal M, et al. (December 2005). "Genome sequence, comparative analysis and haplotype structure of the domestic dog". Nature. 438 (7069): 803–19. Bibcode:2005Natur.438..803L. doi:10.1038/nature04338. PMID 16341006.
- ^ Schumann GG, Gogvadze EV, Osanai-Futahashi M, Kuroki A, Münk C, Fujiwara H, et al. (2010-01-01). Unique functions of repetitive transcriptomes. International Review of Cell and Molecular Biology. Vol. 285. pp. 115–88. doi:10.1016/B978-0-12-381047-2.00003-7. ISBN 9780123810472. PMID 21035099.
- ^ a b c Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (February 2001). "Initial sequencing and analysis of the human genome" (PDF). Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Sheen FM, Sherry ST, Risch GM, Robichaux M, Nasidze I, Stoneking M, et al. (October 2000). "Reading between the LINEs: human genomic variation induced by LINE-1 retrotransposition". Genome Research. 10 (10): 1496–508. doi:10.1101/gr.149400. PMC 310943. PMID 11042149.
- ^ Adams JW, Kaufman RE, Kretschmer PJ, Harrison M, Nienhuis AW (December 1980). "A family of long reiterated DNA sequences, one copy of which is next to the human beta globin gene". Nucleic Acids Research. 8 (24): 6113–28. doi:10.1093/nar/8.24.6113. PMC 328076. PMID 6258162.
- ^ a b Kapitonov VV, Tempel S, Jurka J (December 2009). "Simple and fast classification of non-LTR retrotransposons based on phylogeny of their RT domain protein sequences". Gene. 448 (2): 207–13. doi:10.1016/j.gene.2009.07.019. PMC 2829327. PMID 19651192.
- ^ Heitkam T, Schmidt T (September 2009). "BNR - a LINE family from Beta vulgaris - contains a RRM domain in open reading frame 1 and defines a L1 sub-clade present in diverse plant genomes". The Plant Journal. 59 (6): 872–82. doi:10.1111/j.1365-313x.2009.03923.x. PMID 19473321.
- ^ Zupunski V, Gubensek F, Kordis D (October 2001). "Evolutionary dynamics and evolutionary history in the RTE clade of non-LTR retrotransposons". Molecular Biology and Evolution. 18 (10): 1849–63. doi:10.1093/oxfordjournals.molbev.a003727. PMID 11557792.
- ^ Komatsu M, Shimamoto K, Kyozuka J (August 2003). "Two-step regulation and continuous retrotransposition of the rice LINE-type retrotransposon Karma". The Plant Cell. 15 (8): 1934–44. doi:10.1105/tpc.011809. PMC 167180. PMID 12897263.
- ^ Heitkam T, Holtgräwe D, Dohm JC, Minoche AE, Himmelbauer H, Weisshaar B, et al. (August 2014). "Profiling of extensively diversified plant LINEs reveals distinct plant-specific subclades". The Plant Journal. 79 (3): 385–97. doi:10.1111/tpj.12565. PMID 24862340.
- ^ Smyshlyaev G, Voigt F, Blinov A, Barabas O, Novikova O (December 2013). "Acquisition of an Archaea-like ribonuclease H domain by plant L1 retrotransposons supports modular evolution". Proceedings of the National Academy of Sciences of the United States of America. 110 (50): 20140–5. Bibcode:2013PNAS..11020140S. doi:10.1073/pnas.1310958110. PMC 3864347. PMID 24277848.
- ^ Novikova O, Fet V, Blinov A (February 2009). "Non-LTR retrotransposons in fungi". Functional & Integrative Genomics. 9 (1): 27–42. doi:10.1007/s10142-008-0093-8. PMID 18677522. S2CID 23319640.
- ^ Malik HS, Burke WD, Eickbush TH (June 1999). "The age and evolution of non-LTR retrotransposable elements". Molecular Biology and Evolution. 16 (6): 793–805. doi:10.1093/oxfordjournals.molbev.a026164. PMID 10368957.
- ^ Warren WC, Hillier LW, Marshall Graves JA, Birney E, Ponting CP, Grützner F, et al. (May 2008). "Genome analysis of the platypus reveals unique signatures of evolution". Nature. 453 (7192): 175–83. Bibcode:2008Natur.453..175W. doi:10.1038/nature06936. PMC 2803040. PMID 18464734.
- ^ Smith JD, Gregory TR (June 2009). "The genome sizes of megabats (Chiroptera: Pteropodidae) are remarkably constrained". Biology Letters. 5 (3): 347–51. doi:10.1098/rsbl.2009.0016. PMC 2679926. PMID 19324635.
- ^ Kapitonov VV, Pavlicek A, Jurka J (2006-01-01). Anthology of Human Repetitive DNA. Wiley-VCH Verlag GmbH & Co. KGaA. doi:10.1002/3527600906.mcb.200300166. ISBN 9783527600908.
- ^ Lovsin N, Gubensek F, Kordi D (December 2001). "Evolutionary dynamics in a novel L2 clade of non-LTR retrotransposons in Deuterostomia". Molecular Biology and Evolution. 18 (12): 2213–24. doi:10.1093/oxfordjournals.molbev.a003768. PMID 11719571.
- ^ Malik, HS; Eickbush, TH (September 1998). "The RTE class of non-LTR retrotransposons is widely distributed in animals and is the origin of many SINEs". Molecular Biology and Evolution. 15 (9): 1123–34. doi:10.1093/oxfordjournals.molbev.a026020. PMID 9729877.
- ^ Streva VA, Jordan VE, Linker S, Hedges DJ, Batzer MA, Deininger PL (March 2015). "Sequencing, identification and mapping of primed L1 elements (SIMPLE) reveals significant variation in full length L1 elements between individuals". BMC Genomics. 16 (220): 220. doi:10.1186/s12864-015-1374-y. PMC 4381410. PMID 25887476.
- ^ Bundo M, Toyoshima M, Okada Y, Akamatsu W, Ueda J, Nemoto-Miyauchi T, et al. (January 2014). "Increased l1 retrotransposition in the neuronal genome in schizophrenia". Neuron. 81 (2): 306–13. doi:10.1016/j.neuron.2013.10.053. PMID 24389010.
- ^ Babushok DV, Ostertag EM, Courtney CE, Choi JM, Kazazian HH (February 2006). "L1 integration in a transgenic mouse model". Genome Research. 16 (2): 240–50. doi:10.1101/gr.4571606. PMC 1361720. PMID 16365384.
- ^ Jurka J (March 1997). "Sequence patterns indicate an enzymatic involvement in integration of mammalian retroposons". Proceedings of the National Academy of Sciences of the United States of America. 94 (5): 1872–7. Bibcode:1997PNAS...94.1872J. doi:10.1073/pnas.94.5.1872. PMC 20010. PMID 9050872.
- ^ Luan DD, Korman MH, Jakubczak JL, Eickbush TH (February 1993). "Reverse transcription of R2Bm RNA is primed by a nick at the chromosomal target site: a mechanism for non-LTR retrotransposition". Cell. 72 (4): 595–605. doi:10.1016/0092-8674(93)90078-5. PMID 7679954. S2CID 42587840.
- ^ Yang N, Kazazian HH (September 2006). "L1 retrotransposition is suppressed by endogenously encoded small interfering RNAs in human cultured cells". Nature Structural & Molecular Biology. 13 (9): 763–71. doi:10.1038/nsmb1141. PMID 16936727. S2CID 32601334.
- ^ Ong-Abdullah M, Ordway JM, Jiang N, Ooi SE, Kok SY, Sarpan N, et al. (September 2015). "Loss of Karma transposon methylation underlies the mantled somaclonal variant of oil palm". Nature. 525 (7570): 533–7. Bibcode:2015Natur.525..533O. doi:10.1038/nature15365. PMC 4857894. PMID 26352475.
- ^ Horn AV, Klawitter S, Held U, Berger A, Vasudevan AA, Bock A, et al. (January 2014). "Human LINE-1 restriction by APOBEC3C is deaminase independent and mediated by an ORF1p interaction that affects LINE reverse transcriptase activity". Nucleic Acids Research. 42 (1): 396–416. doi:10.1093/nar/gkt898. PMC 3874205. PMID 24101588.
- ^ Kazazian HH, Wong C, Youssoufian H, Scott AF, Phillips DG, Antonarakis SE (March 1988). "Haemophilia A resulting from de novo insertion of L1 sequences represents a novel mechanism for mutation in man". Nature. 332 (6160): 164–6. Bibcode:1988Natur.332..164K. doi:10.1038/332164a0. PMID 2831458. S2CID 4259071.
- ^ Solyom S, Kazazian HH (February 2012). "Mobile elements in the human genome: implications for disease". Genome Medicine. 4 (2): 12. doi:10.1186/gm311. PMC 3392758. PMID 22364178.
- ^ Carreira PE, Richardson SR, Faulkner GJ (January 2014). "L1 retrotransposons, cancer stem cells and oncogenesis". The FEBS Journal. 281 (1): 63–73. doi:10.1111/febs.12601. PMC 4160015. PMID 24286172.
- ^ a b Kitkumthorn N, Mutirangura A (August 2011). "Long interspersed nuclear element-1 hypomethylation in cancer: biology and clinical applications". Clinical Epigenetics. 2 (2): 315–30. doi:10.1007/s13148-011-0032-8. PMC 3365388. PMID 22704344.
- ^ Estécio MR, Gharibyan V, Shen L, Ibrahim AE, Doshi K, He R, Jelinek J, Yang AS, Yan PS, Huang TH, Tajara EH, Issa JP (May 2007). "LINE-1 hypomethylation in cancer is highly variable and inversely correlated with microsatellite instability". PLOS ONE. 2 (5): e399. Bibcode:2007PLoSO...2..399E. doi:10.1371/journal.pone.0000399. PMC 1851990. PMID 17476321.
- ^ Wolff EM, Byun HM, Han HF, Sharma S, Nichols PW, Siegmund KD, et al. (April 2010). "Hypomethylation of a LINE-1 promoter activates an alternate transcript of the MET oncogene in bladders with cancer". PLOS Genetics. 6 (4): e1000917. doi:10.1371/journal.pgen.1000917. PMC 2858672. PMID 20421991.
- ^ Spadafora C (April 2015). "A LINE-1-encoded reverse transcriptase-dependent regulatory mechanism is active in embryogenesis and tumorigenesis". Annals of the New York Academy of Sciences. 1341 (1): 164–71. Bibcode:2015NYASA1341..164S. doi:10.1111/nyas.12637. PMID 25586649. S2CID 22881053.
- ^ deHaro D, Kines KJ, Sokolowski M, Dauchy RT, Streva VA, Hill SM, et al. (July 2014). "Regulation of L1 expression and retrotransposition by melatonin and its receptor: implications for cancer risk associated with light exposure at night". Nucleic Acids Research. 42 (12): 7694–707. doi:10.1093/nar/gku503. PMC 4081101. PMID 24914052.



