Haplogroup L2(mtDNA)
Haplogroup L2 (mtDNA)| 하플로그룹 L2 | |
|---|---|
| 가능한 출발 시간 | 80,000–111,100 YBP[1] |
| 가능한 원산지 | 서아프리카[2] 또는 중앙아프리카 |
| 조상 | L2─6 |
| 후손 | L2a─d, L2e |
| 돌연변이 정의 | 146, 150, 152, 2416, 8206, 9221, 10115, 13590, 16311!, 16390[3] |
Happlogroup L2는 특히 Subequatorial Africa에서 광범위한 현대적 분포를 가진 인간 미토콘드리아 DNA(mtDNA) 하플로그룹이다. 그것의 L2a 하위 표지는 아프리카계 미국인들뿐만 아니라 대륙에 다소 빈번하고 널리 분포된 mtDNA 클러스터다.
기원
L2는 아프리카에서 흔한 혈통이다. 이것은 8만 7천년에서 10만[4] 7천년 전 사이에 진화했거나 약 9만 YBP라고 여겨진다.[1] 그것의 나이와 대륙 전체에 걸친 광범위한 분포와 다양성은 아프리카 내에서 그것의 정확한 기원을 어떠한 자신감으로도 추적하기 어렵게 만든다.[5] 기니인과 다른 서아프리카 인구에서 관찰된 여러 L2 happlotype은 동아프리카 및 북아프리카와 유전적 일치점을 공유했다.[6] 서아프리카나 중앙아프리카의 L2b, L2c, L2d, L2e의 기원일 가능성이 있어 보인다.[5] L2의 초기 다양성은 아프리카 대륙 전역에서 관찰할 수 있지만, 아래의 서브클라데스 섹션에서 볼 수 있듯이, 서아프리카에서 가장 높은 다양성이 발견된다. 대부분의 하위 건물들은 주로 서부와 중앙 아프리카에 국한되어 있다.[7]
2015년 한 연구에 따르면, "남아프리카의 성단은 최근 서아프리카/중앙아프리카 성단의 라인이라는 것을 보여준다. 반면, 동부 라인은 실질적으로 더 오래된 것처럼 보인다. 중앙아프리카 자원으로부터 3분간의 팽창은 L2와 연관된다: 70-50 ka에서 동부 또는 남부 아프리카로의 이동, 빙하 후의 이동 15-10 ka, 그리고 마지막 5 ka에서 남반투 팽창. 보완적 인구와 L0a 식물학 분석은 이후 이동 동안에 동부와 남부의 인구 사이에 mtDNA 유전자가 흐른다는 유력한 증거가 없다는 것을 나타내며, 동 아프리카 인구와 반투 이주민 사이의 혼화물이 낮음을 시사한다. 이는 적어도 초기 단계에서는 반투 확장이 주로 지역 인구의 거의 통합되지 않은 데미지 확산이었다는 것을 의미한다."[8]
분배
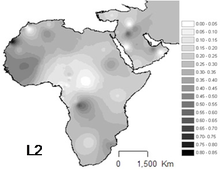
L2는 아프리카에서 가장 흔한 집단으로, 대륙 전역에서 관측되었다. 그것은 아프리카인과 그들의 최근 후손의 약 3분의 1에서 발견된다.
가장 높은 주파수는 음부티 피그미족(64%)[9] 중에서 발생한다. 서아프리카, 특히 세네갈(43~54%)[6]에서 중요한 존재감. 동아프리카의 비반투족 인구([10]44%)에서도 중요하며, 수단과 모잠비크의 인구도 중요하다.
특히 차드와 칸엠부(표본의 38%)에서는 풍부하지만, 유목 아랍인(33%)과 아칸인(약 33%)[11]에서도 비교적 빈번하게 발생하고 있다.
서브클라데스
|
L2에는 L2a, L2b, L2c, L2d, L2e의 5개의 주요 하위 그룹들이 있다. 이들 라인 중 가장 흔한 하위 표지는 L2a로 아프리카와 레반트 양쪽에서 발견된다.
하플로그룹 L2는 초기 기독교 시대(AD 550–800)인 수단 쿨루나티 섬 공동묘지의 표본 가운데 관찰됐다.[12]
하플로그룹 L2a
L2a는 아프리카에 널리 분포하고 있으며 사하라 사막 이남 아프리카 해플로그룹에 분포하고 있으며, 아프리카인의 후손들 사이에서 아메리카 대륙에서도 19%로 다소 빈번하다(Salas et al., 2002). L2a는 대략 48,000 YBP의 발생 가능한 날짜를 가지고 있다.[1]
특히 차드(표본의 38%; 차드 아랍인의 33% 미분화 L2),[13] 동아프리카의 비반투족(케냐, 우간다, 탄자니아)에서 38%[10]로 풍부하다. 모잠비크에서는[14] 약 33%, 가나에서는 약 32%.[11]
이 하위 표지는 2789, 7175, 7274, 7771, 11914, 13803, 14566, 16294에서 돌연변이로 특징지어진다. 전체 L2의 52%를 차지하며, L2의 하위 표지로 아프리카 전역에 널리 퍼져 있다.[15]
L2a와 다양성의 광범위한 분포는 지리적 기원을 식별하는 것을 어렵게 한다. 주요 퍼즐은 거의 어디서나 볼 수 있는 Happlogroup L2a인데, 이것은 마지막 빙하 최대치 이후 북아프리카의 사헬 회랑을 따라 동서로 퍼졌을 수도 있고, 혹은 이러한 팽창의 기원이 후기 석기 시대 초기에 있을 수도 있다.[5][15]
동아프리카의 L2a는 나일계곡-누비아에서 15%, 이집트인 5%, 에티오피아인 14%, 에티오피아인 14%, 세미티아인 암하라인 15%, 구라지인 10%, 티그레이-티그리냐인 6%, 에티오피아인 13%, 예멘인 5%에서 발견되었다.[14]
Happlogroup L2a도 북아프리카에서 나타나며, 가장 높은 주파수는 20% Tuareg, Fulani(14%)이다. 일부 알제리 아랍인들 사이에서도 발견되며, 모로코 아랍인, 일부 모로코인 베르베르인, 튀니지인 베르베르인 사이에서 10%로 발견된다. (1997년) 외, (1991년) 외, 1991년.
HIV를 치료하기 위해 스타부딘이라는 약물을 투여받은 환자들에게, Happlogroup L2a는 부작용으로서 말초 신경증의 낮은 확률과 관련이 있다.[16]
하플로그룹 L2a1
L2a는 16309(Salas et al. 2002)에서 전환기를 유지하며 L2a1로 더 나눌 수 있다.
This subclade is observed at varying frequencies in West Africa among the Malinke, Wolof, and others; among the North Africans;in the Sahel among the Hausa, Fulbe, and others; in Central Africa among the Bamileke, Fali, and others; in South Africa among the Khoisan family including the Khwe and Bantu speakers; and in East Africa among the Kikuyu f롬케냐.
에티오피아에 존재하는 모든 L2 클래드는 주로 L2a1과 L2b라는 두 개의 하위 클래드에서 파생된다. L2a1은 12693, 15784, 16309의 돌연변이에 의해 정의된다. 대부분의 에티오피아 L2a1 시퀀스는 nps 16189와 16309에서 돌연변이를 공유한다. 단, 대다수(33명 중 26명) 아프리카계 미국인이 Happlogroup L2a를 공유하는 반면, 완전한 시퀀스는 nps L2a1e-3495, L2a1a-3918, L2a1f-5581 및 L2a1i-15229로 대체하여 4개의 하위 테이블로 분할할 수 있다. 이러한 시퀀스 중 어떤 것도 에티오피아 16309 L2a1 샘플에서 관찰되지 않았다. (Salas 2002) 등.
하플로그룹 L2a1도 마하라(4.6%)[17]에서 관측됐다.
Happlogroup L2a1은 시리아 텔 할룰라의 신석기 전 신석기 문화와 관련된 고대 화석들에서 발견되었다.[18] 탄자니아 룩스만다의 사바나 목회 신석기 유적지에서 출토된 표본도 L2a1의 쇄골을 운반했다. 혼화재 군집 분석은 또한 개인이 고대 레반트에서 상당한 조상을 가지고 있다는 것을 보여주었고, 사바나 목회 신석기 제작자들과 사전 신석기 제작자들 사이의 조상 관계를 확인시켜 주었다.[19]
하플로그룹 L2a1a
서브클레이드 L2a1a는 3918, 5285, 15244, 15629에서 대체하여 정의된다. 아프리카 남동부 지역에서 잘 표현되는 L2a 클러스터는 L2a1a와 L2a1b로, 둘 다 상당히 안정적인 HVS-I 위치에서 전환에 의해 정의된다. 이 두 가지 모두 서아프리카나 북아프리카에서 기원한 것으로 보인다(매칭이나 이웃 유형의 분포에서 알 수 있듯이), 그리고 동남아프리카에서 또는 오늘날 동남아프리카에서 인구 조상으로 극적인 확장을 겪은 것으로 보인다.
매우 최근의 L2a1a와 L2a2 서브클라드에서의 별 폭발은 페레이라 외 연구진이 제안한 대로 반투 팽창의 시그니처를 암시한다. (2001).
L2a1a는 16286년의 돌연변이에 의해 정의된다. L2a1a 창업자 후보는 2700년 전(SE 1,200년)까지 거슬러 올라간다. 그러나 (np 16286) (Salas 등 2002)에서 대체에 의해 정의된 L2a1a는 현재 코딩-지역 표시기(np 3918)(그림 2A)에 의해 지원되며 예멘 L2a1 라인 6개 중 4개에서 발견되었다. L2a1a는 남아프리카공화국에서 가장 높은 빈도로 발생한다(Pereira et al. 2001; Salas et al. 2002). 예멘인들 사이에서 발견되는 빈번한 설립자 happlotype과 파생된 lineages (16092 돌연변이 포함)는 모두 모잠비크 시퀀스 내에서 정확히 일치한다(Pereira et al. 2001; Salas et al. 2002). L2a1a는 또한 말리와 모리타니아의 모레와 밤바라 사이에서 북아프리카에서 더 작은 주파수에서 발생한다.[20] (Rando et al. 1998; Maca-Meyer et al. 2003)
하플로그룹 L2a1a1a1a1
L2a1a1a1은 6152C, 15391T, 16368C 마커에 의해 정의된다.
하플로그룹 L2a1b
L2a1b는 16189와 10143으로 대체되어 정의된다. 16192는 L2a1b와 L2a1c에서도 흔히 나타난다. 이집트의 북아프리카에서도 나타나며 남동아프리카에서도 나타나기 때문에 반투 확장의 표식이 될 수도 있다.[5]
하플로그룹 L2a1c
L2a1c는 종종 돌연변이 16189를 L2a1b와 공유하지만 3010과 6663으로 자체 표지를 가지고 있다. 16192는 L2a1b와 L2a1c에서도 흔하다. 16192는 동아프리카뿐만 아니라 동남 아프리카에서도 나타난다.[21] 이것은 이 판이 어느 정도 다변화된다는 것을 암시한다.
L2a1 위에 위치 T16209C C16301T C16354T는 키비실드 외 연구진이 L2a1c로 명명하는 작은 하위 표지를 정의한다. (2004, 그림 3) (또한 살라스 외 2002의 그림 6 참조) 투르카나와 서아프리카(예: 카누리) 중에서 주로 동아프리카(수단, 누비아, 에티오피아)에서 나타난다.
차드 분지에서는 동서아프리카 유형으로부터 1, 2개의 서로 다른 L2a1c 유형들이 확인되었다. (키비실드 외). 2004.[21] (9페이지 또는 443페이지의 커미션)[22][23]
Haplogroup L2a1c1
L2a1c1은 북아프리카 출신이다.[24] 표식기 198, 930, 3308, 8604, 16086으로 정의된다. 튀니지 세파르디크, 아슈케나지, 유대인, 히브리인, 모로코인, 이집트인, 누비아인, 예멘인 등에서 관찰된다.
하플로그룹 L2a1f
호시안, 잠비아, 마다가스카르
하플로그룹 L2a1k
L2a1k는 G6722A 및 T12903C 마커에 의해 정의된다. 이전에 유럽 특유의 서브클레이드 L2a1a로 설명되었으며 체코와 슬로바키아에서 검출되었다.[25]
하플로그룹 L2a1l2a
L2a1l2a는 중유럽과 동유럽에서 조상을 가진 아슈케나지 유대인들 사이에서 볼 수 있는 "아슈케나지 특유의" 하플로그그룹으로 인정받고 있다. 표면상으로는 유웨이가 아닌 폴란드 인구에서도 소수로 검출되었는데, 아슈케나지 혼화물에서 온 것으로 추정된다.[26] 그러나, 이 happlotype은 Ashkenazi mitochondrial lineages 중 극히 일부에 불과하다; 다양한 연구(Behar's를 포함)는 그 발생률을 1.4–1.6%로 설정했다.
하플로그룹 L2a2
L2a2는 Mbuti Pygmies의 특징이다.[9]
하플로그룹 L2b'c
L2b'c는 약 62,000년 전에 진화했을 것이다.[1]
하플로그룹 L2b
이 하위 표지는 주로 서아프리카에서 발견되지만 아프리카 전역으로 퍼져 있다.[27]
하플로그룹 L2c
L2c는 서아프리카에서 가장 자주 발생하며, 그곳에서 발생했을 수도 있다.[15] 특히 세네갈에서는 39%, 케이프 베르데 16%, 기니비사우 16%[6]로 나타났다.
하플로그룹 L2d
L2d는 발생 가능성이 있는 서아프리카에서 가장 빈번하다.[15] 예멘, 모잠비크, 수단에서도 발견된다.[14]
하플로그룹 L2e
L2e(구 L2d2)는 서아프리카에서 대표적이다.[5] 튀니지에서도 발견되며,[28] 기니비사우 출신 만딩카족과 아프리카계 미국인 사이에서도 발견된다.[27]
나무
하플로그룹 L2 서브클라드의 이 계통생성 트리는 마니스 판 오븐과 맨프레드 케이서 업데이트의 논문에서 전지구 인간 미토콘드리아 DNA 변형의[3] 종합 계통생성 트리와 이후 발표된 연구에 바탕을 두고 있다.
- 가장 최근의 공통 조상(MRCA)
- L1'2'3'4'5'6"
- L2'3'4'6
- L2
- L2a'b'cd.
- L2a
- L2a1
- L2a1a
- L2a1a1a1
- L2a1a2
- L2a1a2a
- L2a1a2a1a1
- L2a1a2b
- L2a1a2a
- L2a1a3
- 16189 (16192)
- L2a1b
- L2a1b1
- L2a1f
- L2a1f1
- L2a1b
- 143
- L2a1c
- L2a1c1
- L2a1c2
- L2a1c3
- L2a1c4
- L2a1d
- L2a1e
- L2a1e1
- L2a1h
- 16189
- L2a1i
- L2a1j
- L2a1k
- 16192
- L2a1l
- L2a1l1
- L2a1l1a
- L2a1l2
- L2a1l1
- L2a1l
- L2a1c
- L2a1a
- L2a2
- L2a2a
- L2a2a1
- L2a2b
- L2a2b1
- L2a2a
- L2a1
- L2b'c
- L2b
- L2b1
- L2b1a
- L2b1a2
- L2b1a3
- L2b1a
- L2b1
- L2c
- L2c2
- L2c2a
- L2c3
- L2c2
- L2b
- L2d
- L2d1
- L2d1a
- L2d1
- L2a
- L2e
- L2a'b'cd.
- L2
- L2'3'4'6
- L1'2'3'4'5'6"
참고 항목
| 위키미디어 커먼즈에는 해플로그룹 L2(mtDNA)와 관련된 미디어가 있다. |
| 인간 미토콘드리아 DNA(mtDNA) 집단의 계통생성 나무 | |||||||||||||||||||||||||||||||||||||||
| 미토콘드리아 이브 (L) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1-6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | O | A | S | R | I | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | JT 이전 | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
참조
- ^ a b c d Soares, Pedro; Luca Ermini; Noel Thomson; Maru Mormina; Teresa Rito; Arne Röhl; Antonio Salas; Stephen Oppenheimer; Vincent Macaulay; Martin B. Richards (4 Jun 2009). "Correcting for Purifying Selection: An Improved Human Mitochondrial Molecular Clock". The American Journal of Human Genetics. 84 (6): 82–93. doi:10.1016/j.ajhg.2009.05.001. PMC 2694979. PMID 19500773.
- ^ Silva, Marina (2015). "60,000 years of interactions between Central and Eastern Africa documented by major African mitochondrial haplogroup L2". Scientific Reports. Nature. 5: 12526. Bibcode:2015NatSR...512526S. doi:10.1038/srep12526. PMC 4515592. PMID 26211407.
- ^ a b van Oven, Mannis; Manfred Kayser (13 Oct 2008). "Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation". Human Mutation. 30 (2): E386–E394. doi:10.1002/humu.20921. PMID 18853457. S2CID 27566749.
- ^ 티슈코프 외, 고대 아프리카 라인업, 분자 생물학 및 진화의 전체-mtDNA 게놈 시퀀스 분석, 제24권, 제3권(2007), 페이지 757–768.
- ^ a b c d e f 살라스, 안토니오 외 The Makeing of the African mtDNA Georacy, American Journal of Human Genetics, vol. 71, no. 5(2002), 페이지 1082–1111.
- ^ a b c Rosa, Alexandra; Brehm, A; Kivisild, T; Metspalu, E; Villems, R; et al. (2004). "MtDNA Profile of West Africa Guineans: Towards a Better Understanding of the Senegambia Region". Annals of Human Genetics. 68 (Pt 4): 340–352. doi:10.1046/j.1529-8817.2004.00100.x. hdl:10400.13/3044. PMID 15225159. S2CID 15391342.
- ^ 인간 여정의 지도책: Happlogroup L2 Archived 2011-10-06, 웨이백 머신 The Genographic Project, 내셔널 지오그래픽 프로젝트에 보관.
- ^ Silva, Marina; Alshamali, Farida; Silva, Paula; Carrilho, Carla; Mandlate, Flávio; Jesus Trovoada, Maria; Černý, Viktor; Pereira, Luísa; Soares, Pedro (2015). "60,000 years of interactions between Central and Eastern Africa documented by major African mitochondrial haplogroup L2". Scientific Reports. 5: 12526. Bibcode:2015NatSR...512526S. doi:10.1038/srep12526. PMC 4515592. PMID 26211407.
- ^ a b 퀸타나-머시 외 2008년 피그미 수렵-채집자와 반투어를 사용하는 농부들 사이의 깊은 공통 조상과 비대칭 유전자의 모성 흔적 '미국 국립과학원 진행' 105(5): 1599
- ^ a b Sadie Anderson-Mann 2006, 아프리카 미토콘드리아 DNA 변동에 대한 Philogenetic 및 Physlogical 분석. 2011-09-10년 웨이백 머신에 보관
- ^ a b 베라마, 크리슈나 R 외 2010년, 나이지리아의 크로스 리버 지역 사람들의 상당한 언어적 변화가 존재하는 상황에서 단일한 지표에 의해 평가된 작은 유전적 차별화.
- ^ Sirak, Kendra; Frenandes, Daniel; Novak, Mario; Van Gerven, Dennis; Pinhasi, Ron (2016). "Abstract Book of the IUAES Inter-Congress 2016 – A community divided? Revealing the community genome(s) of Medieval Kulubnarti using next-generation sequencing". Abstract Book of the Iuaes Inter-Congress 2016. IUAES: 115.
- ^ Cerezo, María; et al. (2011). "New insights into the Lake Chad Basin population structure revealed by high-throughput genotyping of mitochondrial DNA coding SNPs". PLOS ONE. 6 (4): e18682. Bibcode:2011PLoSO...618682C. CiteSeerX 10.1.1.291.8871. doi:10.1371/journal.pone.0018682. PMC 3080428. PMID 21533064.
- ^ a b c 토오마스 키비실드 외, 에티오피아 미토콘드리아 DNA 유산: Gene Flow Over and Around the Gate of Tears, American Journal of Human Genetics, vol. 75, 5호(2004년 11월), 페이지 752–770.
- ^ a b c d Antonio Torroni 등, MTNA Happlogroup L2의 4개 층이 다른 속도로 진화하는가?, 아메리칸 저널 오브 휴먼 유전학, 제69권(2001), 페이지 348–1356.
- ^ Kampira, E; Kumwenda, J; van Oosterhout, JJ; Dandara, C (Aug 2013). "Mitochondrial DNA subhaplogroups L0a2 and L2a modify susceptibility to peripheral neuropathy in malawian adults on stavudine containing highly active antiretroviral therapy". J Acquir Immune Defic Syndr. 63 (5): 647–652. doi:10.1097/QAI.0b013e3182968ea5. PMC 3815091. PMID 23614993.
- ^ Non, Amy. "ANALYSES OF GENETIC DATA WITHIN AN INTERDISCIPLINARY FRAMEWORK TO INVESTIGATE RECENT HUMAN EVOLUTIONARY HISTORY AND COMPLEX DISEASE" (PDF). University of Florida. Retrieved 2 November 2016.
- ^ Manco, Jean (2013). Ancestral Journeys: The Peopling of Europe from the First Venturers to the Vikings. Thames & Hudson. p. 88. ISBN 978-0500771822. Retrieved 29 September 2017.
- ^ Skoglund; et al. (September 21, 2017). "Reconstructing Prehistoric African Population Structure". Cell. 171 (1): 59–71. doi:10.1016/j.cell.2017.08.049. PMC 5679310. PMID 28938123.
- ^ 곤잘레스, A. M. 외 2006년, 모리타니아와 말리의 미토콘드리아 DNA 변이와 다른 서아프리카 인구와의 유전적 관계
- ^ a b "Wiley Online Library Scientific research articles, journals, books, and reference works". Archived from the original on 2010-08-05. Retrieved 2009-05-19.
- ^ Cˇny, V et al 2006, 사헬-수단 벨트의 양방향 회랑과 차드 유역 인구의 특징: 미토콘드리아 DNA 게놈에 의해 밝혀진 역사.
- ^ Lascaro, Daniela; castelhana, Stefano; Gasparre, Giuseppe; Romeo, Giovanni; Saccone, Cecilia; Attimonelli, Marcella (2008). "The RHNumtS compilation: Features and bioinformatics approaches to locate and quantify Human NumtS". BMC Genomics. 9: 267. doi:10.1186/1471-2164-9-267. PMC 2447851. PMID 18522722.
- ^ Lascaro, Daniela; Castellana, Stefano; Gasparre, Giuseppe; Romeo, Giovanni; Saccone, Cecilia; Attimonelli, Marcella (2008). "The RHNumtS compilation: Features and bioinformatics approaches to locate and quantify Human NumtS". BMC Genomics. 9: 267. doi:10.1186/1471-2164-9-267. PMC 2447851. PMID 18522722.
- ^ 보리스 A 말랴르쿠크, 미로슬라바 데렌코, 마리아 페르코바, 토마스 그르지보우스키, 토마스 베인체크, 얀 라주르, 슬라브에 있는 아프리카 미토콘드리아 DNA 라인의 기형 재구성, 유럽 인간유전학 저널 제16권, 페이지 1091-1096.
- ^ Marta Mielnik-Sikorska, Patrycja Daca, Boris Malyarchuk, Miroslava Derenko, Katarzyna Skonieczna, Maria Perkova, Tadeusz Dobosz, Tomasz Grzybowski, The History of Slavs Inferred from Complete Mitochondrial Genome Sequences PLOSOne January 14, 2013; 10.1371/journal.pone.0054360
- ^ a b Behar 등 2008b, The Dawn of Humilineal Diversity Am J HumGenet. 2008년 5월 9일; 82(5): 1130–1140
- ^ Costa MD 외 2009년, 튀니지 100세 이상 노인들의 완전한 MTDNA 시퀀싱 데이터: 집단 결속 테스트 및 장수에 대한 "황금 평균" ([1])
외부 링크
- 이안 로건의 하플로그룹 L2 미토콘드리아 DNA 사이트
- 이안 로건의 L2bcd. 미토콘드리아 DNA 사이트
- Mannis van Oven's PhyloTree.org – mtDNA 하위 트리 L
- 내셔널 지오그래픽에서 Happlogroup L2 확산