Haplogroup N(mtDNA)
Haplogroup N (mtDNA)| 하플로그룹N | |
|---|---|
 | |
| 가능한 출발 시간 | ~55-70,000 YBP[1][2] 또는 50-65,000 YBP[3] |
| 가능한 원산지 | 아시아[4][5][6][7][8] 또는 동아프리카[9][10][11] |
| 조상 | L3 |
| 후손 | N1'5, N2, N8, N9, N10, N11, N13, N14, N21, N22, A, I, O, R, S, X, Y, W |
| 돌연변이 정의 | 8701, 9540, 10398, 10873, 15301[12] |
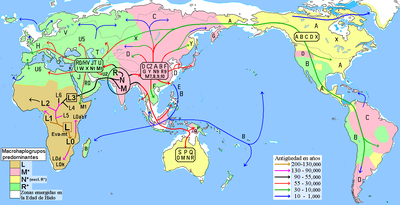
하플로그룹 N은 인간의 미토콘드리아 DNA(mtDNA) 쇄골이다. 매크로합로그 그룹, 그 후예 라인은 많은 대륙에 분포한다. 매크로홀로그그룹 M과 마찬가지로 매크로홀로그그룹 N은 haplogroup L3의 자손이다.
아프리카 밖에서 발견된 모든 mtDNA happlogroups는 happlogroup N의 후손이거나 형제 happlogroup M. M과 N은 현대인류의 최근 아프리카 기원과 이후 세계로의 초기 인간 이동에 대한 이론을 정의하는 대표적인 모성 happlogroups이다. Happlogroup N과 M의 전세계적인 분포는 아프리카에서 인류의 주요 선사시대 이주가 적어도 한 번 있었을 가능성이 있으며, N과 M 둘 다 나중에 대륙 밖에서 진화했을 가능성이 있다는 것을 보여준다.[7]
오리진스
Happlogroup L3(Happlogroup N's parent clade)의 아프리카 조상에 관한 과학계에서는 광범위한 합의가 이루어지고 있다.[13] 그러나, Happlogroup N 자체를 정의하는 돌연변이 자체가 아시아나 아프리카 내에서 처음 일어났는지 여부는 지속적인 논의와 연구의 대상이 되어 왔다.[13]
아프리카에서 나온 가설은 일반화된 합의를 얻었다. 그러나 많은 구체적인 의문점들이 여전히 해결되지 않고 있다. 유라시아를 식민지로 삼은 두 M과 N 매크로합로그 그룹이 출구 전에 이미 아프리카에 존재했는지를 아는 것은 곤혹스럽다.
토로니 외 2006년에 Happlogroups M, N, R이 동아프리카와 페르시아만 사이의 어딘가에서 발생했다고 진술했다.[14]
또한 Happlogroup N의 기원과 관련이 있는 것은 조상의 Happlogroup M, N, R이 아프리카에서 같은 이주의 일부였는지, 아니면 Happlogroup N이 Levant를 거쳐 북방 루트를 통해 아프리카를 떠났는지, M은 아프리카의 Horn을 통해 아프리카를 떠났는지 등이다. 이 이론은 Happlogroup N이 서유라시아에서 단연 우세하고, Happlogroup M이 서유라시아에서는 부재하지만, 인도에서는 우세하고 인도 동부에서 흔히 볼 수 있기 때문에 제시되었다. 그러나 동남아시아와 호주 원주민들 사이에서 고립된 "relict" 인구의 미토콘드리아 DNA 변화는 아프리카로부터 단 한 번의 분산만이 있었다는 견해를 지지한다. 동남아시아의 인구와 호주 원주민들은 모두 집단 M과 N의 뿌리 깊은 집단을 가지고 있다.[15] 따라서 유라시아와 오세아니아에 걸쳐 Happlogroup 내의 초기 지점의 분포는 3개 창업자-mtDNA 시나리오와 아프리카로부터의 단일 이주 경로를 지원한다.[16] 이러한 발견은 또한 인간 정착과 팽창의 초기 유전적 역사에서 인도 아대륙의 중요성을 강조한다.[17]
아시아 기원 가설
Happlogroup N의 원산지로서의 아시아 가설은 다음과 같이 뒷받침된다.
- Happlogroup N은 세계 모든 지역에서 발견되지만 사하라 이남 아프리카에서는 낮은 주파수를 가지고 있다. 여러 연구에 따르면, 아프리카에 Happlogroup N의 존재는 유라시아로부터의 후방 이주의 결과일 가능성이 가장 높다.[6]
- 가장 오래된 거시적 그룹 N의 집단은 아시아와 호주에서 발견된다.
- Happlogroup N이 호주나 신세계까지 먼 거리를 여행했지만 북아프리카와 혼 아프리카인 외에 아프리카 내의 다른 인구에는 영향을 미치지 못했다는 것은 역설적일 것이다.
- 동남아시아의 고립된 "relict" 인구의 미토콘드리아 DNA 변동은 아프리카로부터 단 한 번의 분산만이 있었다는 관점을 뒷받침한다.[15] 유라시아와 오세아니아에 걸친 Happlogroups M, N, R의 초기 지점의 분포는 3개 창업자-mtDNA 시나리오와 아프리카로부터의 단일 이주 경로에 대한 추가적인 증거를 제공한다.[16] 이러한 발견은 또한 인간 정착과 팽창의 초기 유전적 역사에서 인도 아대륙의 중요성을 강조한다.[17] 따라서 N의 역사는 남아시아에서 유래가 가장 유력한 M, R과 유사하다.
한 연구(Vai et al. 2019)는 리비아의 다카르코리 유적지에서 초기 북아프리카 신석기 시대의 모계 집단 N의 기저 분지를 발견하였다. 저자들은 N이 아라비아 반도의 L3에서 갈라져 나중에 북아프리카로 다시 이주할 가능성이 높으며 자매인 하플로그그룹 M도 중동의 L3에서 갈라졌을 가능성이 있다고 제안하지만, N이 북아프리카에서 갈라졌을 가능성이 있다고 제안하고, 더 많은 정보가 확실해지기 위해 필요하다고 진술하고 있다.[3]
아프리카 기원 가설
토오마스 키비실드에 따르면 "인도에서는 M과 N 이외의 L3 라인이 부족하고 비아프리카 미토콘드리아에서는 현대인류의 가장 초기 이주가 아프리카 혼을 넘어 떠나는 길을 통해 이미 이 두 mtDNA 조상들을 실어 나른 것으로 보인다"고 한다.[9]
2019년, Vai 외 연구진의 연구는 신석기 사하라에서 나온 하플로그룹 N의 기저부에 대한 증거를 제시했다. 그들은 N이 근동의 Happlogroup L3에서 이탈하여(아마 아라비아 반도에서는 아프리카에서 L3가 탈퇴한 이후), 북아프리카로 다시 이주했거나, 대신 북아프리카에서 기원했을 가능성이 있다고 제안한다.[3]
분배
하플로그룹 N은 조상의 L3 매크로합로그그룹에서 파생된 것으로, 현대인류의 최근 아프리카 기원 이론에서 논의된 이주를 나타낸다. Happlogroup N은 오늘날 유럽과 오세아니아에 분포하는 거의 모든 집단에 조상들의 하플로그룹이며, 아시아와 아메리카에서 많이 발견된다. 하플로그룹 M과 비슷한 시기에 발생한 것으로 추정된다. Happlogroup U6와 같은 Happlogroup N 하위층도 구석기시대 유럽이나 아시아로부터의 후방 이동으로 인해 북서쪽과 북동쪽 아프리카에서 높은 빈도에서 낮은 빈도로 발견된다. 4만 6천 ybp, 기저 U6*의 추정 연령이다.[18] R0, J, T와 같이 서 유라시아에서 흔한 다른 집단들도 북아프리카와 동아프리카 일부에서 흔하다.
하플로그룹 N의 후손 혈통 U6는 에피팔레오닉에서 유래한 타포랄트 유적지의 이베로마우루스 표본 중에서 발견되었다.[19] 사하라 사막 이남 아프리카에서는 N의 고대 표본이 몇 개 발견되었는데, 지금까지 케냐에서 가장 오래된 것은 기원전 2000년 경으로 거슬러 올라간다.[20] 또한, 이집트 중부의 아부시르 엘-멜렉 고고학 유적지에서 발굴된 고대 이집트 미라들 중 Happlogroup N은 프톨레마이크 이전/후기 신왕국 시대, 프톨레마이크 시대, 로마 시대의 미라들이다.[21]
부분군 분포
Happlogroup N의 파생된 집단은 매크로-Happlogroup R과 그 자손, 그리고 Happlogroup A, I, S, W, X, Y를 포함한다.
카디알과 에피카르디알 문화(카듐 도자기)[22]와 프리포터리 신석기 B에 속하는 화석 중에서 드물게 분류되지 않은 집단 N*이 발견되었다. 현대 알제리에서도 드물게 분류되지 않은 형태의 N이 보고되었다.[23]
- 하플로그룹 N1'5
- 하플로그룹 N2
- Happlogroup N3 – 모든 하위 그룹은 지금까지 벨라루스에서만 발견되었다.
- 하플로그룹 N3a
- Happlogroup N3a1
- 하플로그룹 N3b
- Happlogroup N7 – 모든 하위 그룹은 지금까지 캄보디아에서만 발견되었다.
- 하플로그룹 N7a
- 하플로그룹 N7a1
- 하플로그룹 N7a2
- 하플로그룹 N7b
- 하플로그룹 N7a
- Happlogroup N8 – 중국에서 발견.[34]
- Happlogroup N9 – 극동 지역에서 발견.[24] [TMRCA 45,709.7 ± 7,931.5 ybp; CI=95%[35]]
- Happlogroup N9a [TMRCA 17,520.4 ± 4,389.8 ybp; CI=95%][35]
- Happlogroup N9a12 – Khon Mueang(파이 구)[36][37]
- 하플로그룹 N9a-C16261T
- 하플로그룹 N9a-C16261T* – 베트남(킨)[38][37]
- Happlogroup N9a-A4129G-A4913G-T12354C-A12612G-C12636T-T16311C!!! – 타슈쿠르간(키르기스어)[39][37]
- Happlogroup N9a1'3 [TMRCA 15,007.4 ± 6,060.1 ybp; CI=95%][35]
- Happlogroup N9a1 – 중국어(대만 등에서는 Hakka), She, Tu, Uyghur, Tuvan, 몽골, Khamnigan,[40] Korea,[41][40] Japan [TMRCA 9200 (95% CI 7,100 <-> 11,600) ybp[37]]
- Happlogroup N9a3 – 중국 [TMRCA 11,500 (95% CI 7,500 <-> 16,800) ybp[37]]
- Happlogroup N9a3a – 일본, 한국어(서울), 대만(incl) Paiwan), Thailand (Mon from Lopburi Province and Kanchanaburi Province[36]), China, Uyghur, Kyrgyz (Tashkurgan), Kazakhstan, Buryat, Russia (Belgorod, Chechen Republic, etc.), Ukraine, Moldova, Belarus, Lithuania, Poland, Czech (West Bohemia), Hungary, Austria, Germany [TMRCA 8,280.9 ± 5,124.4 ybp; CI=95%[35]]
- Happlogroup N9a2'4'5'11 [TMRCA 15,305.4 ± 4,022.6 ybp; CI=95%][35]
- Happlogroup N9a2 – 일본, 한국,[41] 중국 (Hulunbuir, Uyghur 등지의 Barghut) [TMRCA 10,700 (95% CI <-> 13,800) ybp[37]]
- Happlogroup N9a2a – 일본, 한국, 위구르 [TMRCA 8,100 (95% CI 6,500 <-> 10,000) ybp[37]]
- Happlogroup N9a2a1 – 일본 [TMRCA 4,200 (95% CI 1,850 <-> 8,400) ybp[37]]
- Happlogroup N9a2a2 – 일본, 한국, 볼가-우랄 지역(타타르)[40] [TMRCA 5,700 (95% CI 3,500 <-> 8,900) ybp[37]]
- Happlogroup N9a2a3 – 일본, 훌룬부아르 지역(Barghut)[40] [TMRCA 4,700 (95% CI 2,400 <-> 8,400) ybp[37]]
- Happlogroup N9a2a4 – 일본 [TMRCA 2,800 (95% CI 600 <-> 7,900) ybp[37]]
- Happlogroup N9a2b – 중국
- Happlogroup N9a2c [TMRCA 7,200 (95% CI 3,600 <-> 12,700) ybp[37]]
- Happlogroup N9a2c* – 일본
- Happlogroup N9a2c1 – 일본, Uyghur [TMRCA 3,700 (95% CI 2,000 <-> 6,100) ybp[37]]
- Happlogroup N9a2d – 일본 [TMRCA 4,900 (95% CI 1,650 <-> 11,400) ybp[37]]
- Happlogroup N9a2e – 중국
- Happlogroup N9a2a – 일본, 한국, 위구르 [TMRCA 8,100 (95% CI 6,500 <-> 10,000) ybp[37]]
- Happlogroup N9a4 – 말레이시아 [TMRCA 7,900 (95% CI 3,900 <-> 14,300) ybp[37]]
- Happlogroup N9a5 [TMRCA 8,700 (95% CI 4,700 <-> 15,000) ybp[37]]
- Happlogroup N9a5* – 한국
- Happlogroup N9a5a – 일본
- Happlogroup N9a5b – 일본 [TMRCA 5,300 (95% CI 1,150 <-> 15,300) ybp[37]]
- Happlogroup N9a11 – 대만(Hakka, Minnan), 라오스(Lao from Luang Prabang[36])
- Happlogroup N9a2 – 일본, 한국,[41] 중국 (Hulunbuir, Uyghur 등지의 Barghut) [TMRCA 10,700 (95% CI <-> 13,800) ybp[37]]
- Happlogroup N9a6 – 태국(Lopburi 성에서 ph,[36] Lamphun 성에서 Khon Mueang,[36]사콘 나콘 성 푸타이,[36] 매홍 손 성 라와,[36] 사콘 나콘[36] 성 소아, 베트남 수마트라 [TMRCA 11,972.5 ± 5,491.7ybp; CI=95%][35]
- Happlogroup N9a6a – 캄보디아(Khmer), 말레이시아(Bydayuh, Jehai, Temuan, Kensiu), 수마트라, 순다네어
- Happlogroup N9a6b – 말레이시아(셀레타)
- Happlogroup N9a7 – 일본
- Happlogroup N9a8 – 일본, 중국, 부랴트
- Happlogroup N9a9 – Chelkans (Biyka, Turochak), Tubalar (북동 알타이), 키르기스스탄 (키르기스스탄), 중국, 우크라이나 (Vinnytsia Obruja), 루마니아 (10세기 AD Dobruja)
- 하플로 그룹 N9a10 – 태국(Khon 므앙 매홍손 주, 치앙 마이 주, 람푼 주, 람빵 Province,[36]Shan 매홍손 Province,[36]Lao 이상 르이 Province,[36]Black 타이 깐짜나Province,[36]Phuan에서 수코타이 주에서 피찟 Province,[36]Mon 깐짜나Province[36]의), 라오스(라오 L.에서uang Prabang,[36]Hmong[38]), 비 엉tnam(Tay Nung), 중국(incl). 충칭의 한)
- Happlogroup N9a10a – 중국, 대만(아미)
- Happlogroup N9a10a1 – 중국어(수저우)
- Happlogroup N9a10a2 – 필리핀(이바탄), 대만(아미)
- Happlogroup N9a10a2a – 대만(아타얄, Tsou)
- Happlogroup N9a10b – 중국
- Happlogroup N9a10a – 중국, 대만(아미)
- Happlogroup N9b – 일본, 우데이지, 나나이, 한국[41] [TMRCA 14,885.6 ± 4,092.5ybp; CI=95%][35]
- 하플로 그룹 Y[43]– Nivkhs, Ulchs, Nanais, Negidals, Ainus, 니아스 섬의 인구 중에 다른 퉁구스 민족 한국인들, 몽골족, 코랴크인, Itelmens, 중국어, 일본어, Tajiks, 섬 동남 아시아(타이완 원주민 등)사이에 중간 빈도의 일부 튀르크 어파 peoples[26]을 발견했다[TMRCA 24,576.4±7,083.2 Y염기쌍, CI=95%[35]]
- Happlogroup Y1 – 한국, 대만(민난), 태국(파야오[44][37] 성 Iu Mien), 폴란드, 슬로바키아, 체코 [TMRCA 14,689.5 ± 5,264.3 ybp; CI=95%][35]
- Happlogroup Y2 – 중국어, 일본어, 한국어, Khamnigan, 남아프리카 공화국(케이프 컬러) [TMRCA 7,279.3 ± 2,894.5 ybp; CI=95%][35]
- Happlogroup N9a [TMRCA 17,520.4 ± 4,389.8 ybp; CI=95%][35]
- Happlogroup N10 – 중국(상하이, 장쑤, 푸젠, 광둥성, 윈난, 하니, 이이, 윈난, 구이저우, 우즈베키스탄, 신장) 및 동남아시아(태국, 인도네시아, 베트남, 말레이시아)[34]에서 발견.
- Happlogroup N11 – 중국(윈난, 쓰촨, 후베이 출신 한족, 시짱 출신 티벳족, 간쑤 출신 둥샹족, 내몽골 출신 오로첸) 및 필리핀에서 발견됨.[34][47]
- 하플로그룹 O 또는 N12-는 인도네시아의 토착 원주민과 플로레아인들 사이에서 발견된다.
- Happlogroup N13 – 오스트레일리아 원주민[48]
- Happlogroup N14 – 오스트레일리아 원주민[49]
- Happlogroup [50]N21 – 말레이시아 및 인도네시아의 말레이계 인종
- Happlogroup N22 – 동남아시아, 방글라데시, 인도, 일본[51]
- Happlogroup A[52] – 중앙아시아와 동아시아뿐만 아니라 미국 원주민들 사이에서 발견된다.
- Happlogroup S – 호주[53] 원주민들 사이에서 확장되었다.
- Happlogroup X[54] – 서유라시아에서 가장 많이 발견되지만, 아메리카 대륙에서도 발견된다.[24]
- Happlogroup X1 – 주로 북아프리카와 레반트의 일부 인구, 특히 Druze 인구에서 발견됨
- Happlogroup X2 – 서유라시아, 시베리아 및 북미 원주민 사이에서 발견됨
- Happlogroup[55] R – 매우 확장되고 다각화된 매크로 Happlogroup.
서브클라데스
나무
이 하플로그룹 N 서브클라드의 계통생성 트리는 마니스 판 오븐과 맨프레드 케이서 업데이트의 논문에서 전지구 인간 미토콘드리아 DNA 변형의[12] 종합적인 계통생성 트리와 이후 발표된 연구에 바탕을 두고 있다.
- N
참고 항목
| 인간 미토콘드리아 DNA(mtDNA) 집단의 계통생성 나무 | |||||||||||||||||||||||||||||||||||||||
| 미토콘드리아 이브 (L) | |||||||||||||||||||||||||||||||||||||||
| L0 | L1-6 | ||||||||||||||||||||||||||||||||||||||
| L1 | L2 | L3 | L4 | L5 | L6 | ||||||||||||||||||||||||||||||||||
| M | N | ||||||||||||||||||||||||||||||||||||||
| CZ | D | E | G | Q | O | A | S | R | I | W | X | Y | |||||||||||||||||||||||||||
| C | Z | B | F | R0 | JT 이전 | P | U | ||||||||||||||||||||||||||||||||
| HV | JT | K | |||||||||||||||||||||||||||||||||||||
| H | V | J | T | ||||||||||||||||||||||||||||||||||||
참조
- ^ Soares, Pedro; Ermini, Luca; Thomson, Noel; Mormina, Maru; Rito, Teresa; Röhl, Arne; Salas, Antonio; Oppenheimer, Stephen; MacAulay, Vincent; Richards, Martin B. (2009). "Correcting for Purifying Selection: An Improved Human Mitochondrial Molecular Clock". The American Journal of Human Genetics. 84 (6): 740–59. doi:10.1016/j.ajhg.2009.05.001. PMC 2694979. PMID 19500773.
- ^ Soares, P; Alshamali, F; Pereira, J. B; Fernandes, V; Silva, N. M; Afonso, C; Costa, M. D; Musilova, E; MacAulay, V; Richards, M. B; Cerny, V; Pereira, L (2011). "The Expansion of mtDNA Haplogroup L3 within and out of Africa". Molecular Biology and Evolution. 29 (3): 915–927. doi:10.1093/molbev/msr245. PMID 22096215.
- ^ a b c Vai S, Sarno S, Lari M, Luiselli D, Manzi G, Gallinaro M, Mataich S, Hübner A, Modi A, Pilli E, Tafuri MA, Caramelli D, di Lernia S (March 2019). "Ancestral mitochondrial N lineage from the Neolithic 'green' Sahara". Sci Rep. 9 (1): 3530. Bibcode:2019NatSR...9.3530V. doi:10.1038/s41598-019-39802-1. PMC 6401177. PMID 30837540.
- ^ MacAulay, V.; Hill, C; Achilli, A; Rengo, C; Clarke, D; Meehan, W; Blackburn, J; Semino, O; et al. (2005). "Single, Rapid Coastal Settlement of Asia Revealed by Analysis of Complete Mitochondrial Genomes" (PDF). Science. 308 (5724): 1034–36. Bibcode:2005Sci...308.1034M. doi:10.1126/science.1109792. PMID 15890885. S2CID 31243109.
Haplogroup L3 (the African clade that gave rise to the two basal non-African clades, haplogroups M and N) is 84,000 years old, and haplogroups M and N themselves are almost identical in age at 63,000 years old, with haplogroup R diverging rapidly within haplogroup N 60,000 years ago.
- ^ Richards, Martin; Bandelt, Hans-Jürgen; Kivisild, Toomas; Oppenheimer, Stephen (2006). "A Model for the Dispersal of Modern Humans out of Africa". In Bandelt, Hans-Jürgen; Macaulay, Vincent; Richards, Martin (eds.). Human Mitochondrial DNA and the Evolution of Homo sapiens. Nucleic Acids and Molecular Biology. 18. pp. 225–65. doi:10.1007/3-540-31789-9_10. ISBN 978-3-540-31788-3.
subclades. L3b d, L3e and L3f, for instance, are clearly of African origin, whereas haplogroup N is of apparently Eurasian origin
- ^ a b Gonder, M. K.; Mortensen, H. M.; Reed, F. A.; De Sousa, A.; Tishkoff, S. A. (2006). "Whole-mtDNA Genome Sequence Analysis of Ancient African Lineages". Molecular Biology and Evolution. 24 (3): 757–68. doi:10.1093/molbev/msl209. PMID 17194802.
- ^ a b Olivieri, A.; Achilli, A.; Pala, M.; Battaglia, V.; Fornarino, S.; Al-Zahery, N.; Scozzari, R.; Cruciani, F.; Behar, D. M.; Dugoujon, J.-M.; Coudray, C.; Santachiara-Benerecetti, A. S.; Semino, O.; Bandelt, H.-J.; Torroni, A. (2006). "The mtDNA Legacy of the Levantine Early Upper Palaeolithic in Africa". Science. 314 (5806): 1767–70. Bibcode:2006Sci...314.1767O. doi:10.1126/science.1135566. PMID 17170302. S2CID 3810151.
The scenario of a back-migration into Africa is supported by another feature of the mtDNA phylogeny. Haplogroup M's Eurasian sister clade, haplogroup N, which has a very similar age to M and no indication of an African origin
- ^ a b c Abu-Amero, Khaled K; Larruga, José M; Cabrera, Vicente M; González, Ana M (2008). "Mitochondrial DNA structure in the Arabian Peninsula". BMC Evolutionary Biology. 8: 45. doi:10.1186/1471-2148-8-45. PMC 2268671. PMID 18269758.
- ^ a b Kivisild T, Rootsi S, Metspalu M,(알.(2003년)."인디언 종족과 caste 인구에 최초의 정착자들의 그 유전적 유산이 지속되".아메리칸 저널 JournalofHumanGenetics.72(2):313–32. doi:10.1086/346068.PMC 379225.PMID 12536373."또한, L3종 M과 N인도에서, 그리고 일반적인 비아프 리카의 미토콘드리아에 비해 다른의 부족이 현대 인류의 초기 migration(s)이미 Africa."의 뿔에 대한 출발 경로를 통해 이 두 mtDNA 조상들을 제시한다.
- ^ Kivisild; et al. (2007). "Genetic Evidence of Modern Human Dispersals in South Asia". The Evolution and History of Human Populations in South Asia. ISBN 9781402055621.
- ^ Osman, Maha M.; et al. "Mitochondrial HVRI and whole mitogenome sequence variations portray similar scenarios on the genetic structure and ancestry of northeast Africans" (PDF). Institute of Endemic Diseases. Meta Gene.
- ^ a b van Oven M, Kayser M (2009). "Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation". Human Mutation. 30 (2): E386–94. doi:10.1002/humu.20921. PMID 18853457. S2CID 27566749.
- ^ a b González, Ana M; Larruga, José M; Abu-Amero, Khaled K; Shi, Yufei; Pestano, José; Cabrera, Vicente M (2007). "Mitochondrial lineage M1 traces an early human backflow to Africa". BMC Genomics. 8: 223. doi:10.1186/1471-2164-8-223. PMC 1945034. PMID 17620140.
- ^ Torroni, A; Achilli, A; MacAulay, V; Richards, M; Bandelt, H (2006). "Harvesting the fruit of the human mtDNA tree". Trends in Genetics. 22 (6): 339–45. doi:10.1016/j.tig.2006.04.001. PMID 16678300.
- ^ a b MacAulay, V.; Hill, C; Achilli, A; Rengo, C; Clarke, D; Meehan, W; Blackburn, J; Semino, O; et al. (2005). "Single, Rapid Coastal Settlement of Asia Revealed by Analysis of Complete Mitochondrial Genomes" (PDF). Science. 308 (5724): 1034–36. Bibcode:2005Sci...308.1034M. doi:10.1126/science.1109792. PMID 15890885. S2CID 31243109.
- ^ a b c Palanichamy MG, Sun C, Agrawal S, et al. (2004). "Phylogeny of mitochondrial DNA macrohaplogroup N in India, based on complete sequencing: implications for the peopling of South Asia". American Journal of Human Genetics. 75 (6): 966–78. doi:10.1086/425871. PMC 1182158. PMID 15467980.
- ^ a b Maji, Suvendu; Krithika, S.; Vasulu, T. S. (2008). "Distribution of Mitochondrial DNA Macrohaplogroup N in India with Special Reference to Haplogroup R and its Sub-Haplogroup U" (PDF). International Journal of Human Genetics. 8 (1–2): 85–96. doi:10.1080/09723757.2008.11886022. S2CID 14231815.
- ^ Hervella, M.; et al. (2016). "The mitogenome of a 35,000-year-old Homo sapiens from Europe supports a Paleolithic back-migration to Africa". Scientific Reports. 6: 25501. Bibcode:2016NatSR...625501H. doi:10.1038/srep25501. PMC 4872530. PMID 27195518.
- ^ Bernard Secher; Rosa Fregel; José M Larruga; Vicente M Cabrera; Phillip Endicott; José J Pestano; Ana M González (2014). "The history of the North African mitochondrial DNA haplogroup U6 gene flow into the African, Eurasian and American continents". Scientific Reports. 14: 109. doi:10.1186/1471-2148-14-109. PMC 4062890. PMID 24885141.
- ^ Prendergast, Mary E.; Lipson, Mark; Sawchuk, Elizabeth A.; Olalde, Iñigo; Ogola, Christine A.; Rohland, Nadin; Sirak, Kendra A.; Adamski, Nicole; Bernardos, Rebecca; Broomandkhoshbacht, Nasreen; Callan, Kimberly; Culleton, Brendan J.; Eccles, Laurie; Harper, Thomas K.; Lawson, Ann Marie; Mah, Matthew; Oppenheimer, Jonas; Stewardson, Kristin; Zalzala, Fatma; Ambrose, Stanley H.; Ayodo, George; Gates, Henry Louis; Gidna, Agness O.; Katongo, Maggie; Kwekason, Amandus; Mabulla, Audax Z. P.; Mudenda, George S.; Ndiema, Emmanuel K.; Nelson, Charles; Robertshaw, Peter; Kennett, Douglas J.; Manthi, Fredrick K.; Reich, David (5 July 2019). "Ancient DNA reveals a multistep spread of the first herders into sub-Saharan Africa". Science. 365 (6448): eaaw6275. Bibcode:2019Sci...365.6275P. doi:10.1126/science.aaw6275. PMC 6827346. PMID 31147405.
- ^ Schuenemann, Verena J.; et al. (2017). "Ancient Egyptian mummy genomes suggest an increase of Sub-Saharan African ancestry in post-Roman periods". Nature Communications. 8: 15694. Bibcode:2017NatCo...815694S. doi:10.1038/ncomms15694. PMC 5459999. PMID 28556824.
- ^ Fernández, Eva; et al. (2014). "Ancient DNA analysis of 8000 BC near eastern farmers supports an early neolithic pioneer maritime colonization of Mainland Europe through Cyprus and the Aegean Islands". PLOS Genetics. 10 (6): e1004401. doi:10.1371/journal.pgen.1004401. PMC 4046922. PMID 24901650.
- ^ Bekada, Asmahan; Arauna, Lara R.; Deba, Tahria; Calafell, Francesc; Benhamamouch, Soraya; Comas, David; Kayser, Manfred (24 September 2015). "Genetic Heterogeneity in Algerian Human Populations". PLOS ONE. 10 (9): e0138453. Bibcode:2015PLoSO..1038453B. doi:10.1371/journal.pone.0138453. PMC 4581715. PMID 26402429.
- ^ a b c "Ian Logan's mtDNA site". Archived from the original on 2011-12-09. Retrieved 2009-07-17.
- ^ A. Stevanovitch; A. Gilles; E. Bouzaid; R. Kefi; F. Paris; R. P. Gayraud; J. L. Spadoni; F. El-Chenawi; E. Béraud-Colomb (January 2004). "Mitochondrial DNA Sequence Diversity in a Sedentary Population from Egypt". Annals of Human Genetics. 68 (1): 23–39. doi:10.1046/j.1529-8817.2003.00057.x. PMID 14748828. S2CID 44901197.
- ^ a b c Derenko, M; Malyarchuk, B; Grzybowski, T; Denisova, G; Dambueva, I; Perkova, M; Dorzhu, C; Luzina, F; Lee, H; Vanecek, Tomas; Villems, Richard; Zakharov, Ilia (2007). "Phylogeographic Analysis of Mitochondrial DNA in Northern Asian Populations". The American Journal of Human Genetics. 81 (5): 1025–41. doi:10.1086/522933. PMC 2265662. PMID 17924343.
- ^ Lipson, Mark; Szécsényi-Nagy, Anna; Mallick, Swapan; Pósa, Annamária; Stégmár, Balázs; Keerl, Victoria; Rohland, Nadin; Stewardson, Kristin; Ferry, Matthew; Michel, Megan; Oppenheimer, Jonas; Broomandkhoshbacht, Nasreen; Harney, Eadaoin; Nordenfelt, Susanne; Llamas, Bastien; Gusztáv Mende, Balázs; Köhler, Kitti; Oross, Krisztián; Bondár, Mária; Marton, Tibor; Osztás, Anett; Jakucs, János; Paluch, Tibor; Horváth, Ferenc; Csengeri, Piroska; Koós, Judit; Sebők, Katalin; Anders, Alexandra; Raczky, Pál; Regenye, Judit; Barna, Judit P.; Fábián, Szilvia; Serlegi, Gábor; Toldi, Zoltán; Gyöngyvér Nagy, Emese; Dani, János; Molnár, Erika; Pálfi, György; Márk, László; Melegh, Béla; Bánfai, Zsolt; Domboróczki, László; Fernández-Eraso, Javier; Antonio Mujika-Alustiza, José; Alonso Fernández, Carmen; Jiménez Echevarría, Javier; Bollongino, Ruth; Orschiedt, Jörg; Schierhold, Kerstin; Meller, Harald; Cooper, Alan; Burger, Joachim; Bánffy, Eszter; Alt, Kurt W.; Lalueza-Fox, Carles; Haak, Wolfgang; Reich, David (8 November 2017). "Parallel palaeogenomic transects reveal complex genetic history of early European farmers". Nature. 551 (7680): 368–372. Bibcode:2017Natur.551..368L. doi:10.1038/nature24476. PMC 5973800. PMID 29144465.
- ^ Kılınç, Gülşah Merve, Omrak, Ayça, Özer, Füsun, 귄터, Torsten, Büyükkarakaya, 알리 Metin, Bıçakçı, 에르한, 베어드, 더글러스, Dönertaş, 한단 Melike, Ghalichi, Ayshin, Yaka, Reyhan, Koptekin, Dilek, Açan, 신안 수 있지, Parvizi, Poorya, Krzewińska, 마하, Daskalaki, Evangelia A;.Yüncü, To. 에렌, Dağtaş, Nihan Dilşad, Fairbairn, 앤드류, 피어슨, 제시카, Mustafaoğlu, 괴크한;Erdal, Yılmaz Selim, Çakan, 야신 괴크한;Togan, İnci, Somel, 메흐멧 2세;Storå, 얀, Jakobsson, Mattias, Götherström, 안데르스(1010월 2016년)."그 인구 개발 제1차 농업인의 아나톨리아에서".현재 생물학. 26(19):2659–2666. doi:10.1016/j.cub.2016.07.057.PMC 5069350.PMID 27498567.-
- ^ a b Lippold, Sebastian; Xu, Hongyang; Ko, Albert; Li, Mingkun; Renaud, Gabriel; Butthof, Anne; Schröder, Roland; Stoneking, Mark (2014). "Human paternal and maternal demographic histories: insights from high-resolution Y chromosome and mtDNA sequences". Investigative Genetics. 5: 13. bioRxiv 10.1101/001792. doi:10.1186/2041-2223-5-13. PMC 4174254. PMID 25254093.
- ^ "Haplogroups I & N". Ianlogan.co.uk. Retrieved 2010-07-29.
- ^ Turchi, Chiara; Buscemi, Loredana; Previderè, Carlo; Grignani, Pierangela; Brandstätter, Anita; Achilli, Alessandro; Parson, Walther; Tagliabracci, Adriano; Ge.f.i., Group (2007). "Italian mitochondrial DNA database: results of a collaborative exercise and proficiency testing". International Journal of Legal Medicine. 122 (3): 199–204. doi:10.1007/s00414-007-0207-1. PMID 17952451. S2CID 11496367.
- ^ "Haplogroup W". Ianlogan.co.uk. Retrieved 2010-07-29.
- ^ Metspalu M, Kivisild T, Metspalu E, et al. (2004). "Most of the extant mtDNA boundaries in south and southwest Asia were likely shaped during the initial settlement of Eurasia by anatomically modern humans". BMC Genetics. 5: 26. doi:10.1186/1471-2156-5-26. PMC 516768. PMID 15339343.
- ^ a b c 콩, 칭펑 외 2011년, 대규모 mtDNA 상영에서 동아시아의 놀라운 모계 복잡성과 그것이 지역 주민에게 미치는 영향을 밝혀냈다.
- ^ a b c d e f g h i j k l m n o p q r s t 베하르 외, 2012b
- ^ a b c d e f g h i j k l m n o p q Soanboon, Pattanawit; Nanakorn, Somsong; Kutanan, Wibhu (June 2016). "Determination of sex difference from fingerprint ridge density in northeastern Thai teenagers". Egyptian Journal of Forensic Sciences. 6 (2): 185–193. doi:10.1016/j.ejfs.2015.08.001.
- ^ a b c d e f g h i j k l m n o p q r s t u v YFull Happlogroup YTree v6.05.11 2018년 9월 25일.
- ^ a b Brandão, Andreia; Eng, Ken Khong; Rito, Teresa; Cavadas, Bruno; Bulbeck, David; Gandini, Francesca; Pala, Maria; Mormina, Maru; Hudson, Bob; White, Joyce; Ko, Tsang-Ming; Saidin, Mokhtar; Zafarina, Zainuddin; Oppenheimer, Stephen; Richards, Martin B.; Pereira, Luísa; Soares, Pedro (2016). "Quantifying the legacy of the Chinese Neolithic on the maternal genetic heritage of Taiwan and Island Southeast Asia". Human Genetics. 135 (4): 363–376. doi:10.1007/s00439-016-1640-3. PMC 4796337. PMID 26875094.
- ^ a b Peng, Min-Sheng; Xu, Weifang; Song, Jiao-Jiao; Chen, Xing; Sulaiman, Xierzhatijiang; Cai, Liuhong; Liu, He-Qun; Wu, Shi-Fang; Gao, Yun; Abdulloevich, Najmudinov Tojiddin; Afanasevna, Manilova Elena; Ibrohimovich, Khudoidodov Behruz; Chen, Xi; Yang, Wei-Kang; Wu, Miao; Li, Gui-Mei; Yang, Xing-Yan; Rakha, Allah; Yao, Yong-Gang; Upur, Halmurat; Zhang, Ya-Ping (29 November 2017). "Mitochondrial genomes uncover the maternal history of the Pamir populations". European Journal of Human Genetics. 26 (1): 124–136. doi:10.1038/s41431-017-0028-8. PMC 5839027. PMID 29187735.
- ^ a b c d Derenko, Miroslava; Malyarchuk, Boris; Denisova, Galina; Perkova, Maria; Rogalla, Urszula; Grzybowski, Tomasz; Khusnutdinova, Elza; Dambueva, Irina; Zakharov, Ilia (21 February 2012). "Complete Mitochondrial DNA Analysis of Eastern Eurasian Haplogroups Rarely Found in Populations of Northern Asia and Eastern Europe". PLOS ONE. 7 (2): e32179. Bibcode:2012PLoSO...732179D. doi:10.1371/journal.pone.0032179. PMC 3283723. PMID 22363811.
- ^ a b c 환영, 유지은, 박명진, 정욱희, 김총열, 신경진, "동아시아 mtDNA 한국인들의 결심: Happlogroup 수준 코딩 영역 SNP 분석 및 하위 그룹 수준 제어 영역 시퀀스 분석." 전기영양증(2006년). DOI 10.1002/엘프.200600151.
- ^ Duong, Nguyen Thuy; Macholdt, Enrico; Ton, Nguyen Dang; Arias, Leonardo; Schröder, Roland; Van Phong, Nguyen; Thi Bich Thuy, Vo; Ha, Nguyen Hai; Thi Thu Hue, Huynh; Thi Xuan, Nguyen; Thi Phuong Oanh, Kim; Hien, Le Thi Thu; Hoang, Nguyen Huy; Pakendorf, Brigitte; Stoneking, Mark; Van Hai, Nong (3 August 2018). "Complete human mtDNA genome sequences from Vietnam and the phylogeography of Mainland Southeast Asia". Scientific Reports. 8 (1): 11651. Bibcode:2018NatSR...811651D. doi:10.1038/s41598-018-29989-0. PMC 6076260. PMID 30076323.
- ^ "Haplogroup Y". Ianlogan.co.uk. 2009-09-22. Retrieved 2010-07-29.
- ^ 위부 쿠타난, 라스미 슈우콩데지, 메타위 스리쿰몰 등(2020년)은 "문화적 변화는 태국의 흐몽미엔과 중-티베탄 그룹의 부계 및 모계 유전자에 영향을 미친다"고 말했다. 유럽 인간 유전학 저널 https://doi.org/10.1038/s41431-020-0693-x
- ^ a b c d e 가족나무 DNA에서의 Y-DNA D 해플로그룹 프로젝트
- ^ 홍빈 야오, 멍게왕, 싱조우 등은 "게놈의 광범위한 유전적 유산을 통해 허시 회랑에 있는 중국 서북부의 인구 혼화의 미세한 규모의 역사에 대한 새로운 통찰"이라고 말했다. MolGenet Genomics 2021년 3월 1일 도이: 10.1007/s00438-021-01767-0.
- ^ Gunnarsdottir, Ellen et al. 2010, 필리핀에서 온 완전한 인간 mtDNA 게놈의 높은 처리량 시퀀싱
- ^ "Hudjashov". Ianlogan.co.uk. Retrieved 2010-07-29.
- ^ Pierson MJ, Martinez-Arias R, Holland BR, Gemmell NJ, Hurles ME, Penny D (2006). "Deciphering past human population movements in Oceania: provably optimal trees of 127 mtDNA genomes". Molecular Biology and Evolution. 23 (10): 1966–75. doi:10.1093/molbev/msl063. PMC 2674580. PMID 16855009.
- ^ Hill, C.; Soares, P.; Mormina, M.; MacAulay, V.; Meehan, W.; Blackburn, J.; Clarke, D.; Raja, J. M.; Ismail, P.; Bulbeck, D.; Oppenheimer, S.; Richards, M. (2006). "Phylogeography and Ethnogenesis of Aboriginal Southeast Asians". Molecular Biology and Evolution. 23 (12): 2480–91. doi:10.1093/molbev/msl124. PMID 16982817.
- ^ Rieux, Adrien; Eriksson, Anders; Li, Mingkun; et al. (2014). "Improved Calibration of the Human Mitochondrial Clock Using Ancient Genomes". Mol Biol Evol. 31 (10): 2780–92. doi:10.1093/molbev/msu222. PMC 4166928. PMID 25100861.
- ^ "Haplogroup A". Ianlogan.co.uk. Retrieved 2010-07-29.
- ^ "Haplogroup S". Ianlogan.co.uk. Retrieved 2010-07-29.
- ^ "Haplogroup X". Ianlogan.co.uk. Retrieved 2010-07-29.
- ^ "Haplogroup R*". Ianlogan.co.uk. Retrieved 2010-07-29.
외부 링크
| 위키미디어 커먼즈에는 해플로그룹 N(mtDNA)과 관련된 미디어가 있다. |
- 하플로그룹N
- Mannis van Oven's – mtDNA 하위 트리 N
- 내셔널 지오그래픽에서 해플로그룹 N의 확산
- Hudjashov, G.; Kivisild, T.; Underhill, P. A.; Endicott, P.; Sanchez, J. J.; Lin, A. A.; Shen, P.; Oefner, P.; Renfrew, C.; Villems, R.; Forster, P. (2007). "Revealing the prehistoric settlement of Australia by Y chromosome and mtDNA analysis". Proceedings of the National Academy of Sciences. 104 (21): 8726–30. Bibcode:2007PNAS..104.8726H. doi:10.1073/pnas.0702928104. PMC 1885570. PMID 17496137.
- 캐서린 보르헤스의 Happlogroup N mtDNA 가족나무 DNA 연구
- 일반
- 이언 로건의 미토콘드리아 DNA 사이트