복사 번호 변동
Copy number variation
CNV(Copy Number Variation)는 게놈의 단면이 반복되고 게놈의 반복 횟수가 [1]개인마다 달라지는 현상이다.복사 번호 변동은 구조 변동의 한 유형입니다. 구체적으로는 상당수의 베이스 쌍에 [2]영향을 주는 복제 또는 삭제 이벤트 유형입니다.전체 인간 게놈의 약 3분의 2가 반복으로[3] 구성될 수 있으며 인간 게놈의 4.8-9.5%는 복사 번호 [4]변이로 분류될 수 있다.포유류에서, 복제 번호의 변화는 질병 [1]표현형뿐만 아니라 개체군에 필요한 변화를 발생시키는 데 중요한 역할을 한다.
복사 번호 변동은 일반적으로 짧은 반복실험과 긴 반복실험의 두 가지 주요 그룹으로 분류할 수 있습니다.그러나 두 그룹 사이에는 명확한 경계가 없으며 분류는 관심장소의 특성에 따라 달라진다.짧은 반복은 주로 디뉴클레오티드 반복(예: A-C-A-C...와 같은 두 개의 반복 뉴클레오티드 반복)과 트리뉴클레오티드 반복을 포함한다.긴 반복은 전체 유전자의 반복을 포함한다.크기는 [5]반복을 일으킬 가능성이 가장 높은 메커니즘의 유형을 조사하는 데 중요한 요소이기 때문에 반복의 크기에 기초한 분류는 가장 명백한 분류 유형이다. 따라서 이러한 반복이 표현형에 미칠 수 있는 영향을 조사한다.
유형 및 염색체 재배열
짧은 복사 번호 변동의 가장 잘 알려진 예 중 하나는 헌팅턴병의 [6]원인이 되는 헌팅틴 유전자의 CAG 염기쌍의 트리뉴클레오티드 반복이다.이 특별한 경우에, 일단 CAG 트리뉴클레오티드가 트리뉴클레오티드의 반복 팽창으로 36회 이상 반복되면, 헌팅턴병은 개인에서 발병하고 그의 혹은 그녀의 [6]자손에게 유전될 가능성이 있다.CAG 트리뉴클레오티드의 반복 횟수는 헌팅턴병의 [7]발병 연령과 관련이 있다.이러한 유형의 짧은 반복은 종종 중합효소 미끄러짐, 템플릿 전환 및 나중에 자세히 설명될 포크 전환을 포함한 복제 중합효소 활성 오류에 기인하는 것으로 생각됩니다.이러한 복사 번호의 짧은 반복 크기는 중합효소에 의해 반복 영역이 잘못 인식되기 쉽고 복제된 영역이 다시 복제될 [8]수 있기 때문에 중합효소에 오류가 발생할 수 있습니다.또한 이들 트리뉴클레오티드 반복이 유전자의 부호화 부분에서 동일한 판독범위 내에 있으면 동일한 아미노산의 긴 사슬을 초래하여 세포 [7]내에 단백질 응집체를 형성할 수 있으며, 이러한 짧은 반복이 유전자의 비부호화 부분에 들어가면 유전자 발현 및 조절에 영향을 미칠 수 있다.반면에, 유전자 전체의 다양한 반복 횟수는 게놈에서 덜 일반적으로 확인된다.전체 유전자 반복의 한 예는 다른 [9]식단을 가진 다른 모집단 간에 유의한 복사수 변화를 갖는 알파 아밀라아제를 코드하는 알파 아밀라아제 1 유전자(AMY1)이다.비록 AMY1 유전자가 복제 수를 증가시키거나 감소시키는 특정한 메커니즘이 여전히 논쟁의 주제이지만, 일부 가설은 비호몰로지 말단 결합 또는 마이크로호몰로지 매개 말단 결합이 이러한 전체 유전자 [9]반복의 원인이 될 수 있음을 시사한다.전체 유전자의 반복은 특정 유전자의 발현에 즉각적인 영향을 미치며, AMI1 유전자의 복제수 변화가 식이요법과 관련이 있다는 사실은 최근의 인간 진화적 [9]적응의 주목할 만한 예이다.복사 번호 변동이 그룹으로 분류되는 일반적인 그룹이지만 복사 번호 변동이 영향을 미치는 정확한 기본 쌍 수는 특정 관심 위치에 따라 달라집니다.현재 보고된 모든 복사 번호 변형 데이터를 사용하여 복사 번호 변형 평균 크기는 약 118kb이고 중앙값은 약 18kb입니다.[10]
카피 번호의 변동의 구조 아키텍처에 관해서, 연구 결과에 의하면, 카피 번호의 변동이 4배 더 [2]풍부한 핫 스팟 영역을 게놈내에서 제안해 정의했습니다.이러한 핫스팟 영역은 분할 복제로 알려진 90-100% 유사한 긴 반복을 포함하는 영역으로 정의되었으며, 가장 중요한 것은 이러한 핫스팟 영역이 염색체 [2]재배열 속도가 증가한다는 점이다.이러한 대규모 염색체 배열은 정상적인 변이와 복사 번호 [1]변이를 포함한 유전적 질병을 발생시킨다고 생각되었다.게다가 이러한 복사 번호 변동 핫스팟은 다른 대륙의 많은 모집단에 걸쳐 일관성이 있으며, 이는 이러한 핫스팟이 모든 모집단에 의해 독립적으로 획득되어 세대를 거쳐 전해졌거나 모집단이 분열되기 전에 초기 인류 진화에 의해 획득되었음을 [1]의미한다.마지막으로, 복사 번호 변화가 가장 조밀하게 분포하는 위치의 공간적 편견이 [1]게놈에서 발생하지 않는 것으로 보인다.원래 형광 현장 교배와 마이크로 위성 분석에 의해 복사 번호 반복이 텔로미어, 동원체, 헤테로크로마틴 [11]등 반복성이 높은 영역에 국한된다는 것이 검출되었지만,[2] 최근의 게놈 전체 연구는 그렇지 않다는 결론을 내렸다.즉, 아색소 영역과 근색소 영역은 대부분의 염색체 재배치 핫스팟이 발견되는 영역이며, 해당 [2]영역에서의 복사수 변화는 현저하게 증가하지 않는다.게다가 이러한 염색체 재배치 핫스팟의 영역은, 다시 유전자수가 감소하지 않고, 카피 번호의 [2]변이의 게놈 위치의 공간적 편향이 최소한인 것을 시사한다.
검출 및 식별
처음에는 세포유전학적 [12]관찰을 통해 복제번호의 변화가 게놈의 극히 작고 무시할 수 있는 부분을 차지하는 것으로 생각되었다.복사 번호의 변동은 일반적으로 작은 탠덤 반복 또는 특정 유전자 [13]장애에만 관련되었으므로, 복사 번호의 변동은 처음에는 특정 궤적의 관점에서만 조사되었다.그러나, 기술의 발달에 의해, 카피 번호의 변형을 특정해 연구하는 고정밀 방법이 많아졌습니다.복사 번호의 변화는 원래 [12]염색체의 물리적 구조를 관찰할 수 있는 기술인 세포유전학 기술에 의해 연구되었다.이러한 기술 중 하나는 결합을 [10]위해 게놈에 높은 상보성을 필요로 하는 형광 탐침을 삽입하는 것을 포함하는 형광 현장 교배(FISH)이다.비교 유전체 교배는 또한 일반적으로 형광체를 시각화하고 [10]염색체의 길이를 비교함으로써 복사 번호의 변화를 검출하기 위해 사용되었다.이러한 초기 기술의 한 가지 큰 결점은 게놈 분해능이 상대적으로 낮고 전체 유전자 반복과 같은 큰 반복만 검출될 수 있다는 것이다.
최근 게놈 기술의 진보는 게놈 분해능이 매우 높은 많은 중요한 방법을 낳았고, 그 결과 게놈의 복사 번호 변이가 증가하고 있는 것으로 [10]보고되고 있다.처음에 이러한 진보는 [14]유전자 전체에 걸쳐 약 1메가베이스의 간격을 갖는 세균성 인공염색체(BAC) 어레이를 사용하는 것을 포함하며, BAC는 또한 재배열 핫스팟에서 복사 번호 변화를 검출할 수 있어 119개의 새로운 복사 번호 [2]변화를 검출할 수 있다.높은 처리량 게놈 염기서열 분석은 인간 유전체학 분야에 혁명을 일으켰고,[2] 실리콘 연구는 게놈의 복사 번호 변화를 검출하기 위해 수행되었습니다.참조 시퀀스는 fosmid 클론을 40kb로 [15]엄격하게 제어함으로써 fosmid를 사용한 다른 관심 시퀀스와 비교되었습니다.시퀀싱 종료 판독은 참조 시퀀스를 관심 시퀀스에 맞추기 위한 충분한 정보를 제공할 수 있으며, 오정렬은 쉽게 눈에 띄기 때문에 [15]클론의 해당 영역 내에서 복사 번호의 변동으로 결론지어집니다.이러한 유형의 검출 기술은 게놈에서 높은 게놈 분해능과 반복의 정확한 위치를 제공하며,[10] 또한 반전 같은 다른 유형의 구조적 변화를 검출할 수 있다.
또, 카피 번호의 변동을 검출하는 다른 방법은, 단일 뉴클레오티드 다형(SNP)[10]을 사용하는 것입니다.인간 SNP 데이터가 풍부하기 때문에, 이러한 SNP를 [16]이용하기 위해서 카피 번호의 변동을 검출하는 방향이 바뀌었습니다.인간 재조합이 비교적 드물고 재조합 핫스팟으로 알려진 게놈의 특정 영역에서 많은 재조합 사건이 발생한다는 사실에 의존하여 결합불평형을 사용하여 복사번호의 [16]변화를 식별할 수 있다.연결불균형을 분석함으로써 특정 하플로타입 SNP와 카피번호의 차이를 관련짓기 위한 노력이 이루어지고 있으며, 이러한 연관성을 이용하여 SNP를 마커로 하여 게놈 내의 카피번호의 변화를 인식할 수 있다.최근에는 읽기 시퀀싱과 읽기 시퀀싱을 포함한 차세대 시퀀싱 기술이 점점 더 많이 사용되고 있으며, 복사 [17][18]번호의 변동을 검출하는 어레이 기반 기술을 대체하기 시작했습니다.배열 기반 기법과 달리 시퀀스 기반 검출 방법은 반전 및 이동과 같은 다른 종류의 구조적 변동을 쉽게 식별한다.
분자 메커니즘
카피 번호의 변동 형성을 위한 분자 메커니즘에는, 상동 베이스와 비상동 베이스의 [5]2개의 주요한 타입이 있습니다.많은 제안이 제기되었지만, 이러한 이론의 대부분은 추측과 추측이다.특정 복사 번호의 변동을 특정 메커니즘과 연관짓는 결정적인 증거는 없습니다.
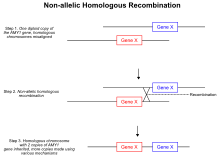
복사 번호의 변화뿐만 아니라 삭제 및 반전을 초래하는 가장 잘 알려진 이론 중 하나는 비알레르 상동 [19]재조합입니다.감수생물 재조합 동안, 상동 염색체들은 짝을 이루어 홀리데이 접합으로 이어지는 두 개의 끝 이중 가닥 단절을 형성한다.그러나 홀리데이 접합 형성 중 이상 메커니즘에서는 이중가닥 절단이 잘못 정렬되어 교차점이 동일한 염색체 상에 비알렐 위치에 착지한다.홀리데이 접합이 해결되면, 불균등한 교차이벤트는 두 개의 상동 염색체 사이의 유전 물질의 이동을 허용하고, 그 결과, 양쪽 상동 염색체의 DNA의 일부가 [19]반복된다.반복된 영역은 더 이상 독립적으로 분리되지 않기 때문에 염색체의 중복된 영역은 유전된다.카피 번호의 변동을 일으킬 수 있는 상동 재조합 베이스의 메카니즘의 또 다른 타입은, 브레이크 유도 [20]복제라고 알려져 있습니다.게놈에서 예기치 않게 이중가닥 파단이 발생하면 세포는 파열의 [20]복구를 중재하는 경로를 활성화한다.비알레르기 상동 재조합과 유사한 파손 복구 오류는 게놈의 특정 영역의 복사 수를 증가시킬 수 있습니다.이중가닥 절단을 수리하는 동안, 절단된 끝은 원래의 [20]가닥에 다시 합류하는 대신 그것의 상동 염색체를 침범할 수 있다.비알레르기 상동재조합기구와 마찬가지로 특정 영역의 여분의 복사가 다른 염색체에 전달되어 복제 이벤트를 일으킨다.또한 코게틴 단백질은 염색체 [21]간 침입을 방지하는 양끝을 근접하게 클램핑함으로써 이중가닥절단 복구시스템에 도움이 되는 것으로 밝혀졌다.리보솜 RNA의 활성화와 같은 어떤 이유로든 응집 활성에 영향을 받는 경우 이중 가닥 절단 복구 [21]오류의 국소적 증가가 있을 수 있다.
카피 번호의 변동으로 이어지는 것으로 가정되는 다른 종류의 가능한 메커니즘은 비호몰로지 기반입니다.이 메커니즘과 상동성 기반 메커니즘을 구별하려면 상동성의 개념을 이해해야 합니다.서로 매우 유사한(~97%) DNA 가닥을 사용하는 염색체의 상동성 쌍은 짧지만 매우 유사한 [5]쌍을 피하기 위해 특정 길이보다 길어야 한다.반면, 비상동성 쌍은 두 가닥 사이의 유사성의 소수의 염기 쌍에만 의존하므로, 비상동성 기반 이중 가닥 [5]수복 과정에서 유전자 물질이 교환되거나 복제될 수 있다.
비호몰로지 기반 메커니즘의 1가지 유형은 비호몰로지 엔드 결합 [22]또는 마이크로호몰로지 엔드 결합 메커니즘입니다.이러한 메커니즘은 이중 가닥 절단 수리에도 관여하지만 호몰로지나 제한된 마이크로 [5]호몰로지가 필요하지 않습니다.이러한 가닥을 수리할 때 종종 수리된 가닥에 작은 결손이나 삽입이 추가됩니다.이 복구 시스템을 [22]통해 역트랜스포존이 게놈에 삽입될 수 있다.염색체상의 비알렐 위치에 역트랜스포존을 삽입하면 감수생물 재조합은 삽입을 같은 영역의 기존 복사본과 동일한 가닥으로 재결합하도록 구동할 수 있다.또 다른 메커니즘은 이중 [23]가닥 파단으로 인해 텔로미어 영역을 잃은 자매 염색체와 관련된 브레이크-퓨전-브릿지 사이클입니다.이 자매 염색체들은 하나의 이심 염색체를 형성하기 위해 함께 융합하고, 그리고 나서 두 [23]개의 다른 핵으로 분리될 것이라고 제안됩니다.이심 염색체를 분리하면 이중 가닥이 끊어지기 때문에 끝부분은 다른 이중 가닥이 끊어지고 그 주기를 [23]반복할 수 있다.두 자매 염색체의 융합은 역전 복제를 일으킬 수 있으며, 이러한 현상이 주기 내내 반복되면 반전 영역이 반복되어 복사 [23]수가 증가하게 됩니다.복사 번호의 변화를 초래할 수 있는 마지막 메커니즘은 중합효소 미끄러짐이며, 이는 템플릿 [24]전환이라고도 합니다.정상적인 DNA 복제 중에 복제 영역을 지속적으로 [24]언클램핑 및 재클램핑하려면 지연된 가닥의 중합효소가 필요합니다.DNA 염기서열에서 작은 규모의 반복이 이미 존재하는 경우, 중합효소는 복제를 계속하기 위해 재클램핑할 때 '혼란'될 수 있으며 올바른 염기쌍으로 클램핑하는 대신 몇 개의 염기쌍을 이동시키고 반복된 영역의 일부를 [24]다시 복제할 수 있습니다.이것은 실험적으로 관찰되어 널리 받아들여지고 있는 메커니즘이지만, 이 오류를 초래한 분자 상호작용은 알려지지 않은 채로 남아 있습니다.또한 이러한 유형의 메커니즘은 중합효소가 DNA 가닥 주위를 점프하는 것을 필요로 하며, 중합효소가 몇 킬로베이스 떨어진 다른 궤적에서 재클램핑될 가능성이 낮기 때문에 디뉴클레오티드 또는 트리뉴클레오티드 [25]반복과 같은 짧은 반복에 더 적용할 수 있다.
알파아밀라아제유전자
아밀라아제는 전분을 단당류로 분해하는 타액 중의 효소이며, 한 가지 유형의 아밀라아제는 알파 아밀라아제 유전자(AMY1)[9]에 의해 암호화된다.아밀라아제 효소뿐만 아니라 AMY1 궤적은 인간 게놈에서 가장 광범위하게 연구되고 배열된 유전자 중 하나이다.그것의 상동성은 다른 영장류에서도 발견되며, 따라서 영장류 AMY1 유전자는 인간 AMY1 유전자의 조상이고 영장류 [9]진화 초기에 적응되었을 가능성이 있다.AMY1은 다양한 인간 [9]집단에 걸쳐 다양한 수의 복사를 가진 가장 잘 연구된 유전자 중 하나이다.AMY1 유전자는 또한 단백질 기능과 복제 [9]번호를 연관시키는 설득력 있는 증거를 보여준 몇 안 되는 유전자 중 하나이다.복제 번호는 특정 유전자의 번역 수준뿐만 아니라 전사를 변화시키는 것으로 알려져 있지만, 연구는 단백질 수치와 복제 번호 사이의 관계가 [26]가변적이라는 것을 보여주었다.유럽계 미국인의 AMY1 유전자에서 타액 아밀라아제 농도는 AMY1 [9]유전자의 복사 번호와 밀접한 관련이 있는 것으로 밝혀졌다.그 결과, AMY1 유전자의 카피 번호가 [9]전분을 소화시키는 단백질 기능과 밀접하게 관련되어 있다는 가설을 세웠다.
AMY1 유전자 복제 번호는 다른 [9]집단의 식단에서 다른 수준의 녹말과 관련이 있는 것으로 밝혀졌다.다른 대륙의 8개 집단은 고분자 식단과 저분자 식단으로 분류되었으며, 이들의 AMY1 유전자 복사 번호는 고해상도 FISH와 [9]qPCR을 사용하여 시각화되었다.일본인, 하다, 유럽계 미국인 인구로 구성된 고분 섭취 인구에서 비아카,[9] 음부티, 데이토그, 야쿠트 인구를 포함한 저분 섭취 인구보다 평균 AMY1 복사 수가 유의미하게 높은 것으로 나타났다.규칙적인 식단의 녹말 수준인 AMY1의 기질이 AMY1 [9]유전자의 복제 수에 직접적인 영향을 미칠 수 있다는 가설이 있었다.AMY1의 복사 수는 타액 [9]아밀라아제와 직접적인 상관관계가 있다는 결론이 나왔기 때문에, 인구의 일상 식단에 존재하는 전분이 많을수록, AMY1 유전자의 여러 복사가 있는 것이 진화적으로 더 유리하다.AMI1 유전자는 분자 유전학 수준에서 [26]진화에 대한 강력한 증거를 제공한 최초의 유전자였다.또, 비교 게놈 하이브리드화를 이용해, 일본 인구의 전체 게놈의 카피 번호의 변화를 야쿠트 [9]인구의 카피 번호와 비교했다.AMY1 유전자의 복사 번호 변동은 게놈의 다른 유전자 또는 영역의 복사 번호 변동과 유의하게 다른 것으로 밝혀졌으며, 이는 AMY1 유전자가 다른 복사 번호 [9]변동에 거의 또는 전혀 영향을 미치지 않는 강한 선택적 압력을 받고 있음을 시사한다.마지막으로, 두 모집단 사이의 783개의 마이크로 위성 길이의 변동성은 AMI1 유전자의 복사 번호 변동성과 비교되었다.조사된 마이크로 위성 중 97% 이상에서 [9]AMI1 유전자 복사 수 범위가 더 큰 것으로 밝혀졌다.이것은 자연 선택이 이 두 [9]집단에서 평균적인 수의 AMY1 유전자를 형성하는데 상당한 역할을 했다는 것을 암시한다.그러나 6개의 모집단만이 연구되었기 때문에, 그들의 식단이나 배양에 녹말 이외의 다른 요소들이 있을 가능성을 고려하는 것이 중요하다.
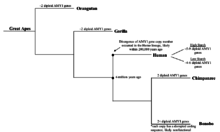
AMY1 유전자 복제 수가 언제부터 증가하기 시작했는지는 불분명하지만, 초기 영장류에 AMY1 유전자가 존재했다는 것이 알려져 확인되었습니다.인간과 가장 가까운 진화적 친척인 침팬지들은 인간 AMY1 유전자와 길이가 같은 두 개의 이배체 유전자를 가지고 있는 것으로 밝혀졌는데, 이는 인간 AMY1 [9]유전자에 비해 상당히 적은 것이다.반면에, 현생인류의 가까운 친척인 보노보스는 AMI1 [9]유전자의 두 개 이상의 이배체를 가지고 있는 것으로 밝혀졌다.그럼에도 불구하고, 보노보 AMY1 유전자의 염기서열 분석 및 염기서열 분석 결과, AMY1 유전자의 염기서열이 교란되어 기능성 침샘아밀라아제 [9]생성으로 이어질 수 있다.보노보 AMY1 카피수의 증가는 그들의 식단에서 녹말의 양과 관련이 없을 가능성이 높다는 것을 그 결과로부터 추론할 수 있다.복제수 증가는 초기 인류의 진화 과정에서 시작되었는데, 어떤 유인원도 기능성 [9]단백질을 생성하는 AMY1 유전자의 복제수 이상을 가지고 있지 않았기 때문이다.게다가, AMI1 카피수의 증가는 인간이 수렵 채집 생활에서 농업 사회로 옮겨갔을 때 약 20,000년 전에 시작되었고, 이것은 또한 인간이 [9]녹말이 많이 함유된 뿌리 채소에 많이 의존했을 때라고 추측되었다.이 가설은 논리적이긴 하지만 인간의 식생활, 특히 녹말 함량이 높은 뿌리 채소에 대한 정보를 수집하기 어렵기 때문에 실험적인 증거가 부족하다.최근 DNA 염기서열 분석의 획기적인 발전으로 연구원들은 네안데르탈인과 같은 오래된 DNA의 염기서열을 어느 정도 정확하게 배열할 수 있게 되었다.아마도 네안데르탈인의 DNA 염기서열을 분석하면 언제 AMI1 유전자 복제 수가 증가했는지에 대한 시간표시를 제공할 수 있고 인간의 식단과 유전자 진화에 대한 통찰력을 제공할 수 있을 것이다.
현재 어떤 메커니즘이 아밀라아제 유전자의 초기 복제를 발생시켰는지는 알려지지 않았으며, 레트로바이러스 배열의 삽입은 AMY1 [27]유전자의 복제를 야기한 비호몰로지 말단 결합에 의한 것임을 암시할 수 있다.하지만, 현재 이 이론을 뒷받침할 증거가 없기 때문에 이 가설은 추측으로 남아 있다.다중복사형 AMY1 유전자의 최근 기원은 환경에 따라 AMY1 유전자 복제 [26]수가 환경과 직접적으로 상호작용하지 않는 유전자에 비해 매우 빠르게 증가하고 감소할 수 있다는 것을 암시한다.AMI1 유전자는 유전자 복용량이 주어진 환경에서 유기체의 생존에 어떻게 영향을 미치는지 보여주는 훌륭한 예이다.AMI1 유전자의 다중 복제는 고분자 식단에 더 많이 의존하는 사람들에게 진화적인 이점을 준다. 따라서 높은 유전자 복제 수치는 [26]모집단에서 지속된다.
뇌세포
인간의 뇌 신경 세포 중, 신체적으로 파생된 복사 번호의 변이가 [28]빈번하다.복사 번호의 변화는 광범위한 변동성을 보여준다(다른 연구에서 뇌 뉴런의 9-100%).대부분의 변경은 2~10Mb 사이이며 삭제는 [28]증폭보다 훨씬 많습니다.복사 번호의 변화는 뇌세포에서 다른 세포 [28]유형보다 더 높은 것으로 보입니다.카피 번호의 변동의 원인으로서는, DNA 손상의 잘못된 수복을 생각할 수 있습니다.
유전자 복제와 유전자의 삼중 복사는 [29]점 돌연변이보다 더 흔하지만 파킨슨병의 드문 원인인 것으로 보인다.
RCL1 유전자의 복사 번호 변이는 [30]아동의 다양한 신경정신학적 표현형과 관련되어 있다.
유전자 패밀리 및 자연 선택
최근 복제 번호의 변이를 유전자 패밀리와 연관짓는 논의가 있었다.유전자 패밀리는 비슷한 기능을 하지만 작은 시간적 또는 공간적 차이를 가지고 있는 일련의 연관된 유전자로 정의되며 이러한 유전자들은 하나의 조상 [26]유전자에서 유래한 것으로 보인다.복제 번호의 변화가 유전자 패밀리와 연결되는 주된 이유는 가족 내 유전자가 다른 [26]복제로 복제되는 하나의 조상 유전자로부터 파생되었을 가능성이 있기 때문이다.돌연변이는 시간이 지남에 따라 축적되고 자연선택이 유전자에 작용함에 따라 일부 돌연변이는 유전자가 유전될 수 있는 환경적 이점을 가져오고 결국 명확한 유전자 패밀리가 분리된다.카피 번호의 변이에 의해 생성되었을 가능성이 있는 유전자 패밀리의 예는 글로빈 유전자 패밀리다.글로빈 유전자 패밀리는 알파와 베타 글로빈 유전자로 구성된 정교한 유전자 네트워크로,[31] 유사 유전자뿐만 아니라 배아와 성인 모두에서 발현되는 유전자를 포함합니다.글로빈 계열의 이러한 글로빈 유전자는 모두 잘 보존되어 있고 유전자의 작은 부분만 다를 뿐이며, 아마도 초기 글로빈 [31]유전자의 복제로 인해 공통 조상 유전자에서 파생되었음을 나타낸다.
연구는 복사 번호의 변화가 기본적인 세포 [32]활동에 관여하는 단백질보다 환경과 직접적으로 상호작용하는 단백질을 코드하는 유전자에서 훨씬 더 흔하다는 것을 보여주었다.카피 번호 변화에 수반하는 유전자 용량 효과는 필수 세포 기능이 파괴될 경우 유해한 영향을 초래할 수 있으므로 세포 경로에 관여하는 단백질은 강력한 정제 [32]선택을 받게 된다.또한 단백질은 함께 기능하고 다른 경로의 단백질과 상호작용하기 때문에 개별 단백질보다는 생물 분자 경로에 대한 자연 선택의 영향을 보는 것이 중요하다.앞에서 설명한 바와 같이 경로의 주변 단백질은 복사 번호 변동으로 농축되는 반면 경로의 중앙 단백질은 복사 번호 [33]변동으로 고갈된다는 것이 확인되었다.경로의 말초 단백질은 더 적은 단백질과 상호작용하기 때문에 복사 번호의 변화에 의해 영향을 받는 단백질 용량 변화는 세포 [33]경로의 전체 결과에 더 작은 영향을 미칠 수 있다고 설명되었다.
지난 몇 년 동안 연구자들은 카피 번호의 변이를 검출, 특정 및 시퀀싱하는 것에서 인간 게놈과 자연에서 카피 번호의 변이의 역할에 대한 심층 분석으로 초점을 옮긴 것으로 보입니다.이러한 관계와 변화를 형성하는 데 자연 선택이 수행하는 역할뿐만 아니라 복사 번호의 변동과 유전자 패밀리 사이의 관계를 더욱 검증하기 위해 증거가 필요하다.게다가, 연구원들은 또한 복사 번호의 변화에 관련된 분자 메커니즘을 밝히는 것을 목표로 하고 있다. 왜냐하면 그것은 일반적으로 구조적인 변화에 관한 중요한 정보를 드러낼 수 있기 때문이다.한 걸음 물러서면 인간 게놈의 구조적 변이 영역은 빠르게 성장하고 있는 연구 주제인 것 같다.이러한 연구 데이터는 진화와 자연 도태에 대한 추가 증거를 제공할 수 있을 뿐만 아니라 광범위한 유전 질환에 대한 치료법을 개발하는 데도 사용될 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c d e McCarroll SA, Altshuler DM (July 2007). "Copy-number variation and association studies of human disease". Nature Genetics. 39 (7 Suppl): S37-42. doi:10.1038/ng2080. PMID 17597780. S2CID 8521333.
- ^ a b c d e f g h Sharp AJ, Locke DP, McGrath SD, Cheng Z, Bailey JA, Vallente RU, et al. (July 2005). "Segmental duplications and copy-number variation in the human genome". American Journal of Human Genetics. 77 (1): 78–88. doi:10.1086/431652. PMC 1226196. PMID 15918152.
- ^ de Koning AP, Gu W, Castoe TA, Batzer MA, Pollock DD (December 2011). "Repetitive elements may comprise over two-thirds of the human genome". PLOS Genetics. 7 (12): e1002384. doi:10.1371/journal.pgen.1002384. PMC 3228813. PMID 22144907.
- ^ Zarrei M, MacDonald JR, Merico D, Scherer SW (March 2015). "A copy number variation map of the human genome". Nature Reviews. Genetics. 16 (3): 172–83. doi:10.1038/nrg3871. hdl:2027.42/146425. PMID 25645873. S2CID 19697843.
- ^ a b c d e Hastings PJ, Lupski JR, Rosenberg SM, Ira G (August 2009). "Mechanisms of change in gene copy number". Nature Reviews. Genetics. 10 (8): 551–64. doi:10.1038/nrg2593. PMC 2864001. PMID 19597530.
- ^ a b "A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington's disease chromosomes. The Huntington's Disease Collaborative Research Group" (PDF). Cell. 72 (6): 971–83. March 1993. doi:10.1016/0092-8674(93)90585-e. hdl:2027.42/30901. PMID 8458085. S2CID 802885.
- ^ a b Myers RH (April 2004). "Huntington's disease genetics". NeuroRx. 1 (2): 255–62. doi:10.1602/neurorx.1.2.255. PMC 534940. PMID 15717026.
- ^ Albertini AM, Hofer M, Calos MP, Miller JH (June 1982). "On the formation of spontaneous deletions: the importance of short sequence homologies in the generation of large deletions". Cell. 29 (2): 319–28. doi:10.1016/0092-8674(82)90148-9. PMID 6288254. S2CID 36657944.
- ^ a b c d e f g h i j k l m n o p q r s t u v w Perry GH, Dominy NJ, Claw KG, Lee AS, Fiegler H, Redon R, et al. (October 2007). "Diet and the evolution of human amylase gene copy number variation". Nature Genetics. 39 (10): 1256–60. doi:10.1038/ng2123. PMC 2377015. PMID 17828263.
- ^ a b c d e f Freeman JL, Perry GH, Feuk L, Redon R, McCarroll SA, Altshuler DM, et al. (August 2006). "Copy number variation: new insights in genome diversity". Genome Research. 16 (8): 949–61. doi:10.1101/gr.3677206. PMID 16809666.
- ^ Bailey JA, Gu Z, Clark RA, Reinert K, Samonte RV, Schwartz S, et al. (August 2002). "Recent segmental duplications in the human genome". Science. 297 (5583): 1003–7. Bibcode:2002Sci...297.1003B. doi:10.1126/science.1072047. PMID 12169732. S2CID 16501865.
- ^ a b Jacobs PA, Browne C, Gregson N, Joyce C, White H (February 1992). "Estimates of the frequency of chromosome abnormalities detectable in unselected newborns using moderate levels of banding". Journal of Medical Genetics. 29 (2): 103–8. doi:10.1136/jmg.29.2.103. PMC 1015848. PMID 1613759.
- ^ Inoue K, Lupski JR (2002). "Molecular mechanisms for genomic disorders". Annual Review of Genomics and Human Genetics. 3: 199–242. doi:10.1146/annurev.genom.3.032802.120023. PMID 12142364.
- ^ Iafrate AJ, Feuk L, Rivera MN, Listewnik ML, Donahoe PK, Qi Y, et al. (September 2004). "Detection of large-scale variation in the human genome". Nature Genetics. 36 (9): 949–51. doi:10.1038/ng1416. PMID 15286789.
- ^ a b Tuzun E, Sharp AJ, Bailey JA, Kaul R, Morrison VA, Pertz LM, et al. (July 2005). "Fine-scale structural variation of the human genome". Nature Genetics. 37 (7): 727–32. doi:10.1038/ng1562. PMID 15895083. S2CID 14162962.
- ^ a b Conrad B, Antonarakis SE (2007). "Gene duplication: a drive for phenotypic diversity and cause of human disease". Annual Review of Genomics and Human Genetics. 8: 17–35. doi:10.1146/annurev.genom.8.021307.110233. PMID 17386002.
- ^ Alkan C, Coe BP, Eichler EE (May 2011). "Genome structural variation discovery and genotyping". Nature Reviews. Genetics. 12 (5): 363–76. doi:10.1038/nrg2958. PMC 4108431. PMID 21358748.
- ^ Sudmant PH, Rausch T, Gardner EJ, Handsaker RE, Abyzov A, Huddleston J, et al. (October 2015). "An integrated map of structural variation in 2,504 human genomes". Nature. 526 (7571): 75–81. Bibcode:2015Natur.526...75.. doi:10.1038/nature15394. PMC 4617611. PMID 26432246.
- ^ a b Pâques F, Haber JE (June 1999). "Multiple pathways of recombination induced by double-strand breaks in Saccharomyces cerevisiae". Microbiology and Molecular Biology Reviews. 63 (2): 349–404. doi:10.1128/MMBR.63.2.349-404.1999. PMC 98970. PMID 10357855.
- ^ a b c Bauters M, Van Esch H, Friez MJ, Boespflug-Tanguy O, Zenker M, Vianna-Morgante AM, et al. (June 2008). "Nonrecurrent MECP2 duplications mediated by genomic architecture-driven DNA breaks and break-induced replication repair". Genome Research. 18 (6): 847–58. doi:10.1101/gr.075903.107. PMC 2413152. PMID 18385275.
- ^ a b Kobayashi T, Ganley AR (September 2005). "Recombination regulation by transcription-induced cohesin dissociation in rDNA repeats". Science. 309 (5740): 1581–4. Bibcode:2005Sci...309.1581K. doi:10.1126/science.1116102. PMID 16141077. S2CID 21547462.
- ^ a b Lieber MR (January 2008). "The mechanism of human nonhomologous DNA end joining". The Journal of Biological Chemistry. 283 (1): 1–5. doi:10.1074/jbc.R700039200. PMID 17999957.
- ^ a b c d McCLINTOCK B (1951). "Chromosome organization and genic expression". Cold Spring Harbor Symposia on Quantitative Biology. 16: 13–47. doi:10.1101/sqb.1951.016.01.004. PMID 14942727.
- ^ a b c Smith CE, Llorente B, Symington LS (May 2007). "Template switching during break-induced replication". Nature. 447 (7140): 102–5. Bibcode:2007Natur.447..102S. doi:10.1038/nature05723. PMID 17410126. S2CID 7427921.
- ^ Bi X, Liu LF (January 1994). "recA-independent and recA-dependent intramolecular plasmid recombination. Differential homology requirement and distance effect". Journal of Molecular Biology. 235 (2): 414–23. doi:10.1006/jmbi.1994.1002. PMID 8289271.
- ^ a b c d e f Korbel JO, Kim PM, Chen X, Urban AE, Weissman S, Snyder M, Gerstein MB (June 2008). "The current excitement about copy-number variation: how it relates to gene duplications and protein families". Current Opinion in Structural Biology. 18 (3): 366–74. doi:10.1016/j.sbi.2008.02.005. PMC 2577873. PMID 18511261.
- ^ Samuelson LC, Wiebauer K, Snow CM, Meisler MH (June 1990). "Retroviral and pseudogene insertion sites reveal the lineage of human salivary and pancreatic amylase genes from a single gene during primate evolution". Molecular and Cellular Biology. 10 (6): 2513–20. doi:10.1128/mcb.10.6.2513. PMC 360608. PMID 1692956.
- ^ a b c Rohrback S, Siddoway B, Liu CS, Chun J (November 2018). "Genomic mosaicism in the developing and adult brain". Developmental Neurobiology. 78 (11): 1026–1048. doi:10.1002/dneu.22626. PMC 6214721. PMID 30027562.
- ^ Singleton AB, Farrer M, Johnson J, Singleton A, Hague S, Kachergus J, et al. (October 2003). "alpha-Synuclein locus triplication causes Parkinson's disease". Science. 302 (5646): 841. doi:10.1126/science.1090278. PMID 14593171. S2CID 85938327.
- ^ Brownstein, CA; Smith, RS; Rodan, LH; Gorman, MP; Hojlo, MA; Garvey, EA; Li, J; Cabral, K; Bowen, JJ; Rao, AS; Genetti, CA; Carroll, D; Deaso, EA; Agrawal, PB; Rosenfeld, JA; Bi, W; Howe, J; Stavropoulos, DJ; Hansen, AW; Hamoda, HM; Pinard, F; Caracansi, A; Walsh, CA; D'Angelo, EJ; Beggs, AH; Zarrei, M; Gibbs, RA; Scherer, SW; Glahn, DC; Gonzalez-Heydrich, J (17 February 2021). "RCL1 copy number variants are associated with a range of neuropsychiatric phenotypes". Molecular Psychiatry. 26 (5): 1706–1718. doi:10.1038/s41380-021-01035-y. PMC 8159744. PMID 33597717.
- ^ a b Goodman M, Koop BF, Czelusniak J, Weiss ML (December 1984). "The eta-globin gene. Its long evolutionary history in the beta-globin gene family of mammals". Journal of Molecular Biology. 180 (4): 803–23. doi:10.1016/0022-2836(84)90258-4. PMID 6527390.
- ^ a b Redon R, Ishikawa S, Fitch KR, Feuk L, Perry GH, Andrews TD, et al. (November 2006). "Global variation in copy number in the human genome". Nature. 444 (7118): 444–54. Bibcode:2006Natur.444..444R. doi:10.1038/nature05329. PMC 2669898. PMID 17122850.
- ^ a b Kim PM, Korbel JO, Gerstein MB (December 2007). "Positive selection at the protein network periphery: evaluation in terms of structural constraints and cellular context". Proceedings of the National Academy of Sciences of the United States of America. 104 (51): 20274–9. Bibcode:2007PNAS..10420274K. doi:10.1073/pnas.0710183104. PMC 2154421. PMID 18077332.
추가 정보
- Pollack JR, Perou CM, Alizadeh AA, Eisen MB, Pergamenschikov A, Williams CF, Jeffrey SS, Botstein D, Brown PO (September 1999). "Genome-wide analysis of DNA copy-number changes using cDNA microarrays". Nature Genetics. 23 (1): 41–6. doi:10.1038/12640. PMID 10471496. S2CID 997032.
- "Huge genetic variation in healthy people". New Scientist. 7 August 2004.
- Carter NP (September 2004). "As normal as normal can be?". Nature Genetics. 36 (9): 931–2. doi:10.1038/ng0904-931. PMID 15340426.
- Check E (October 2005). "Human genome: patchwork people". Nature. 437 (7062): 1084–6. Bibcode:2005Natur.437.1084C. doi:10.1038/4371084a. PMID 16237414. S2CID 8211641.
- "Gene duplications may define who you are". New Scientist. 22 November 2006.
- "DNA varies more widely from person to person, Genetic maps reveal". National Geographic. 22 November 2006.
- "Finding the right lenses" (PDF). Nature Genetics. 1 July 2007.
- Lam HY, Mu XJ, Stütz AM, Tanzer A, Cayting PD, Snyder M, et al. (January 2010). "Nucleotide-resolution analysis of structural variants using BreakSeq and a breakpoint library". Nature Biotechnology. Nature Biotechnoloty. 28 (1): 47–55. doi:10.1038/nbt.1600. PMC 2951730. PMID 20037582.
- "New Research Sheds Light on Autism's Genetic Causes". Singularity Hub. 15 June 2010. Archived from the original on 18 June 2010. Retrieved 15 June 2010.
외부 링크
- 복사 번호 변경 프로젝트, Sanger Institute
- 청구 내용:일란성 쌍둥이는 동일한 DNA를 가지고 있다.
- 인간의 복사 번호 변화를 위한 통합 주석 플랫폼
- 복사 번호 변동에 대한 참고 문헌 목록
- 인간 게놈의 구조적 변종 데이터베이스인 게놈 변종 데이터베이스
- 고밀도 SNP 유전자형을 통한 복사 번호 변동 검출
- 옥스퍼드 진 테크놀로지
- 바이오 디스커버리 Nexus 복사 번호
- 2,026명의 건강한 사용자를 대상으로 한 복사 번호의 고해상도 매핑
- IGSR: 국제 게놈 샘플 자원
- cn.FARS: 오검출률이 낮은 마이크로 어레이 데이터의 복사 번호 변동을 검출하는 잠재 변수 모델, R 패키지: 소프트웨어
- cn.MOPS: 차세대 시퀀싱 데이터의 복사 번호 변동을 검출하기 위한 Poisson 혼합 소프트웨어