TMEM267
TMEM267| TMEM267 | |||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||||||||||||||||||||||
| 에일리어스 | TMEM267, C5orf28, 막투과단백질 267 | ||||||||||||||||||||||||||||||
| 외부 ID | MGI: 3648543 HomoloGene: 49708 GenCards: TMEM267 | ||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
| 위키데이터 | |||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||
TMEM267은 인간의 TMEM267 유전자에 의해 암호화되는 단백질이다.그것은 막 통과 단백질을 코드하는 가능한 종양 유전자이다.TMEM267의 기능은 운반에 관련된 모티브와 영역의 존재가 정형외과에서 보존되었기 때문에 세포에서 분자의 운반을 수반할 가능성이 높다.TMEM267은 많은 종에서 정형어를 가지고 있으며 갑상선에서 가장 높은 수준으로 발현된다.
진
TMEM267 의 기존의 에일리어스에는, C5orf28, B2RDA6, 및 Q9H6Z2 [5]가 포함됩니다.TMEM267은 염기쌍 43,444,252와 43,485,178 사이의 역사슬의 염색체 5, 세포대 p12에서 발견되며, 이는 염기쌍 길이가 40,[6]927임을 의미한다.TMEM267은 13개의 서로 다른 gt-ag 인트론 및 12개의 다른 mRNA를 생성하며, 9개의 교대로 스플라이스된 변형과 3개의 비교차적 형태를 가진다.2개의 대체 프로모터와 7개의 검증된 폴리아데닐화 [7]사이트가 있습니다.6명의 다양한 [8]길이의 예상 프로모터가 있습니다.
| 이름. | 등록 번호 | 엑손 수 | 사이즈(bp) |
|---|---|---|---|
| 트랜스크립트 바리안트 1 | NM_022483 | 3 | 2736 |
| 트랜스크립트 바리안트 2 | NM_001377394.1 | 4 | 2887 |
| 트랜스크립트 바리안트 X2 | XM_011514075 | 4 | 3574 |
| 트랜스크립트 바리안트 3 | NM_001377395.1 | 5 | 3210 |
| 트랜스크립트 바리안트 4 | NM_001377396.1 | 3 | 2960 |
| 트랜스크립트 바리안트 5 | NM_001377397.1 | 4 | 3108 |
| 트랜스크립트 바리안트 6 | NM_001377398.1 | 5 | 3283 |
| 트랜스크립트 바리안트7 | NM_001377399.1 | 3 | 3377 |
| 트랜스크립트 바리안트8 | NM_001377400.1 | 4 | 3528 |
| 트랜스크립트 바리안트9 | NM_001377401.1 | 4 | 2834 |
| 트랜스크립트 바리안트 10 | NM_001377402.1 | 3 | 2809 |
| 트랜스크립트 바리안트 11 | NM_001377403.1 | 5 | 2979 |
단백질
일반 정보
모든 아이소폼의 TMEM267 단백질은 길이가 [9]215개 아미노산이다.모든 아이소폼은 24.2kDa의 예측 분자량과 8.[10][11]91의 이론적인 등전점을 가지고 있다.히스티딘 및 트립토판 잔류물의 평균 백분율이 높았다.아스파라긴, 글루탐산, 티로신의 비율은 평균 미만이었다.남극 Yellowbelly Rockcod의 분석 후, 열대, 그리고 태평양, 트립 토판의 평균%작곡과 아스파라긴과 글루타민산 펩타이드의 평균%구성 아래에 양서류, 어류, 포유류, 새, 파충류를 가로질러 보전했던 것 그 이상의 돌고래 orthologs white-sided 개구리, 윌로우는 딱새, 일반적인 벽 도마뱀을 허우적거렸다.[12]
Isoforms
| 이름. | 등록 번호 | 크기(aa) |
|---|---|---|
| 트랜스크립트 바리안트 2 | NP_001364323.1 | 215 |
| 트랜스크립트 바리안트 X2 | XM_011514075 | 215 |
| 트랜스크립트 바리안트 3 | NP_001364324 | 215 |
| 트랜스크립트 바리안트 4 | NP_001364325 | 215 |
| 트랜스크립트 바리안트 5 | NP_001364326 | 215 |
| 트랜스크립트 바리안트 6 | NP_001364327 | 215 |
| 트랜스크립트 바리안트7 | NP_001364328 | 215 |
| 트랜스크립트 바리안트8 | NP_001364329 | 215 |
| 트랜스크립트 바리안트9 | NP_001364330 | 215 |
| 트랜스크립트 바리안트 10 | NP_001364331 | 215 |
| 트랜스크립트 바리안트 11 | NP_001364332 | 215 |
| 트랜스크립트 바리안트 12 | NP_071928 | 215 |
막 통과 영역
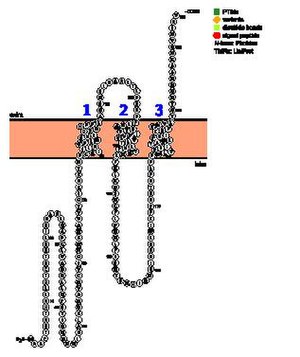
TMEM267의 막 통과 영역을 둘러싼 예측은 명확하지 않다.일부 예측 도구에서는 막 통과 [13]영역이나 소수성 영역이 없다고 주장합니다.다른 사람들은 2에서 5개의 막 통과 [14]도메인을 예측한다.[15] 113-135와 176-198 부근의 아미노산에는 적어도 두 개의 막 통과 영역이 있을 가능성이 높다는 것이 일치된 의견이다.NCBI Gene에 의해 주어진 3개의 막 통과 영역의 헬리컬 휠 다이어그램은 [16]특이한 막 통과 영역에 염기성 및 산성인 극성 아미노산의 존재를 나타낸다.
도메인
TMEM267에는 2개의 도메인이 예측되었습니다.첫 번째는 LaxA 결합, 내막 관련 돌연변이 가수분해효소이며, 이는 TMEM267이 포스포리파아제 역할을 할 수 있는 막 결합 금속의존성 가수분해효소 그룹으로 관여할 수 있음을 나타낸다.다른 하나는 TMEM267이 리소좀으로의 분자 전달 및 세포 [17][18]시그널링에 관여할 수 있음을 나타내는 Vacuolar sorting protein 9(VPS9) 도메인으로 예측된다.
| 도메인 | 위치 | Isoforms에 보존 | 맞춤법 보존 |
|---|---|---|---|
| LaxA 바인딩 사이트 | 55-161 | 네. | 네. |
| 액포분류단백질9(VSP9)결합부위 | 175-199 | 네. | 아니요. |
모티브
예측된 모티브는 표준 아르기닌 함유 포스포펩타이드 모티브로, [19]많은 세포 과정에 관여하는 14-3-3 단백질의 수송에 관여할 수 있다.바이러스 및 미생물 [20]감염의 존재 하에서 IRF-3를 작용시키는 시그널링 경로에 Interferon Regulatory Factor 3(IRF-3) 단백질의 결합 부위를 포함할 수 있다.호몰로지 도메인에 대한 테더링을 가능하게 하는 트립토판 기반 모티브는 TMEM267이 골지로부터 ER로의 수송을 매개하는 역할을 할 수 있다는 것을 의미할 수 있다.코토머 서브유닛 델타(delta-COP)는 모티브에 결합하고 ER 및 [21]골지로부터의 단백질 운반에 관여하는 소포와 관련된 세포질 단백질 복합체이다.LC3-Interacting Region(LIR) 모티브가 예측되었다.이는 TMEM267이 세포의 [22]건전성을 유지하기 위해 리소좀에 대한 오토파고솜의 세포질 물질 전달 및 독성 고분자 및 유기 제거에 관여하는 오토파지 경로에 관여할 수 있음을 나타낸다.Class 2 PDZ 결합 모티브는 TMEM267이 교통, 재활용 및 세포 내 [23]분류에 영향을 미치는 PDZ 단백질과 관련될 수 있다고 예측한다.Wxx[FY] 모티브는 TMEM267이 Pex14와 Pex5 [24][25]단백질의 상호작용에 관여할 수 있음을 나타낸다.
| 모티브 | 위치 | Isoforms에 보존 | 맞춤법 보존 | 셀룰러 위치 |
|---|---|---|---|---|
| 표준 아르기닌 함유 포스포펩티드 모티브 | 49-56 | 네. | 네. | 세포 |
| Interferon Regulatory Factor 3(IRF-3) 결합 부위 | 139-146 | 네. | 네. | 세포 |
| 호몰로지 도메인에 대한 테더링을 가능하게 하는 트립토판 기반 모티브 | 144-151 | 네. | 네. | 세포 |
| LIR 모티브 | 64-68 | 네. | 네. | 세포 |
| PDZ 결합 모티브 | 210-215 | 네. | 네. | 세포 |
| Wxx[FY] 모티브 | 166-170 | 네. | 네. | 세포 |
현지화와 풍부함
전반적으로 TMEM267은 세포질에서 발견될 가능성이 높다.TMEM267 단백질은 세포의 [26]핵체에 국소화되었다고 주장되었다.또 다른 도구는 라인하르트의 세포질/핵 [27]판별법에서 신뢰도 94.1로 세포질(69.6%)과 미토콘드리아(13%)에서 발견될 것으로 예측했다.TMEM267은 0.05ppm으로 [28]인체에 풍부하지 않다.
이차 구조 예측
2차 구조 예측은 NCBI Gene에 의해 주어진 TMEM267의 막 통과 영역을 이용하여 이루어졌다.예측 서버는 아미노산 1-76이 본질적으로 나선형임을 나타냅니다.TMEM267 단백질의 세포외 및 세포내 영역은 알파 나선과 베타 시트의 조합으로 예측되지만, [29][30][31]합의는 이루어지지 않는다.
번역 후 수정
조직에서 [32]발견된 TMEM267 단백질의 번역 후 수정에 대한 증거는 없다.단백질 배열 분석에 따르면 팔미토일화 사이트 1개, SMO 인터랙션 사이트 1개, 섬모일화 사이트 [33][34]2개가 예측된다.AGC, CKII 및 케이스 키나아제 [35][36]II를 포함한 다양한 단백질 키나제를 가진 비막 통과 영역에는 예측된 인산화 부위가 많다.TMEM267의 [37]N 말단에서 1개의 부위가 아세틸화될 것으로 예측된다.TMEM267은 7개의 O-beta-GlycNAc [38][39]사이트뿐만 아니라 4개의 예측 당화 사이트를 가지고 있다.
표현
TMEM267 단백질은 체내 100개 이상의 조직에서 발현되며, 이는 조직의 특이성이 낮지만 갑상선, 뇌하수체, 췌장에 주로 존재한다.NCBI Geo의 데이터에 따르면 주로 갑상선에서 발현 수준이 높지만 다른 조직들은 각 샘플에 대해 다양한 발현을 보인다.평균적으로 갑상선, 난소, 고환, 뇌하수체, 췌장의 발현 수치가 가장 높은 것으로 보입니다.TMEM267은 TMEM267에 [40]비해 RPKM이 거의 3배인 베타 액틴에 비해 매우 높은 수준으로 발현되지 않는다.TMEM267은 [41]5번 염색체 평균 유전자의 1.6배에서 발현된다.
상호 작용
전사 계수
아래 표는 Genomatix 예측 TMEM267 프로모터 [42]영역에서 결합할 것으로 예상되는 일련의 전사인자를 설명한다.
| 문자 변환 계수 | 상세 정보 |
|---|---|
| RU49 | 아연 손가락 증식 1-Zipro |
| 소리 | SOX-SOY-sex/testis 결정 및 관련 HMG 박스 요인 |
| FKHD | 포크헤드 도메인 요인 |
| 브라크 | 중배엽발달인자 완두유전자 |
| EVI1 | EVI1-myleoid변환단백질 |
| VTBP | 척추동물TATA결합단백질인자 |
| 서두르다 | 링핑거 DNA 결합 모티브를 가진 SWI/SNF 관련 누셀로인산단백질 |
| NKXH | NKX 호메오도메인 인자 |
| BRN5 | Brn-5 POU 도메인 |
| PDX1 | 췌장 호메오도메인 TF |
| CEBPA/B | CCAAT/강화제결합단백질 |
| 카아카 | CCAAT 결합 계수 |
| SPZ1 | 테스트 고유의 bHLH-Zip TF |
| 하지 않다 | 활성화된 T세포의 핵인자 |
| DMRT | DM 도메인 포함 TF |
| ZF01 | C2H2 아연 핑거 TF1 |
| 크레브 | cAMP반응성 요소결합단백질 |
| XBBF | X박스 결합 계수 |
| GCNR | 생식세포핵수용체 |
| PLZF | C2H2 아연핑거단백질 PLZF |
상호작용단백질
TMEM267은 아래 표의 단백질과 상호작용할 것으로 예측되었다.
| 단백질명 | UniProt 라벨 | 기능. | 위치 |
|---|---|---|---|
| TMEM52B | Q4KMG9 | 기능이 연구되지 않았습니다. | 세포막의 일체성분, 세포외영역 |
| TMEM14B | Q9NUH8 | 발달하는 신피질의 피질 확장 및 접힘에 관여하며, 핵이전을 통해 신경 전구체의 증식을 촉진할 수 있습니다. | 막의 일체성분 |
| RTP2(수용체수송단백질2) | Q5 QGT7 | 후각 수용체의 기능 세포 표면 발현 촉진 | 플라즈마막 |
| SAR1A | Q9NR31 | ER에서 골지 기기로의 수송에 관여하며, ER 막에서 SEC16A의 SAR1S-GTP 의존적인 조립체는 ER 출구 부위를 정의하는 조직화된 골격을 형성한다. | ER, 골지 장치 |
| STX7(Syntaxin-7) | Q15400 | 혈장막에서 초기 엔도솜으로의 단백질 전달에 관여할 수 있으며, 초기부터 후기 엔도솜 및 리소솜으로의 전달을 매개한다. | 엔도솜, 혈장막 |
| CPLX4(Complexin-4) | Q7Z7G2 | SNARE 단백질 복합체 매개 시냅스 소포 융합 조절 | 플라즈마막 |
| APP(아밀로이드β 전구체 단백질 P4) | P05067 | 뉴런의 세포 표면 수용체로서 기능하며, 세포 이동성 및 전사 조절에 관여한다. | 세포외 영역/분비물, 플라즈마막, 엔도솜, 핵, 세포질 소포 |
| EGFR | P00533 | 리간드 결합은 핵심 세포질 잔류물에 대한 호모/헤테로다이머화 및 자동인산화를 트리거한다; 이 인산화 수용체는 다운스트림 시그널링 캐스케이드를 활성화하는 어댑터 단백질을 모집한다. | 핵막, ER막 |
| LNPEP(루실시티닐아미노펩티드가수분해효소) | Q9UIQ6 | N 말단 아미노산 방출, 시스테인 및 류신보다 먼저 갈라짐, 임신 중 항상성 유지에 도움 | 혈장막, 세포외 영역 |
| ECM29 | Q5VYK3 | 특정 단백질에 결합하는 어댑터/스카폴딩 단백질. 프로테아솜을 ER, 엔도솜 및 중심체와 결합할 수 있습니다. | 핵, 중심체, ER, 엔도솜, 세포질 소포 |
호몰로지와 진화
정형외과와 병변외과
TMEM267은 유방, 파충류, 양서류, 몰루스카, 절지동물, 가지종, 트리코플락스, 오균류, 세균 등에 정형어를 가지고 있지만 평행어는 없다.선택한 맞춤법 표는 [43]다음과 같습니다.
| 속과 종 | 공통명 | 분류학 그룹 | 추정 분기일(MYA) | 등록 번호 | 시퀀스 길이(aa) | 시퀀스 얼라인먼트 | 시퀀스 유사성 |
|---|---|---|---|---|---|---|---|
| 난노스팔락스갈릴리 | 북부 이스라엘 맹인 지하 두더지 | 쥐목:짐승 강의 한 목. | 90 | XP_008831143.1 | 215 | 85.19% | 93% |
| Opisthocomus hoazin | 파충류 조류 | Opisthocomiformes | 312 | XP_009941974.1 | 215 | 83.26% | 92% |
| Protobothrops mucrosquamatus | 살모사 독 | 뱀목 | 312 | XP_015672610.1 | 219 | 82.16% | 89% |
| 제노 Tropicalis | 열대성 발톱 개구리 | 무미류 | 351.8 | XP_031750178.1 | 215 | 74.88% | 86% |
| Notothenia coriiceps | 남극 Yellowbelly Rockcod | 퍼시폼스과 | 435 | XP_010772860.1 | 244 | 67.21% | 82% |
| 아가미꽃차례 | 가지종 | 양악상목 | 684 | XP_019622820.1 | 215 | 32.09% | 63% |
| 강직성 자반충 | 청개구리 | 에키노데르마타 | 684 | XP_030828106.1 | 221 | 36.3% | 59% |
| 아에티나투미다 | 작은 벌집 비틀 | 콜롭테라속 | 797 | XP_019869992.1 | 201 | 35.82% | 49% |
| 크라소스트레아기가스 | 퍼시픽 오이스터 | 오스트레오이드과 | 797 | XP_011423125.1 | 209 | 34.93% | 52% |
진화
TMEM267은 피브리노겐 알파 사슬보다는 느리게, 시토크롬 [44]C보다는 빠르게 진화할 것으로 예측된다.
기능.
임상적 의의
이 단백질은 포유동물 세포에서 필터를 구성하는 단백질의 큰 그룹의 구성원으로 확인되었으며,[45] 이는 단백질을 섬모 밖으로 선택적으로 통과시켜 내용물을 조절한다.TMEM267은 아토피(피부 표면의 심한 가려움증, 홍조, 스케일링, 손실 부위로 특징지어지는 피부 질환)에 대한 훈련 데이터의 2-샘플 t-테스트와 윌콕슨 만-화이트니 분석을 통해 대조군과 경험군 사이의 분리에 대한 가장 많은 정보를 제공하는 10개의 유전자 중 하나였다.정신 [46]집단TMEM267은 두 개의 miRNA의 하향조절과 관련된 기사에서 언급되고 있으며, 그 중 하나는 증식, 아포토시스, 이동 및 분화와 같은 광범위한 세포 기능을 조절하는 데 포함되며, 이들 모두는 심장 [47][48]세포의 정상적인 발달에 필수적이다.
암
TMEM267 단백질은 대장암,[49] 위암, 폐암, 자궁내막암, 신장암, 유방암의 0.1~0.9%에서 돌연변이가 나타났다.TMEM267은 NUDT21 유전자의 증가된 수치의 영향을 받았으며, 3'-UTR이 짧아지면 제어할 수 없는 세포 [50]성장을 일으킬 수 있는 큰 그룹의 가능한 종양유전자의 일부로 확인되었다.그것은 PNI+[51] 혀암 환자의 생존율을 확인할 수 있는 유전자 그룹의 일부이다.TMEM267은 5p 염색체에서 과잉 발현된 26개의 유전자 중 하나로 나타났으며, 이는 암세포가 성장 및 주변 세포의 [52]침입에 이점을 제공할 가능성이 있는 유전자 그룹에 속한다는 것을 의미한다.또한 존스홉킨스 대학 연구진은 대조군에 비해 HMGA1 단백질 존재 하에서 발현이 증가한 TMEM267을 포함한 63개 유전자에 대한 특허를 출원했는데, 이들은 HMGA1 억제제를 [53]사용하여 암 줄기세포를 억제하는 방법에 유용하다고 생각한다.
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG00000151881 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG000074634 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ TMEM267의 GeneCards.org 엔트리
- ^ TMEM267의 GeneCards.org 엔트리
- ^ Q0VDI3의 UniProtKB 엔트리(TM267_HUMAN)
- ^ 제노마틱스의 엘도라도
- ^ 국립생명공학정보센터C5orf28의 AceView엔트리 [1]
- ^ TMEM267의 GeneCards.org 엔트리
- ^ ExPASy 계산 pI/MW 예측 변수 [2]/
- ^ TMEM267 위의 SAPS 엔트리
- ^ TMEM267 위의 SAPS 엔트리
- ^ TMEM267의 ELM 엔트리
- ^ TMEM267에서의 Prodom 엔트리
- ^ TMEM267용 NetWheels 헬리컬 투영
- ^ TMEM267의 MotifFinder 엔트리
- ^ Hislop JN, Marley A, von Zastrow M. Lysosomes에 대한 비유기화 G 단백질 결합 수용체의 내구성 운반에서 포유동물 포경 단백질 정렬 단백질의 역할.J Biol Chem 2004; 279:22522–22531.[3]
- ^ 표준 아르기닌 함유 포스포펩타이드 모티브의 ELM 상세
- ^ IRF3 정보
- ^ 델타 COP단백질
- ^ Worth, M., Zhang, W., Razi, M. 등ATG8 단백질에 대한 자동 파지 어댑터와 수용체의 선택적 결합을 조절하는 분자 결정인자.NAT Community 10, 2055 (2019)[4]
- ^ Romero, G., Von Zastrow, M. 및 P. A. 프리드먼. (2011년)GPCR의 트래픽, 시그널링 및 기능을 조절하는 PDZ 단백질의 역할: 수단, 모티브 및 기회.약리학의 진보(캘리포니아 샌디에이고), 62, 279–314.[5]
- ^ LIG_Pex14의 ELM 상세
- ^ TMEM267의 ELM 엔트리
- ^ TMEM267의 Human Protein Atlas 항목
- ^ TMEM267의 PSORT 예측
- ^ PaxDb 단백질 풍부도
- ^ TMEM267에 대한 Chau-Fasman 예측
- ^ TMEM267의 GOR4 예측
- ^ TMEM267의 Phyre 2 예측
- ^ TMEM267의 Human Protein Atlas 항목
- ^ TMEM267용 CSS-Palm
- ^ TMEM267의 SMOsp 예측
- ^ TMEM267용 GPS
- ^ TMEM267의 NetPhos 예측
- ^ TMEM267의 NetACET 예측
- ^ TMEM267의 NetGlycate 예측
- ^ TMEM267에 대한 InoYANG 예측
- ^ 국립생명공학정보센터C5orf28의 AceView엔트리 [6]
- ^ 국립생명공학정보센터C5orf28의 AceView엔트리 [7]
- ^ TMEM267용 GenoMatix MatInspector의 El Dorado
- ^ 국립생명공학정보센터C5orf28의 AceView엔트리 [8]
- ^ 분기 날짜의 TimeTree 데이터
- ^ 섬모형 생물 파라메슘 테트라우렐리아에서 발렌타인, 메간 스미스, "폴리시스틴-2(PKD2), 편심(XNTA), 메켈린(MKS3)"(2015년).대학원 논문과 논문. 419. [9]
- ^ 테세, 다윗(2018).Transcriptome Data에서 후보 유전자 선택을 위한 선택된 파라메트릭과 비모수 통계적 접근법의 비교[10]
- ^ Han, S., Wang, W., Duan, L., Hou, Z., Z., Zeng, J., L., …Jang, L.(2019).산발적인 심방중격결손환자의 마이크로RNA 프로파일링.바이오테크놀로지 & 바이오테크놀로지 기기, 33(1), 510~519.[11]
- ^ Sun, Xiaoyan & Song, Zhenhua & Si, Yawei & Wang, Jin-Hui. (2018년)스트레스 유발 우울증과 복원력과 관련된 복측 피개 영역의 마이크로RNA 및 mRNA 프로파일.신경심리약리학 및 생물정신학의 진보.86.10.16 [12]악의 측면
- ^ Yao, Z., Darowski, K., St-Denis, N., Wong, V., Offensperger, F., Villedieu, A., …Stagljar, I. (2017년)티로신인산화효소-단백질인산가수분해효소 인터랙텀의 글로벌 분석분자 세포, 65(2), 347~360.[13]
- ^ 시옹, M., 천, L., 저우, L., 딩, Y., 카조빙카, G., 천, Z. 및 호, T.(2019).NUDT21은 대체 폴리아데닐화에 의해 ANSA2와 LIMK2를 통해 방광암의 진행을 억제한다.시라노스틱스, 9(24), 7156-7167. [14]
- ^ Reddy RB, Khora SS, 수레쉬 A(2019) 머리와 목 편평상피세포암의 임상적 및 병리적으로 구별되는 코호트의 분자 예측인자- 메타 분석 접근법.PLoS ONE 14(7): e0218989.[15]
- ^ 스코토, L., 나라얀, G., 난둘라, SV. 등자궁경부암의 5p 염색체 증가의 통합 유전체 분석 결과, Drosha를 포함한 표적 과잉 발현 유전자가 밝혀졌다.Mol Cancer 7, 58 (2008).[16]
- ^ 구글 특허