인간백혈구항원
Human leukocyte antigen| 인간백혈구항원 | |
|---|---|
 MHC 클래스 I의 개략적 표현 | |
| 식별자 | |
| 기호. | HLA |
| 인터프로 | IPR037055 |
| 막질 | 63 |
인간 백혈구 항원(HLA) 체계 또는 복합체는 면역계의 [1]조절을 담당하는 세포 표면 단백질을 코드하는 인간의 6번 염색체 유전자 복합체이다.HLA 시스템은 또한 많은 [2]동물에서 발견되는 주요 조직적합성 복합체(MHC)의 인간 버전으로 알려져 있다.
HLA 유전자의 돌연변이는 I형 당뇨병과 같은 자가면역질환과 관련이 있을 수 있다.HLA 유전자 복합체는 6번 염색체 내 3Mbp 스트레치, p-arm 21.3에 존재한다.HLA 유전자는 매우 다형성이며, 이것은 그들이 적응 면역 체계를 미세 조정할 수 있게 해주는 많은 다른 대립 유전자를 가지고 있다는 것을 의미한다.특정 유전자에 의해 암호화된 단백질은 또한 항원으로 알려져 있는데, 장기 [3]이식의 인자로서의 역사적 발견의 결과이다.
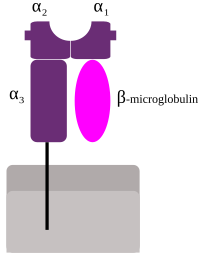
모두 HLA Class1 그룹인 MHC Class I(A, B, C)에 해당하는 HLA는 세포 내부에서 펩타이드를 나타낸다.예를 들어, 세포가 바이러스에 감염되면, HLA 시스템은 면역 체계에 의해 세포가 파괴될 수 있도록 바이러스의 파편을 세포 표면에 가져온다.이 펩타이드들은 프로테아솜에서 분해되는 소화 단백질로부터 생산된다.일반적으로 이러한 특정 펩타이드는 길이가 [4]약 8-10개의 아미노산인 작은 중합체이다.MHC 클래스 I에 의해 제시된 외래 항원은 세포를 파괴하는 킬러 T 세포(CD8 양성 또는 세포독성 T 세포라고도 함)라고 불리는 T 림프구를 끌어들인다.일부 새로운 연구는 세포독성 [5]T세포 반응을 유도하는 MHC I에 10개 이상의 아미노산, 11-14개의 아미노산이 제시될 수 있다는 것을 제안했다.MHC 클래스 I 단백질은 HLA 단백질과 달리 15번 염색체의 유전자에 의해 암호화되는 β2-마이크로글로불린과 관련된다.
MHC 클래스 II(DP, DM, DO, DQ 및 DR)에 대응하는 HLA는 세포 외부에서 T림프구로 항원을 나타낸다.이 특별한 항원은 T-도움 세포(CD4 양성 T 세포라고도 함)의 증식을 자극하고, 이것은 항체를 생산하는 B-세포를 자극하여 그 특정 항원에 대한 항체를 생산한다.자가항원은 조절 T세포에 의해 억제된다.어떤 항원이 특정 HLA 유형에 의해 면역체계에 나타날지 예측하는 것은 어렵지만 관련된 기술은 [6]발전하고 있다.
MHC 클래스 III에 대응하는 HLA는 보체계의 컴포넌트를 부호화한다.
HLA에는 다른 역할이 있습니다.그것들은 질병 방어에 중요하다.그것들은 장기이식 거부반응의 주요 원인이다.그들은 [7]암으로부터 보호하거나 (감염에 의해 억제된 경우) 보호하지 못할 수 있다.HLA는 또한 다른 사람의 냄새에 대한 사람들의 인식과도 관련이 있을 수 있으며, 적어도 한 연구는 고립된 [8]공동체에서 배우자 간의 HLA 유사성의 비율이 예상보다 낮다는 것을 발견했기 때문에 배우자 선택에 관여할 수도 있다.
6개의 주요 항원 제시 단백질을 코드하는 유전자 외에도, 면역 기능에 관련된 많은 다른 유전자들이 HLA 복합체에 위치하고 있습니다.인간 인구에서 HLA의 다양성은 질병 방어의 한 측면이며, 그 결과 모든 위치에 동일한 HLA 분자를 가진 두 개의 관련 없는 개인이 존재할 가능성은 매우 낮다.HLA 유전자는 역사적으로 HLA와 유사한 [citation needed]개인들 간에 장기를 성공적으로 이식할 수 있는 능력의 결과로 확인되었다.
기능들
HLA에 의해 암호화되는 단백질은 신체세포의 바깥쪽 부분에 있는 단백질로, 그 사람에게 고유한 단백질이다.면역체계는 자기 세포와 비자기 세포를 구별하기 위해 HLA를 사용한다.그 사람의 HLA 유형을 표시하는 모든 셀은 그 사람의 것이므로 침입자가 아닙니다.
전염병
외래 병원체가 체내에 들어오면, 항원 제시 세포라고 불리는 특정 세포들이 식세포증이라고 불리는 과정을 통해 병원체를 삼켜버린다.병원체로부터의 단백질은 작은 조각(펩타이드)으로 소화되어 HLA 항원(구체적으로는 MHC 클래스 II)에 적재된다.그런 다음 항원 제시 세포에 의해 CD4+ 도우미 T [9]세포에 표시되며, CD4+ 도우미 T 세포는 병원체를 제거하기 위해 다양한 효과와 세포 간 상호작용을 일으킨다.
비슷한 과정을 통해, 대부분의 세포에서 생성된 단백질(바이러스의 단백질과 같은 토종과 외래 단백질)은 세포 표면의 HLAs(구체적으로 MHC 클래스 I)에 표시된다.감염된 세포는 CD8+T [9]세포에 의해 인식되고 파괴될 수 있다.
측면의 이미지는 HLA-DR1 분자의 결합 균열 부분에 결합된 독성 박테리아 단백질(SEI 펩타이드) 조각을 보여줍니다.아래 멀리 있는 그림에서는 다른 관점에서 볼 때 유사한 틈새에 결합된 펩타이드가 있는 전체 DQ를 볼 수 있다.질병 관련 펩타이드는 손이 장갑에 끼는 것처럼 이러한 "슬롯"에 잘 맞습니다.결합 시 펩타이드는 T세포에 제시된다.T세포는 외부 항원을 인식하기 위해 MHC 분자를 통해 제시해야 합니다. 이는 MHC 제한으로 알려져 있습니다.T세포는 B세포 수용체와 유사한 수용체를 가지고 있으며, 각각의 T세포는 소수의 MHC 클래스 II-펩타이드 조합만을 인식한다.일단 T세포가 MHC 클래스 II 분자 내에서 펩타이드를 인식하면, 그것은 또한 그들의 B세포 수용체에서 같은 분자를 인식하는 B세포를 자극할 수 있다.따라서, T세포는 B세포가 동일한 외래 항원에 대한 항체를 만드는 것을 돕는다.각 HLA는 많은 펩타이드를 결합할 수 있으며, 각 사람은 3개의 HLA 타입을 가지고 있으며, 총 12개의 ISO 폼에 대해 4개의 DP, 4개의 DQ, 4개의 ISO 폼(DRB1의 2개 및 DRB3, DRB4 또는 DRB5)을 가질 수 있다.이러한 헤테로 접합체에서는 질병 관련 단백질이 검출을 면하기 어렵다.
이식 거부 반응
어떤 다른 HLA 타입을 나타내는 세포는 "비자기"이며 신체의 면역 체계에 의해 침입자로 보여지며, 결과적으로 그러한 세포를 가진 조직의 거부를 초래한다.이것은 이식된 조직의 경우 이식 거부반응을 일으킬 수 있기 때문에 특히 중요합니다.이식 시 HLA의 중요성 때문에 HLA 위치는 혈청학 및 PCR에 의해 가장 자주 입력되는 항목 중 하나입니다.고분해능 HLA 타입(HLA-A, HLA-B, HLA-C, HLA-DRB1, HLA-DQB1 및 HLA-DPB1)은 [10]기증자가 관련된 경우에도 완전한 일치를 식별하기 위해 이식 시 관련될 수 있는 것으로 나타났다.
| HLA 대립 유전자 | 리스크가 높은 질환 | 상대 리스크 |
|---|---|---|
| HLA-B27 | 강직성 척추염 | 12개[11] |
| 반응성 관절염 | 열네[11] 개 | |
| 급성 전방 포도막염 | 15개[11] | |
| HLA-B47 | 21-히드록실화효소결핍증 | 15개[11] |
| HLA-DR2 | 전신성 홍반성 낭창 | 2[12] ~ 3 |
| HLA-DR3 | 자가면역간염 | 열네[11] 개 |
| 일차 셰그렌 증후군 | 10개[11] | |
| 제1형 당뇨병 | 5개[11] | |
| 전신성 홍반성 낭창 | 2[12] ~ 3 | |
| HLA-DR4 | 류마티스 관절염 | 4개[11] |
| 제1형 당뇨병 | 6개[11] | |
| HLA-DR3 및 (DR4의 조합 | 제1형 당뇨병 | 15개[11] |
| HLA-DQ2 및 HLA-DQ8 | 강실성 질환 | 7개[13] |
자가면역 중
HLA형은 유전되며, 그 중 일부는 자가면역장애나 다른 질병과 관련이 있다.특정 HLA 항원을 가진 사람들은 I형 당뇨병, 강직성 척추염, 류마티스 관절염,[14] 셀리악 질환, SLE(시스템성 루푸스 홍반증), 근력증,[15] 포접체 근염, 쇼그렌 증후군, 기면증과 같은 특정 자가면역 질환에 걸릴 가능성이 더 높다.HLA 타이핑은 셀리악 질환과 제1형 당뇨병의 진단에 어느 정도 개선과 가속화를 가져왔지만, DQ2 타이핑이 유용하려면 고해상도 B1*타이핑(*02:02에서 *02:01 해결), DQ1*타이핑 또는 DR 혈청형이 필요합니다.현재 혈청형성은 DQ8을 한 단계로 해결할 수 있습니다. 자가면역 HLA 타이핑은 진단 도구로 점점 더 많이 사용되고 있습니다.소아지방병에서는 알레르기나 2차 자가면역질환과 같이 때때로 돌이킬 수 없는 증상이 나타나기 전에 위험에 처한 1급 친척과 위험하지 않은 친척을 구별할 수 있는 유일한 효과적인 수단이다.
암에서
일부 HLA 매개 질환은 암의 촉진과 직접적으로 관련되어 있다.글루텐 감수성 장질환은 장질환 관련 T세포 림프종의 유병률 증가와 관련이 있으며, DR3-DQ2 호모 접합체는 글루텐 감수성 장질환 관련 T세포 림프종 사례의 거의 80%와 함께 가장 높은 위험군 내에 있다.하지만, 더 자주, HLA 분자는 정상 상태에서의 낮은 수치 때문에 용인되지 않는 항원의 증가를 인식하면서 보호 역할을 합니다.비정상적인 세포는 진단 전에 많은 암을 중재하는 것으로 생각되는 아포토시스의 표적이 될 수 있다.
배우자선택중
특정 유전적 [16][17]특성과 관련하여 무작위로 짝짓기를 선택한다는 증거가 있다.이것은 유전자 중매로 알려진 분야로 이어졌다.
분류
MHC 클래스 I 단백질은 [1]신체의 대부분의 핵세포에서 기능성 수용체를 형성한다.
HLA에는 3개의 주요 MHC Class I 유전자와 3개의 마이너 MHC Class I 유전자가 있습니다.
메이저 MHC 클래스 I
마이너 유전자는 HLA-E, HLA-F 및 HLA-G이다.β-마이크로글로불린은2 메이저 및 마이너 유전자 서브유닛과 결합하여 헤테로디머를 생성한다.
HLA에 의해 코드되는 3개의 주요 MHC Class II 단백질과 2개의 마이너 MHC Class II 단백질이 있습니다.클래스 II의 유전자는 결합되어 전형적으로 항원 제시 세포의 표면에 발현되는 헤테로 이성질체(αβ) 단백질 수용체를 형성한다.
주요 MHC 클래스 II 단백질은 항원 제시 세포, B 세포 및 T [1]세포에서만 발생합니다.
- HLA-DP
- HLA-DPA1 궤적으로 부호화된 α 사슬
- HLA-DPB1 궤적으로 부호화된 β 사슬
- HLA-DQ
- HLA-DR
- HLA-DRA 궤적으로 부호화된 α 사슬
- HLA-DRB1, DRB3, DRB4, DRB5 loci에 의해 부호화된 β체인×4(1인당 3개만 가능)
다른 MHC Class II 단백질인 DM과 DO는 항원의 내부 처리에 사용되며, 병원균에서 생성된 항원 펩타이드를 항원 제시 세포의 HLA 분자에 적재한다.
명명법
현대의 HLA 대립 유전자는 일반적으로 다양한 수준의 세부사항으로 나타납니다.대부분의 지정은 HLA-와 궤적 이름으로 시작하여 *와 대립 유전자를 지정하는 일부(짝수) 자리수로 시작합니다.처음 두 자리는 슈퍼타입이라고도 하는 대립 유전자 그룹을 지정합니다.이전의 타이핑 방법론에서는 대립 유전자를 완전히 구별할 수 없었기 때문에 이 수준에서 멈췄다.세 번째에서 네 번째 숫자는 익명이 아닌 대립 유전자를 지정합니다.숫자 5에서 숫자 6은 유전자의 코드 프레임 내의 동의어 돌연변이를 나타냅니다.일곱 번째와 여덟 번째 자릿수는 부호화 영역 밖의 돌연변이를 구분합니다.L, N, Q 또는 S와 같은 문자는 발현 수준 또는 그에 대해 알려진 다른 비게놈 데이터를 지정하기 위해 대립 유전자의 지정을 따를 수 있습니다.따라서 완전히 기술된 대립 유전자는 HLA 프리픽스 및 궤적 [18]표기를 포함하지 않고 최대 9자리 길이일 수 있습니다.
가변성
MHC자리는 포유류에서 유전적으로 가장 가변적인 코드 위치 중 하나이며, 인간의 HLA자리는 예외는 아닙니다.인류는 역사상 여러 번 많은 위치를 고칠 수 있는 제약을 겪었음에도 불구하고, HLA의 흔적은 많은 [19]변화들과 함께 그러한 제약을 견뎌낸 것으로 보인다.위에 언급된 9개의 위치 중 대부분은 각 위치에 대해 12개 이상의 대립 유전자 그룹을 유지했으며, 이는 인간의 위치보다 훨씬 더 많이 보존된 변화이다.이는 이러한 위치에 대한 헤테로 접합 또는 균형 선택 계수와 일치합니다.또한 일부 HLA 위치는 인간 게놈에서 가장 빠르게 진화하는 코딩 영역 중 하나입니다.각 HLA 유전자 [20]클래스 내에서 가변 대립 유전자와 위치 사이의 치열한 유전자 변환을 거친 것으로 보이는 남아메리카의 아마존 부족의 연구에서 다양화의 한 메커니즘이 언급되었습니다.HLA 유전자를 통한 장기적인 생산적 재조합은 키메라 유전자를 생성하는 경우가 적습니다.
6개의 위치에는 100개가 넘는 대립 유전자가 존재하며 인간 개체에서 검출되었다.그 중 가장 가변적인 것은 HLA B와 HLA DRB1입니다.2012년 현재 확인된 대립 유전자의 수는 아래 표에 나와 있습니다.이 표를 해석하기 위해서는 각 대립 유전자가 적어도 하나의 위치(단일 뉴클레오티드 다형성, SNP)에서 다른 모든 대립 유전자와 다르도록 위치(위치에 있는 DNA) 배열의 변종임을 고려할 필요가 있다.이러한 변화의 대부분은 아미노산 배열의 변화를 초래하여 단백질에 약간의 또는 주요한 기능적 차이를 초래한다.
이 변동을 제한하는 문제가 있습니다.DQA1*05:01 및 DQA1*05:05와 같은 특정 대립 유전자는 동일한 처리 산물로 단백질을 인코딩합니다.DQB1*0201 및 DQB1*0202와 같은 다른 대립 유전자는 기능적으로 유사한 단백질을 생성한다.클래스 II(DR, DP 및 DQ)의 경우, 수용체의 펩타이드 결합 구개열 내의 아미노산 변이체는 서로 다른 결합 능력을 가진 분자를 생성하는 경향이 있다.
다만, 유전자 다양성 연구에서의 유형화·순서화·조절을 실시한 남미산 HLA-A, -B, -C, HLA-DPA1, -DPB1, -DQA1, -DRB1의 가장 일반적인 대립 유전자 빈도(>5%)는 보고되고 있다.또, 유럽 인구의 HLA-I 및 HLA-II 유전자의 대립 유전자 빈도에 관한 정보도 작성했다.[22][23]두 경우 모두 대립 유전자 빈도의 분포는 모집단의 역사와 관련된 지역적 변동을 나타낸다.
변종 대립 유전자 표
IMGT-HLA 데이터베이스에 따른 클래스 I 위치에서의 변종 대립 유전자 수, 2018년 10월 최종 갱신:
| MHC 클래스 I | |
|---|---|
| 궤적 | #[24][25] |
| 메이저 안티겐스 | |
| HLA A | 4,340 |
| HLA B | 5,212 |
| HLA C | 3,930 |
| 소항원 | |
| HLA E | 27 |
| HLA F | 31 |
| HLA G | 61 |
클래스 II 위치(DM, DO, DP, DQ 및 DR)에서의 변종 대립 유전자의 수:
| MHC 클래스 II | ||||
|---|---|---|---|---|
| HLA | - A1 | - B1 | - B3 ~ - B51 | 이론. 가능. |
| 궤적 | #[25] | #[25] | #[25] | 조합 |
| DM- | 7 | 13 | 91 | |
| 실행하다 | 12 | 13 | 156 | |
| DP- | 67 | 1,014 | 16,036 | |
| DQ- | 95 | 1,257 | 34,528 | |
| DR- | 7 | 2,593 | 312 | 11,431 |
| 1DRB3, DRB4, DRB5는 사람에게 다양한 존재감을 가진다. | ||||
시퀀스 피쳐 바리안트타입(SFVT)
HLA 유전자의 큰 가변성은 질병에서 HLA 유전자 변이의 역할을 조사하는데 있어 중대한 문제를 제기한다.질병 관련 연구는 일반적으로 각 HLA 대립 유전자를 질병과 관련된 분자의 일부를 비추지 않는 하나의 완전한 단위로 취급한다.Karp D. R. 등은 HLA 단백질을 생물학적으로 관련된 작은 배열 특징(SF)과 그 변형 유형(VT)[26]으로 분류하는 HLA 유전자 분석을 위한 새로운 배열 특징 변형 유형(SFVT) 접근방식을 설명한다.배열 특성은 구조 정보(예: 베타 시트 1), 기능 정보(예: 펩타이드 항원 결합) 및 다형성을 기반으로 정의된 아미노산 부위의 조합이다.이러한 시퀀스 피쳐는 선형 시퀀스에서 겹치고 연속적이거나 불연속적일 수 있습니다.각 시퀀스 기능의 배리언트타입은 설명되는 HLA 궤적 내의 기존의 모든 폴리모피즘에 근거해 정의됩니다.여러 HLA 대립 유전자가 공유하는 에피토프의 효과와 역할을 파악할 수 있도록 HLA의 SFVT 분류를 유전자 연관 분석에 적용한다.모든 고전적인 HLA 단백질에 대해 염기서열 특징과 그 변종 유형이 설명되었다. HLA SFVT의 국제적 저장소는 IMGT/HLA 데이터베이스에 [27]유지된다.HLA 대립 유전자를 구성 요소 SFVT로 변환하는 도구는 ImmPort([28]Immology Database and Analysis Port) 웹 사이트에서 찾을 수 있습니다.
일반적이고 문서화된 희귀 대립 유전자
확인된 개별 HLA 대립 유전자의 수는 많지만, 이러한 대립 유전자의 약 40%는 단일 [29][30]개체에서만 확인되는 독특한 것으로 보입니다.대략 3분의 1의 대립 유전자가 관련이 없는 [30][31]개체에서 3번 이상 보고되었다.개별 HLA 대립 유전자가 검출되는 속도의 변화 때문에 발현되는 각 HLA 궤적에서의 대립 유전자의 유병률로 분류하려는 시도가 있었다.그 결과 일반적이고 잘 문서화된 HLA 대립 유전자 [31][32]목록과 희귀하고 매우 희귀한 HLA [29][30]대립 유전자 목록이 작성되었습니다.
일반적인 HLA 대립 유전자는 최소 1500명의 [31][32]기준 모집단에서 최소 0.001의 빈도로 관찰된 것으로 정의된다.같은 적어도에 세번 관계 없는 individuals,[31일]에서 보고되는 것 Well-documented HLAalleles는 원래 지금 정의된다 같은 적어도 5배수에서 혈연이 아닌 사람들에서 SBT 메서드를 통해sequence-based을 입력하(SBT)메서드 또는 적어도 3번 및 활용 u에서 특별한 단상형을 통해 탐지되고 정의되었다nre라떼가 있는 사람들.[32]희귀 대립 유전자는 1~4회 보고된 유전자로 정의되며, 매우 희귀한 대립 유전자는 [29][30]1회 보고된 유전자로 정의된다.
각 유병률 범주별 HLA 대립 유전자 표
현재 CWD와 희귀 또는 매우 희귀한 명칭이 서로 다른 데이터셋과 IMGT/[30][32]HLA 데이터베이스의 다른 버전을 사용하여 개발되었지만, 각 범주의 각 HLA 궤적에서 대립 유전자의 대략적인 비율은 다음과 같다.
| HLA 궤적 | 일반적인 번호 대립하다[32] | %공통 대립하다[32] | 아니요. 잘 문서화되어 있습니다. 대립하다[32] | 적절한 비율(%) 대립하다[32] | 아니요, 레어 대립하다[30] | 희박도(%) 대립하다[30] | 아니요. 매우 희귀합니다. 대립하다[30] | 매우 드문 비율(%) 대립하다[30] | 대립 유전자 비율 분류된 |
|---|---|---|---|---|---|---|---|---|---|
| A | 68 | 3.4% | 178 | 8.8% | 145 | 21.5% | 280 | 41.6% | ~75% |
| B | 125 | 4.8% | 242 | 9.3% | 190 | 17.6% | 468 | 43.5% | ~75% |
| C | 44 | 2.8% | 102 | 6.6% | 77 | 21.4% | 154 | 42.8% | ~74% |
| DRB1 | 79 | 6.8% | 147 | 12.7% | 133 | 22.7% | 206 | 35.2% | ~77% |
| DRB3 | 5 | 8.6% | 7 | 12.1% | ~21% | ||||
| DRB4 | 6 | 40.0% | 2 | 13.3% | ~53% | ||||
| DRB5 | 5 | 25.0% | 3 | 15.0% | ~40% | ||||
| DQA1 | 15 | 31.9% | 4 | 8.5% | 9 | 26.5% | 7 | 20.6% | ~88% |
| DQB1 | 22 | 12.5% | 8 | 4.5% | 26 | 28.9% | 42 | 45.2% | ~91% |
| DPA1 | 6 | 17.6% | 0 | 0.0% | 4 | 14.8% | 15 | 55.6% | ~88% |
| DPB1 | 40 | 28.8% | 14 | 9.0% | 29 | 22.7% | 29 | 32.8% | ~90% |
| 모든 로키 | 415 | 5.3% | 707 | 9.0% | 613 | 20.6% | 1214 | 40.8% | ~76% |
HLA 유형 검사
혈청형 및 대립 유전자 이름
HLA에 적용되는 두 개의 병렬 명명 시스템이 있습니다.최초의 가장 오래된 시스템은 혈청학적(항체 기반) 인식에 기초하고 있습니다.이 시스템에서 항원은 결국 문자와 숫자(예: HLA-B27 또는 줄여서 B27)를 할당받았다.대립 유전자의 보다 정교한 정의를 가능하게 하는 병렬 시스템이 개발되었습니다.본 발명의 시스템에서는 문자 * 및 4자리 이상의 숫자(예를 들어 HLA-B*08:01, A*68:01, A*24:02:01N=N=N=N)와 함께 HLA를 사용하여 주어진 HLA 궤적에서의 특정 대립 유전자를 지정한다.HLA 궤적은 MHC 클래스 I 및 MHC 클래스 II(또는 드물게 D 궤적)로 더 분류할 수 있다.2년마다 혈청형을 대립 [24]유전자로 해석하는 연구자들을 돕기 위해 명명법이 발표된다.
혈청형성
타이핑 시약을 만들기 위해 동물이나 사람의 혈액을 채취하고 혈청을 혈청에서 분리하며 혈청을 최적의 감도로 희석하여 다른 사람이나 동물의 세포를 타이핑하는 데 사용합니다.따라서 혈청형은 HLA 수용체와 수용체 아이소폼을 대략적으로 식별하는 방법이 되었다.수년간 혈청형 항체는 민감도를 높이는 기술이 개선되고 새로운 혈청형 항체가 계속 등장함에 따라 더욱 정교해졌다.혈청형 분석의 목적 중 하나는 분석의 공백을 메우는 것이다.'제곱근', '최대우도' 방법 또는 적절한 유형의 대립 유전자를 설명하기 위한 가족성 하플로타입 분석을 바탕으로 예측할 수 있다.혈청형성 기법을 사용한 이러한 연구는 특히 비유럽인 또는 북동아시아인 모집단에 대해 많은 null 또는 공백 혈청형이 자주 밝혀졌다.이것은 최근까지 Cw 궤적에 특히 문제가 되어 1991년 인구 조사에서 Cw 혈청형의 거의 절반이 타이핑되지 않았다.
혈청형에는 몇 가지 종류가 있다.광범위한 항원 혈청형은 세포의 동일성에 대한 대략적인 척도이다.예를 들어 HLA A9 혈청형은 A23과 A24를 가진 개체의 세포를 인식한다.또한 A23과 A24가 작은 변화 때문에 놓치는 세포를 인식할 수도 있습니다.A23과 A24는 분할 항원이지만, 일반적으로 두 항원에 특화된 항체가 넓은 항원에 대한 항체보다 더 자주 사용된다.
셀룰러 타이핑
대표적인 세포측정법은 혼합림프구배양(MLC)으로 HLA클래스II [33]타입을 결정하는 데 사용된다.세포 검사는 혈청형 검사보다 HLA 차이를 감지하는 데 더 민감합니다.이것은 알로안티세라가 인식하지 못하는 작은 차이가 T세포를 자극할 수 있기 때문이다.이 입력은 Dw 유형으로 지정됩니다.혈청형 DR1은 다른 혈청형 DR에 대해 Dw1 또는 Dw20 중 하나로 세포적으로 정의되어 있습니다.표에는[34] DR 대립 유전자에 대한 관련 세포 특이성이 나와 있습니다.그러나 세포 타입은 세포 타입의 개인들 간의 반응에 일관성이 없으며, 때로는 예측과 다르게 나타난다.세포형 시약의 생성 및 유지보수의 어려움과 함께 세포형 시약은 DNA 기반의 타이핑 [33]방법으로 대체되고 있다.
유전자 염기서열
혈청형 그룹의 대립 유전자 생성물에 대해 다른 유형과 유사성을 보이는 하위 영역에 대한 경미한 반응을 관찰할 수 있다.항원의 배열은 항체 반응성을 결정하며, 따라서 좋은 염기서열 분석 능력(또는 염기서열 기반 타이핑)을 갖는 것은 혈청학적 반응을 필요로 하지 않는다.따라서, 다른 혈청형 반응은 새로운 유전자 서열을 결정하기 위해 사람의 HLA 서열을 결정할 필요성을 나타낼 수 있다.
많은 미확인 HLA 대립 유전자(아프리카, 아라비아,[35] 이란 남동부[36], 파키스탄, 인도[37])를 가진 매우 다양한 모집단을 입력하는 것과 같은 광범위한 항원 유형은 여전히 유용하다.아프리카, 이란 남부, 아라비아는 이전에 정착된 지역을 타이핑하는 데 어려움을 보여준다.대립 유전자의 다양성은 혈청형성 기술에 의해 잘못 식별될 위험이 증가하기 때문에 광범위한 항원 유형화 후 유전자 염기서열 분석을 사용할 필요가 있다.
결국, 워크샵은 시퀀스를 기반으로 어떤 새로운 대립 유전자가 어떤 혈청 그룹에 들어갈지를 시퀀스 또는 반응성으로 결정한다.시퀀스가 확인되면 번호가 할당됩니다.예를 들어 B44의 새로운 대립 유전자는 혈청형(즉, B44)과 대립 유전체 ID(즉, B*44:65)를 얻을 수 있다.마쉬 등(2005)[24]는 HLA 혈청형 및 유전자형 코드북으로 간주되며, 조직항원(Tissue Antigens)에서 매월 갱신되는 새로운 책으로 간주됩니다.
표현형
유전자 타입은 유전자 배열이나 혈청 타입과는 다르다.이 전략에서는 DNA의 변이 영역에 고유한 PCR 프라이머가 사용됩니다(배열 특이 프라이머라고 함).적절한 크기의 산물이 발견되면 HLA 대립 유전자가 식별된 것으로 가정합니다.새로운 유전자 배열은 종종 모호한 모습을 증가시킨다.유전자 타입은 SSP-PCR에 기초하기 때문에 새로운 변종, 특히 클래스 I와 DRB1의 위치가 누락될 수 있다.
예를 들어 임상 상황 내의 SSP-PCR은 HLA 표현형을 식별하기 위해 자주 사용된다.개인에 대한 확장 표현형의 예는 다음과 같습니다.
A*01:01/*03:01, C*07:01, B*07:02/*08:01, DRB1*03:01/*15:01, DQA1*05:01/*01:02, DQB1*06:02
일반적으로 확장 혈청형 A1, A3, B7, B8, DR3, DR15(2), DQ2, DQ6(1)과 동일합니다.
일본이나 유럽 인구와 같은 많은 인구에서, 많은 환자들이 타입을 이루었기 때문에 새로운 대립 유전자는 상대적으로 드물기 때문에 SSP-PCR은 대립 유전자의 분해에 충분합니다.하플로타입은 SSP-PCR이 대립 유전자를 인식할 수 없고 새로운 대립 유전자의 염기서열을 필요로 하는 세계의 지역에서 가족 구성원을 입력하면 얻을 수 있다.SSP-PCR 또는 혈청형이 불충분한 세계 지역은 중앙 아프리카, 동아프리카, 남아프리카의 일부, 아라비아, S. 이란, 파키스탄, 인도를 포함한다.
하플로타입
HLA 하플로타입은 염색체에 의한 일련의 HLA "유전자" (Loci-allees)로, 하나는 어머니로부터, 다른 하나는 아버지로부터 물려받습니다.
위에서 살펴본 표현형은 아일랜드에서 가장 흔한 것 중 하나이며, 두 가지 일반적인 유전자 하플로타입의 결과이다.
A*01:01; C*07:01; B*08:01; DRB1*03:01; DQB1*05:01, DQB1*02:01(A1-Cw7-B8-DR3-DQ2)
이것은 '슈퍼 B8' 또는 '조골 하플로타입'으로 불린다.
A*03:01; C*07:02; B*07:02; DRB1*15:01; DQB1*01:02; DQB1*06:02 (A3-Cw7-B7-DR15-DQ6 또는 이전 버전 "B2-3B"를 입력함으로써)
이러한 하플로타입은 종종 진화 과정에서 일어난 사건의 지문과 비슷하기 때문에 인구 이동을 추적하는데 사용될 수 있다.Super-B8 하플로타입은 서부 아일랜드에서 풍부하고, 그 지역에서 떨어진 구배를 따라 감소하며, 서유럽인들이 이주한 세계 지역에서만 발견된다.'A3-B7-DR2-DQ1'은 동아시아에서 이베리아까지 광범위하게 보급되어 있다.Super-B8 하플로타입은 다이어트와 관련된 많은 자가면역질환과 관련되어 있다.10만 개의 확장된 하플로타입이 있지만, 소수의 사람들만이 눈에 보이고 결절된 특징을 보인다.
대립 유전자 변이의 역할
인간과 동물에 대한 연구는 이 변동성에 [38]대한 설명으로서 이러한 위치에서 작동하는 헤테로 접합 선택 메커니즘을 암시한다.제안된 메커니즘 중 하나는 암컷이 자신의 [39]유형에 대해 다른 HLA를 가진 남성을 발견할 수 있는 성적 선택이다.DQ 및 DP 부호화 궤적은 대립 유전자가 적지만 A1:B1의 조합은 각각 7,755DQ 및 5,270DPαβ 헤테로디머의 이론 전위를 생성할 수 있다.인간 인구에는 이 숫자에 가까운 동소체들이 존재하지 않지만, 각 개인은 4개의 가변 DQ와 DP 동소체들을 운반할 수 있고, 이러한 수용체들이 면역체계에 나타낼 수 있는 잠재적 항원 수를 증가시킨다.
DP, DR 및 DQ의 가변 위치에 대한 연구는 Class II 분자의 펩타이드 항원 접촉 잔류물이 단백질 1차 구조의 변동 부위가 가장 자주 발생한다는 것을 보여준다.따라서 강력한 대립 유전자 변이 및/또는 서브유닛 페어링의 조합을 통해 클래스 II 펩타이드 수용체는 9개의 아미노산 이상의 길이가 거의 무한히 변화하는 펩타이드를 결합할 수 있으며, 교배 아집단을 초기 또는 유행병으로부터 보호할 수 있다.집단 내의 개인들은 종종 다른 하플로 타입을 가지고 있으며, 이것은 작은 집단에서도 많은 조합을 낳는다.이러한 다양성은 그러한 그룹의 생존을 향상시키고, 병원균의 에피토프의 진화를 방해하며, 그렇지 않으면 면역 체계로부터 보호될 수 있을 것이다.
항체
HLA 항체는 일반적으로 자연적으로 발생하지 않으며, 수혈, 임신(태아 유전 항원), 장기 또는 조직 이식을 통해 비자기 HLA를 포함하는 이물질에 대한 면역학적 도전의 결과로 거의 예외 없이 형성된다.
중증 자가면역질환의 [40]치료제로서 질병관련 HLA 하플로타입에 대한 항체가 제안되었다.
기증자 특이적 HLA 항체는 신장, 심장, 폐 및 간이식에서의 이식 부전과 관련이 있는 것으로 밝혀졌다.이러한 기증자 특이적 HLA 항체는 이전 이식이나 임신을 통한 감작의 결과로 이식 전 존재할 수 있지만 이식 후 발생하기도 한다.HLA 항체 감작 위험과 기증자-수령자 HLA(분자) 불일치 사이에는 분명한 연관성이 있다.[41]
아픈 형제자매의 HLA 매칭
조혈줄기세포이식을 필요로 하는 일부 질병에서는 윤리적 [42]고려가 있지만, 이식 전 유전자 진단을 사용하여 일치하는 HLA를 가진 형제자매를 낳을 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c Choo SY (February 2007). "The HLA system: genetics, immunology, clinical testing, and clinical implications". Yonsei Medical Journal. 48 (1): 11–23. doi:10.3349/ymj.2007.48.1.11. PMC 2628004. PMID 17326240.
- ^ "Human leukocyte antigen (HLA)". MS Trust. December 2020. Retrieved 7 June 2021.
{{cite web}}: CS1 maint :url-status (링크) - ^ Nakamura T, Shirouzu T, Nakata K, Yoshimura N, Ushigome H (September 2019). "The Role of Major Histocompatibility Complex in Organ Transplantation- Donor Specific Anti-Major Histocompatibility Complex Antibodies Analysis Goes to the Next Stage". International Journal of Molecular Sciences. 20 (18): 4544. doi:10.3390/ijms20184544. PMC 6769817. PMID 31540289.
- ^ Matsumura M, Fremont DH, Peterson PA, Wilson IA (August 1992). "Emerging principles for the recognition of peptide antigens by MHC class I molecules". Science. 257 (5072): 927–34. Bibcode:1992Sci...257..927M. doi:10.1126/science.1323878. PMID 1323878.
- ^ Burrows SR, Rossjohn J, McCluskey J (January 2006). "Have we cut ourselves too short in mapping CTL epitopes?". Trends in Immunology. 27 (1): 11–6. doi:10.1016/j.it.2005.11.001. PMID 16297661.
- ^ Bouzid R, de Beijer MT, Luijten RJ, Bezstarosti K, Kessler AL, Bruno MJ, Peppelenbosch MP, Demmers JA, Buschow SI (May 2021). "Empirical Evaluation of the Use of Computational HLA Binding as an Early Filter to the Mass Spectrometry-Based Epitope Discovery Workflow". Cancers. 13 (10): 2307. doi:10.3390/cancers13102307. PMC 8150281. PMID 34065814.
- ^ Galbraith W, Wagner MC, Chao J, Abaza M, Ernst LA, Nederlof MA, et al. (1991). "Imaging cytometry by multiparameter fluorescence". Cytometry. 12 (7): 579–96. doi:10.1002/cyto.990120702. PMID 1782829.
- ^ Brennan PA, Kendrick KM (December 2006). "Mammalian social odours: attraction and individual recognition". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 361 (1476): 2061–78. doi:10.1098/rstb.2006.1931. PMC 1764843. PMID 17118924.
- ^ a b Taylor CJ, Bolton EM, Bradley JA (August 2011). "Immunological considerations for embryonic and induced pluripotent stem cell banking". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 366 (1575): 2312–22. doi:10.1098/rstb.2011.0030. PMC 3130422. PMID 21727137.
- ^ Agarwal RK, Kumari A, Sedai A, Parmar L, Dhanya R, Faulkner L (September 2017). "The Case for High Resolution Extended 6-Loci HLA Typing for Identifying Related Donors in the Indian Subcontinent". Biology of Blood and Marrow Transplantation. 23 (9): 1592–1596. doi:10.1016/j.bbmt.2017.05.030. PMID 28603069.
- ^ a b c d e f g h i j 표 5-7인치: 제8판
- ^ a b 백인에 대한 값은 다음과 같은 61페이지(오른쪽 열)에 따라 제공됩니다.
- ^ Margaritte-Jeannin P, Babron MC, Bourgey M, Louka AS, Clot F, Percopo S, et al. (June 2004). "HLA-DQ relative risks for coeliac disease in European populations: a study of the European Genetics Cluster on Coeliac Disease". Tissue Antigens. 63 (6): 562–7. doi:10.1111/j.0001-2815.2004.00237.x. PMID 15140032.
- ^ Kurkó J, Besenyei T, Laki J, Glant TT, Mikecz K, Szekanecz Z (October 2013). "Genetics of rheumatoid arthritis - a comprehensive review". Clinical Reviews in Allergy & Immunology. 45 (2): 170–9. doi:10.1007/s12016-012-8346-7. PMC 3655138. PMID 23288628.
- ^ Mignot E, Lin L, Rogers W, Honda Y, Qiu X, Lin X, et al. (March 2001). "Complex HLA-DR and -DQ interactions confer risk of narcolepsy-cataplexy in three ethnic groups". American Journal of Human Genetics. 68 (3): 686–99. doi:10.1086/318799. PMC 1274481. PMID 11179016.
- ^ Jokiniemi A, Magris M, Ritari J, Kuusipalo L, Lundgren T, Partanen J, Kekäläinen J (August 2020). "Post-copulatory genetic matchmaking: HLA-dependent effects of cervical mucus on human sperm function". Proceedings. Biological Sciences. 287 (1933): 20201682. doi:10.1098/rspb.2020.1682. PMC 7482290. PMID 32811307.
- ^ Jokiniemi A, Kuusipalo L, Ritari J, Koskela S, Partanen J, Kekäläinen J (November 2020). "Gamete-level immunogenetic incompatibility in humans-towards deeper understanding of fertilization and infertility?". Heredity. 125 (5): 281–289. doi:10.1038/s41437-020-0350-8. PMC 7553942. PMID 32747723. S2CID 220947699.
- ^ "HLA Nomenclature @ hla.alleles.org". hla.alleles.org. Archived from the original on 2 May 2018. Retrieved 2 May 2018.
- ^ Shennan DH (2006). Evolution and the Spiral of Technology. Trafford Publishing. ISBN 978-1-55212-518-2.
- ^ 를 클릭합니다Parham P, Ohta T (April 1996). "Population biology of antigen presentation by MHC class I molecules". Science. 272 (5258): 67–74. Bibcode:1996Sci...272...67P. doi:10.1126/science.272.5258.67. PMID 8600539. S2CID 22209086..
- ^ Requena D, Médico A, Chacón RD, Ramírez M, Marín-Sánchez O (2020). "Identification of Novel Candidate Epitopes on SARS-CoV-2 Proteins for South America: A Review of HLA Frequencies by Country". Frontiers in Immunology. 11: 2008. doi:10.3389/fimmu.2020.02008. PMC 7494848. PMID 33013857.
- ^ Nunes JM, Buhler S, Roessli D, Sanchez-Mazas A (May 2014). "The HLA-net GENE[RATE] pipeline for effective HLA data analysis and its application to 145 population samples from Europe and neighbouring areas". Tissue Antigens. 83 (5): 307–23. doi:10.1111/tan.12356. PMID 24738646.
- ^ Sanchez-Mazas A, Buhler S, Nunes JM (2013). "A new HLA map of Europe: Regional genetic variation and its implication for peopling history, disease-association studies and tissue transplantation". Human Heredity. 76 (3–4): 162–77. doi:10.1159/000360855. PMID 24861861.
- ^ a b c Marsh SG, Albert ED, Bodmer WF, Bontrop RE, Dupont B, Erlich HA, et al. (April 2010). "Nomenclature for factors of the HLA system, 2010". Tissue Antigens. 75 (4): 291–455. doi:10.1111/j.1399-0039.2010.01466.x. PMC 2848993. PMID 20356336.
- ^ a b c d EBI Web Services. "Statistics < IMGT/HLA < IPD < EMBL-EBI". www.ebi.ac.uk. Archived from the original on 20 September 2012. Retrieved 2 May 2018.
- ^ Karp DR, Marthandan N, Marsh SG, Ahn C, Arnett FC, Deluca DS, et al. (February 2010). "Novel sequence feature variant type analysis of the HLA genetic association in systemic sclerosis". Human Molecular Genetics. 19 (4): 707–19. doi:10.1093/hmg/ddp521. PMC 2807365. PMID 19933168.
- ^ "IMGT/HLA Database". Archived from the original on 24 September 2006.
- ^ "Immunology Database and Analysis Portal (ImmPort)". Archived from the original on 26 July 2011.
- ^ a b c Middleton D, Gonzalez F, Fernandez-Vina M, Tiercy JM, Marsh SG, Aubrey M, et al. (December 2009). "A bioinformatics approach to ascertaining the rarity of HLA alleles". Tissue Antigens. 74 (6): 480–5. doi:10.1111/j.1399-0039.2009.01361.x. PMID 19793314.
- ^ a b c d e f g h i Gonzalez-Galarza FF, Mack SJ, Hollenbach J, Fernandez-Vina M, Setterholm M, Kempenich J, et al. (February 2013). "16(th) IHIW: extending the number of resources and bioinformatics analysis for the investigation of HLA rare alleles". International Journal of Immunogenetics. 40 (1): 60–5. doi:10.1111/iji.12030. PMID 23198982. S2CID 205192491.
- ^ a b c d Cano P, Klitz W, Mack SJ, Maiers M, Marsh SG, Noreen H, et al. (May 2007). "Common and well-documented HLA alleles: report of the Ad-Hoc committee of the american society for histocompatiblity and immunogenetics". Human Immunology. 68 (5): 392–417. doi:10.1016/j.humimm.2007.01.014. PMID 17462507.
- ^ a b c d e f g h Mack SJ, Cano P, Hollenbach JA, He J, Hurley CK, Middleton D, et al. (April 2013). "Common and well-documented HLA alleles: 2012 update to the CWD catalogue". Tissue Antigens. 81 (4): 194–203. doi:10.1111/tan.12093. PMC 3634360. PMID 23510415.
- ^ a b 헐리 CK(1997)."DNA 기반 이식용 HLA 타이핑"Leffell MS, Donnenberg AD, Rose NR, eds. (1997) 인간면역학 핸드북. 페이지 521-55, Boca Raton: CRC Press, ISBN 0-8493-0134-3.
- ^ Bodmer JG, Marsh SG, Albert ED, Bodmer WF, Dupont B, Erlich HA, et al. (May 1992). "Nomenclature for factors of the HLA system, 1991". Human Immunology. 34 (1): 4–18. doi:10.1016/0198-8859(92)90079-3. PMID 1399721.
- ^ Valluri V, Valluei V, Mustafa M, Santhosh A, Middleton D, Alvares M, et al. (August 2005). "Frequencies of HLA-A, HLA-B, HLA-DR, and HLA-DQ phenotypes in the United Arab Emirates population". Tissue Antigens. 66 (2): 107–13. doi:10.1111/j.1399-0039.2005.00441.x. PMID 16029430.
- ^ Farjadian S, Naruse T, Kawata H, Ghaderi A, Bahram S, Inoko H (November 2004). "Molecular analysis of HLA allele frequencies and haplotypes in Baloch of Iran compared with related populations of Pakistan". Tissue Antigens. 64 (5): 581–7. doi:10.1111/j.1399-0039.2004.00302.x. PMID 15496201.
- ^ Shankarkumar U, Prasanavar D, Ghosh K, Mohanty D (May 2003). "HLA A*02 allele frequencies and B haplotype associations in Western Indians". Human Immunology. 64 (5): 562–6. doi:10.1016/S0198-8859(03)00032-6. PMID 12691707.
- ^ 를 클릭합니다Apanius V, Penn D, Slev PR, Ruff LR, Potts WK (1997). "The nature of selection on the major histocompatibility complex". Critical Reviews in Immunology. 17 (2): 179–224. doi:10.1615/critrevimmunol.v17.i2.40. PMID 9094452..
- ^ Wedekind C, Seebeck T, Bettens F, Paepke AJ (June 1995). "MHC-dependent mate preferences in humans". Proceedings. Biological Sciences. 260 (1359): 245–9. Bibcode:1995RSPSB.260..245W. doi:10.1098/rspb.1995.0087. PMID 7630893. S2CID 34971350.
- ^ Oshima M, Deitiker P, Ashizawa T, Atassi MZ (May 2002). "Vaccination with a MHC class II peptide attenuates cellular and humoral responses against tAChR and suppresses clinical EAMG". Autoimmunity. 35 (3): 183–90. doi:10.1080/08916930290022270. PMID 12389643. S2CID 5690960.
- ^ Senev A, Coemans M, Lerut E, Van Sandt V, Kerkhofs J, Daniëls L, Driessche MV, Compernolle V, Sprangers B, Van Loon E, Callemeyn J, Claas F, Tambur AR, Verbeke G, Kuypers D, Emonds MP, Naesens M (September 2020). "Eplet Mismatch Load and De Novo Occurrence of Donor-Specific Anti-HLA Antibodies, Rejection, and Graft Failure after Kidney Transplantation: An Observational Cohort Study". J Am Soc Nephrol. 31 (9): 2193–204. doi:10.1681/ASN.2020010019. PMC 7461684. PMID 32764139.
- ^ Verlinsky Y, Rechitsky S, Schoolcraft W, Strom C, Kuliev A (June 2001). "Preimplantation diagnosis for Fanconi anemia combined with HLA matching". JAMA. 285 (24): 3130–3. doi:10.1001/jama.285.24.3130. PMID 11427142.
참고 문헌
- Daniel M. Davis, 호환성 유전자, 런던, Penguin Books, 2014(ISBN 978-0-241-95675-5).
- Zsolt Harsanyi와 Richard Hutton, Richard 유전예언: 런던, 이중 나선 너머: 그라나다, 1982 (ISBN 0-246-11760-5).
외부 링크
- 유럽생물정보학연구소 IMGT/HLA 배열 데이터베이스
- hla.alleles.org
- Anthony Nolan Trust의 HLA 정보학 그룹
- 영국조직적합성면역유전학회
- 미국조직적합성면역유전학회
- 유럽면역유전학연맹
- 장기 및 줄기세포 이식을 위한 HistoCheck HLA 매칭 도구
- 가변 면역 관련 위치의 대립 유전자 빈도
- 미국 국립 의학 도서관 의학 주제 제목(MeSH)의 인간+백혈구+항원
- 주요 조직적합성 복합체에 대한 NCBI 데이터베이스인 dbMHC Home
- AFND(AlleleFrequencies Net Database)에서의 희귀알레알레알레알레스 프로젝트
- 일반 및 Well-Documented(CWD) 대립 유전자 카탈로그




