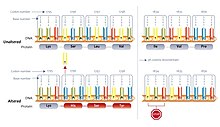
코돈 정지
Stop codon분자생물학(구체적으로 단백질 생합성)에서 정지 코돈(또는 종단 코돈)은 [1]현재 단백질의 번역 과정의 종료를 알리는 코돈(메신저 RNA 내의 뉴클레오티드 트리플렛)이다.메신저 RNA의 대부분의 코돈은 성장하는 폴리펩타이드 사슬에 아미노산을 추가하는 것에 해당하며, 이는 궁극적으로 단백질이 될 수 있다.; 코돈은 리보솜 서브유닛의 연결을 해제시키고 아미노산 사슬을 방출하는 결합 방출 인자에 의해 이 과정의 종료를 신호한다.
start codon은 변환을 시작하기 위해 가까운 시퀀스 또는 시작 계수가 필요하지만 종료를 시작하기 위해서는 stop codon만으로 충분합니다.
특성.
표준 코돈
표준 유전자 코드에는 세 가지 다른 종단 코돈이 있습니다.
| 코돈 | 표준코드 (번역표 1) | 이름. | ||
|---|---|---|---|---|
| DNA | RNA | |||
TAG | UAG | STOP = Ter (*) | "실패" | |
TAA | UAA | STOP = Ter (*) | 황토색 | |
TGA | UGA | STOP = Ter (*) | "opal"(또는 "umber") | |
대체 정지 코돈
표준 유전자 코드에는 변이가 있으며, 척추동물의 [2]미토콘드리아 게놈인 Scenedesmus obliquus,[3] Thraustchytrium에서 [4]대체 정지 코돈이 발견되었다.
| 유전자 코드 | 번역. 테이블 | 코돈 | 번역. 이 코드를 사용하여 | 표준 번역 | ||||
|---|---|---|---|---|---|---|---|---|
| DNA | RNA | |||||||
| 척추동물 미토콘드리아 | 2 | AGA | AGA | STOP = Ter (*) | Arg (R) | |||
AGG | AGG | STOP = Ter (*) | Arg (R) | |||||
| 미토콘드리아 오블리쿠스 | 22 | TCA | UCA | STOP = Ter (*) | Ser (S) | |||
| 미토콘드리아 트라우스토키튬 | 23 | TTA | UUA | STOP = Ter (*) | Leu (L) | |||
| 아미노산 생화학적 특성 | 논폴라 | 북극의 | 기본의 | 산성 | 종료: 코돈 정지 |
재할당된 중지 코드
핵유전자 코드는 표준 [5]정지 코돈을 아미노산에 재할당하는 변형 유전자 코드에 의해 설명되었듯이 유연하다.
| 유전자 코드 | 번역. 테이블 | 코돈 | 조건부 번역. | 표준 번역 | ||||
|---|---|---|---|---|---|---|---|---|
| DNA | RNA | |||||||
| 카리오레플란트 핵 | 27 | TGA | UGA | Ter (*) | 또는 | Trp (W) | Ter (*) | |
| 콘딜로스토마 핵 | 28 | TAA | UAA | Ter (*) | 또는 | Gln (Q) | Ter (*) | |
TAG | UAG | Ter (*) | 또는 | Gln (Q) | Ter (*) | |||
TGA | UGA | Ter (*) | 또는 | Trp (W) | Ter (*) | |||
| 블라스토크리티디아 핵 | 31 | TAA | UAA | Ter (*) | 또는 | Glu (E) | Ter (*) | |
TAG | UAG | Ter (*) | 또는 | Glu (E) | Ter (*) | |||
번역.
1986년에 셀레노시스테인(Sec)이 공역적으로 통합되었다는 설득력 있는 증거가 제공되었다.또한 폴리펩타이드 사슬에 부분적으로 혼입되는 코돈은 오팔 종단 [6]코돈으로도 알려진 UGA로 식별되었다.이 코돈의 종단 기능을 오버라이드하기 위한 다른 메커니즘이 원핵생물 [7]및 진핵생물에서 확인되었다.이 왕국들 사이의 특별한 차이점은 시스 원소가 원핵생물에서는 UAG 코돈 근처에 제한적으로 보이는 반면 진핵생물에서는 이러한 제한이 존재하지 않는다는 것이다.대신에 그러한 장소는 금지되지는 않았지만 별로 탐탁지 않은 것처럼 보인다.[8]
2003년에 한 획기적인 논문이 인간에게 알려진 모든 셀레노프로틴의 식별을 설명했습니다. [9]총 25개입니다.다른 유기체에도 유사한 분석이 실행되었다.
UAG 코돈은 유사한 방법으로 필로리신(Pyl)으로 변환될 수 있습니다.
게놈 분포
유기체의 게놈 내에서 정지 코돈의 분포는 랜덤하지 않으며 GC [10][11]함량과 상관관계가 있을 수 있다.예를 들어 대장균 K-12 게놈은 2705 TAA(63%), 1257 TGA(29%), 326 TAG(8%) 스톱 코돈(GC 함량 50.8%)[12]을 함유하고 있다.또한 스톱코돈 방출인자 1 또는 릴리스인자 2의 기판은 스톱코돈의 [11]풍부함과 강한 상관관계가 있다.광범위한 GC 함량을 가진 박테리아에 대한 대규모 연구는 TAA의 발생 빈도가 GC 함량과 음의 상관관계가 있고 TGA의 발생 빈도가 GC 함량과 양의 상관관계가 있는 반면, 종종 게놈에서 최소로 사용되는 코드인 TAG 스톱 코돈의 발생 빈도는 GC 함량과 양의 상관관계가 있음을 보여준다.는 GC [13]콘텐츠의 영향을 받지 않습니다.
인식
박테리아에서 정지 코돈의 인식은 RF1(PxT) 및 RF2(SPF)에서 고도로 보존된 아미노산 모티브인 이른바 '트리펩타이드 안티코돈'[14]과 관련되어 있다.이것은 구조적인 연구에 의해 뒷받침되고 있지만, 삼중의 반코돈 가설은 지나치게 [15]단순화된 것으로 나타났다.
명명법
스톱 코돈은 역사적으로 많은 다른 이름이 붙었는데, 각각이 비슷한 방식으로 행동하는 다른 종류의 돌연변이들에 대응했기 때문이다.이 돌연변이들은 박테리아 대장균을 감염시키는 바이러스인 박테리오파지(T4와 람다)에서 처음 분리되었다.바이러스 유전자의 돌연변이는 때때로 특정 종류의 대장균 내에서만 감염되고 자랄 수 있는 바이러스를 만들면서 그들의 감염 능력을 약화시켰다.
황색 돌연변이(UAG)
그들은 리처드 H. 엡스타인과 찰스 스타인버그에 의해 격리되고 그들의 친구이자 대학원생인 Harris Bernstein의 이름을 따서 명명된, 말도 안 되는 돌연변이가 발견된 첫 번째 세트였다. Harris Bernstein의 성은 독일어로 "황색"을 의미한다.번스타인).[16][17]
호박 돌연변이를 가진 바이러스는 호박 억제제로 알려진 특정 균주만을 감염시키는 능력을 특징으로 한다.이 박테리아들은 돌연변이 바이러스의 기능을 회복시킬 수 있는 그들만의 돌연변이를 가지고 있다.예를 들어, 호박 정지 코돈을 인식하는 tRNA의 돌연변이는 번역이 코돈을 "독해"하여 전장 단백질을 생성하도록 하여 단백질의 정상적인 형태를 회복하고 호박 [18]돌연변이를 "억제"한다.따라서, 호박 돌연변이는 호박 억제 돌연변이를 포함하는 박테리아에서 자랄 수 있는 전체 종류의 바이러스 돌연변이입니다.유사한 억제제는 황토 및 오팔 정지 코돈으로도 알려져 있습니다.
부자연스러운 아미노산을 운반하는 tRNA 분자는 박테리아 RNA의 호박 정지 코돈을 인식하도록 설계되었다.이 기술은 표적 단백질의 특정 위치에 직교 아미노산(예: p-아지도페닐알라닌)의 통합을 가능하게 한다.
황토색 돌연변이(UAA)
코돈 돌연변이가 발견된 것은 두 번째였다.호박과 관련된 일반적인 황-오렌지-갈색을 연상시키는 이 두 번째 멈춤 코돈은 오렌지-레디시-갈색 광물 [17]색소인 "ochre"라는 이름을 얻었다.
황토색 돌연변이 바이러스는 특정 억제제 균주 내에서 감염력을 회복했다는 점에서 호박 돌연변이와 유사한 성질을 가지고 있었다.황토 억제제 세트는 호박 억제제와 구별되었기 때문에 황토 돌연변이는 다른 뉴클레오티드 트리플렛에 해당하는 것으로 추정되었다.시드니 브레너는 이 돌연변이들을 서로 및 다른 알려진 아미노산 코돈들과 비교하는 일련의 돌연변이 실험을 통해 호박과 황토색 돌연변이가 뉴클레오티드 세쌍둥이 "UAG"와 "UAA"[19]에 해당한다고 결론지었다.
오팔 또는 황색 돌연변이(UGA)
표준 유전자 코드의 세 번째이자 마지막 정지 코돈은 곧 발견되었으며, 뉴클레오티드 트리플렛 "UGA"[20]에 해당한다.
착색 광물을 테마로 한 세 번째 난센스 코돈은 다양한 [17]색을 나타내는 실리카의 일종인 '오팔'로 알려지게 되었다.이 조기 정지 코돈을 만든 말도 안 되는 돌연변이는 나중에 오팔 돌연변이 또는 황색 돌연변이로 불렸습니다.
돌연변이와 질병
말도 안돼
난센스 돌연변이는 DNA 염기서열의 변화로 인해 생기는 모든 단백질이 비정상적으로 짧아지는 것을 야기합니다.아미노산 사슬의 중요한 부분이 더 이상 조립되지 않기 때문에 이것은 종종 단백질의 기능 상실을 야기합니다.이 용어 때문에 stop codon은 난센스 코돈이라고도 불립니다.
논스톱
논스톱 변환(stop-loss variant라고도 함)은 정지 코돈 내에서 발생하는 포인트 변환입니다.중단 없는 돌연변이는 mRNA 가닥이 번역되지 않은 영역으로 계속 변환되도록 합니다.중단 없는 돌연변이를 가진 유전자에 의해 생성된 대부분의 폴리펩타이드는 그들의 극단적인 길이와 정상적인 접힘에 대한 영향을 잃는다.논스톱 돌연변이는 정지 코돈을 생성하지 않고 삭제한다는 점에서 넌센스 돌연변이와 다릅니다.논스톱 돌연변이는 또한 단일 뉴클레오티드가 다른 아미노산으로 치환되는 점 돌연변이인 미스센스 돌연변이와 다르다.끊임없는 돌연변이는 내분비 장애,[21] 눈병,[22] 신경 발달 [23][24]장애를 포함한 많은 유전 질환과 연관되어 왔다.
숨김 정지
숨김 중지란 프레임 이동 시 +1 또는 -1인 경우 정지 코돈으로 읽히는 논스톱 코돈입니다.숨겨진 중지 전에 해당하는 프레임 이동(리보솜 RNA 슬립 등으로 인해)이 발생하면 변환이 조기에 종료됩니다.이것은 비기능성 단백질에 대한 자원 낭비와 잠재적 세포독소의 생산을 감소시킨다는 가설이다.루이지애나 주립 대학의 연구원들은 숨겨진 정류장이 선택된다는 매복 가설을 제안합니다.숨겨진 멈춤을 형성할 수 있는 코돈은 같은 아미노산을 코드화할 수 있는 동의어 코돈에 비해 게놈에서 더 자주 사용됩니다.유기체의 불안정한 rRNA는 숨겨진 정지 [25]빈도가 높은 것과 관련이 있다.그러나 더 큰 데이터 집합에서는 이 [26]가설을 검증할 수 없습니다.
정지 코돈과 숨김 정지를 합친 것을 정지 신호라고 합니다.멤피스 대학의 연구원들은 유전자 함량에서 큰 차이에도 불구하고 유전적으로 관련된 박테리아 게놈의 세 가지 읽기 프레임에 있는 정지 신호들의 비율이 매우 비슷하다는 것을 발견했습니다.유전적으로 관련된 박테리아의 거의 동일한 게놈-TSSR 값은 박테리아 [27]게놈 확장이 박테리아 종에 대한 독특한 정지 신호 편향에 의해 제한된다는 것을 암시할 수 있다.
번역판 읽기
stop codon 억제 또는 translational readthrough는 번역에서 stop codon이 센스 코돈으로 해석될 때, 즉 standard 아미노산이 stop codon에 의해 'encodon'될 때 발생합니다.돌연변이된 tRNA는 판독의 원인이 될 수 있지만 정지 코돈에 가까운 특정 뉴클레오티드 모티브도 될 수 있다.번역 판독은 바이러스와 박테리아에서 매우 흔하며, 인간, 효모,[28][29] 박테리아 및 드로소필라에서 유전자 조절 원리로도 발견되었습니다.이런 종류의 내인성 번역 판독은 유전자 코드의 변형을 구성합니다. 왜냐하면 정지 코돈은 아미노산을 위한 코드이기 때문입니다.인간말레이트탈수소효소의 경우 스톱코돈은 [30]약 4%의 빈도로 읽힌다.정지 코돈에 삽입된 아미노산은 정지 코돈 자체의 동일성에 따라 달라집니다.UAA와 UAG 코돈의 경우 Gln, Tyr, Lys가 발견되었으며, UGA 코돈의 경우 Cys, Trp 및 Arg가 질량 [31]분석으로 확인되었다.포유류의 판독 범위는 매우 다양하며, 프로테옴을 광범위하게 다양화하고 암 [32]진행에 영향을 미칠 수 있습니다.
워터마크로 사용
2010년 크레이그 벤터는 합성 DNA에 의해 제어되는 최초의 완전한 기능을 가진 복제 세포를 공개했을 때, 어떻게 그의 팀이 RNA와 DNA에 워터마크를 생성하기 위해 빈번히 정지 코돈을 사용하여 결과가 합성인지(그리고 오염되지 않았거나 다른 것이 아님) 확인하는 데 도움을 주었는지 설명하고, 저자의 이름과 웹사이트 [33]주소를 암호화하기 위해 그것을 사용했습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, Gelbart WM (2000). "Chapter 10 (Molecular Biology of Gene Function): Genetic code: Stop codons". An Introduction to Genetic Analysis. W.H. Freeman and Company.
- ^ Barrell, B. G.; Bankier, A. T.; Drouin, J. (1979-11-08). "A different genetic code in human mitochondria". Nature. 282 (5735): 189–194. Bibcode:1979Natur.282..189B. doi:10.1038/282189a0. ISSN 0028-0836. PMID 226894. S2CID 4335828.
- ^ A. M. Nedelcu, R. W. Lee, G. Lemieux, M. W. Gray, G. Burger (June 2000). "The complete mitochondrial DNA sequence of Scenedesmus obliquus reflects an intermediate stage in the evolution of the green algal mitochondrial genome". Genome Research. 10 (6): 819–831. doi:10.1101/gr.10.6.819. PMC 310893. PMID 10854413.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Wideman, Jeremy G.; Monier, Adam; Rodríguez-Martínez, Raquel; Leonard, Guy; Cook, Emily; Poirier, Camille; Maguire, Finlay; Milner, David S.; Irwin, Nicholas A. T.; Moore, Karen; Santoro, Alyson E. (2019-11-25). "Unexpected mitochondrial genome diversity revealed by targeted single-cell genomics of heterotrophic flagellated protists". Nature Microbiology. 5 (1): 154–165. doi:10.1038/s41564-019-0605-4. hdl:10871/39819. ISSN 2058-5276. PMID 31768028. S2CID 208279678.
- ^ Swart, Estienne Carl; Serra, Valentina; Petroni, Giulio; Nowacki, Mariusz (2016). "Genetic Codes with No Dedicated Stop Codon: Context-Dependent Translation Termination". Cell. 166 (3): 691–702. doi:10.1016/j.cell.2016.06.020. PMC 4967479. PMID 27426948.
- ^ Zinoni, F; Birkmann, A; Stadtman, T; Böck, A (1986). "Nucleotide sequence and expression of the selenocysteine-containing polypeptide of formate dehydrogenase (formate-hydrogen-lyase-linked) from Escherichia coli". Proceedings of the National Academy of Sciences. 83 (13): 4650–4654. Bibcode:1986PNAS...83.4650Z. doi:10.1073/pnas.83.13.4650. PMC 323799. PMID 2941757.
- ^ Böck, A (2013). "Selenoprotein Synthesis". Encyclopedia of Biological Chemistry. pp. 210–213. doi:10.1016/B978-0-12-378630-2.00025-6. ISBN 9780123786319. Retrieved 23 August 2021.
- ^ Mix, H; Lobanov, A; Gladyshev, V (2007). "SECIS elements in the coding regions of selenoprotein transcripts are functional in higher eukaryotes". Nucleic Acids Research. 35 (2): 414–423. doi:10.1093/nar/gkl1060. PMC 1802603. PMID 17169995.
- ^ Kryukov, G; Gladyshev, V (2003). "Characterization of mammalian selenoproteomes". Science. 300 (5624): 1439–1443. Bibcode:2003Sci...300.1439K. doi:10.1126/science.1083516. PMID 12775843. S2CID 10363908.
- ^ Povolotskaya IS, Kondrashov FA, Ledda A, Vlasov PK (2012). "Stop codons in bacteria are not selectively equivalent". Biology Direct. 7: 30. doi:10.1186/1745-6150-7-30. PMC 3549826. PMID 22974057.
- ^ a b Korkmaz, Gürkan; Holm, Mikael; Wiens, Tobias; Sanyal, Suparna (2014). "Comprehensive Analysis of Stop Codon Usage in Bacteria and Its Correlation with Release Factor Abundance". The Journal of Biological Chemistry. 289 (44): 775–806. doi:10.1074/jbc.M114.606632. PMC 4215218. PMID 25217634.
- ^ "Escherichia coli str. K-12 substr. MG1655, complete genome [Genbank Accession Number: U00096]". GenBank. NCBI. Retrieved 2013-01-27.
- ^ Wong, Tit-Yee; Fernandes, Sanjit; Sankhon, Naby; Leong, Patrick P; Kuo, Jimmy; Liu, Jong-Kang (2008). "Role of Premature Stop Codons in Bacterial Evolution". Journal of Bacteriology. 190 (20): 6718–6725. doi:10.1128/JB.00682-08. PMC 2566208. PMID 18708500.
- ^ Ito, Koichi; Uno, Makiko; Nakamura, Yoshikazu (1999). "A tripeptide 'anticodon' deciphers stop codons in messenger RNA". Nature. 403 (6770): 680–684. doi:10.1038/35001115. PMID 10688208. S2CID 4331695.
- ^ Korkmaz, Gürkan; Sanyal, Suparna (2017). "R213I mutation in release factor 2 (RF2) is one step forward for engineering an omnipotent release factor in bacteria Escherichia coli". Journal of Biological Chemistry. 292 (36): 15134–15142. doi:10.1074/jbc.M117.785238. PMC 5592688. PMID 28743745.
- ^ Stahl FW (1995). "The amber mutants of phage T4". Genetics. 141 (2): 439–442. doi:10.1093/genetics/141.2.439. PMC 1206745. PMID 8647382.
- ^ a b c Lewin, Benjamin; Krebs, Jocelyn E.; Goldstein, Elliott S.; Kilpatrick, Stephen T. (2011-04-18). Lewin's Essential GENES. Jones & Bartlett Publishers. ISBN 978-1-4496-4380-5.
- ^ Robin Cook. "Amber, Ocher, and Opal Mutations Summary". World of Genetics. Gale.
- ^ Brenner, S.; Stretton, A. O. W.; Kaplan, S. (1965). "Genetic Code: The 'Nonsense' Triplets for Chain Termination and their Suppression". Nature. 206 (4988): 994–8. Bibcode:1965Natur.206..994B. doi:10.1038/206994a0. PMID 5320272. S2CID 28502898.
- ^ Brenner, S.; Barnett, L.; Katz, E. R.; Crick, F. H. C. (1967). "UGA: A Third Nonsense Triplet in the Genetic Code". Nature. 213 (5075): 449–50. Bibcode:1967Natur.213..449B. doi:10.1038/213449a0. PMID 6032223. S2CID 4211867.
- ^ Pang S.; Wang W.; et al. (2002). "A novel nonstop mutation in the stop codon and a novel missense mutation in the type II 3beta-hydroxysteroid dehydrogenase (3beta-HSD) gene causing, respectively, nonclassic and classic 3beta-HSD deficiency congenital adrenal hyperplasia". J Clin Endocrinol Metab. 87 (6): 2556–63. doi:10.1210/jcem.87.6.8559. PMID 12050213.
- ^ Doucette, L.; et al. (2011). "A novel, non-stop mutation in FOXE3 causes an autosomal dominant form of variable anterior segment dysgenesis including Peters anomaly". European Journal of Human Genetics. 19 (3): 293–299. doi:10.1038/ejhg.2010.210. PMC 3062009. PMID 21150893.
- ^ Torres-Torronteras, J.; Rodriguez-Palmero, A.; et al. (2011). "A novel nonstop mutation in TYMP does not induce nonstop mRNA decay in a MNGIE patient with severe neuropathy" (PDF). Hum. Mutat. 32 (4): E2061–E2068. doi:10.1002/humu.21447. PMID 21412940. S2CID 24446773.
- ^ Spaull, R; Steel, D; Barwick, K; Prabhakar, P; Wakeling, E; Kurian, MA (2022-07-23). "STXBP1 Stop‐Loss Mutation Associated with Complex Early Onset Movement Disorder without Epilepsy". Movement Disorders Clinical Practice: mdc3.13509. doi:10.1002/mdc3.13509. ISSN 2330-1619.
- ^ Seligmann, Hervé; Pollock, David D. (2004). "The Ambush Hypothesis: Hidden Stop Codons Prevent Off-Frame Gene Reading". DNA and Cell Biology. 23 (10): 701–5. doi:10.1089/1044549042476910. PMID 15585128.
- ^ Cavalcanti, Andre; Chang, Charlotte H.; Morgens, David W. (2013). "Ambushing the ambush hypothesis: predicting and evaluating off-frame codon frequencies in Prokaryotic Genomes". BMC Genomics. 14 (418): 1–8. doi:10.1186/1471-2164-14-418. PMC 3700767. PMID 23799949.
- ^ Wong, Tit-Yee; Schwartzbach, Steve (2015). "Protein mis-termination initiates genetic diseases, cancers, and restricts bacterial genome expansion". Journal of Environmental Science and Health, Part C. 33 (3): 255–85. doi:10.1080/10590501.2015.1053461. PMID 26087060. S2CID 20380447.
- ^ Namy O, Rousset JP, Napthine S, Brierley I (2004). "Reprogrammed genetic decoding in cellular gene expression". Molecular Cell. 13 (2): 157–68. doi:10.1016/S1097-2765(04)00031-0. PMID 14759362.
- ^ Schueren F, Lingner T, George R, Hofhuis J, Gartner J, Thoms S (2014). "Peroxisomal lactate dehydrogenase is generated by translational readthrough in mammals". eLife. 3: e03640. doi:10.7554/eLife.03640. PMC 4359377. PMID 25247702.
- ^ Hofhuis J, Schueren F, Nötzel C, Lingner T, Gärtner J, Jahn O, Thoms S (2016). "The functional readthrough extension of malate dehydrogenase reveals a modification of the genetic code". Open Biol. 6 (11): 160246. doi:10.1098/rsob.160246. PMC 5133446. PMID 27881739.
- ^ Blanchet S, Cornu D, Argentini M, Namy O (2014). "New insights into the incorporation of natural suppressor tRNAs at stop codons in Saccharomyces cerevisiae". Nucleic Acids Res. 42 (15): 10061–72. doi:10.1093/nar/gku663. PMC 4150775. PMID 25056309.
- ^ Ghosh, Souvik; Guimaraes, Joao C; Lanzafame, Manuela; Schmidt, Alexander; Syed, Afzal Pasha; Dimitriades, Beatrice; Börsch, Anastasiya; Ghosh, Shreemoyee; Mittal, Nitish; Montavon, Thomas; Correia, Ana Luisa; Danner, Johannes; Meister, Gunter; Terracciano, Luigi M; Pfeffer, Sébastien; Piscuoglio, Salvatore; Zavolan, Mihaela (15 September 2020). "Prevention of dsRNA‐induced interferon signaling by AGO1x is linked to breast cancer cell proliferation". The EMBO Journal. 39 (18): e103922. doi:10.15252/embj.2019103922. PMC 7507497. PMID 32812257.
- ^ "Watch me unveil "synthetic life"".