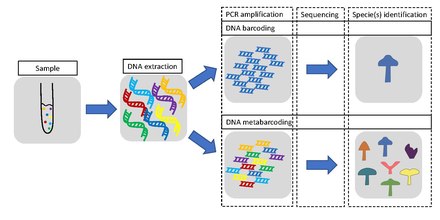
메타바코딩
Metabarcoding| 시리즈의 일부 |
| DNA바코딩 |
|---|
 |
| 분류별 |
| 다른. |
메타바코딩은 DNA/RNA(또는 eDNA/eRNA)의 바코딩으로 동일한 샘플 내에서 많은 분류군을 동시에 식별할 수 있습니다.바코딩과 메타바코딩의 주요 차이점은 메타바코딩은 하나의 특정 유기체에 초점을 맞추는 것이 아니라 샘플 내의 종 구성을 결정하는 것을 목표로 한다는 것이다.
바코드는 짧은 가변 유전자 영역(예를 들어 다른 마커/바코드 참조)으로 구성되며 프라이머 [1]설계에 사용할 수 있는 고도로 보존된 유전자 영역으로 측면 배치되는 분류학적 할당에 유용하다.일반적인 바코딩에 대한 이 아이디어는 2003년 [2]겔프 대학의 연구원들에 의해 시작되었다.
메타바코딩 절차는 일반적인 바코딩과 마찬가지로 DNA 추출, PCR 증폭, 시퀀싱 및 데이터 분석 단계를 거쳐 순차적으로 진행됩니다.하나의 종을 바코드화하는 것이 목적인지 아니면 여러 종을 메타암호화하는 것이 목적인지에 따라 다른 유전자가 사용된다.후자의 경우, 보다 보편적인 유전자가 사용된다.메타바코딩은 단일 종 DNA/RNA를 출발점으로 사용하지 않고 하나의 환경 또는 벌크 샘플에서 파생된 여러 다른 유기체의 DNA/RNA를 사용한다.
환경 DNA
환경 DNA 또는 eDNA는 전체 세포, 세포외 DNA 및 잠재적으로 전체 [3][4]유기체를 포함한 침전물, 물, 공기와 같은 환경 샘플에 존재하는 유전 물질을 말한다. eDNA는 환경 샘플에서 포착되어 [5]그 순서에 따라 보존, 추출, 증폭, 배열 및 분류될 수 있다.이 정보를 통해, 생물 종의 탐지 및 분류. 미생물들은 전부 얻을 수 있eDNA 좀 더 큰 생물체의 피부, 점막, 침, 정자, 분비물, 달걀, 대변, 소변, 혈액, 뿌리, 잎, 과일, 꽃가루, 그리고 썩어 가는 몸에서 오는 것도 가능할 것이다.[6][7][4]eDNA 생산 바이오매스, 나이와 먹이 먹는 행동을 하게 의존하고 있다.생리학, 생활사, 공간 [4][8][9][10]사용뿐만 아니라 유기체의 ivity.
2019년까지 eDNA 연구의 방법은 단일 표본에서 전체 지역사회를 평가할 수 있도록 확장되었다.이 과정은 메타바코딩을 포함하며, 메타바코딩은 모든 기원의 혼합 DNA 샘플에 일반 또는 범용 중합효소 연쇄반응(PCR) 프라이머를 사용한 후 시료의 종 구성을 결정하기 위해 높은 처리량 차세대 염기서열처리(NGS)를 사용하는 것으로 정밀하게 정의될 수 있습니다.이 방법은 미생물학에서 몇 년 동안 보편화되어 왔지만, 2020년 현재, 그것은 이제 겨우 [11][12][13]미생물의 평가에 기반을 두고 있다.eDNA 메타암코딩의 생태계 전체 애플리케이션은 지역사회와 생물 다양성을 설명할 뿐만 아니라 오염이나 기타 [7][14][12][9]오류로 인한 잘못된 판독에 의해 제한될 수 있지만 큰 공간적 규모로 상호작용과 기능 생태학을 탐지할 수 있는 잠재력을 가지고 있다.전체적으로, eDNA 메타암코딩은 기존의 바코드보다 속도, 정확도 및 식별을 향상시키고 비용을 절감하지만, 완전한 생태학적 [11][15][16][17][9][10]연구를 위해 분류법과 분자 방법을 통합하고 표준화 및 통일해야 한다.
eDNA 메타암코딩은 모든 서식지와 분류학 그룹, 고대 생태계 재구성, 식물-오염자 상호작용, 식단 분석, 침입종 검출, 오염 반응 및 대기질 모니터링에 응용된다. eDNA 메타암코딩은 아직 개발 중인 고유한 방법이며 유동성을 유지할 가능성이 높다.테크놀로지의 진보와 절차가 표준화됨에 따라 얼마간의 시간이 걸릴 수도 있습니다.그러나 메타코딩이 최적화되고 그 사용이 확산됨에 따라 생태 모니터링 및 지구 보존 [10]연구에 필수적인 도구가 될 것으로 보인다.
커뮤니티 DNA
HTS([18]High Throughput Sequencing)가 시작된 이래 생물다양성 검출 도구로서의 메타코드 사용은 큰 [12][19]관심을 끌었다.그러나 메타암호화 분석을 수행하기 위해 어떤 소스 물질이 사용되는지에 대해서는 아직 명확하지 않다(예: 환경 DNA 대 지역사회 DNA).이 두 소스 물질 사이의 명확성이 없다면 표본 추출의 차이와 실험실 절차의 차이는 데이터 처리에 사용되는 후속 생물 정보학 파이프라인에 영향을 미칠 수 있으며 공간 및 시간 생물 다양성 패턴의 해석을 복잡하게 할 수 있다.여기서는 널리 사용되는 소스 재료와 동식물의 환경 DNA 메타카코딩에 대한 하류 분석 및 해석에 미치는 영향을 커뮤니티 DNA [13]메타카코딩과 비교하여 명확하게 구별하려고 한다.
동식물의 지역사회 DNA 메타코딩을 통해 대상 그룹은 대량으로 수집되는 경우가 가장 많고(예: 토양, 병균 트랩 또는 그물), 개체는 대량 DNA [12]추출 전에 다른 샘플 잔해에서 제거되고 함께 뭉친다.이와는 대조적으로 매크로 유기체 eDNA는 샘플에서 개별 유기체 또는 식물 재료를 사전에 분리하지 않고 환경 물질(예를 들어 토양 또는 물)에서 직접 분리되며 전체 유기체가 샘플에 존재하지 않는다고 암묵적으로 가정한다.물론, 군집 DNA 샘플은 다른 유기체의 조직, 세포 및 소기관(예: 내장 내용물, 피부 세포 내 또는 세포 외 DNA)의 일부에서 DNA를 포함할 수 있다.마찬가지로, 매크로 유기 eDNA 샘플은 의도치 않게 전체 현미경 비표적 생물(예: 프로토리스트, 박테리아)을 포착할 수 있다.따라서 이러한 차이는 [13]실무상 최소한 부분적으로 분해될 수 있다.
군집 DNA와 매크로 유기 eDNA의 또 다른 중요한 차이점은 군집 DNA 메타암호화에서 생성된 염기서열이 추출 과정에서 파괴되지 않았을 때 분류학적으로 검증될 수 있다는 것이다.여기서 Sanger 시퀀싱을 사용하여 바우처 샘플에서 시퀀스를 생성할 수 있습니다.eDNA 메타암호화 샘플은 전체 유기체가 부족하기 때문에 현장 비교는 할 수 없다.Taxonomic 유사성만 직접, 분류학상으로. 미국 국가 생물 공학 센터의 GenBanknucleotide database,[20]BOLD,[21]이나self‐generated 참조 데이터베이스에 Sanger‐sequenced DNA.[22][에서 주석이 달린은 배열에 결합한 시퀀스)(눈을 통해 코끼리(조작 분류 단위 발생하 MOTUs)를 비교하여 설립할 수 있다.23][24](molecular operational competryic unit(MOTU)는 클러스터 알고리즘과 사전 정의된 비율 시퀀스 유사성(97%)[25][13]을 사용하여 식별되는 그룹입니다.그런 다음 결과 분류군의 목록을 최소한 부분적으로 확인하기 위해 동시에 수행된 기존의 물리적, 음향 또는 시각 기반 조사 방법과 비교하거나 위치에 대한 조사의 과거 기록과 비교한다(표 [13]1 참조).
따라서 군집 DNA와 eDNA 사이의 소스 물질의 차이는 검출된 생물 다양성에 대한 시공간 추론의 규모를 해석하는 데 뚜렷한 영향을 미친다.공동체 DNA로 볼 때 개별 종들이 저 시간과 장소에 있으나 eDNA에, DNA생산한 유기체 샘플링된 location,[26]는 일이나 DNA가 더 활동적 약탈적인 종(예를 들어의 대변에 운반되었을지도 모를 가장 수 있다는 것이 분명하다 새들 물고기 eDNA,[27]또는 기탁 이전에, 하지만 아냐. 참석했다 더 긴지역 사회에서 활동하며 검출은 수년에서 수십 년 전에 [28]유출된 DNA에서 나온 것입니다.후자는 eDNA를 [13]기반으로 지역사회에서 종의 존재를 추론할 때 공간과 시간 모두에서 추론의 규모를 신중하게 고려해야 한다는 것을 의미한다.
메타바코딩 단계
DNA 바코딩과 메타암코딩에는 6단계 또는 단계가 있습니다.동물(특히 박쥐)의 DNA 바코딩은 오른쪽 다이어그램과 바로 아래의 논의에서 예로 사용됩니다.
먼저, 몇 가지 특정 연구 질문에 답하기 위해 적합한 DNA 바코딩 영역을 선택합니다.동물에게 가장 일반적으로 사용되는 DNA 바코드 영역은 미토콘드리아 유전자 시토크롬 산화효소 I(CO1)[2]의 약 600쌍의 염기쌍이다.이 흔적은 종들 간의 큰 배열 변화를 제공하지만 [30]종들 내에서의 비교적 적은 양의 변화를 제공합니다.동물의 종 식별에 일반적으로 사용되는 다른 바코드 영역은 16S, 18S, 12S와 같은 리보솜 DNA(rDNA) 영역과 시토크롬 [31][32][33][34]B와 같은 미토콘드리아 영역이다.이러한 마커는 장점과 단점이 있으며 다양한 [35][36]용도로 사용됩니다.정확한 종 구분, 특히 가까운 친척을 구별하기 위해 더 긴 바코드 영역(최소 600개의 염기쌍)이 필요한 경우가 많다.분변, 털, 타액 등의 유기체 유해 생산자 식별은 생태계 내 종의 부재/존재를 검증하는 대용 수단으로 사용될 수 있다.이들 유골의 DNA는 보통 품질과 양이 낮기 때문에 약 100쌍의 염기쌍의 짧은 바코드를 사용한다.비슷하게, 똥에 남아 있는 DNA도 종종 분해되기 때문에,[29] 먹잇감을 식별하기 위해 짧은 바코드가 필요하다.
둘째, 기준 데이터베이스는 연구에서 발생할 가능성이 있는 모든 DNA 바코드로 구축되어야 한다.이상적으로는, 이러한 바코드는, 예를 들면 자연사 박물관이나 다른 연구 [29]기관과 같이, 공공이 접근할 수 있는 장소에 보관된 보증된 표본으로부터 생성되어야 한다.이러한 참조 데이터베이스의 구축은 현재 전 세계에서 이루어지고 있습니다.파트너 조직은 International Barcode of Life Project(iBOL) 및 Consortium for the Barcode of Life(CBOL)와 같은 국제 프로젝트에서 협력하여 세계 바이오옴의 DNA 기반 식별의 기반이 되는 DNA 바코드 참조를 구축하는 것을 목표로 합니다.잘 알려진 바코드 저장소는 NCBI GenBank와 BOLD(Barcode of Life Data System)[29]입니다.
셋째, 관심 있는 DNA를 가진 세포는 DNA를 노출시키기 위해 분해되어야 한다.이 단계인 DNA 추출 및 정화는 조사 대상 기질에서 수행해야 합니다.여기에는 [37]몇 가지 절차를 사용할 수 있습니다.DNA가 부분적으로 분해된 기질(예: 화석 샘플)과 혈액, 분변 및 토양과 같은 억제제를 포함하는 샘플)에서 DNA를 분리하는 특정 기술을 선택해야 합니다.DNA 수율이나 품질이 낮을 것으로 예상되는 추출은 현대 [38][39]DNA에 의한 오염을 방지하기 위해 확립된 프로토콜과 함께 고대 DNA 시설에서 수행되어야 한다.실험은 항상 양성 대조군이 [29]포함된 상태에서 중복하여 수행되어야 한다.
넷째, 1단계에서 선택한 DNA 바코드에 기초한 단일 시료 또는 프라이머와의 복합혼합물에서 추출된 DNA에서 앰피콘을 생성해야 한다.기원을 추적하기 위해 메타암코딩의 경우 라벨이 부착된 뉴클레오티드(분자 ID 또는 MID 라벨)를 추가해야 합니다.이러한 라벨은 분석 후반부에서 벌크 데이터 세트의 판독치를 원래 위치로 [29]추적하기 위해 필요합니다.
다섯째, DNA 염기서열 분석을 위해 적절한 기술을 선택해야 한다.전형적인 생어 연쇄 종단 방법은 DNA 복제 중 DNA 중합효소의 연쇄-인자 억제제의 선택적 배합에 의존한다.이들 4개의 염기는 전기영동을 사용하여 크기에 따라 분리되며 나중에 레이저 검출로 식별됩니다.Sanger 방법은 제한적이며 동시에 단일 판독치를 생성할 수 있으므로 단일 [29]종만을 포함하는 기질에서 DNA 바코드를 생성하는 데 적합합니다.나노포어 배열 분석과 같은 신기술로 인해 DNA 배열 분석 비용이 2002년 메가바이트당 약 3만 달러에서 2016년 [42][43]약 0.60달러로 감소했습니다.현대의 차세대 시퀀싱(NGS) 기술은 수천에서 수백만 개의 읽기를 병렬 처리할 수 있기 때문에 메타암코딩으로 [29]요약되는 기판에 존재하는 다양한 종의 혼합물을 대량 식별하는데 적합합니다.
마지막으로, [44]참조 라이브러리의 바코드 색인 번호(BIN)로 얻은 DNA 바코드를 일치시키기 위해 생체 정보 분석을 수행해야 한다.때 그것은 DNA바코드는 종 선물에 대한 참조 도서관에서 연결되어 있을 때나 종 수준으로 분류 식별은 종의 그룹(즉 종, 가족이나 더 높은 타소를 지칭하는 조작 분류 단위(작전 훈련 부대), 부족하다 높은(>97%)용어 색인을 보여 주각 BIN또는 BIN클러스터, 종 수준으로 확인될 수 있다.아니요.마이크 [29]랭크).('비닝(메타게노믹스)' 참조).생체정보학 파이프라인의 결과는 예를 들어 신뢰할 수 없는 싱글톤, 불필요한 중복, 저품질 판독 및/또는 키메라 판독을 걸러냄으로써 제거되어야 한다.이 작업은 일반적으로 자동 필터링 및 트리밍 [45]스크립트와 함께 직렬 BLAST 검색을 수행하여 수행됩니다.다른 종이나 올바른 식별과 잘못된 [29]식별을 구별하려면 표준화된 임계값이 필요하다.
메타바코딩 워크플로우
접근법의 명백한 힘에도 불구하고, eDNA 메타암코딩은 현장, 실험실 및 [46]키보드의 워크플로우 전체에 분산된 정밀도 및 정확도 과제에 영향을 받는다.오른쪽 다이어그램에 설명된 바와 같이, 초기 연구 설계(hypothesis/질문, 표적 분류군 등)에 따라 현재 eDNA 워크플로우는 현장,[13] 실험실 및 생물 정보학의 세 가지 요소로 구성됩니다.필드 구성요소는 DNA 추출 전에 보존되거나 냉동되는 샘플 채취(예: 물, 침전물, 공기)로 구성됩니다.실험실 구성요소는 4가지 기본 단계로 구성된다. (i) DNA가 농축되고(현장에서 수행되지 않는 경우), (ii) PCR을 사용하여 표적 유전자 또는 영역을 증폭하고, (ii) "인덱스"라고 불리는 고유한 뉴클레오티드 배열("바코드")을 PCR을 사용하여 통합하거나 다른 PCR 생성한다.ary"는 여러 샘플을 함께 풀링할 수 있으며 (iv) 풀링된 라이브러리는 높은 처리량 기계에서 시퀀싱됩니다.샘플의 실험실 처리 후 마지막 단계는 견고한 생체 정보학 [13]파이프라인을 사용하여 시퀀서의 출력 파일을 계산적으로 처리하는 것이다.

OTU와 종 개념
방법 및 시각화

b) [47]QIIME에서 수행된 주요 좌표 분석을 통해 설명되는 베타 다양성 패턴. 여기서 각 점은 표본을 나타내며 색상은 표본의 다른 클래스를 구분한다.3D 공간에서 두 개의 샘플 포인트가 가까울수록 커뮤니티 어셈블리가 더 유사합니다.
c) GraPhal 환경 데이터의 계통발생학적 시각화, 정량적 분류 [48]특성을 전달하는 데 사용되는 원형 열 지도 및 풍성 막대를 사용한다.
d) 엣지 PCA: 다른 PCA [49][50]축에 분산된 샘플에서 관찰된 커뮤니티 변화에 가장 큰 기여를 하는 특정 라인어지(녹색/주황색 브랜치)를 식별하는 트리 기반의 다이버시티 메트릭.
이 방법에서는 수집된 각 DNA를 일반적인 수집 데이터 외에 해당하는 "유형 표본" (각 분류군마다 하나씩)과 함께 보관해야 합니다.이러한 유형은 각 국가의 특정 기관(박물관, 분자 실험실, 대학, 동물원, 식물원, 허브리아 등)에 1개씩 보관되며, 일부 국가가 기술 또는 재정 자원을 보유하고 있지 않은 경우 동일한 기관이 국가 이상의 유형을 포함하도록 지정된다.o 그렇게 합니다.
이와 같이, 유전자 코드의 형식 표본의 작성은 전통적인 분류법에 의해 행해진 것과 평행한 방법론을 나타낸다.
첫 번째 단계에서는 바코드를 만드는 데 사용될 DNA 영역이 정의되었습니다.짧고 독특한 시퀀스의 높은 비율을 달성해야 했습니다.동물, 조류 및 곰팡이에 대해서는 시토크롬산화효소 CO1의 서브유닛1을 코드하는 미토콘드리아 유전자의 일부가 648개 [51]염기쌍의 영역인 높은 비율(95%)을 제공한다.
식물의 경우, CO1의 사용은 다배체, 침입, 그리고 교잡의 빈번한 효과로 인해 발생하는 어려움과 더불어 그 지역에서 낮은 수준의 변동성을 가지고 있기 때문에 효과적이지 않아, 그래서 엽록체 게놈이 더 [52][53]적합해 보인다.
적용들
폴리네이터 네트워크
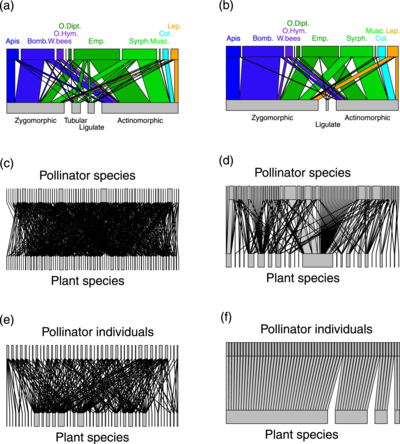
오른쪽 그림은 DNA 메타코딩에 기초한 수분 네트워크와 곤충의 식물 방문에 대한 직접적인 관찰에 기초한 보다 전통적인 네트워크를 비교한 것이다.메타코드 데이터는 수많은 숨겨진 상호작용을 탐지함으로써 방문 조사에 비해 수분 네트워크의 속성을 크게 변경합니다.분자 데이터는 방문 조사에서 예상했던 것보다 꽃가루 매개자가 훨씬 더 일반적이라는 것을 보여준다.그러나 꽃가루 매개자 종은 비교적 전문화된 개체로 구성되었고 꽃 [54]형태에 고도로 특화된 기능군을 형성하였다.
계속되는 세계적인 변화의 결과로, 꽃가루 매개자와 동물에 의해 수분되는 식물 종들의 극적인 그리고 병행적인 감소가 [55]관찰되었습니다.이러한 감소에 대한 수분 네트워크의 반응을 이해하는 것은 생태계가 발생할 수 있는 위험을 진단하고 보존 조치의 [56]효과성을 설계 및 평가하기 위해 시급하다.동물 수분에 대한 초기 연구는 단순화된 시스템, 즉 특정 쌍별 상호작용을 다루거나 식물-동물 공동체의 작은 하위 집합을 다루었다.그러나 장애의 영향은 매우 복잡한 상호 작용 네트워크를 통해 발생하며, 오늘날 이러한 복잡한 시스템은 주요 연구 초점이다.표본 추출 효과의 대상이 되는 현장 조사에서 진정한 네트워크(생태 프로세스에 의해 결정됨)를 평가하는 것은 [58][54]여전히 과제를 제공한다.
최근의 연구 연구는 대규모 종 [59]집합의 상호작용 패턴을 연구하기 위한 네트워크 개념과 도구로부터 분명히 이익을 얻고 있다.그들은 식물-공해자 네트워크가 랜덤 [60]연관성에서 크게 벗어나 고도로 구조화되었음을 보여주었다.일반적으로 네트워크는 낮은 수준의 일반화를 시사하는 낮은 연결성(공동체 내 모든 잠재적 연결의 실현된 부분)을 가진다. (2) 높은 중첩성(더 전문적인 유기체는 더 일반적인 유기체가 상호작용하는 종의 하위 집합과 상호작용할 가능성이 더 높다.) 더 전문적인 종만 상호작용한다.일반론자와 상호작용하는 적절한 하위 집합,[61] (3) 우연히 예상보다 더 많은 연결을 가진 소수의 슈퍼 제너럴리스트와 다수의 전문가에 의해 특징지어지는 멱함수 또는 잘린 멱함수를 따르는 연결성의 누적 분포(종당 링크 수, s). (4) 모듈러 조직.모듈은 모듈 내 연결성이 높고 다른 그룹의 [63][54]종과 연결이 잘 되지 않는 식물 및 꽃가루 매개자 종 그룹입니다.
접속성이 낮고 수분 네트워크 전문가 비율이 높은 것은 [64][65]네트워크에서는 전문화가 아니라 일반화가 표준이라는 견해와 대조됩니다.사실, 대부분의 식물 종들은 다양한 식물 [66][67]종들의 꽃 자원을 이용하는 다양한 종류의 꽃가루 매개자들에 의해 방문됩니다.이러한 명백한 모순을 설명하기 위해 유발되는 주요 원인은 상호작용의 [68]불완전한 표본 추출이다.실제로 대부분의 네트워크 속성은 샘플링 강도 및 네트워크 [60]크기에 매우 민감합니다.네트워크 연구는 기본적으로 식물 중심적, 즉 꽃가루 매개자의 꽃 방문 관찰에 기초한다.그럼에도 불구하고 이 식물 중심의 접근방식은 지역사회의 모임과 생물다양성 패턴에 기여하는 메커니즘의 이해를 저해할 수 있는 내재적 한계로 인해 어려움을 겪는다.첫째, 난초와 같은 특정 분류군에 대한 꽃가루 매개자 방문의 직접적인 관찰은 종종 드물고 일반적으로 [56][57][58][59]현장에서 드문 상호작용을 감지하는 것은 매우 어렵다.꽃가루 매개자와 식물 군집은 보통 소수의 풍부한 종과 방문 조사에서 [70][71]잘 기록되지 않은 많은 희귀 종으로 구성되어 있습니다.이 희귀종들은 전문가로 나타나지만, 사실 그들은 전형적인 일반학자일 수 있다.인터랙션 빈도(f)와 접속(s)의 관계가 정의되어 있기 때문에, 언더샘플링된 인터랙션은 네트워크의 [72]전문화 정도를 과대평가하는 원인이 될 수 있습니다.둘째, 네트워크 분석은 대부분 종 수준에서 작동해 왔다.네트워크는 기능 그룹으로 확장되거나 개별 기반 네트워크로 [73]축소되는 경우는 거의 없습니다.대부분의 네트워크는 1~2종에만 초점을 맞추고 있습니다.개체 또는 군집의 행동은 일반적으로 무시되지만,[73] 종 네트워크의 구조에 영향을 미칠 수 있습니다.따라서 종 네트워크에서 일반론자로 간주되는 종들은 수수께끼 같은 전문화된 개체나 군락을 수반할 수 있다.셋째, 꽃 방문자들은 동종 꽃가루 및/또는 많은 [74][75]이종 꽃가루를 축적할 수 없기 때문에 항상 효과적인 꽃가루 매개자는 아니다.방문객과 식물 스티그마에 대한 꽃가루 부하 조사에 기초한 동물 중심 접근법이 식물과 꽃가루 매개자의 [74][75][54]상호작용을 밝히는 데 더 효율적일 수 있다.
먹이사슬의 얽힘 제거

(A) 영양 네트워크:
절지동물과 척추동물의 포식자 – 화살표는 포식자와 먹이 사이의 바이오매스 흐름을 나타낸다.
(B) 기관 내 상호작용: *절지동물 포식자 *절지동물 기생충: * 식충척추동물:[76]
메타암호화는 먹이망 안에서 [77][78]포식자와 먹이 사이의 영양적 연결을 해독할 수 있는 새로운 기회를 제공한다.현미경이나 혈청학적 분석과 같은 전통적인 시간 소모적인 방법과 달리, DNA 메타암호화의 개발은 포식자의 먹이 범위에 대한 사전 지식 없이 먹이 종을 식별할 수 있게 해준다.또한 메타바코딩은 단일 PCR 반응에서 다수의 종을 특성화하고 수백 개의 샘플을 동시에 [6]분석하기 위해 사용될 수 있다.이러한 접근법은 농업 [78][79][80][81][82]생태계에서 먹이망의 기능적 다양성과 구조를 탐구하기 위해 점점 더 많이 사용되고 있다.다른 분자 기반 접근법과 마찬가지로 메타암호화는 내장 또는 분변 [83]샘플에서 먹이 종의 존재/존재 여부에 대한 질적 결과만 제공한다.그러나, 주어진 환경에서 같은 종의 포식자들이 소비하는 먹잇감의 정체성에 대한 이러한 지식은 "진정한 양적 [84][76]정보를 위한 체계적이고 유용한 대리인"을 가능하게 한다.
먹이 웹 생태학에서, "누가 누구를 먹느냐"는 주어진 [35][85]생태계 내에서 해충과 그들의 천적 사이에 존재하는 복잡한 영양 상호작용을 더 잘 이해하기 위한 근본적인 문제이다.절지동물과 척추동물 포식자의 식이요법 분석은 절지동물 해충의 자연 방제에 관여하는 주요 포식자를 식별할 수 있도록 하며, 그들의 식이요법의 폭(일반주의자 대 전문가)과 조직 내 [76]포식자에 대한 통찰력을 제공한다.
오른쪽 그림은 메타암호를 사용하여 세네갈의 두 개의 기장 밭과 관련된 먹이 그물의 기능적 다양성과 구조를 푼 2020년 연구에서 얻은 결과를 요약한 것이다.확인된 OTU를 종으로 지정한 후, 9개의 절지동물 포식자로부터 27개의 절지동물 먹이 분류군이 확인되었다.표본당 평균 먹이 분류군 수는 딱정벌레, 개미, 거미에서 가장 많았으며 안토코리드 벌레, 오각류 벌레, 귓발톱 등 나머지 포식자에서 가장 적었다.포식성 절지동물 전반에 걸쳐, 절지동물 먹이의 다양성이 거미, 딱정벌레, 개미, 그리고 개미과 곤충에서 관찰되었습니다.반면, 귓불과 오원충에서 확인된 먹잇감의 다양성은 상대적으로 낮았다.레피도프테라, 헤미프테라, 딥테라, 콜롭테라는 포식성 절지동물에서 [76]발견된 가장 흔한 곤충 먹이 분류군이었다.
특히 천적을 이용해 해충을 제어함으로써 기능적인 생물 다양성과 관련된 생태계 서비스를 보호하는 것은 식량 생산 [86][87][88]시스템의 지속 가능한 강화에 대한 도전에 대처할 수 있는 새로운 방법을 제공한다.절지동물과 척추동물을 포함한 일반주의 포식자에 의한 농작물 해충의 포식은 자연 해충 [89]방제의 주요 구성요소이다.대부분의 일반 포식자들의 특히 중요한 특성은 그들이 계절 [90][91]초기에 대체 먹이를 먹음으로써 농작물을 군집화할 수 있다는 것입니다.하지만, "일반주의" 식단의 폭은 길드 내 [89][92][93]포식과 같은 해충 방제에 몇 가지 단점을 수반합니다.따라서 비살충 먹잇감의 포식자를 포함한 일반론 포식자의 식단 폭에 대한 조정된 진단은 먹이 그물(예: 착취 경쟁과 명백한 경쟁)을 더 잘 풀고 궁극적으로 농업 생태계에서 자연 병충해 방제의 핵심 동인을 식별하기 위해 필요하다.그러나 먹이망에서 일반론 포식자의 중요성은 일반적으로 개별 포식자와 먹이와의 [92][94]상호작용의 일시적인 특성 때문에 평가하기가 어렵다.포식자의 유일한 결정적인 증거는 먹이 소비의 직접적인 관찰, 포식자의 [92]내장 내 먹이 잔류물의 확인, 역류나[95] [96][76]대변의 분석에서 비롯된다.
해양 생물 보안
비토종종(NIS)의 확산은 [98]생태계에 중대한 위험과 증가하는 위험을 나타냅니다.해양 시스템에서, 수송에서 살아남아 새로운 장소에 적응하는 국정원은 토종 종의 이동과 생물 군집과 관련 먹이 [99][100]그물의 이동 등 지역 생물 다양성에 상당한 악영향을 미칠 수 있다.일단 국정원이 설립되면,[101][102] 근절하는 것은 매우 어렵고 비용이 많이 들며, 자연분산이나 인위적인 운송 [102][103][104]경로를 통해 더 이상의 지역 확산이 일어날 수 있다.선체 오염과 선박의 밸러스트 물은 [98][105][106][107]NIS의 국제적 확산에 중요한 인공적인 경로로 잘 알려져 있지만, 지역 통과 선박이 빌지 물 이동을 [97]통해 해양 해충의 2차 확산에 기여할 가능성에 대해서는 비교적 거의 알려져 있지 않다.
최근 연구에 따르면 소형 선박의 빌지 공간(20m 미만)에 포함된 물과 관련 파편이 지역 규모로 [108][109][110][111][112][113]NIS 확산의 매개체 역할을 할 수 있다.빌지 워터는 (밸러스트가 아닌) 선박에 유지되는 모든 물로 정의되며, 선내에서 의도적으로 펌핑되지 않습니다.이는 파동 작용, 누출, 프로펠러 선미샘을 통한 누출 및 다이빙, 어업, 양식 또는 과학 장비 등의 [114]하중을 통해 선박 갑판(예: 바닥 패널 아래) 위 또는 아래에 축적될 수 있다.따라서 빌지 물은 해수뿐만 아니라 다양한 생활 단계의 생물, 세포 잔해 및 오염물질(예: 기름, 오염물, 세제 등)을 포함할 수 있으며, 이들 모두는 보통 자동 빌지 펌프를 사용하여 배출되거나 오리빌 밸브를 사용하여 자체 배수된다.여과 시스템, 원심 분리 또는 탄소 [114][115]흡수를 사용하여 기름과 물을 분리해야 하는 대형 선박과는 대조적으로 소형 선박에서 펌핑된 빌지 물은 보통 바다로 배출되기 전에 처리되지 않는다.이 과정을 통해 전파가 가능할 경우 빌지 워터의 방류로 인해 [97]NIS가 확산될 수 있습니다.
2017년에는 Fletcher et al.는 소규모 해안 [113]선박에서 채취한 빌지 물 샘플에 포함된 생물학적 물질의 다양성, 풍부성 및 생존을 조사하기 위해 실험실 및 현장 실험을 조합하여 사용했다.그들의 실험실 실험 결과, 백시디언 군락이나 파편, 브리오조아 유충은 여과되지 않은 펌프 시스템을 통해 거의 다치지 않고 생존할 수 있는 것으로 나타났습니다.그들은 또한 다양한 기원과 항해 시간을 가진 30개의 소형 선박(배와 모터보트)에서 빌지 물이 배출될 때 발생하는 생물 보안 위험에 대한 첫 번째 형태 분자 평가(eDNA 메타암코딩 사용)를 실시했다.그들은 eDNA 메타암호화를 사용하여 연구 지역에서 [97]비원주민으로 인식된 5종의 검출을 포함하여 기존의 현미경 방법을 통해 약 3배 더 많은 분류군을 특징지었다.
서로 다른 NIS 도입 벡터와 관련된 위험을 이해하는 데 도움을 주기 위해 기존의 현미경 생물 다양성 평가는 eDNA 메타암코딩으로 [116][117][118][119]점점 더 보완되고 있다.이를 통해 많은 생명 단계에서 다양한 분류학적 집합이 식별될 수 있다.또한 기존 방법으로 간과했을 수 있는 NIS를 탐지할 수도 있습니다.광범위한 분류학적 [120][121]선별을 위한 eDNA 메타암코딩 도구의 큰 잠재력에도 불구하고, 해양 해충의 환경 모니터링의 맥락에서, 특히 일부 빌지 공간이나 밸러스트 탱크와 같은 밀폐된 환경을 모니터링할 때 eDNA의 핵심 과제는 죽은 생물과 생존 가능한 [122]생물을 구별하는 것이다.세포외 DNA는 오랜 기간(수개월에서 수년)[123][124] 동안 어두운/차가운 환경에서 지속될 수 있으며, 따라서 eDNA 메타암호화를 사용하여 검출된 많은 유기체가 며칠 또는 몇 주 동안 샘플 채취 위치에서 생존하지 못했을 수 있다.이와는 대조적으로, 리보핵산(RNA)은 세포사망 후 빠르게 악화되어 생존 가능한 [125]공동체의 보다 정확한 표현을 제공할 수 있다.최근 metabarcoding 연구와 때 eR를 사용하고 물리 화학 생물학적 변수 간 영향 그라데이션을 따라 약간 더 강한 상관 관계를 발견했다co-extracted eDNA과 eRNA 분자의 해양 물고기 양식장과 석유 시추 sites,[126][127][128][129][130]주변 해저 침전물 샘플들을 감시하기 위해 사용을 탐사하였습니다.NA.해양생물보안 분야에서는 살아있는 국정원의 탐지가 DNA 신호만으로 국정원을 탐지하는 것보다 더 심각하고 즉각적인 위협이 될 수 있다.따라서 환경 RNA는 [97]샘플에서 살아있는 유기체를 식별하기 위한 유용한 방법을 제공할 수 있습니다.
여러가지 종류의
유전자 바코드 라이브러리의 건설은 처음에는 물고기와 [132][133][134]조류에 초점을 맞췄고, 나비와 다른 무척추동물이 [135]그 뒤를 따랐다.조류의 경우 DNA 샘플은 보통 가슴에서 채취된다.
연구자들은 이미 벌, 새, 포유류 또는 물고기와 같은 큰 동물군을 위한 구체적인 카탈로그를 개발했다.또 다른 용도는 지구 양극 지역에 사는 모든 유기체의 유전적 특성을 수집하는 것을 목표로 하는 "북극 생명 바코드" 프로젝트와 같은 특정 지역의 완전한 동물성 축소를 분석하는 것입니다.이 형태와 관련된 것은 브라질 [136][137]북동부 리오 상프란시스코에서 발달하기 시작한 수계 분지의 모든 어류 동물군을 코딩하는 것이다.
바코드의 사용 가능성은 매우 넓다. 수많은 암호종이 발견되었기 때문에(이미 수많은 긍정적인 [138]결과를 낳았다), 그들의 삶의 어느 단계에서나 종의 식별, 불법적으로 거래되는 보호종의 경우 안전한 식별 등.[139][140]
장점과 단점
가능성
DNA 바코딩은 전문가가 아닌 사람도 사용할 [141]수 있는 적합한 종을 구별하는 방법으로 제안되어 왔다.
단점
일반적으로 DNA 바코딩의 단점은 메타암코딩에도 유효합니다.메타암호화 연구의 한 가지 특별한 단점은 eDNA 메타암호화에 [142]적용될 최적의 실험 설계와 생물 정보학 기준에 대한 합의가 아직 없다는 것이다.그러나, EU COST 네트워크 DNAqua-Net과 [143]같이, 바이오모니터링의 [144]베스트 프랙티스 표준을 확립하기 위한 경험과 지식을 교환하는 등, 현재 협력하고 있는 시도가 있다.
이른바 바코드는 시토크롬c산화효소 유전자 내의 미토콘드리아 DNA 영역이다.바코드 오브 라이프 데이터 시스템(BOD) 데이터베이스에는 190,000여 [145][146]종의 DNA 바코드 시퀀스가 포함되어 있습니다.하지만, 롭 드살과 같은 과학자들은 종들을 [147]다르게 구분하기 때문에, 그들이 잘못된 명칭으로 여기는 고전적인 분류법과 DNA 바코딩이 조화를 이룰 필요가 있다고 우려를 표명했다.내심비온트와 다른 벡터에 의해 매개되는 유전적 침입은 [148]종의 식별에 있어 바코드를 더욱 효과적이지 않게 만들 수 있다.
바코드종 현황
미생물학에서 유전자는 먼 친척 박테리아 사이에서도 자유롭게 이동할 수 있으며, 아마도 전체 박테리아 영역까지 확장될 수 있다.경험적으로 미생물학자들은 서로 97% 이상 유사한 16S 리보솜 RNA 유전자 서열을 가진 박테리아나 고세균의 종류가 같은 종에 속하는지 [149]여부를 결정하기 위해 DNA-DNA 교잡에 의해 확인되어야 한다고 가정했다.이 개념은 2006년에 98.7%[150]의 유사성으로 좁혀졌다.
DNA-DNA 교배는 시대에 뒤떨어져 있고, 그 결과는 때때로 포마린과 큰 [151][152]스쿠아처럼 종에 대한 잘못된 결론을 이끌어냈다.최신 접근법은 계산 [153]방법을 사용하여 시퀀스 유사성을 비교한다.
「 」를 참조해 주세요.
- 바코드 오브 라이프 데이터 시스템(BOD)
- 바코드 오브 라이프(CBOL) 컨소시엄
- 국제 뉴클레오티드 배열 데이터베이스 콜라보레이션(INSDC)
- 분자 마커
- 분류학적 장애
레퍼런스
- ^ Pierre, Taberlet (2 February 2018). Environmental DNA : for biodiversity research and monitoring. Bonin, Aurelie, 1979-. Oxford. ISBN 9780191079993. OCLC 1021883023.
- ^ a b Hebert, Paul D. N.; Cywinska, Alina; Ball, Shelley L.; Dewaard, Jeremy R. (2003). "Biological identifications through DNA barcodes". Proceedings of the Royal Society of London. Series B: Biological Sciences. 270 (1512): 313–321. doi:10.1098/rspb.2002.2218. PMC 1691236. PMID 12614582.
- ^ Ficetola, Gentile Francesco; Miaud, Claude; Pompanon, François; Taberlet, Pierre (2008). "Species detection using environmental DNA from water samples". Biology Letters. 4 (4): 423–425. doi:10.1098/rsbl.2008.0118. PMC 2610135. PMID 18400683.
- ^ a b c Barnes, Matthew A.; Turner, Cameron R. (2016). "The ecology of environmental DNA and implications for conservation genetics". Conservation Genetics. 17: 1–17. doi:10.1007/s10592-015-0775-4. S2CID 14914544.
- ^ Deiner, Kristy; Walser, Jean-Claude; Mächler, Elvira; Altermatt, Florian (2015). "Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA". Biological Conservation. 183: 53–63. doi:10.1016/j.biocon.2014.11.018.
- ^ a b Taberlet, Pierre; Coissac, Eric; Pompanon, François; Brochmann, Christian; Willerslev, Eske (2012). "Towards next-generation biodiversity assessment using DNA metabarcoding". Molecular Ecology. 21 (8): 2045–2050. doi:10.1111/j.1365-294X.2012.05470.x. PMID 22486824. S2CID 41437334.
- ^ a b Bohmann, Kristine; Evans, Alice; Gilbert, M. Thomas P.; Carvalho, Gary R.; Creer, Simon; Knapp, Michael; Yu, Douglas W.; De Bruyn, Mark (2014). "Environmental DNA for wildlife biology and biodiversity monitoring". Trends in Ecology & Evolution. 29 (6): 358–367. doi:10.1016/j.tree.2014.04.003. PMID 24821515.
- ^ Goldberg, Caren S.; Turner, Cameron R.; Deiner, Kristy; Klymus, Katy E.; Thomsen, Philip Francis; Murphy, Melanie A.; Spear, Stephen F.; McKee, Anna; Oyler‐Mccance, Sara J.; Cornman, Robert Scott; Laramie, Matthew B.; Mahon, Andrew R.; Lance, Richard F.; Pilliod, David S.; Strickler, Katherine M.; Waits, Lisette P.; Fremier, Alexander K.; Takahara, Teruhiko; Herder, Jelger E.; Taberlet, Pierre (2016). "Critical considerations for the application of environmental DNA methods to detect aquatic species". Methods in Ecology and Evolution. 7 (11): 1299–1307. doi:10.1111/2041-210X.12595.
- ^ a b c Hering, Daniel; Borja, Angel; Jones, J.Iwan; Pont, Didier; Boets, Pieter; Bouchez, Agnes; Bruce, Kat; Drakare, Stina; Hänfling, Bernd; Kahlert, Maria; Leese, Florian; Meissner, Kristian; Mergen, Patricia; Reyjol, Yorick; Segurado, Pedro; Vogler, Alfried; Kelly, Martyn (2018). "Implementation options for DNA-based identification into ecological status assessment under the European Water Framework Directive". Water Research. 138: 192–205. doi:10.1016/j.watres.2018.03.003. PMID 29602086. S2CID 5008250.
- ^ a b c d e Ruppert, 크리스타 M., 클라인, RichardJ., 라만, Md Saydur(2019년)."과거, 현재, 환경 DNA(EDNA)metabarcoding의 미래 관점:.방법, 감시 및 글로벌 eDNA"의 응용 프로그램의 체계적인 논평.글로벌 생태 학부와 보존.17:e00547.doi:10.1016/j.gecco.2019.e00547.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ a b Coissac, Eric; Riaz, Tiayyba; Puillandre, Nicolas (2012). "Bioinformatic challenges for DNA metabarcoding of plants and animals". Molecular Ecology. 21 (8): 1834–1847. doi:10.1111/j.1365-294X.2012.05550.x. PMID 22486822. S2CID 24398174.
- ^ a b c d e f g Creer, 사이먼, Deiner, 크리스티, 프레이, Serita, Porazinska, Dorota, Taberlet, 피에르,.토마스 W. 켈리, 포터, 케이틀린;Bik, 홀리 M.(2016년)."생물 다양성의sequence‐based 확인에 그 생태 학자의 필드 가이드".생태학과 진화의. 7(9):1008–1018. doi:10.1111/2041-210X.12574.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ a b c d e f g h i Deiner, 크리스티, Bik, 홀리 M.;Mächler, 엘비라, 시모어, 매튜, Lacoursière‐Roussel, Anaïs, Altermatt, 플로리안, Creer, 사이먼, Bista, Iliana, 로지, 데이비드 맥도웰, 베르, 나타샤, Pfrender, 마이클 E.;Bernatchez, 루이(2017년)."환경 DNAmetabarcoding:우리가 어떻게 동물과 식물 communities" 조사를 변환합니다.분자 생태. 26(21):5872–5895. doi:10.1111/mec.14350.PMID 28921802.S2CID 8001074.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ Ficetola, Gentile Francesco; Taberlet, Pierre; Coissac, Eric (2016). "How to limit false positives in environmental DNA and metabarcoding?". Molecular Ecology Resources. 16 (3): 604–607. doi:10.1111/1755-0998.12508. PMID 27062589. S2CID 785279.
- ^ Yu, Douglas W.; Ji, Yinqiu; Emerson, Brent C.; Wang, Xiaoyang; Ye, Chengxi; Yang, Chunyan; Ding, Zhaoli (2012). "Biodiversity soup: Metabarcoding of arthropods for rapid biodiversity assessment and biomonitoring". Methods in Ecology and Evolution. 3 (4): 613–623. doi:10.1111/j.2041-210X.2012.00198.x.
- ^ Cristescu, Melania E. (2014). "From barcoding single individuals to metabarcoding biological communities: Towards an integrative approach to the study of global biodiversity". Trends in Ecology & Evolution. 29 (10): 566–571. doi:10.1016/j.tree.2014.08.001. PMID 25175416.
- ^ Gibson, Joel F.; Shokralla, Shadi; Curry, Colin; Baird, Donald J.; Monk, Wendy A.; King, Ian; Hajibabaei, Mehrdad (2015). "Large-Scale Biomonitoring of Remote and Threatened Ecosystems via High-Throughput Sequencing". PLOS ONE. 10 (10): e0138432. Bibcode:2015PLoSO..1038432G. doi:10.1371/journal.pone.0138432. PMC 4619546. PMID 26488407.
- ^ Margulies, Marcel; et al. (2005). "Genome sequencing in microfabricated high-density picolitre reactors". Nature. 437 (7057): 376–380. Bibcode:2005Natur.437..376M. doi:10.1038/nature03959. PMC 1464427. PMID 16056220.
- ^ Hajibabaei, Mehrdad; Shokralla, Shadi; Zhou, Xin; Singer, Gregory A. C.; Baird, Donald J. (2011). "Environmental Barcoding: A Next-Generation Sequencing Approach for Biomonitoring Applications Using River Benthos". PLOS ONE. 6 (4): e17497. Bibcode:2011PLoSO...617497H. doi:10.1371/journal.pone.0017497. PMC 3076369. PMID 21533287.
- ^ Benson, Dennis A.; Cavanaugh, Mark; Clark, Karen; Karsch-Mizrachi, Ilene; Lipman, David J.; Ostell, James; Sayers, Eric W. (2012). "Gen Bank". Nucleic Acids Research. 41 (Database issue): D36–D42. doi:10.1093/nar/gks1195. PMC 3531190. PMID 23193287.
- ^ Pruesse, E.; Quast, C.; Knittel, K.; Fuchs, B. M.; Ludwig, W.; Peplies, J.; Glockner, F. O. (2007). "SILVA: A comprehensive online resource for quality checked and aligned ribosomal RNA sequence data compatible with ARB". Nucleic Acids Research. 35 (21): 7188–7196. doi:10.1093/nar/gkm864. PMC 2175337. PMID 17947321.
- ^ o'Donnell, James L.; Kelly, Ryan P.; Lowell, Natalie C.; Port, Jesse A. (2016). "Indexed PCR Primers Induce Template-Specific Bias in Large-Scale DNA Sequencing Studies". PLOS ONE. 11 (3): e0148698. Bibcode:2016PLoSO..1148698O. doi:10.1371/journal.pone.0148698. PMC 4780811. PMID 26950069.
- ^ Sønstebø, J. H.; Gielly, L.; Brysting, A. K.; Elven, R.; Edwards, M.; Haile, J.; Willerslev, E.; Coissac, E.; Rioux, D.; Sannier, J.; Taberlet, P.; Brochmann, C. (2010). "Using next‐generation sequencing for molecular reconstruction of past Arctic vegetation and climate". Molecular Ecology Resources. 10 (6): 1009–1018. doi:10.1111/j.1755-0998.2010.02855.x. PMID 21565110. S2CID 23029234.
- ^ Willerslev, Eske; et al. (2014). "Fifty thousand years of Arctic vegetation and megafaunal diet" (PDF). Nature. 506 (7486): 47–51. Bibcode:2014Natur.506...47W. doi:10.1038/nature12921. PMID 24499916. S2CID 4461741.
- ^ Blaxter, Mark; Mann, Jenna; Chapman, Tom; Thomas, Fran; Whitton, Claire; Floyd, Robin; Abebe, Eyualem (2005). "Defining operational taxonomic units using DNA barcode data". Philosophical Transactions of the Royal Society B: Biological Sciences. 360 (1462): 1935–1943. doi:10.1098/rstb.2005.1725. PMC 1609233. PMID 16214751.
- ^ Deiner, Kristy; Altermatt, Florian (2014). "Transport Distance of Invertebrate Environmental DNA in a Natural River". PLOS ONE. 9 (2): e88786. Bibcode:2014PLoSO...988786D. doi:10.1371/journal.pone.0088786. PMC 3921251. PMID 24523940.
- ^ Merkes, Christopher M.; McCalla, S. Grace; Jensen, Nathan R.; Gaikowski, Mark P.; Amberg, Jon J. (2014). "Persistence of DNA in Carcasses, Slime and Avian Feces May Affect Interpretation of Environmental DNA Data". PLOS ONE. 9 (11): e113346. Bibcode:2014PLoSO...9k3346M. doi:10.1371/journal.pone.0113346. PMC 4234652. PMID 25402206.
- ^ Yoccoz, N. G.; Bråthen, K. A.; Gielly, L.; Haile, J.; Edwards, M. E.; Goslar, T.; von Stedingk, H.; Brysting, A. K.; Coissac, E.; Pompanon, F.; Sønstebø, J. H.; Miquel, C.; Valentini, A.; De Bello, F.; Chave, J.; Thuiller, W.; Wincker, P.; Cruaud, C.; Gavory, F.; Rasmussen, M.; Gilbert, M. T. P.; Orlando, L.; Brochmann, C.; Willerslev, E.; Taberlet, P. (2012). "DNA from soil mirrors plant taxonomic and growth form diversity". Molecular Ecology. 21 (15): 3647–3655. doi:10.1111/j.1365-294X.2012.05545.x. PMID 22507540.
- ^ a b c d e f g h i j Haarsma, Anne‐Jifke; Siepel, Henk; Gravendeel, Barbara (2016). "Added value of metabarcoding combined with microscopy for evolutionary studies of mammals". Zoologica Scripta. 45: 37–49. doi:10.1111/zsc.12214. S2CID 89048681.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ Kress, W. John; García-Robledo, Carlos; Uriarte, Maria; Erickson, David L. (2015). "DNA barcodes for ecology, evolution, and conservation". Trends in Ecology & Evolution. 30 (1): 25–35. doi:10.1016/j.tree.2014.10.008. PMID 25468359.
- ^ Galan, Maxime; Pagès, Marie; Cosson, Jean-François (2012). "Next-Generation Sequencing for Rodent Barcoding: Species Identification from Fresh, Degraded and Environmental Samples". PLOS ONE. 7 (11): e48374. Bibcode:2012PLoSO...748374G. doi:10.1371/journal.pone.0048374. PMC 3492341. PMID 23144869.
- ^ Ivanova, Natalia V.; Clare, Elizabeth L.; Borisenko, Alex V. (2012). "DNA Barcoding in Mammals". DNA Barcodes. Methods in Molecular Biology. Vol. 858. pp. 153–182. doi:10.1007/978-1-61779-591-6_8. ISBN 978-1-61779-590-9. PMID 22684956.
- ^ Clare, Elizabeth L. (2014). "Molecular detection of trophic interactions: Emerging trends, distinct advantages, significant considerations and conservation applications". Evolutionary Applications. 7 (9): 1144–1157. doi:10.1111/eva.12225. PMC 4231602. PMID 25553074.
- ^ Orlando, Ludovic; Hänni, Catherine; Douady, Christophe J. (2007). "Mammoth and Elephant Phylogenetic Relationships: Mammut Americanum, the Missing Outgroup". Evolutionary Bioinformatics. 3: 45–51. doi:10.1177/117693430700300019. PMC 2674638. PMID 19430604.
- ^ a b Pompanon, Francois; Deagle, Bruce E.; Symondson, William O. C.; Brown, David S.; Jarman, Simon N.; Taberlet, Pierre (2012). "Who is eating what: Diet assessment using next generation sequencing". Molecular Ecology. 21 (8): 1931–1950. doi:10.1111/j.1365-294X.2011.05403.x. PMID 22171763. S2CID 10013333.
- ^ Deagle, Bruce E.; Jarman, Simon N.; Coissac, Eric; Pompanon, François; Taberlet, Pierre (2014). "DNA metabarcoding and the cytochrome c oxidase subunit I marker: Not a perfect match". Biology Letters. 10 (9). doi:10.1098/rsbl.2014.0562. PMC 4190964. PMID 25209199.
- ^ Dhaliwal, Anandika (2013). "DNA Extraction and Purification". Materials and Methods. 3. doi:10.13070/mm.en.3.191.
- ^ Cooper, A.; Poinar, H. N. (2000). "Ancient DNA: Do It Right or Not at All". Science. 289 (5482): 1139b–1139. doi:10.1126/science.289.5482.1139b. PMID 10970224. S2CID 11030200.
- ^ Willerslev, Eske; Cooper, Alan (2005). "Review Paper. Ancient DNA". Proceedings of the Royal Society B: Biological Sciences. 272 (1558): 3–16. doi:10.1098/rspb.2004.2813. PMC 1634942. PMID 15875564.
- ^ Ficetola, Gentile F.; Pansu, Johan; Bonin, Aurélie; Coissac, Eric; Giguet-Covex, Charline; De Barba, Marta; Gielly, Ludovic; Lopes, Carla M.; Boyer, Frédéric; Pompanon, François; Rayé, Gilles; Taberlet, Pierre (2015). "Replication levels, false presences and the estimation of the presence/Absence from eDNA metabarcoding data". Molecular Ecology Resources. 15 (3): 543–556. doi:10.1111/1755-0998.12338. PMID 25327646. S2CID 24432585.
- ^ Yang, Aimin; Zhang, Wei; Wang, Jiahao; Yang, Ke; Han, Yang; Zhang, Limin (2020). "Review on the Application of Machine Learning Algorithms in the Sequence Data Mining of DNA". Frontiers in Bioengineering and Biotechnology. 8: 1032. doi:10.3389/fbioe.2020.01032. PMC 7498545. PMID 33015010.
- ^ Shendure, Jay; Aiden, Erez Lieberman (2012). "The expanding scope of DNA sequencing". Nature Biotechnology. 30 (11): 1084–1094. doi:10.1038/nbt.2421. PMC 4149750. PMID 23138308.
- ^ Panda, Darshan; Molla, Kutubuddin Ali; Baig, Mirza Jainul; Swain, Alaka; Behera, Deeptirekha; Dash, Manaswini (2018). "DNA as a digital information storage device: Hope or hype?". 3 Biotech. 8 (5): 239. doi:10.1007/s13205-018-1246-7. PMC 5935598. PMID 29744271.
- ^ Ratnasingham, S. (2013). "A DNA-based registry for all animal species: the Barcode Index Number (BIN) system". PLOS ONE. 8 (7): e66213. Bibcode:2013PLoSO...866213R. doi:10.1371/journal.pone.0066213. PMC 3704603. PMID 23861743.
- ^ Nelson, Karen E. (3 January 2015). Encyclopedia of Metagenomics: Genes, Genomes and Metagenomes. Basics, Methods, Databases and Tools. ISBN 9781489974778.
- ^ Abarenkov, Kessy; Henrik Nilsson, R.; Larsson, Karl-Henrik; Alexander, Ian J.; Eberhardt, Ursula; Erland, Susanne; Høiland, Klaus; Kjøller, Rasmus; Larsson, Ellen; Pennanen, Taina; Sen, Robin; Taylor, Andy F. S.; Tedersoo, Leho; Ursing, Björn M.; Vrålstad, Trude; Liimatainen, Kare; Peintner, Ursula; Kõljalg, Urmas (2010). "The UNITE database for molecular identification of fungi - recent updates and future perspectives". New Phytologist. 186 (2): 281–285. doi:10.1111/j.1469-8137.2009.03160.x. PMID 20409185.
- ^ Caporaso, J Gregory; Kuczynski, Justin; Stombaugh, Jesse; Bittinger, Kyle; Bushman, Frederic D.; Costello, Elizabeth K.; Fierer, Noah; Peña, Antonio Gonzalez; Goodrich, Julia K.; Gordon, Jeffrey I.; Huttley, Gavin A.; Kelley, Scott T.; Knights, Dan; Koenig, Jeremy E.; Ley, Ruth E.; Lozupone, Catherine A.; McDonald, Daniel; Muegge, Brian D.; Pirrung, Meg; Reeder, Jens; Sevinsky, Joel R.; Turnbaugh, Peter J.; Walters, William A.; Widmann, Jeremy; Yatsunenko, Tanya; Zaneveld, Jesse; Knight, Rob (2010). "QIIME allows analysis of high-throughput community sequencing data". Nature Methods. 7 (5): 335–336. doi:10.1038/nmeth.f.303. PMC 3156573. PMID 20383131.
- ^ Asnicar, Francesco; Weingart, George; Tickle, Timothy L.; Huttenhower, Curtis; Segata, Nicola (2015). "Compact graphical representation of phylogenetic data and metadata with GraPhl An". PeerJ. 3: e1029. doi:10.7717/peerj.1029. PMC 4476132. PMID 26157614.
- ^ Matsen Iv, Frederick A.; Evans, Steven N. (2013). "Edge Principal Components and Squash Clustering: Using the Special Structure of Phylogenetic Placement Data for Sample Comparison". PLOS ONE. 8 (3): e56859. Bibcode:2013PLoSO...856859M. doi:10.1371/journal.pone.0056859. PMC 3594297. PMID 23505415.
- ^ Darling, Aaron E.; Jospin, Guillaume; Lowe, Eric; Matsen, Frederick A.; Bik, Holly M.; Eisen, Jonathan A. (2014). "Phylo Sift: Phylogenetic analysis of genomes and metagenomes". PeerJ. 2: e243. doi:10.7717/peerj.243. PMC 3897386. PMID 24482762.
- ^ Stoeckle, M. Y. & Hebert, P. D. (2008)엘 코디고 데 바라스 데 라 비다조사관, (387), 42-47
- ^ 뉴마스터 S. G. 등(2007).Myristicaceae의 후보 식물 바코드 영역 테스트.분자생태학주. 1-11.
- ^ Jaén-Molina, R., Caujapé-Castells, J., 페르난데스-Palacios, O., de Paz, J. P., Febles, R., Bramwell, D., ... 및 K.필로게니아 분자 드 라스 마티올레 마카로네시카스 세군 라 인포마시온 데 라 레지온 ITS.
- ^ a b c d e Pornon, 앙드레, Andalo, 크리스토프;Burrus, 이름, Escaravage, 나탈리(2017년)."DNAmetabarcoding 데이터 unveils 보이지 않는 수분 네트워크".과학 보고서. 7(1):16828.Bibcode:2017NatSR...716828P.doi:10.1038/s41598-017-16785-5.PMC5715002.PMID 29203872.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ Biesmeijer, J. C.; Roberts, S. P.; Reemer, M.; Ohlemüller, R.; Edwards, M.; Peeters, T.; Schaffers, A. P.; Potts, S. G.; Kleukers, R.; Thomas, C. D.; Settele, J.; Kunin, W. E. (2006). "Parallel Declines in Pollinators and Insect-Pollinated Plants in Britain and the Netherlands". Science. 313 (5785): 351–354. Bibcode:2006Sci...313..351B. doi:10.1126/science.1127863. PMID 16857940. S2CID 16273738.
- ^ a b Hegland, Stein Joar; Dunne, Jennifer; Nielsen, Anders; Memmott, Jane (2010). "How to monitor ecological communities cost-efficiently: The example of plant–pollinator networks". Biological Conservation. 143 (9): 2092–2101. doi:10.1016/j.biocon.2010.05.018.
- ^ a b Jordano, Pedro (2016). "Chasing Ecological Interactions". PLOS Biology. 14 (9): e1002559. doi:10.1371/journal.pbio.1002559. PMC 5025190. PMID 27631692.
- ^ a b Vázquez, Diego P.; Chacoff, Natacha P.; Cagnolo, Luciano (2009). "Evaluating multiple determinants of the structure of plant–animal mutualistic networks". Ecology. 90 (8): 2039–2046. doi:10.1890/08-1837.1. hdl:11336/20750. PMID 19739366.
- ^ a b Bartomeus, Ignasi (2013). "Understanding Linkage Rules in Plant-Pollinator Networks by Using Hierarchical Models That Incorporate Pollinator Detectability and Plant Traits". PLOS ONE. 8 (7): e69200. Bibcode:2013PLoSO...869200B. doi:10.1371/journal.pone.0069200. PMC 3698228. PMID 23840909.
- ^ a b Blüthgen, Nico; Menzel, Florian; Hovestadt, Thomas; Fiala, Brigitte; Blüthgen, Nils (2007). "Specialization, Constraints, and Conflicting Interests in Mutualistic Networks". Current Biology. 17 (4): 341–346. doi:10.1016/j.cub.2006.12.039. PMID 17275300. S2CID 17241040.
- ^ Bascompte, J.; Jordano, P.; Melian, C. J.; Olesen, J. M. (2003). "The nested assembly of plant-animal mutualistic networks". Proceedings of the National Academy of Sciences. 100 (16): 9383–9387. Bibcode:2003PNAS..100.9383B. doi:10.1073/pnas.1633576100. PMC 170927. PMID 12881488.
- ^ Jordano, Pedro; Bascompte, Jordi; Olesen, Jens M. (2002). "Invariant properties in coevolutionary networks of plant-animal interactions". Ecology Letters. 6: 69–81. doi:10.1046/j.1461-0248.2003.00403.x. hdl:10261/41710.
- ^ Olesen, J. M.; Bascompte, J.; Dupont, Y. L.; Jordano, P. (2007). "The modularity of pollination networks". Proceedings of the National Academy of Sciences. 104 (50): 19891–19896. Bibcode:2007PNAS..10419891O. doi:10.1073/pnas.0706375104. PMC 2148393. PMID 18056808.
- ^ Ollerton, Jeff (1996). "Reconciling Ecological Processes with Phylogenetic Patterns: The Apparent Paradox of Plant--Pollinator Systems". Journal of Ecology. 84 (5): 767–769. doi:10.2307/2261338. JSTOR 2261338.
- ^ Johnson, Steven D.; Steiner, Kim E. (2000). "Generalization versus specialization in plant pollination systems". Trends in Ecology & Evolution. 15 (4): 140–143. doi:10.1016/S0169-5347(99)01811-X. PMID 10717682.
- ^ Waser, Nikolas M.; Chittka, Lars; Price, Mary V.; Williams, Neal M.; Ollerton, Jeff (1996). "Generalization in Pollination Systems, and Why it Matters". Ecology. 77 (4): 1043–1060. doi:10.2307/2265575. JSTOR 2265575.
- ^ Fenster, Charles B.; Armbruster, W. Scott; Wilson, Paul; Dudash, Michele R.; Thomson, James D. (2004). "Pollination Syndromes and Floral Specialization". Annual Review of Ecology, Evolution, and Systematics. 35: 375–403. doi:10.1146/annurev.ecolsys.34.011802.132347.
- ^ Bosch, Jordi; Martín González, Ana M.; Rodrigo, Anselm; Navarro, David (2009). "Plant-pollinator networks: Adding the pollinator's perspective". Ecology Letters. 12 (5): 409–419. doi:10.1111/j.1461-0248.2009.01296.x. PMID 19379135.
- ^ Widmer, A.; Cozzolino, S.; Pellegrino, G.; Soliva, M.; Dafni, A. (2000). "Molecular analysis of orchid pollinaria and pollinaria-remains found on insects". Molecular Ecology. 9 (11): 1911–1914. doi:10.1046/j.1365-294x.2000.01103.x. PMID 11091327. S2CID 13242534.
- ^ Petanidou, T. & Potts, S. G. (2006)"지중해 식물-화분자 공동체에서의 자원 상호 사용:수분 웹은 얼마나 전문화되어 있습니까?인: Waser N. M., Olerton J. (eds) Plant-Polinator 상호작용, 전문화에서 일반화까지, 시카고 대학 출판부, 221~244쪽.
- ^ Gómez, José M.; Bosch, Jordi; Perfectti, Francisco; Fernández, Juande; Abdelaziz, Mohamed (2007). "Pollinator diversity affects plant reproduction and recruitment: The tradeoffs of generalization". Oecologia. 153 (3): 597–605. Bibcode:2007Oecol.153..597G. doi:10.1007/s00442-007-0758-3. PMID 17576602. S2CID 4219676.
- ^ Vázquez, Diego P.; Aizen, Marcelo A. (2003). "Null Model Analyses of Specialization in Plant–Pollinator Interactions". Ecology. 84 (9): 2493–2501. doi:10.1890/02-0587.
- ^ a b Dupont, Yoko L.; Trøjelsgaard, Kristian; Hagen, Melanie; Henriksen, Marie V.; Olesen, Jens M.; Pedersen, Nanna M. E.; Kissling, W. Daniel (2014). "Spatial structure of an individual-based plant-pollinator network" (PDF). Oikos. 123 (11): 1301–1310. doi:10.1111/oik.01426.
- ^ a b King, Caroline; Ballantyne, Gavin; Willmer, Pat G. (2013). "Why flower visitation is a poor proxy for pollination: Measuring single-visit pollen deposition, with implications for pollination networks and conservation". Methods in Ecology and Evolution. 4 (9): 811–818. doi:10.1111/2041-210X.12074.
- ^ a b Lopezaraiza–Mikel, Martha E.; Hayes, Richard B.; Whalley, Martin R.; Memmott, Jane (2007). "The impact of an alien plant on a native plant–pollinator network: An experimental approach". Ecology Letters. 10 (7): 539–550. doi:10.1111/j.1461-0248.2007.01055.x. PMID 17542933.
- ^ a b c d e f 뿌려라, Ahmadou. 하란, 쥘리앵;브노와는, Laure, 갈란, 막심, Brévault, 티에리(2020년)."DNAMetabarcoding 도구 Disentangling 식품 웨브에 Agroecosystems에".곤충들은. 11(5):294.doi:10.3390/insects11050294.PMC7290477.PMID 32403224.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ Galan, Maxime; Pons, Jean-Baptiste; Tournayre, Orianne; Pierre, Éric; Leuchtmann, Maxime; Pontier, Dominique; Charbonnel, Nathalie (2018). "Metabarcoding for the parallel identification of several hundred predators and their prey: Application to bat species diet analysis". Molecular Ecology Resources. 18 (3): 474–489. doi:10.1111/1755-0998.12749. PMID 29288544. S2CID 1004450.
- ^ a b Paula, Débora P.; Linard, Benjamin; Crampton-Platt, Alex; Srivathsan, Amrita; Timmermans, Martijn J. T. N.; Sujii, Edison R.; Pires, Carmen S. S.; Souza, Lucas M.; Andow, David A.; Vogler, Alfried P. (2016). "Uncovering Trophic Interactions in Arthropod Predators through DNA Shotgun-Sequencing of Gut Contents". PLOS ONE. 11 (9): e0161841. Bibcode:2016PLoSO..1161841P. doi:10.1371/journal.pone.0161841. PMC 5021305. PMID 27622637.
- ^ Firlej, Annabelle; Doyon, Josée; Harwood, James D.; Brodeur, Jacques (2013). "A Multi-Approach Study to Delineate Interactions Between Carabid Beetles and Soybean Aphids". Environmental Entomology. 42 (1): 89–96. doi:10.1603/EN11303. PMID 23339789. S2CID 7903342.
- ^ Kartzinel, Tyler R.; Chen, Patricia A.; Coverdale, Tyler C.; Erickson, David L.; Kress, W. John; Kuzmina, Maria L.; Rubenstein, Daniel I.; Wang, Wei; Pringle, Robert M. (2015). "DNA metabarcoding illuminates dietary niche partitioning by African large herbivores". Proceedings of the National Academy of Sciences. 112 (26): 8019–8024. Bibcode:2015PNAS..112.8019K. doi:10.1073/pnas.1503283112. PMC 4491742. PMID 26034267.
- ^ Lopes, C. M.; De Barba, M.; Boyer, F.; Mercier, C.; Da Silva Filho, P J S.; Heidtmann, L. M.; Galiano, D.; Kubiak, B. B.; Langone, P.; Garcias, F. M.; Gielly, L.; Coissac, E.; De Freitas, T R O.; Taberlet, P. (2015). "DNA metabarcoding diet analysis for species with parapatric vs sympatric distribution: A case study on subterranean rodents". Heredity. 114 (5): 525–536. doi:10.1038/hdy.2014.109. PMC 4815513. PMID 25649502. S2CID 5077455.
- ^ Mollot, Gregory; Duyck, Pierre-François; Lefeuvre, Pierre; Lescourret, Françoise; Martin, Jean-François; Piry, Sylvain; Canard, Elsa; Tixier, Philippe (2014). "Cover Cropping Alters the Diet of Arthropods in a Banana Plantation: A Metabarcoding Approach". PLOS ONE. 9 (4): e93740. Bibcode:2014PLoSO...993740M. doi:10.1371/journal.pone.0093740. PMC 3973587. PMID 24695585.
- ^ Greenstone, Matthew H.; Payton, Mark E.; Weber, Donald C.; Simmons, Alvin M. (2014). "The detectability half-life in arthropod predator-prey research: What it is, why we need it, how to measure it, and how to use it". Molecular Ecology. 23 (15): 3799–3813. doi:10.1111/mec.12552. PMID 24303920. S2CID 41032815.
- ^ Symondson, William O. C.; Harwood, James D. (2014). "Special issue on molecular detection of trophic interactions: Unpicking the tangled bank" (PDF). Molecular Ecology. 23 (15): 3601–3604. doi:10.1111/mec.12831. PMID 25051891. S2CID 26082315.
- ^ Valentini, Alice; Pompanon, François; Taberlet, Pierre (2009). "DNA barcoding for ecologists". Trends in Ecology & Evolution. 24 (2): 110–117. doi:10.1016/j.tree.2008.09.011. PMID 19100655.
- ^ Begg, Graham S.; Cook, Samantha M.; Dye, Richard; Ferrante, Marco; Franck, Pierre; Lavigne, Claire; Lövei, Gábor L.; Mansion-Vaquie, Agathe; Pell, Judith K.; Petit, Sandrine; Quesada, Nora; Ricci, Benoit; Wratten, Stephen D.; Birch, A.Nicholas E. (2017). "A functional overview of conservation biological control". Crop Protection. 97: 145–158. doi:10.1016/j.cropro.2016.11.008.
- ^ Gurr, G.M.; Van Emden, H.F.; Wratten, S.D. (1998). "Habitat manipulation and natural enemy efficiency". Conservation Biological Control. pp. 155–183. doi:10.1016/B978-012078147-8/50055-4. ISBN 9780120781478.
- ^ Wyckhuys, Kris A.G.; Lu, Yanhui; Morales, Helda; Vazquez, Luis L.; Legaspi, Jesusa C.; Eliopoulos, Panagiotis A.; Hernandez, Luis M. (2013). "Current status and potential of conservation biological control for agriculture in the developing world". Biological Control. 65: 152–167. doi:10.1016/j.biocontrol.2012.11.010.
- ^ a b Symondson, W. O. C.; Sunderland, K. D.; Greenstone, M. H. (2002). "Can Generalist Predators be Effective Biocontrol Agents?". Annual Review of Entomology. 47: 561–594. doi:10.1146/annurev.ento.47.091201.145240. PMID 11729085.
- ^ Harwood, J. D.와 Obrycki, J. J.(2005).포식자 집단을 지탱하는 대체 먹잇감의 역할.인: M. S. Hoddle (Ed.) 절지동물의 생물학적 방제에 관한 제2차 국제 심포지엄, 제2권, 453-462쪽.
- ^ Harwood, James D.; Desneux, Nicolas; Yoo, HO Jung S.; Rowley, Daniel L.; Greenstone, Matthew H.; Obrycki, John J.; o′Neil, Robert J. (2007). "Tracking the role of alternative prey in soybean aphid predation by Orius insidiosus: A molecular approach". Molecular Ecology. 16 (20): 4390–4400. doi:10.1111/j.1365-294X.2007.03482.x. PMID 17784913. S2CID 21211301.
- ^ a b c Furlong, Michael J. (2015). "Knowing your enemies: Integrating molecular and ecological methods to assess the impact of arthropod predators on crop pests". Insect Science. 22 (1): 6–19. doi:10.1111/1744-7917.12157. PMID 25081301. S2CID 27851198.
- ^ Jonsson, Mattias; Wratten, Steve D.; Landis, Doug A.; Gurr, Geoff M. (2008). "Recent advances in conservation biological control of arthropods by arthropods". Biological Control. 45 (2): 172–175. doi:10.1016/j.biocontrol.2008.01.006.
- ^ Pompanon, Francois; Deagle, Bruce E.; Symondson, William O. C.; Brown, David S.; Jarman, Simon N.; Taberlet, Pierre (2012). "Who is eating what: Diet assessment using next generation sequencing". Molecular Ecology. 21 (8): 1931–1950. doi:10.1111/j.1365-294X.2011.05403.x. PMID 22171763. S2CID 10013333.
- ^ Waldner, Thomas; Traugott, Michael (2012). "DNA‐based analysis of regurgitates: A noninvasive approach to examine the diet of invertebrate consumers". Molecular Ecology Resources. 12 (4): 669–675. doi:10.1111/j.1755-0998.2012.03135.x. PMID 22443278. S2CID 21254959.
- ^ Bohmann, Kristine; Monadjem, Ara; Lehmkuhl Noer, Christina; Rasmussen, Morten; Zeale, Matt R. K.; Clare, Elizabeth; Jones, Gareth; Willerslev, Eske; Gilbert, M. Thomas P. (2011). "Molecular Diet Analysis of Two African Free-Tailed Bats (Molossidae) Using High Throughput Sequencing". PLOS ONE. 6 (6): e21441. Bibcode:2011PLoSO...621441B. doi:10.1371/journal.pone.0021441. PMC 3120876. PMID 21731749.
- ^ a b c d e 포천, 사비에르;Zaiko, Anastasija, 플레처, 로렌 M.;Laroche, 올리비에, 우드, 수잔나 A(2017년)." 되길 원했다 사망 선고를 받았거나 살아 있나요?biosecurity applications"의 생활 무더기를 구별하기 위해 환경 DNA과 RNAmetabarcoding이 사용됩니다.PLOS다. 12(11):e0187636.Bibcode:2017년PLoSO..1287636P. doi:10.1371/journal.pone.0187636.PMC 5667844.PMID 29095959.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ a b Molnar, Jennifer L.; Gamboa, Rebecca L.; Revenga, Carmen; Spalding, Mark D. (2008). "Assessing the global threat of invasive species to marine biodiversity". Frontiers in Ecology and the Environment. 6 (9): 485–492. doi:10.1890/070064.
- ^ Olenin, Sergej; Minchin, Dan; Daunys, Darius (2007). "Assessment of biopollution in aquatic ecosystems". Marine Pollution Bulletin. 55 (7–9): 379–394. doi:10.1016/j.marpolbul.2007.01.010. PMID 17335857.
- ^ Katsanevakis, Stelios; Wallentinus, Inger; Zenetos, Argyro; Leppäkoski, Erkki; Çinar, Melih Ertan; Oztürk, Bayram; Grabowski, Michal; Golani, Daniel; Cardoso, Ana Cristina (2014). "Impacts of invasive alien marine species on ecosystem services and biodiversity: A pan-European review". Aquatic Invasions. 9 (4): 391–423. doi:10.3391/ai.2014.9.4.01.
- ^ Pimentel, David; Zuniga, Rodolfo; Morrison, Doug (2005). "Update on the environmental and economic costs associated with alien-invasive species in the United States". Ecological Economics. 52 (3): 273–288. doi:10.1016/j.ecolecon.2004.10.002.
- ^ a b Harvey, Chad T.; Qureshi, Samir A.; MacIsaac, Hugh J. (2009). "Detection of a colonizing, aquatic, non-indigenous species". Diversity and Distributions. 15 (3): 429–437. doi:10.1111/j.1472-4642.2008.00550.x. S2CID 53602450.
- ^ Leppäkoski, Erkki; Gollasch, Stephan; Olenin, Sergej (29 June 2013). Invasive Aquatic Species of Europe. Distribution, Impacts and Management. ISBN 9789401599566.
- ^ Hulme, Philip E. (2009). "Trade, transport and trouble: Managing invasive species pathways in an era of globalization". Journal of Applied Ecology. 46: 10–18. doi:10.1111/j.1365-2664.2008.01600.x.
- ^ Inglis G, Floerl O, Ahyong S, Cox S, Unwin M, Ponder-Sutton A, et al. (2010) "뉴질랜드에 도착하는 국제 선박의 바이오 파울링과 관련된 생물 보안 위험: 파울링 패턴의 요약 및 예측 변수"바이오시큐리티 뉴질랜드, 테크니컬 리포트 No: 2008.
- ^ Ruiz, Gregory M.; Rawlings, Tonya K.; Dobbs, Fred C.; Drake, Lisa A.; Mullady, Timothy; Huq, Anwarul; Colwell, Rita R. (2000). "Global spread of microorganisms by ships". Nature. 408 (6808): 49–50. Bibcode:2000Natur.408...49R. doi:10.1038/35040695. PMID 11081499. S2CID 205010602.
- ^ Gollasch, S. (2002). "The Importance of Ship Hull Fouling as a Vector of Species Introductions into the North Sea". Biofouling. 18 (2): 105–121. doi:10.1080/08927010290011361. S2CID 85620063.
- ^ Mineur, Frédéric; Johnson, Mark P.; Maggs, Christine A. (2008). "Macroalgal Introductions by Hull Fouling on Recreational Vessels: Seaweeds and Sailors". Environmental Management. 42 (4): 667–676. Bibcode:2008EnMan..42..667M. doi:10.1007/s00267-008-9185-4. PMID 18704562. S2CID 25680517.
- ^ Johnson, Ladd E.; Ricciardi, Anthony; Carlton, James T. (2001). "Overland Dispersal of Aquatic Invasive Species: A Risk Assessment of Transient Recreational Boating". Ecological Applications. 11 (6): 1789. doi:10.1890/1051-0761(2001)011[1789:ODOAIS]2.0.CO;2. ISSN 1051-0761.
- ^ Darbyson, Emily (2009). "Marine boating habits and the potential for spread of invasive species in the Gulf of St. Lawrence". Aquatic Invasions. 4: 87–94. doi:10.3391/ai.2009.4.1.9.
- ^ Acosta, Hernando; Forrest, Barrie M. (2009). "The spread of marine non-indigenous species via recreational boating: A conceptual model for risk assessment based on fault tree analysis". Ecological Modelling. 220 (13–14): 1586–1598. doi:10.1016/j.ecolmodel.2009.03.026.
- ^ McMahon, Robert (2011). "Quagga mussel (Dreissena rostriformis bugensis) population structure during the early invasion of Lakes Mead and Mohave January-March 2007". Aquatic Invasions. 6 (2): 131–140. doi:10.3391/ai.2011.6.2.02.
- ^ a b Fletcher, Lauren M.; Zaiko, Anastasija; Atalah, Javier; Richter, Ingrid; Dufour, Celine M.; Pochon, Xavier; Wood, Susana A.; Hopkins, Grant A. (2017). "Bilge water as a vector for the spread of marine pests: A morphological, metabarcoding and experimental assessment". Biological Invasions. 19 (10): 2851–2867. doi:10.1007/s10530-017-1489-y. S2CID 25513146.
- ^ a b Sainer, Jim, Barrie Forrest, Mark Newton, Graeme Inglis, Chris Woods, Don Morrisey, Aurélie Castinel 및 Rowan Strickland(2013) "유해 해양 생물 국내 확산 관리" Part B: 법적 프레임워크 및 분석 옵션.코프론 연구소
- ^ Inglis G, Morrisey D, Woods C, Cain J, Newton M. (2013) "유해 해양생물의 국내 확산 관리", 파트 A: 관리를 위한 운영 도구.뉴질랜드 1차 산업부, 보고서 No. 2013/xx.
- ^ Comtet, Thierry; Sandionigi, Anna; Viard, Frédérique; Casiraghi, Maurizio (2015). "DNA (Meta)barcoding of biological invasions: A powerful tool to elucidate invasion processes and help managing aliens". Biological Invasions. 17 (3): 905–922. doi:10.1007/s10530-015-0854-y. S2CID 12799342.
- ^ Zaiko, Anastasija; Schimanski, Kate; Pochon, Xavier; Hopkins, Grant A.; Goldstien, Sharyn; Floerl, Oliver; Wood, Susanna A. (2016). "Metabarcoding improves detection of eukaryotes from early biofouling communities: Implications for pest monitoring and pathway management". Biofouling. 32 (6): 671–684. doi:10.1080/08927014.2016.1186165. PMID 27212415. S2CID 46842367.
- ^ Brown, Emily A.; Chain, Frédéric J. J.; Zhan, Aibin; MacIsaac, Hugh J.; Cristescu, Melania E. (2016). "Early detection of aquatic invaders using metabarcoding reveals a high number of non-indigenous species in Canadian ports". Diversity and Distributions. 22 (10): 1045–1059. doi:10.1111/ddi.12465.
- ^ Zaiko, Anastasija; Martinez, Jose L.; Schmidt-Petersen, Julia; Ribicic, Deni; Samuiloviene, Aurelija; Garcia-Vazquez, Eva (2015). "Metabarcoding approach for the ballast water surveillance – an advantageous solution or an awkward challenge?". Marine Pollution Bulletin. 92 (1–2): 25–34. doi:10.1016/j.marpolbul.2015.01.008. PMID 25627196.
- ^ Shokralla, Shadi; Spall, Jennifer L.; Gibson, Joel F.; Hajibabaei, Mehrdad (2012). "Next-generation sequencing technologies for environmental DNA research". Molecular Ecology. 21 (8): 1794–1805. doi:10.1111/j.1365-294X.2012.05538.x. PMID 22486820. S2CID 5944083.
- ^ Ji, Yinqiu; Ashton, Louise; Pedley, Scott M.; Edwards, David P.; Tang, Yong; Nakamura, Akihiro; Kitching, Roger; Dolman, Paul M.; Woodcock, Paul; Edwards, Felicity A.; Larsen, Trond H.; Hsu, Wayne W.; Benedick, Suzan; Hamer, Keith C.; Wilcove, David S.; Bruce, Catharine; Wang, Xiaoyang; Levi, Taal; Lott, Martin; Emerson, Brent C.; Yu, Douglas W. (2013). "Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding". Ecology Letters. 16 (10): 1245–1257. doi:10.1111/ele.12162. PMID 23910579.
- ^ Darling, John A.; Frederick, Raymond M. (2018). "Nucleic acids-based tools for ballast water surveillance, monitoring, and research". Journal of Sea Research. 133: 43–52. Bibcode:2018JSR...133...43D. doi:10.1016/j.seares.2017.02.005. PMC 6104837. PMID 30147432.
- ^ Corinaldesi, C.; Beolchini, F.; Dell'Anno, A. (2008). "Damage and degradation rates of extracellular DNA in marine sediments: Implications for the preservation of gene sequences". Molecular Ecology. 17 (17): 3939–3951. doi:10.1111/j.1365-294X.2008.03880.x. PMID 18643876. S2CID 22062643.
- ^ Dell'Anno, A.; Danovaro, R. (2005). "Extracellular DNA Plays a Key Role in Deep-Sea Ecosystem Functioning". Science. 309 (5744): 2179. doi:10.1126/science.1117475. PMID 16195451. S2CID 39216262.
- ^ Mengoni, Alessio; Tatti, Enrico; Decorosi, Francesca; Viti, Carlo; Bazzicalupo, Marco; Giovannetti, Luciana (2005). "Comparison of 16S rRNA and 16S rDNA T-RFLP Approaches to Study Bacterial Communities in Soil Microcosms Treated with Chromate as Perturbing Agent". Microbial Ecology. 50 (3): 375–384. doi:10.1007/s00248-004-0222-4. PMID 16254761. S2CID 23943691.
- ^ Pawlowski, Jan; Esling, Philippe; Lejzerowicz, Franck; Cedhagen, Tomas; Wilding, Thomas A. (2014). "Environmental monitoring through protist next-generation sequencing metabarcoding: Assessing the impact of fish farming on benthic foraminifera communities". Molecular Ecology Resources. 14 (6): 1129–1140. doi:10.1111/1755-0998.12261. PMID 24734911. S2CID 2303206.
- ^ Visco, Joana Amorim; Apothéloz-Perret-Gentil, Laure; Cordonier, Arielle; Esling, Philippe; Pillet, Loïc; Pawlowski, Jan (2015). "Environmental Monitoring: Inferring the Diatom Index from Next-Generation Sequencing Data". Environmental Science & Technology. 49 (13): 7597–7605. Bibcode:2015EnST...49.7597V. doi:10.1021/es506158m. PMID 26052741.
- ^ Dowle, Eddy; Pochon, Xavier; Keeley, Nigel; Wood, Susanna A. (2015). "Assessing the effects of salmon farming seabed enrichment using bacterial community diversity and high-throughput sequencing". FEMS Microbiology Ecology. 91 (8): fiv089. doi:10.1093/femsec/fiv089. PMID 26207046.
- ^ Pochon, X.; Wood, S.A.; Keeley, N.B.; Lejzerowicz, F.; Esling, P.; Drew, J.; Pawlowski, J. (2015). "Accurate assessment of the impact of salmon farming on benthic sediment enrichment using foraminiferal metabarcoding". Marine Pollution Bulletin. 100 (1): 370–382. doi:10.1016/j.marpolbul.2015.08.022. PMID 26337228.
- ^ Laroche, Olivier; Wood, Susanna A.; Tremblay, Louis A.; Ellis, Joanne I.; Lejzerowicz, Franck; Pawlowski, Jan; Lear, Gavin; Atalah, Javier; Pochon, Xavier (2016). "First evaluation of foraminiferal metabarcoding for monitoring environmental impact from an offshore oil drilling site". Marine Environmental Research. 120: 225–235. doi:10.1016/j.marenvres.2016.08.009. PMID 27595900.
- ^ 형태학적, 유전적 방법에 의한 애벌레의 Cazarez Carrillo, D. E. Descripcion de la levar de "Eucinostomus jonesi"(피시스, Gerreidae)
- ^ Kerr, K. C., Lijtmaer, D. A. A. S., Hebert, P. D. 및 Tubaro, P. L. (2009)DNA 바코드를 통해 신열대 조류의 진화 패턴을 조사합니다.PLOS ONE, 4(2), e4379.
- ^ Lijtmaer, D. A., K. C., Barreira, A. S., Hebert, P. D. 및 Tubaro, P. L. (2011년)DNA 바코드 라이브러리는 조류 다양화의 대륙적 패턴에 대한 통찰력을 제공합니다.PLOS ONE, 6(7), e20744.
- ^ Lijtmaer, D. A., Ker, K. C., Stoeckle, M. Y. 및 Tubaro, P. L.(2012).DNA 바코드 버드: 필드 수집부터 데이터 분석까지.DNA 바코드 (p. 127-152).휴마나 프레스
- ^ 가르시아 모랄레스, A.E. (2013)코디고 데 바라스 이 아나리스 필로게오그라피코 데 로티페로스 델 수레스테 멕시카노.
- ^ 데 카르발류, 다니엘 카르도소, 세실리아 곤티조 릴, 파울로 도스 산토스 폼페우, 호세 반데르발 멜로 주니어, 데니스 A.A. de Oliveira (2011)Aplicaess da técnica de identificassao genética - DNA 바코드 - nos peixes da bacia do Rio So Francisco.Boletim Societade Brasileira de Ictiologia N°104.모폴로지아 학부, 비오첸시아 연구소, 보투카투, 상파울루, 브라질.브라질.
- ^ 데 카르발류, 다니엘 카르도소, 파울로 도스 산토스 폼페우, 세실리아 곤티조 릴, 데니스 A.A. de Oliveira & Hanner, R. (2011년)브라질 민물고기의 딥 바코드 확산:So Francisco River Basin의 경우.미토콘드리아 DNA, 2011년
- ^ 에르난데스, 에스메랄다 살가도(2008).El codigo de Barras gen icto (DNA 바코딩) como herramienta en la identifacion de es 종.헤레리아나.레뉴가시온 데 라 시엔시아의 레비스타.제4권 (1)
- ^ Hebert P.와 G. Ryan(2005).DNA 바코딩이 택사노미에게 주는 약속입니다.계통생물학. 54(5): 852-859.
- ^ 홀링스워스, P. (2007)DNA 바코드: 잠재적인 사용자.유전체학, 사회, 정책. 3(2): 44-47.
- ^ "What Is DNA Barcoding?". Barcode of Life. Archived from the original on 1 July 2017. Retrieved 11 October 2017.
- ^ Evans, Darren M.; Kitson, James J. N.; Lunt, David H.; Straw, Nigel A.; Pocock, Michael J. O. (2016). "Merging DNA metabarcoding and ecological network analysis to understand and build resilient terrestrial ecosystems" (PDF). Functional Ecology. 30 (12): 1904–1916. doi:10.1111/1365-2435.12659. ISSN 1365-2435.
- ^ https://dnaqua.net/
- ^ Pawlowski, Jan; Kelly-Quinn, Mary; Altermatt, Florian; Apothéloz-Perret-Gentil, Laure; Beja, Pedro; Boggero, Angela; Borja, Angel; Bouchez, Agnès; Cordier, Tristan (2018). "The future of biotic indices in the ecogenomic era: Integrating (e)DNA metabarcoding in biological assessment of aquatic ecosystems". Science of the Total Environment. 637–638: 1295–1310. Bibcode:2018ScTEn.637.1295P. doi:10.1016/j.scitotenv.2018.05.002. PMID 29801222.
- ^ Ratnasingham, Sujeevan; Hebert, Paul D. N. (2007). "BOLD: The Barcode of Life Data System (http://www.barcodinglife.org)". Molecular Ecology Notes. 7 (3): 355–364. doi:10.1111/j.1471-8286.2007.01678.x. PMC 1890991. PMID 18784790.
- ^ Stoeckle, Mark (November–December 2013). "DNA Barcoding Ready for Breakout". GeneWatch. 26 (5).
- ^ DeSalle, R.; Egan, M. G.; Siddall, M. (2005). "The unholy trinity: taxonomy, species delimitation and DNA barcoding". Philosophical Transactions of the Royal Society B: Biological Sciences. 360 (1462): 1905–1916. doi:10.1098/rstb.2005.1722. PMC 1609226. PMID 16214748.
- ^ Whitworth, T. L.; Dawson, R. D.; Magalon, H.; Baudry, E. (2007). "DNA barcoding cannot reliably identify species of the blowfly genus Protocalliphora (Diptera: Calliphoridae)". Proceedings of the Royal Society B: Biological Sciences. 274 (1619): 1731–1739. doi:10.1098/rspb.2007.0062. PMC 2493573. PMID 17472911.
- ^ Stackebrandt, E.; Goebel, B. M. (1994). "Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology". Int. J. Syst. Bacteriol. 44 (4): 846–849. doi:10.1099/00207713-44-4-846.
- ^ Stackebrandt, E.; Ebers, J. (2006). "Taxonomic parameters revisited: tarnished gold standards" (PDF). Microbiology Today. 33 (4): 152–155. Archived (PDF) from the original on 25 March 2018.
- ^ Newton, Ian (2003). Speciation and Biogeography of Birds. Academic Press. p. 69. ISBN 978-0-08-092499-1. Archived from the original on 7 February 2018.
- ^ Andersson, Malte (1999). Hybridization and skua phylogeny. Proceedings of the Royal Society B. Vol. 266. pp. 1579–1585. doi:10.1098/rspb.1999.0818. ISBN 9780080924991. PMC 1690163. Archived from the original on 7 February 2018.
- ^ Keswani, J.; Whitman, W. B. (2001). "Relationship of 16S rRNA sequence similarity to DNA hybridization in prokaryotes". International Journal of Systematic and Evolutionary Microbiology. 51 (2): 667–78. doi:10.1099/00207713-51-2-667. PMID 11321113.
기타 참고 자료
- ^ Santoferrara, Luciana; Burki, Fabien; Filker, Sabine; Logares, Ramiro; Dunthorn, Micah; McManus, George B. (2020). "Perspectives from Ten Years of Protist Studies by High‐Throughput Metabarcoding". Journal of Eukaryotic Microbiology. 67 (5): 612–622. doi:10.1111/jeu.12813. hdl:10261/223228. PMID 32498124. S2CID 219331807.