조류 DNA 바코드
Algae DNA barcoding| 다음에 대한 시리즈 일부 |
| DNA 바코드 |
|---|
 |
| by taxa |
| 기타 |
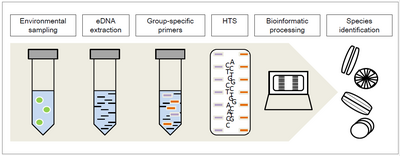
조류에 대한 DNA 바코드는 종 식별과 유전학 연구에 일반적으로 사용된다. 조류는 유전학적으로 이질적인 그룹을 형성하는데, 이는 종 구분용 단일 범용 바코드/마커의 적용이 불가능하기 때문에 다른 조류 그룹에서 이 목적을 위해 서로 다른 표식기/바코드를 적용한다는 것을 의미한다.
디아톰스
디아톰 DNA 바코딩은 종 수준까지 디아톰을 분류학적으로 식별하는 방법이다. 그것은 DNA나 RNA를 이용하여 행해지고, 그 다음, 규소 게놈의 특정 보존 부위의 증폭과 염기서열화에 이어 분류학 배정이 이루어진다.
규조류를 식별하는 데 있어 가장 큰 난제 중 하나는 그것이 여러 종의 규조류를 혼합하여 수집되는 경우가 많다는 것이다. DNA 메타코딩은 토양이나 물 샘플과 같은 환경에서 바로 추출한 DNA인 환경 DNA의 혼합 샘플(eDNA라고도 함)에서 개별 종을 식별하는 과정이다.
새롭게 적용된 방법은 특정 생태학적 조건에 대한 규조류의 구체적인 반응 때문에 하천과 하천에 대한 생태학적 품질 평가에 사용되는 규조류 DNA 메타코딩이다. 형태학을 통한 종 식별은 상대적으로 어렵고 많은 시간과 전문지식이 필요하기 때문에 고투과 염기서열(HTS) DNA 메타코딩은 이전의 DNA 증폭을 위해 선택된 그룹별 프라이머에 대한 분류학적 할당과 완전한 샘플 식별을 가능하게 한다.[1][2]
지금까지 이미 여러 개의 DNA 표지가 개발되었으며, 주로 18S rRNA를 목표로 하고 있다.[3] 리보솜 소단위 DNA(SSU rDNA)의 V4 고변량 영역을 사용하여 DNA 기반 식별이 고전적인 형태학 기반 접근법보다 더 효율적인 것으로 밝혀졌다.[4] 표식 유전자로 자주 사용되는 게놈의 보존 영역으로는 리불로스-1-5-비스포산염 카르복실라제(rbcL), 시토크롬 산화효소 I(cox1, COI),[5] ITS[6], 28S 등이 있다.[7] diatom eDNA 메타카코딩으로 얻은 분자 데이터가 형태학 기반의 생물학적 이원자 지수를 상당히 충실하게 반영해 생태계 상태에 대한 유사한 평가를 제공한다는 사실이 반복적으로 드러났다.[8][9] 그 동안, 규조류는 다른 담수 생태계의 생태적 품질 평가를 위해 일상적으로 사용된다.[7] 수생 무척추동물과 함께 그들은 수류의 물리적, 화학적 또는 생물학적 조건과 관련된 교란의 가장 좋은 지표로 여겨진다. 많은 연구들이 생체모미토리엄을 위해 벤트히크 디아톰을 사용하고 있다.[10][11][12][13] 이상적인 diatom DNA 바코드가 발견되지 않았기 때문에, 다른 목적으로 다른 마커를 사용하는 것이 제안되었다. 실제로, 고도로 가변적인 cox1, ITS, 28S 유전자는 분류학 연구에 더 적합하다고 여겨졌고, 보존이 완료된 18S와 rbcL 유전자는 생물역학 연구에 더 적합해 보였다.
이점
디엔에이 바코딩 개념을 디아톰에 적용하면 부정확한 종 식별 문제를 해결하고 따라서 환경 샘플의 생물다양성 분석을 용이하게 할 수 있는 큰 잠재력을 약속한다.[14]
NGS 기술에 기반한 분자 방법은 거의 항상 더 많은 수의 식별된 세사로 이어지며, 그 존재는 나중에 가벼운 현미경으로 검증될 수 있다.[4] 이 연구의 결과는 규조류의 바코딩이 수질 평가에 적합하고 전통적인 방법을 보완하거나 개선할 수 있다는 증거를 제공한다. Stoeck 외 연구진은 또한 eDNA 바코딩이 규조 다양성 또는 다른 원자 공동체에 대한 더 많은 통찰력을 제공하므로 지구 다양성의 생태학적 투영에 사용될 수 있다는 것을 보여주었다.[15] 다른 연구들은 다른 결과를 보여주었다. 예를 들어, 분자 기반 방법에서 얻은 재고는 풍부한 종들이 집중되어 있을 때 형태학 기반 방법으로 얻은 재고에 더 가까웠다.[5]
DNA 메타코딩은 또한 형태학적 문자만을 사용하는 것이 어려운 지리적 영역 간 분류학적 분해능과 비교 가능성을 증가시킬 수 있다. 더욱이 DNA 기반 식별은 특정 스트레스 요인에 매우 민감하거나 내성적일 수 있는 눈에 띄지 않는 분류학 그룹을 포함하여 잠재적인 생물학적 인디케이터의 범위를 확장할 수 있다. 간접적으로, 분자법은 또한 처리 시간의 감소(가성비)와 함께 처리된 샘플의 수를 증가시킴과 동시에 종/MOTU 발생과 환경 요인 간의 상관관계의 정확성과 정밀도를 증가시킴으로써 종 생태학의 지식 격차를 메우는 데 도움을 줄 수 있다.[16]
과제들
현재 DNA 보존과 격리를 위한 방법, DNA 바코드 및 PCR 프라이머의 선택, MOTU 클러스터링의 매개변수 및 분류학적 할당에 관한 합의는 없다.[16] 샘플링과 분자 단계는 개발 연구를 통해 표준화할 필요가 있다.[5] 주요 제한사항 중 하나는 규조류 종에 대한 기준 바코드의 가용성이다. 많은 종들이 여전히 바코드 정보가 부족한 수많은 국가 바코드화 이니셔티브들의 끊임없는 노력에도 불구하고 생물인디케이터 택사의 참조 데이터베이스는 완성도와는 거리가 멀다. 더욱이 기존의 메타바코딩 데이터는 대부분 국지적으로만 이용할 수 있고 지리적으로 분산되어 있어 세계적으로 유용한 도구의 개발을 방해하고 있다.[16] 비스코 외 연구진은 현재 유럽 규조류 종의 30% 이하가 참조 데이터베이스에 표현되어 있다고 추정했다.[17] 예를 들어, 페노칸디안 공동체(특히 은오티아 잉시사와 같은 산호필성 규조류)의 여러 종에 대한 중요한 부족이 있다. DNA 바코드를 이용한 분류학적 식별이 종 수준 이상에서 정확하지 않은 것으로 나타나, 예를 들어 품종을 구별하는 것(참조 누락)도 나타났다.
분류학 식별을 위한 바코딩의 또 다른 잘 알려진 제한은 분류학 할당 전에 사용되는 클러스터링 방법이다. 그것은 종종 유전 정보의 엄청난 손실을 초래하고 다른 클러스터링과 다른 분류법 할당 과정의 영향을 평가하는 신뢰할 수 있는 유일한 방법은 동일한 참조 데이터베이스를 사용할 때 다른 파이프라인에서 생성된 종 목록을 비교하는 것이다. 이것은 유럽 내 이원자 공동체의 분자 평가에 사용되는 다양한 파이프라인에 대해 아직 수행되지 않았다.[16] 액세스 가능한 쿠폰을 포함하는 분류학적으로 검증된 데이터베이스는 NGS를 통한 신뢰할 수 있는 세금 식별에도 매우 중요하다.[18]
또한 프라이머 편향은 바코딩의 주요 변동의 원천인 경우가 많고 PCR 프라이머 효율은 디아톰 종마다 다를 수 있다. 즉, 일부 프라이머는 다른 프라이머에 비해 한 택스톤을 우선 증폭시킨다.[16]
메타스코딩 데이터로부터의 풍요에 대한 추론은 환경 이용에서 가장 어려운 문제 중 하나로 간주된다.[19][20] HTS에 의해 생성된 시퀀스 수는 시료나 바이오매스의 수와 직접적으로 일치하지 않으며, 다른 종들이 다른 양의 읽기를 생성할 수 있다(예를 들어, rbcL 마커와 엽록체 크기의 차이로 인해). 바셀론 등은 최근 rbcL 마커를 사용할 때 바이오 볼륨 보정 계수를 만들었다.[21] 예를 들어, Achanantidium minutissimum은 작은 생체량을 가지기 때문에 더 큰 종에 비해 rbcL 파편(엽록체 내에 위치)을 더 적게 생성하게 된다. 그러나 이 보정 계수는 각 종 자체의 생체 부피에 대한 광범위한 교정을 필요로 하며, 지금까지 몇 가지 종에서만 시험되었다. 18S 마커와 같은 다른 마커에 대한 유전자 복사 번호의 변동은 종에 특정한 것으로 보이지는 않지만, 아직 테스트되지 않았다.
Diatom 대상 영역
바코딩 마커는 대개 게놈의 고변량 영역(종간의 구분을 허용하기 위해)과 매우 보존된 영역(대상 유기체에 대한 특수성을 보장하기 위해)을 결합한다. 핵, 미토콘드리아 및 엽록체 게놈(rbcL, COI, ITS+5.8S,[22][23][6] SSU, 18S...)에 속하는 여러 개의 DNA 표지는 NGS와의 규광체 식별에 설계되어 성공적으로 사용되었다.
18S 및 V4 서브 유닛
18S 유전자 영역은 다른 양성자 그룹에서[24][25] 표식자로 널리 사용되어 왔고 얀 [26]외 연구진은 18S 유전자 영역에서 diatoms 바코딩을 시험한 첫 번째였다. 짐머만 외 [7]연구진은 HTS로 환경 샘플 분석을 위한 바코드 마커로 1800bp 길이 18S rRNA 유전자 위치 390–410bp의 긴 파편을 제안하고 이원자 식별에 대한 사용과 한계에 대해 논의한다. 이 파편에는 18S 위치 내에서 가장 크고 가장 복잡한 V4 서브 유닛이 포함된다.[27] 그들은 18S 유전자의 이 고변량 영역이 대규모로 양성자 다양성을 연구할 수 있는 큰 잠재력을 가지고 있지만 종 수준 이하나 암호종 이하로 식별하는 데 효율성이 제한되어 있다고 강조했다.
rbcL
rbcl 유전자는 분류학 연구에 사용된다(Trobajo et al. 2009) 어떤 편익에는 거의 어떠한 유전적 변화도 포함하지 않으며 그것들은 매우 쉽게 조정되고 비교된다. R-Sysst::diatom이라는 개방형 액세스 참조 라이브러리에는 두 개의 바코드(18S 및 rbcL)에 대한 데이터가 포함되어 있다. 웹사이트를 통해 자유롭게 접근할 수 있다.[28] Kermmarec 등은 또한 [5]diatoms의 생태학적 평가에 rbcL 유전자를 성공적으로 사용했다. rbcL 마커도 쉽게 정렬하고 비교한다.
모니즈와 카츠마르스카는 SSU, COI, ITS2 마커의 증폭 성공률을 조사한 결과 300~400 BP ITS-2 + 5.8S 파편이 가장 높은 증폭 성공률과 우수한 종 분해능을 제공한다는 것을 알아냈다. 이 표식은 이후 99.5%의 성공률로 형태학적으로 정의된 종을 분리하는 데 사용되었다. 이러한 증폭 성공에도 불구하고 짐머만 외 연구진은 개인 내 이질성으로 인한 ITS-2의 사용을 비판했다.[7] SSU[7] 또는 rbcL(Mann et al., 2010)은 개인 간의 이질성이 덜하므로 종을 구별할 때 더 유리하다고 제안되었다.
적용들
바이오모니터링 및 생물평가를 위한 유전자 도구
디아톰은 유럽 물 프레임워크 지침의 일부로 감시되어야 하는 생물 모노미토링 도구 모음의 일부로 일상적으로 사용된다.[29] 규조류는 어디에나 있고 물리 화학적 매개 변수의 변화에 직접 영향을 받고 무척추동물 등 환경변수와의 관계가 좋아 전반적으로 수질을 더 잘 파악할 수 있기 때문에 담수지의 생태계 건강 지표로 사용된다.[30]

수생 생태계의
최근 몇 년 동안, 연구자들은 현미경을 이용한 전통적인 평가를 보완하기 위해 디아톰의 메타바코딩과 염기서열을 위한 도구를 개발, 표준화하여 수생 시스템을 위한 생물모노네토링의 새로운 길을 열었다.[31] 강 바이오모니터링에 대한 차세대 염기서열 접근법을 통해 벤트히크 디아톰을 사용한 것은 그 안에서 좋은 잠재력을 드러냈다.[5] 많은 연구들이 메타스코딩과 HTS(고투과 시퀀싱)를 활용하여 신선한 화구의 품질 상태와 다양성을 추정할 수 있다는 것을 보여주었다. 환경청의 일원으로 켈리 외 연구원이 있다.[32] 영국을 위해 강에 있는 규조류 공동체를 평가하기 위한 DNA 기반 메타코딩 접근법을 개발했다. 바셀론 외 [33]연구진은 규조류에 대한 형태학적 접근법과 HTS 접근법을 비교한 결과 HTS가 특정 오염 민감도 지수(SPI) 측면에서 대부분의 하천에 대한 품질 상태를 신뢰할 수 있는 지표로 제시한 것을 발견했다. 바셀론 등은 열대섬 마요테(프랑스령 DOM-TOM)의 강 감시망에도 디아톰 공동체의 DNA 메타바코딩을 적용했다.[34]
리멧 외 연구진은 또한 이원자 다양성을 평가하기 위해 HTS를 사용할 가능성을 조사했고 HTS와 현미경 분석의 다양성 지수가 완벽하지는 않지만 상관관계가 있다는 것을 보여주었다.[35]
DNA 바코딩과 메타바코딩은 분자 측정 기준과 지수를 설정하는데 사용될 수 있으며, 이는 잠재적으로 수생태계의 생태적, 환경적 상태에 대한 전통적인 접근방식의 그것과 유사한 결론을 제공한다.[36]
포렌식
디아톰은 법의학적 관행에 익사하는 진단 도구로 사용된다. 이산화질소 검사는 물에서 폐로 이산화질소를 흡입해 체내에 분포하고 침전시키는 원리에 기초한다. DNA 방법은 사망원인이 실제로 익사했는지 확인하고 익사 원인을 규명하는 데 사용할 수 있다.[37] 디아톰 DNA 메타코딩은 체내에 존재하는 디아톰 공동체를 신속하게 분석하여 익사 원점을 찾아내고 시신이 한 장소에서 다른 곳으로 옮겨졌을 수 있는지를 조사할 수 있는 기회를 제공한다.
암호화 종 및 데이터 저장
디아톰 메타스코딩은 현미경으로 식별하기 어려운 암호종을 구분하는 데 도움이 될 수 있으며, 형태학적 조립물을 메타스코딩 데이터와 비교함으로써 참조 데이터베이스를 완성하는 데 도움이 될 수 있다.[35]
기타미갈개
엽록소는 지구식물을 포함한 고대인과 분류학적으로 매우 다양한 혈통을 가지고 있다(Fang et al. 2014). 구조 및 초구조적 기준에 따라 14,000종 이상이 설명되었음에도 불구하고(Hall et al. 2010) 형태학적 식별은 종종 제한적이다.
엽록소에 대한 여러 바코드가 DNA 기반 식별을 위해 제안되었다. 시토크롬 산화효소 I(COI, COX) 코딩유전자(링크)는 동물에게는 표준 바코드지만, 이 유전자가 이 조류군(Turmel et al. 2002)에 여러 개의 인트론을 포함하고 있기 때문에 엽록소에게는 만족스럽지 못하다는 것이 입증되었다. 엽록소에 사용된 핵마커 유전자는 SSU rDNA, LSU rDNA, rDNA ITS(Leiaert et al. 2014)이다.
마크로알개
분류학적 그룹화보다는 형태학적 그룹화인 매크로개는 단순한 형태학, 표현적 가소성, 대체 라이프사이클 단계 때문에 식별하기가 매우 어려울 수 있다. 따라서 조류학 및 식별은 DNA 바코딩과 같은 유전적/분자적 도구에 크게 의존하게 되었다.[38][39] SSU rDNA 유전자는 매크로 게에 대한 유전학 연구에 흔히 사용되는 바코드다.[40] 그러나 SSU rDNA는 보존율이 높은 지역이며 일반적으로 종 식별에 대한 분해능이 부족하다.
지난 20년 동안 종 식별을 목적으로 한 DNA 바코딩에 대한 특정 표준이 매크로의 주요 그룹별로 개발되었다.[41][38][42][43][44] 시토크롬 c 산화효소 서브유닛 I(COI) 유전자는 홍조류와 갈색조류의 바코드로, 녹조에는 tufA(플라스티드 신장 계수), rbcL(루비스코 대형 서브유닛), ITS(내부 전사 스페이서) 유전자가 주로 사용된다.[40][44] 이 바코드는 일반적으로 600~700bp 길이다.
바코드는 진화의 유산이 매우 다양하기 때문에 일반적으로 매크로의 3대 그룹(빨간색, 녹색, 갈색) 사이에 차이가 있다.[45] 마크로알개는 다혈질 집단으로, 집단 내에서 모두 최근의 공통 조상을 공유하지 않는다는 뜻으로, 종 식별을 위해 충분히 가변적일 뿐 모든 것 중에서 보존되는 유전자를 찾기가 쉽지 않다.
대상 지역
| 분류군 | 마커유전자 | ||
| 핵의 | 미토콘드리아 | 엽록체 | |
| 엽록소 | SSU rDNA, LSU rDNA, rDNA ITS | tufA, rbcL | |
| 로도피테스 | 피코에린, 신장 계수, LSU rDNA | cox1, cox2-3 스페이서 | rbcL, 루비스코 스페이서 |
| 파에오피스트 | RDNA ITS | cox1, cox3 | psbA, rbcL, 루비스코 스페이서 |
| 번데기와 시누로피테스 | SSU rDNA, rDNA ITS | 콕스1 | psaA, rbcL |
| 암호학 | SSU rDNA, LSU rDNA, rDNA ITS | 콕스1 | 루비스코 스페이서 |
| 바킬라리오피테스 | SSU rDNA, LSU rDNA, rDNA ITS | 콕스1 | rbcL |
| 다이노피스 | LSU RDNA, RDNA ITS | cox1, cob | PsbAncr, 23S rDNA |
| 햅트로피스트 | SSU rDNA, LSU rDNA, rDNA, rDNA ITS | cx1b-atp4 | 투파 |
| 라피도피테스 | SSU rDNA, LSU rDNA, rDNA, rDNA ITS | 콕스1 | psaA, rbcL |
| 크산토피테스 | RDNA ITS | RbcL, psbA-rbcL 스페이서 | |
| 클로로라치니오피스류 | 핵 rDNA ITS, 핵형 rDNA ITS | ||
| 우글레노피스 | SSU rDNA, LSU rDNA | SSU rDNA, LSU rDNA | |
적응 대상
참고 항목
다양한 유기체의 DNA 바코딩에 대한 자세한 정보는 여기에서 확인할 수 있다.
참조
- ^ Lobo, Eduardo A.; Heinrich, Carla Giselda; Schuch, Marilia; Wetzel, Carlos Eduardo; Ector, Luc (2016), Necchi JR, Orlando (ed.), "Diatoms as Bioindicators in Rivers", River Algae, Springer International Publishing, pp. 245–271, doi:10.1007/978-3-319-31984-1_11, ISBN 9783319319834
- ^ Stevenson, R. Jan; Pan, Yangdong; van Dam, Herman (2010), Smol, John P.; Stoermer, Eugene F. (eds.), "Assessing environmental conditions in rivers and streams with diatoms", The Diatoms (2 ed.), Cambridge University Press, pp. 57–85, doi:10.1017/cbo9780511763175.005, ISBN 9780511763175
- ^ Taberlet, Pierre; Bonin, Aurélie; Zinger, Lucie; Coissac, Eric (2018-03-22). Environmental DNA. Vol. 1. Oxford University Press. doi:10.1093/oso/9780198767220.001.0001. ISBN 9780198767220.
- ^ a b Zimmermann, Jonas; Glöckner, Gernot; Jahn, Regine; Enke, Neela; Gemeinholzer, Birgit (2015). "Metabarcoding vs. morphological identification to assess diatom diversity in environmental studies". Molecular Ecology Resources. 15 (3): 526–542. doi:10.1111/1755-0998.12336. PMID 25270047. S2CID 27291997.
- ^ a b c d e Kermarrec, Lenaïg; Franc, Alain; Rimet, Frédéric; Chaumeil, Philippe; Frigerio, Jean-Marc; Humbert, Jean-François; Bouchez, Agnès (2014). "A next-generation sequencing approach to river biomonitoring using benthic diatoms". Freshwater Science. 33 (1): 349–363. doi:10.1086/675079. ISSN 2161-9549. S2CID 85771495.
- ^ a b Hamsher, Sarah E.; Evans, Katharine M.; Mann, David G.; Poulíčková, Aloisie; Saunders, Gary W. (2011). "Barcoding Diatoms: Exploring Alternatives to COI-5P". Protist. 162 (3): 405–422. doi:10.1016/j.protis.2010.09.005. PMID 21239228.
- ^ a b c d e Zimmermann, Jonas; Jahn, Regine; Gemeinholzer, Birgit (2011). "Barcoding diatoms: evaluation of the V4 subregion on the 18S rRNA gene, including new primers and protocols". Organisms Diversity & Evolution. 11 (3): 173–192. doi:10.1007/s13127-011-0050-6. ISSN 1439-6092. S2CID 39047583.
- ^ Apothéloz-Perret-Gentil, Laure; Cordonier, Arielle; Straub, François; Iseli, Jennifer; Esling, Philippe; Pawlowski, Jan (2017). "Taxonomy-free molecular diatom index for high-throughput eDNA biomonitoring". Molecular Ecology Resources. 17 (6): 1231–1242. doi:10.1111/1755-0998.12668. PMID 28296259. S2CID 206947826.
- ^ Pawlowski, J.; Lejzerowicz, F.; Apotheloz-Perret-Gentil, L.; Visco, J.; Esling, P. (2016). "Protist metabarcoding and environmental biomonitoring: Time for change". European Journal of Protistology. 55 (Pt A): 12–25. doi:10.1016/j.ejop.2016.02.003. PMID 27004417.
- ^ Almeida, Salomé F.P.; Elias, Carmen; Ferreira, João; Tornés, Elisabet; Puccinelli, Camilla; Delmas, François; Dörflinger, Gerald; Urbanič, Gorazd; Marcheggiani, Stefania (2014). "Water quality assessment of rivers using diatom metrics across Mediterranean Europe: A methods intercalibration exercise". Science of the Total Environment. 476–477: 768–776. Bibcode:2014ScTEn.476..768A. doi:10.1016/j.scitotenv.2013.11.144. PMID 24342490.
- ^ Lavoie, Isabelle; Campeau, Stéphane; Darchambeau, François; Cabana, Gilbert; Dillon, Peter J. (2008). "Are diatoms good integrators of temporal variability in stream water quality?". Freshwater Biology. 53 (4): 827–841. doi:10.1111/j.1365-2427.2007.01935.x. hdl:2268/62412. ISSN 0046-5070.
- ^ Martin, Gonzalo; Reyes Fernandez, Maria de los (2012-05-16), Voudouris, Kostas (ed.), "Diatoms as Indicators of Water Quality and Ecological Status: Sampling, Analysis and Some Ecological Remarks", Ecological Water Quality - Water Treatment and Reuse, InTech, doi:10.5772/33831, ISBN 9789535105084
- ^ Rimet, Frédéric; Bouchez, Agnès (2012). "Biomonitoring river diatoms: Implications of taxonomic resolution". Ecological Indicators. 15 (1): 92–99. doi:10.1016/j.ecolind.2011.09.014.
- ^ Vasselon, Valentin; Domaizon, Isabelle; Rimet, Frédéric; Kahlert, Maria; Bouchez, Agnès (2017). "Application of high-throughput sequencing (HTS) metabarcoding to diatom biomonitoring: Do DNA extraction methods matter?" (PDF). Freshwater Science. 36 (1): 162–177. doi:10.1086/690649. ISSN 2161-9549. S2CID 59043627.
- ^ Stoeck, Thorsten; Behnke, Anke; Christen, Richard; Amaral-Zettler, Linda; Rodriguez-Mora, Maria J; Chistoserdov, Andrei; Orsi, William; Edgcomb, Virginia P (2009). "Massively parallel tag sequencing reveals the complexity of anaerobic marine protistan communities". BMC Biology. 7 (1): 72. doi:10.1186/1741-7007-7-72. ISSN 1741-7007. PMC 2777867. PMID 19886985.
- ^ a b c d e Pawlowski, Jan; Kelly-Quinn, Mary; Altermatt, Florian; Apothéloz-Perret-Gentil, Laure; Beja, Pedro; Boggero, Angela; Borja, Angel; Bouchez, Agnès; Cordier, Tristan (2018). "The future of biotic indices in the ecogenomic era: Integrating (e)DNA metabarcoding in biological assessment of aquatic ecosystems" (PDF). Science of the Total Environment. 637–638: 1295–1310. Bibcode:2018ScTEn.637.1295P. doi:10.1016/j.scitotenv.2018.05.002. PMID 29801222.
- ^ Visco, Joana Amorim; Apothéloz-Perret-Gentil, Laure; Cordonier, Arielle; Esling, Philippe; Pillet, Loïc; Pawlowski, Jan (2015-07-07). "Environmental Monitoring: Inferring the Diatom Index from Next-Generation Sequencing Data". Environmental Science & Technology. 49 (13): 7597–7605. Bibcode:2015EnST...49.7597V. doi:10.1021/es506158m. ISSN 0013-936X. PMID 26052741.
- ^ Zimmermann, Jonas; Abarca, Nelida; Enk, Neela; Skibbe, Oliver; Kusber, Wolf-Henning; Jahn, Regine (2014-09-29). Schierwater, Bernd (ed.). "Taxonomic Reference Libraries for Environmental Barcoding: A Best Practice Example from Diatom Research". PLOS ONE. 9 (9): e108793. Bibcode:2014PLoSO...9j8793Z. doi:10.1371/journal.pone.0108793. ISSN 1932-6203. PMC 4180937. PMID 25265556.
- ^ Shaw, Jennifer L. A.; Weyrich, Laura; Cooper, Alan (2017). "Using environmental (e)DNA sequencing for aquatic biodiversity surveys: a beginner's guide". Marine and Freshwater Research. 68 (1): 20. doi:10.1071/MF15361. ISSN 1323-1650.
- ^ Edgar, Graham J.; Alexander, Timothy J.; Lefcheck, Jonathan S.; Bates, Amanda E.; Kininmonth, Stuart J.; Thomson, Russell J.; Duffy, J. Emmett; Costello, Mark J.; Stuart-Smith, Rick D. (2017). "Abundance and local-scale processes contribute to multi-phyla gradients in global marine diversity". Science Advances. 3 (10): e1700419. Bibcode:2017SciA....3E0419E. doi:10.1126/sciadv.1700419. ISSN 2375-2548. PMC 5647131. PMID 29057321.
- ^ Vasselon, Valentin; Bouchez, Agnès; Rimet, Frédéric; Jacquet, Stéphan; Trobajo, Rosa; Corniquel, Méline; Tapolczai, Kálmán; Domaizon, Isabelle (2018). Mahon, Andrew (ed.). "Avoiding quantification bias in metabarcoding: Application of a cell biovolume correction factor in diatom molecular biomonitoring" (PDF). Methods in Ecology and Evolution. 9 (4): 1060–1069. doi:10.1111/2041-210X.12960. hdl:20.500.12327/161.
- ^ Evans, Katharine M.; Wortley, Alexandra H.; Mann, David G. (2007). "An Assessment of Potential Diatom "Barcode" Genes (cox1, rbcL, 18S and ITS rDNA) and their Effectiveness in Determining Relationships in Sellaphora (Bacillariophyta)". Protist. 158 (3): 349–364. doi:10.1016/j.protis.2007.04.001. PMID 17581782.
- ^ a b Moniz, Mónica B.J.; Kaczmarska, Irena (2010). "Barcoding of Diatoms: Nuclear Encoded ITS Revisited". Protist. 161 (1): 7–34. doi:10.1016/j.protis.2009.07.001. PMID 19674931.
- ^ Scicluna, Stephanie M.; Tawari, Blessing; Clark, C. Graham (2006). "DNA Barcoding of Blastocystis". Protist. 157 (1): 77–85. doi:10.1016/j.protis.2005.12.001. PMID 16431158.
- ^ Utz, Laura R. P.; Eizirik, Eduardo (2007). "Molecular Phylogenetics of Subclass Peritrichia (Ciliophora: Oligohymenophorea) Based on Expanded Analyses of 18S rRNA Sequences". The Journal of Eukaryotic Microbiology. 54 (3): 303–305. doi:10.1111/j.1550-7408.2007.00260.x. ISSN 1066-5234. PMID 17552986. S2CID 31422075.
- ^ Jahn, R. (2007-12-11). "Diatoms and DNA barcoding: a pilot study on an environmental sample". Proceedings of the 1st Central European Diatom Meeting 2007. Botanic Garden and Botanical Museum Berlin-Dahlem: 63–68. doi:10.3372/cediatom.113. ISBN 9783921800638.
- ^ Nickrent, Daniel L.; Sargent, Malcolm L. (1991). "An overview of the secondary structure of the V4 region of eukaryotic small-subunit ribosomal RNA". Nucleic Acids Research. 19 (2): 227–235. doi:10.1093/nar/19.2.227. ISSN 0305-1048. PMC 333584. PMID 2014163.
- ^ Wilde, Erik (1999), "Hypertext Transfer Protocol (HTTP)", Wilde's WWW, Springer Berlin Heidelberg, pp. 53–135, doi:10.1007/978-3-642-95855-7_4, ISBN 9783642958571
- ^ Directive 2000/60/EC of the European Parliament and of the Council of 23 October 2000 establishing a framework for Community action in the field of water policy, vol. OJ L, 2000-12-22, retrieved 2019-03-28
- ^ Rimet, Frédéric (2012-03-01). "Recent views on river pollution and diatoms". Hydrobiologia. 683 (1): 1–24. doi:10.1007/s10750-011-0949-0. ISSN 1573-5117. S2CID 18668007.
- ^ Kermarrec, L.; Franc, A.; Rimet, F.; Chaumeil, P.; Humbert, J. F.; Bouchez, A. (2013). "Next-generation sequencing to inventory taxonomic diversity in eukaryotic communities: a test for freshwater diatoms". Molecular Ecology Resources. 13 (4): 607–619. doi:10.1111/1755-0998.12105. PMID 23590277. S2CID 2706722.
- ^ "Kelly et al" (PDF).
- ^ Vasselon, Valentin; Domaizon, Isabelle; Rimet, Frédéric; Kahlert, Maria; Bouchez, Agnès (2017). "Application of high-throughput sequencing (HTS) metabarcoding to diatom biomonitoring: Do DNA extraction methods matter?" (PDF). Freshwater Science. 36 (1): 162–177. doi:10.1086/690649. ISSN 2161-9549. S2CID 59043627.
- ^ Vasselon, Valentin; Rimet, Frédéric; Tapolczai, Kálmán; Bouchez, Agnès (2017). "Assessing ecological status with diatoms DNA metabarcoding: Scaling-up on a WFD monitoring network (Mayotte island, France)". Ecological Indicators. 82: 1–12. doi:10.1016/j.ecolind.2017.06.024.
- ^ a b Rimet, Frédéric; Vasselon, Valentin; A.-Keszte, Barbara; Bouchez, Agnès (2018). "Do we similarly assess diversity with microscopy and high-throughput sequencing? Case of microalgae in lakes". Organisms Diversity & Evolution. 18 (1): 51–62. doi:10.1007/s13127-018-0359-5. ISSN 1439-6092. S2CID 3879922.
- ^ Hering, Daniel; Borja, Angel; Jones, J.Iwan; Pont, Didier; Boets, Pieter; Bouchez, Agnes; Bruce, Kat; Drakare, Stina; Hänfling, Bernd (2018). "Implementation options for DNA-based identification into ecological status assessment under the European Water Framework Directive". Water Research. 138: 192–205. doi:10.1016/j.watres.2018.03.003. PMID 29602086. S2CID 5008250.
- ^ Seo, Yasuhisa; Ichida, Daisuke; Sato, Shingo; Kuroki, Kohji; Kishida, Tetsuko (2014). "An Improved Method for the Diatom Test Utilizing DNA Binding Ability of Silica". Journal of Forensic Sciences. 59 (3): 779–784. doi:10.1111/1556-4029.12390. PMID 24502836. S2CID 1057761.
- ^ a b Saunders, Gary W (2005-10-29). "Applying DNA barcoding to red macroalgae: a preliminary appraisal holds promise for future applications". Philosophical Transactions of the Royal Society B: Biological Sciences. 360 (1462): 1879–1888. doi:10.1098/rstb.2005.1719. ISSN 0962-8436. PMC 1609223. PMID 16214745.
- ^ a b Leliaert, Frederik; Verbruggen, Heroen; Vanormelingen, Pieter; Steen, Frederique; López-Bautista, Juan M.; Zuccarello, Giuseppe C.; De Clerck, Olivier (2014-04-03). "DNA-based species delimitation in algae". European Journal of Phycology. 49 (2): 179–196. doi:10.1080/09670262.2014.904524. ISSN 0967-0262.
- ^ a b Harper & Saunders (2001). "The application of sequences of the ribosomal cistron to the systematics and classification of the florideophyte red algae (Florideophyceae, Rhodophyta)". Cahiers de Biologie Marine. 42(1/2): 25–38.
- ^ McDevit, Daniel C.; Saunders, Gary W. (2009). "On the utility of DNA barcoding for species differentiation among brown macroalgae (Phaeophyceae) including a novel extraction protocol". Phycological Research. 57 (2): 131–141. doi:10.1111/j.1440-1835.2009.00530.x. S2CID 84811166.
- ^ Saunders & Kucera (2010). "An evaluation of rbcL, tufA, UPA, LSU and ITS as DNA barcode markers for the marine green macroalgae". Cryptogamie Algologie. 31 (4).
- ^ Valero, Myriam; Küpper, Frithjof C.; Tsiamis, Konstantinos; Couceiro, Lucía; Peters, Akira F. (2015). "Barcoding of Cryptic Stages of Marine Brown Algae Isolated from Incubated Substratum Reveals High Diversity in Acinetosporaceae (Ectocarpales, Phaeophyceae)1". Cryptogamie, Algologie. 36 (1): 3–29. doi:10.7872/crya.v36.iss1.2015.3. ISSN 0181-1568. S2CID 84276804.
- ^ a b Saunders, Gary W.; McDevit, Daniel C. (2012), Kress, W. John; Erickson, David L. (eds.), "Methods for DNA Barcoding Photosynthetic Protists Emphasizing the Macroalgae and Diatoms", DNA Barcodes, Humana Press, vol. 858, pp. 207–222, doi:10.1007/978-1-61779-591-6_10, ISBN 9781617795909, PMID 22684958
- ^ Baldauf, S. L. (2003-06-13). "The Deep Roots of Eukaryotes". Science. 300 (5626): 1703–1706. Bibcode:2003Sci...300.1703B. doi:10.1126/science.1085544. ISSN 0036-8075. PMID 12805537. S2CID 32788100.