CUL4A
CUL4ACullin-4A는 인간에게 CUL4A 유전자에 의해 암호화된 단백질이다.[4][5]CUL4A는 유비퀴틴 리가아제 단백질의 컬린 계열에 속하며, CUL4B 단백질과 매우 동질적이다.CUL4A는 DNA 수리, 염색질 리모델링, 정조세포, 조혈모피, 그리고 유사세포 순환과 같은 수많은 핵심 과정을 규제한다.그 결과, CUL4A는 여러 암과 HIV를 포함한 특정 바이러스의 병원균에 연루되었다.CUL4A 복합체의 성분인 세레블론이 테라토마이제 탈리도마이드의 주요 표적이 된 것으로 밝혀졌다.
구조
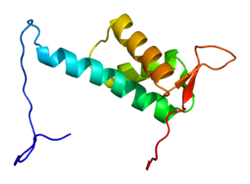
CUL4A 단백질은 759개의 아미노산으로 주로 알파헬리크로 구성된 확장되고 견고한 구조를 형성한다.N-terminus에서, CUL4A는 수많은 DDB1-CUL4-관련 요인(DCAF)과 상호작용하는 DDB1 어댑터 단백질의 베타-프로펠러에 결합한다.그 결과 유비퀴틴 리게아제 복합체의 기질채용에 N-terminus가 결정적이다.C-터미널 끝에서 CUL4A는 RING 도메인을 통해 RBX1/ROC1 단백질과 상호작용한다.RBX1은 CRL(Cullin-Ring ubiquitin ligase) 복합체의 핵심 성분으로 E2 유비퀴틴 결합 효소를 모집하는 기능이다.따라서, CUL4A의 C-단자 - RBX1 및 활성화된 E2 효소 - CRL4 복합체의 촉매 코어를 구성한다.CUL4A는 또한 C-단자 지역의 고도로 보존된 리신 잔여물에 NEDD8 분자를 공동 부착함으로써 수정된다.이 수정은 Cullin 단백질의 RING 영역에 유연성을 촉진하고 강화된 유비퀴틴 리게아제 활동을 촉진하는 순응적 변화를 유도하는 것으로 보인다.[6]
전체적으로 CRL4A 복합체는 셀에 의한 정교한 규제와 셀 내 수많은 기판과 프로세스에 대한 영향을 가능하게 하는 모듈형 구조를 가지고 있다.비록 개별적인 부분은 다르지만, 모든 컬린 기반의 유비퀴틴 연대는 이러한 특성을 나타낸다.[7]
함수
DNA 손상 및 수리
DDB1 어댑터 단백질은 초기에 손상된 DNA를 인식하고 뉴클레오티드 절연 보수(NER)라고 알려진 수리 형태에 참여하는 것으로 밝혀진 이질 복합체(UV-DDB)의 큰 서브 유닛으로 특징지어졌다.이 손상된 DNA 결합 단백질 복합체의 더 작은 하위 단위는 DDB2로 알려져 있으며 UV-방사선과 관련된 DNA 병변을 직접 결합할 수 있다.DDB2는 DCAF 단백질로 CRL4 콤플렉스의 편재성 기질이며, 또한 손상 부위 근처의 XPC와 히스톤(다음 섹션 참조)과 같은 다른 기질에 대한 E3 리기아제 단백질 역할도 한다.[8]DNA 손상을 인식하는 단백질인 DDB2와 XPC의 보편화 때문에, CULL4A는 NER 활동의 음성 조절기로 설명되어 왔다.[9][10]CRL4A 콤플렉스는 '글로벌형' NER 외에도 코카인 신드롬 A 단백질과 연계해 '전송-커플링' NER의 역할을 하는 것으로 보인다.[11]CRL4A 복합체는 특정 유형의 DNA 손상(가장 두드러지게 UV-방사선)에 의해 활성화되는 것으로 보이며, DNA 손상 유도 후 여러 기판이 우선 보편화된다.
크로마틴 리모델링
크로마틴을 변형하는 CUL4A의 역할은 주로 DNA 수리 활동과 관련이 있으며, DNA 손상 유도 후에 발생한다.CUL4A와 그 밀접하게 연관된 동음이의 CUL4B 모두 히스톤 H2A, H3 및 H4를 보편화할 수 있다.[12][13]CUL4A, Rtt101의 효모 호몰로로그는 히스톤 H3를 편재하고 뉴클레오솜 조립을 촉진하며 CRL4A 콤플렉스는 인간 세포에서 유사한 기능을 수행한다.[14]CRL4 콤플렉스는 히스톤 메틸전달효소 규제를 통해 히스톤 메틸화 이벤트와 염색질 구조에도 영향을 미친다.[15]히스톤 H4 monomethylase PR-Set7/SET8은 S상 동안 그리고 PCNA 의존적인 방식으로 DNA 손상에 따라 CRL4(Cdt2) 콤플렉스에 의해 염색질에 보편화된다.[16][17][18]
세포주기와 DNA복제 규정
CRL4A 복합체들은 복제허용인자 단백질 Cdt1과 사이클린 의존성 키나제 억제제 p21의 단백질 발현 수준을 조절함으로써 유사체 주기의 DNA 합성 단계 즉 S상 진입을 규제한다.두 경우 모두 CRL4A는 Cdt2를 DCAF로 활용하여 두 기판을 PCNA 종속 방식으로 바인딩한다.방해받지 않는 세포 주기 진행 동안, CRL4A에Cdt2 의한 이러한 단백질의 편재화 및 하향 조절은 DNA 복제가 시작될 때 발생한다.UV 조사와 같은 DNA 손상은 또한 CRL4A 매개Cdt2 단백질 파괴를 유발한다.두 기판 모두 SCFSkp2 단지에 의해 규제된다.
CRL4 매개 p21 파괴는 사이클린 E-Cdk2 억제를 완화시키고 S상 진입을 촉진한다.Cdt2 발현 상실은 세포 내 p21 발현을 증가시키고 UV-방사선에 이어 p21을 안정화시킨다.[19]CUL4A를 삭제하면 마우스 배아 섬유화합물의 S상 입력이 지연되고 p21을 삭제하여 구조된다.[10]인간의 망막색소 상피세포에서도 Cdt2 표현의 상실은 p21 종속적인 S상 입력을 지연시키고, S상에서는 p21의 재표현을 초래하며, 불완전한 복제의 사이클, p21의 장기집적, 그리고 어떤 경우에는 사멸의 유도를 초래한다.[20]
원점에서 진핵 DNA 복제의 시작을 촉진한 후, Cdt1은 제미니인에 의해 비활성화되고 SCF와Skp2 CRL4Cdt2 콤플렉스에 의해 분해 대상이 된다.Cdt1 표현은 DDB1의 RNAi 매개 녹다운 또는 CUL4A와 CUL4B 둘 다에 의해 안정화되며, Cdt1 조절을 위해 두 개의 CUL4 단백질의 중복 또는 중복 기능을 제안한다.[21][22]제미닌 발현 감소만이 Cdt1과 과다압축 세포에서 재복제를 유도하는 것으로 보인다.
또한 CRL4는 Cdt2와 PCNA를 활용하여 S상 및 UV 조사 후 DNA 중합효소 Δ의 p12 서브단위를 저하시킨다.[23]
해마토피시스
CRL4A 콤플렉스는 조혈모세포의 필수 조절제인 HOX 전사 계열의 수많은 구성원의 저하를 유도하는 것으로 보인다.[24]CRL4A 매개 분해의 대상으로 확인된 HOX 계열의 첫 번째 구성원은 HOXA9로, 조혈모세포 유지에 필수적이며 골수성 백혈병의 하위집합에 관여해왔다.[25][26]HOXA9 디그론은 DNA 결합에 중요한 동역자 안에 있다.시퀀스 정렬 연구 결과, 홈오도메인 중 하나의 나선 내부에 보존성이 높은 "LEXE" 모티브가 있는 것으로 나타났다.이 모티프 내의 여러 아미노산이 변이되었을 때, HOXB4는 CRL4A 매개 분해에 내성이 생겼다.[24]HOX 단백질 분해에 필요한 기질 수용체 또는 DCAF는 여전히 알려져 있지 않다.
정자생식과 감수분열
Cul4a 유전자는 쥐의 수컷 세균 세포에서 정상적인 정조세포 생성과 감수분열을 위해 필요하다.[27][28]cul4a−/− 수컷은 비정상적인 정자를 생산하고 불임이다.CUL4A와 CUL4B는 모두 남성 게이메트로 표현되는 반면, CUL4A는 파키테네와 디플롯테네로 표현되는 비율이 높다.CUL4A 결핍 남성 세균 세포가 높은 수준의 세포사멸, 부적절한 DNA 수리, CRL4 기질 Cdt1 축적을 보이는 것은 이러한 단계에 있다.
조절곤란
암
CUL4A 유전자를 포함하는 염색체 부위 ch13q34는 유방암, 자궁암, 폐암, 위암, 대장암을 포함한 특정 암의 3-6%에서 증폭된다.[29]또한 CUL4A는 멜라노마 약 4%에서 돌연변이 또는 증폭된다(변이가 분산되어 개별 돌연변이가 산발적으로 발생함).
마우스 모델에서 Cul4a 녹아웃은 UV로 인한 피부 발암에 대한 내성을 두드러지게 만들었다.[10]생쥐 폐조직의 Cul4a 과다압박은 과대성장을 촉진시켰다.[30]
여러 발암에서 CUL4A의 증폭이 관찰되고 CRL4 콤플렉스가 복수의 DNA 수리 및 종양 억제기 유전자를 대상으로 한다는 사실 때문에 CUL4A는 특정 맥락에서 유전자로 볼 수 있다.
바이러스성 병원체
CRL4A 콤플렉스는 강력한 표현(특히 DNA 복제 중)과 모듈성 때문에 포유류 세포의 바이러스 증식을 촉진하기 위해 CRL4A 콤플렉스를 공동 선택 또는 '하이잭트(hijacked)'할 수 있다.
특정 파라믹소바이러스는 STAT1을 목표로 하고 신호를 방해하여 세포의 인터페론 반응을 피한다.Simian virus 5와 타입 II 인간 파라인플루엔자 바이러스는 "V"라는 단백질을 표현하는데, 이는 기질 수용체 역할을 하며 DDB1과 STAT 단백질 사이의 상호작용을 연결한다(CRL4ASV5V 복합체의 구조가 inset에 그려져 있음). 따라서 STAT1의 편재와 열화를[31][32] 유도한다.
DCAF1은 또한 HIV-1 단백질 Vpr과의 상호작용 때문에 VPRBP로 명명된다.DCAF1/VPRBP는 종양 억제, DNA 복제 및 배아 발달에 있어 중요한 기능을 하는 것으로 보이지만, HIV-1은 유비퀴틴 리가제 콤플렉스를 "히잭스"하여 G2상에서의 세포 주기 억제를 유도한다.[33][34][35]CRL4A는DCAF1-Vpr 우라실-DNA 글리코실라아제의 핵 등소 형태의 보편화를 유도한다.[36][37]또한 HIV-2는 SAMHD1이라는 이름의 렌즈 바이러스 억제 디옥시뉴클레오시드 삼인산수소효소의 Vpx 단백질 파괴를 통해 CRL4A를DCAF1 이용하는 것으로 보인다.[38][39]
탈리도마이드 처리
2010년 이토 외 연구진은 DCAF 단백질인 세레블론이 기형성 화합물 탈리도마이드의 주요 표적이었다고 보고했다.[40]탈리도마이드와 포말리도마이드, 레날리도마이드와 같은 다른 파생상품은 면역억제제(또는 IMiDs)로 알려져 있으며 자가면역질환과 여러 암, 특히 골수종 등의 치료제로 연구되어 왔다.최근 보고에 따르면 IMiDs는 CRL4에CRBN 바인딩되어 일반적으로 CRL4 단지의 대상이 아닌 IKZF1과 IKZF3 전사 인자의 저하를 촉진한다고 한다.[41][42]
교호작용 및 기질
인간 CUL4A는 다음과 직접적인 상호작용을 형성한다.
인간 CUL4A-DDB1-RBX1 복합체는 다음 요소의 편재화를 촉진한다.
†단백질은 바이러스성 단백질에 의해 지시되었을 때만 CRL4A 기질이다.
§단백질은 IMiDs가 지시하는 경우에만 CRL4A 기질이다.
메모들
이 글의 2015년 버전은 이중 출판 모델에 따라 외부 전문가에 의해 갱신되었다.해당 학술적 동료 검토 기사는 진에 게재되었으며, 다음과 같이 인용할 수 있다. Jeffrey Hannah; Pengbo Zhou (15 November 2015). "Distinct and overlapping functions of the cullin E3 ligase scaffolding proteins CUL4A and CUL4B". Gene. Gene Wiki Review Series. 573 (1): 33–45. doi:10.1016/J.GENE.2015.08.064. ISSN 0378-1119. PMC 5110433. PMID 26344709. Wikidata Q28086765. |
참조
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG000031446 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Kipreos ET, Lander LE, Wing JP, He WW, Hedgecock EM (Jun 1996). "cul-1 is required for cell cycle exit in C. elegans and identifies a novel gene family". Cell. 85 (6): 829–39. doi:10.1016/S0092-8674(00)81267-2. PMID 8681378. S2CID 15805562.
- ^ "Entrez Gene: CUL4A Cullin 4A".
- ^ Duda DM, Borg LA, Scott DC, Hunt HW, Hammel M, Schulman BA (Sep 2008). "Structural insights into NEDD8 activation of cullin-RING ligases: conformational control of conjugation". Cell. 134 (6): 995–1006. doi:10.1016/j.cell.2008.07.022. PMC 2628631. PMID 18805092.
- ^ Bosu DR, Kipreos ET (2008). "Cullin-RING ubiquitin ligases: global regulation and activation cycles". Cell Division. 3: 7. doi:10.1186/1747-1028-3-7. PMC 2266742. PMID 18282298.
- ^ a b Sugasawa K, Okuda Y, Saijo M, Nishi R, Matsuda N, Chu G, Mori T, Iwai S, Tanaka K, Tanaka K, Hanaoka F (May 2005). "UV-induced ubiquitylation of XPC protein mediated by UV-DDB-ubiquitin ligase complex". Cell. 121 (3): 387–400. doi:10.1016/j.cell.2005.02.035. PMID 15882621. S2CID 16773579.
- ^ Chen X, Zhang J, Lee J, Lin PS, Ford JM, Zheng N, Zhou P (May 2006). "A kinase-independent function of c-Abl in promoting proteolytic destruction of damaged DNA binding proteins". Molecular Cell. 22 (4): 489–99. doi:10.1016/j.molcel.2006.04.021. PMID 16713579.
- ^ a b c d Liu L, Lee S, Zhang J, Peters SB, Hannah J, Zhang Y, Yin Y, Koff A, Ma L, Zhou P (May 2009). "CUL4A abrogation augments DNA damage response and protection against skin carcinogenesis". Molecular Cell. 34 (4): 451–60. doi:10.1016/j.molcel.2009.04.020. PMC 2722740. PMID 19481525.
- ^ Hannah J, Zhou P (Apr 2009). "Regulation of DNA damage response pathways by the cullin-RING ubiquitin ligases". DNA Repair. 8 (4): 536–43. doi:10.1016/j.dnarep.2009.01.011. PMC 2858918. PMID 19231300.
- ^ Guerrero-Santoro J, Kapetanaki MG, Hsieh CL, Gorbachinsky I, Levine AS, Rapić-Otrin V (Jul 2008). "The cullin 4B-based UV-damaged DNA-binding protein ligase binds to UV-damaged chromatin and ubiquitinates histone H2A". Cancer Research. 68 (13): 5014–22. doi:10.1158/0008-5472.CAN-07-6162. PMID 18593899.
- ^ Wang H, Zhai L, Xu J, Joo HY, Jackson S, Erdjument-Bromage H, Tempst P, Xiong Y, Zhang Y (May 2006). "Histone H3 and H4 ubiquitylation by the CUL4-DDB-ROC1 ubiquitin ligase facilitates cellular response to DNA damage". Molecular Cell. 22 (3): 383–94. doi:10.1016/j.molcel.2006.03.035. PMID 16678110.
- ^ Han J, Zhang H, Zhang H, Wang Z, Zhou H, Zhang Z (Nov 2013). "A Cul4 E3 ubiquitin ligase regulates histone hand-off during nucleosome assembly". Cell. 155 (4): 817–29. doi:10.1016/j.cell.2013.10.014. PMC 3994564. PMID 24209620.
- ^ Higa LA, Wu M, Ye T, Kobayashi R, Sun H, Zhang H (Nov 2006). "CUL4-DDB1 ubiquitin ligase interacts with multiple WD40-repeat proteins and regulates histone methylation". Nature Cell Biology. 8 (11): 1277–83. doi:10.1038/ncb1490. hdl:10397/34293. PMID 17041588. S2CID 22180568.
- ^ a b Jørgensen S, Eskildsen M, Fugger K, Hansen L, Larsen MS, Kousholt AN, Syljuåsen RG, Trelle MB, Jensen ON, Helin K, Sørensen CS (Jan 2011). "SET8 is degraded via PCNA-coupled CRL4(CDT2) ubiquitylation in S phase and after UV irradiation". The Journal of Cell Biology. 192 (1): 43–54. doi:10.1083/jcb.201009076. PMC 3019552. PMID 21220508.
- ^ a b Tardat M, Brustel J, Kirsh O, Lefevbre C, Callanan M, Sardet C, Julien E (Nov 2010). "The histone H4 Lys 20 methyltransferase PR-Set7 regulates replication origins in mammalian cells". Nature Cell Biology. 12 (11): 1086–93. doi:10.1038/ncb2113. PMID 20953199. S2CID 6710289.
- ^ a b Oda H, Hübner MR, Beck DB, Vermeulen M, Hurwitz J, Spector DL, Reinberg D (Nov 2010). "Regulation of the histone H4 monomethylase PR-Set7 by CRL4(Cdt2)-mediated PCNA-dependent degradation during DNA damage". Molecular Cell. 40 (3): 364–76. doi:10.1016/j.molcel.2010.10.011. PMC 2999913. PMID 21035370.
- ^ a b Abbas T, Sivaprasad U, Terai K, Amador V, Pagano M, Dutta A (Sep 2008). "PCNA-dependent regulation of p21 ubiquitylation and degradation via the CRL4Cdt2 ubiquitin ligase complex". Genes & Development. 22 (18): 2496–506. doi:10.1101/gad.1676108. PMC 2546691. PMID 18794347.
- ^ Barr, Alexis R.; Cooper, Samuel; Heldt, Frank S.; Butera, Francesca; Stoy, Henriette; Mansfeld, Jörg; Novák, Béla; Bakal, Chris (2017). "DNA damage during S-phase mediates the proliferation-quiescence decision in the subsequent G1 via p21 expression". Nature Communications. 8: 14728. Bibcode:2017NatCo...814728B. doi:10.1038/ncomms14728. PMC 5364389. PMID 28317845.
- ^ a b Higa LA, Mihaylov IS, Banks DP, Zheng J, Zhang H (Nov 2003). "Radiation-mediated proteolysis of CDT1 by CUL4-ROC1 and CSN complexes constitutes a new checkpoint". Nature Cell Biology. 5 (11): 1008–15. doi:10.1038/ncb1061. PMID 14578910. S2CID 26070877.
- ^ a b Hu J, Xiong Y (Feb 2006). "An evolutionarily conserved function of proliferating cell nuclear antigen for Cdt1 degradation by the Cul4-Ddb1 ubiquitin ligase in response to DNA damage". The Journal of Biological Chemistry. 281 (7): 3753–6. doi:10.1074/jbc.C500464200. PMID 16407242.
- ^ a b Zhang S, Zhao H, Darzynkiewicz Z, Zhou P, Zhang Z, Lee EY, Lee MY (2013). "A novel function of CRL4(Cdt2): regulation of the subunit structure of DNA polymerase δ in response to DNA damage and during the S phase". J. Biol. Chem. 288 (41): 29550–61. doi:10.1074/jbc.M113.490466. PMC 3795253. PMID 23913683.
- ^ a b c Lee J, Shieh JH, Zhang J, Liu L, Zhang Y, Eom JY, Morrone G, Moore MA, Zhou P (May 2013). "Improved ex vivo expansion of adult hematopoietic stem cells by overcoming CUL4-mediated degradation of HOXB4". Blood. 121 (20): 4082–9. doi:10.1182/blood-2012-09-455204. PMC 3656448. PMID 23520338.
- ^ a b Zhang Y, Morrone G, Zhang J, Chen X, Lu X, Ma L, Moore M, Zhou P (Nov 2003). "CUL-4A stimulates ubiquitylation and degradation of the HOXA9 homeodomain protein". The EMBO Journal. 22 (22): 6057–67. doi:10.1093/emboj/cdg577. PMC 275435. PMID 14609952.
- ^ Lawrence, H. J.; Christensen, J.; Fong, S.; Hu, Y. L.; Weissman, I.; Sauvageau, G.; Humphries, R. K.; Largman, C. (2005). "Loss of expression of the Hoxa-9 homeobox gene impairs the proliferation and repopulating ability of hematopoietic stem cells". Blood. 106 (12): 3988–3994. doi:10.1182/blood-2005-05-2003. PMC 1895111. PMID 16091451.
- ^ Yin Y, Lin C, Kim ST, Roig I, Chen H, Liu L, Veith GM, Jin RU, Keeney S, Jasin M, Moley K, Zhou P, Ma L (2011). "The E3 ubiquitin ligase Cullin 4A regulates meiotic progression in mouse spermatogenesis". Dev. Biol. 356 (1): 51–62. doi:10.1016/j.ydbio.2011.05.661. PMC 3130830. PMID 21624359.
- ^ Kopanja D, Roy N, Stoyanova T, Hess RA, Bagchi S, Raychaudhuri P (2011). "Cul4A is essential for spermatogenesis and male fertility". Dev. Biol. 352 (2): 278–87. doi:10.1016/j.ydbio.2011.01.028. PMC 3065526. PMID 21291880.
- ^ "cBioPortal for Cancer Genomics". Archived from the original on 2015-05-21.
- ^ Li T, Hung MS, Wang Y, Mao JH, Tan JL, Jahan K, Roos H, Xu Z, Jablons DM, You L (Mar 2011). "Transgenic mice for cre-inducible overexpression of the Cul4A gene". Genesis. 49 (3): 134–41. doi:10.1002/dvg.20708. PMC 3285554. PMID 21381181.
- ^ a b Ulane CM, Kentsis A, Cruz CD, Parisien JP, Schneider KL, Horvath CM (Aug 2005). "Composition and assembly of STAT-targeting ubiquitin ligase complexes: paramyxovirus V protein carboxyl terminus is an oligomerization domain". Journal of Virology. 79 (16): 10180–9. doi:10.1128/JVI.79.16.10180-10189.2005. PMC 1182666. PMID 16051811.
- ^ a b Precious B, Childs K, Fitzpatrick-Swallow V, Goodbourn S, Randall RE (Nov 2005). "Simian virus 5 V protein acts as an adaptor, linking DDB1 to STAT2, to facilitate the ubiquitination of STAT1". Journal of Virology. 79 (21): 13434–41. doi:10.1128/JVI.79.21.13434-13441.2005. PMC 1262611. PMID 16227264.
- ^ McCall CM, Miliani de Marval PL, Chastain PD, Jackson SC, He YJ, Kotake Y, Cook JG, Xiong Y (Sep 2008). "Human immunodeficiency virus type 1 Vpr-binding protein VprBP, a WD40 protein associated with the DDB1-CUL4 E3 ubiquitin ligase, is essential for DNA replication and embryonic development". Molecular and Cellular Biology. 28 (18): 5621–33. doi:10.1128/MCB.00232-08. PMC 2546929. PMID 18606781.
- ^ Le Rouzic E, Belaïdouni N, Estrabaud E, Morel M, Rain JC, Transy C, Margottin-Goguet F (Jan 2007). "HIV1 Vpr arrests the cell cycle by recruiting DCAF1/VprBP, a receptor of the Cul4-DDB1 ubiquitin ligase". Cell Cycle. 6 (2): 182–8. doi:10.4161/cc.6.2.3732. PMID 17314515.
- ^ Wen, Xiaoyun; Duus, Karen M.; Friedrich, Thomas D.; De Noronha, Carlos M. C. (2007). "The HIV1 Protein Vpr Acts to Promote G2Cell Cycle Arrest by Engaging a DDB1 and Cullin4A-containing Ubiquitin Ligase Complex Using VprBP/DCAF1 as an Adaptor". Journal of Biological Chemistry. 282 (37): 27046–27057. doi:10.1074/jbc.M703955200. PMID 17620334.
- ^ a b Ahn J, Vu T, Novince Z, Guerrero-Santoro J, Rapic-Otrin V, Gronenborn AM (Nov 2010). "HIV-1 Vpr loads uracil DNA glycosylase-2 onto DCAF1, a substrate recognition subunit of a cullin 4A-ring E3 ubiquitin ligase for proteasome-dependent degradation". The Journal of Biological Chemistry. 285 (48): 37333–41. doi:10.1074/jbc.M110.133181. PMC 2988339. PMID 20870715.
- ^ a b Wen X, Casey Klockow L, Nekorchuk M, Sharifi HJ, de Noronha CM (2012). "The HIV1 protein Vpr acts to enhance constitutive DCAF1-dependent UNG2 turnover". PLOS ONE. 7 (1): e30939. Bibcode:2012PLoSO...730939W. doi:10.1371/journal.pone.0030939. PMC 3265533. PMID 22292079.
- ^ a b Hofmann H, Logue EC, Bloch N, Daddacha W, Polsky SB, Schultz ML, Kim B, Landau NR (Dec 2012). "The Vpx lentiviral accessory protein targets SAMHD1 for degradation in the nucleus". Journal of Virology. 86 (23): 12552–60. doi:10.1128/JVI.01657-12. PMC 3497686. PMID 22973040.
- ^ a b Ahn J, Hao C, Yan J, DeLucia M, Mehrens J, Wang C, Gronenborn AM, Skowronski J (Apr 2012). "HIV/simian immunodeficiency virus (SIV) accessory virulence factor Vpx loads the host cell restriction factor SAMHD1 onto the E3 ubiquitin ligase complex CRL4DCAF1". The Journal of Biological Chemistry. 287 (15): 12550–8. doi:10.1074/jbc.M112.340711. PMC 3321004. PMID 22362772.
- ^ Ito, T.; Ando, H.; Suzuki, T.; Ogura, T.; Hotta, K.; Imamura, Y.; Yamaguchi, Y.; Handa, H. (2010). "Identification of a Primary Target of Thalidomide Teratogenicity". Science. 327 (5971): 1345–1350. Bibcode:2010Sci...327.1345I. doi:10.1126/science.1177319. PMID 20223979. S2CID 17575104.
- ^ a b c Lu G, Middleton RE, Sun H, Naniong M, Ott CJ, Mitsiades CS, Wong KK, Bradner JE, Kaelin WG Jr (2014). "The myeloma drug lenalidomide promotes the cereblon-dependent destruction of Ikaros proteins". Science. 343 (6168): 305–9. Bibcode:2014Sci...343..305L. doi:10.1126/science.1244917. PMC 4070318. PMID 24292623.
- ^ a b c Krönke J, Udeshi ND, Narla A, Grauman P, Hurst SN, McConkey M, Svinkina T, Heckl D, Comer E, Li X, Ciarlo C, Hartman E, Munshi N, Schenone M, Schreiber SL, Carr SA, Ebert BL (2014). "Lenalidomide causes selective degradation of IKZF1 and IKZF3 in multiple myeloma cells". Science. 343 (6168): 301–5. Bibcode:2014Sci...343..301K. doi:10.1126/science.1244851. PMC 4077049. PMID 24292625.
- ^ Shiyanov P, Nag A, Raychaudhuri P (Dec 1999). "Cullin 4A associates with the UV-damaged DNA-binding protein DDB". The Journal of Biological Chemistry. 274 (50): 35309–12. doi:10.1074/jbc.274.50.35309. PMID 10585395.
- ^ Dias DC, Dolios G, Wang R, Pan ZQ (Dec 2002). "CUL7: A DOC domain-containing cullin selectively binds Skp1.Fbx29 to form an SCF-like complex". Proceedings of the National Academy of Sciences of the United States of America. 99 (26): 16601–6. doi:10.1073/pnas.252646399. PMC 139190. PMID 12481031.
- ^ Ohta T, Michel JJ, Schottelius AJ, Xiong Y (Apr 1999). "ROC1, a homolog of APC11, represents a family of cullin partners with an associated ubiquitin ligase activity". Molecular Cell. 3 (4): 535–41. doi:10.1016/s1097-2765(00)80482-7. PMID 10230407.
- ^ Min KW, Hwang JW, Lee JS, Park Y, Tamura TA, Yoon JB (May 2003). "TIP120A associates with cullins and modulates ubiquitin ligase activity". The Journal of Biological Chemistry. 278 (18): 15905–10. doi:10.1074/jbc.M213070200. PMID 12609982.
- ^ Serino G, Deng XW (2003). "The COP9 signalosome: regulating plant development through the control of proteolysis" (PDF). Annual Review of Plant Biology. 54: 165–82. doi:10.1146/annurev.arplant.54.031902.134847. PMID 14502989.
- ^ Chen X, Zhang Y, Douglas L, Zhou P (Dec 2001). "UV-damaged DNA-binding proteins are targets of CUL-4A-mediated ubiquitination and degradation". The Journal of Biological Chemistry. 276 (51): 48175–82. doi:10.1074/jbc.M106808200. PMID 11673459.
- ^ Nishitani H, Shiomi Y, Iida H, Michishita M, Takami T, Tsurimoto T (Oct 2008). "CDK inhibitor p21 is degraded by a proliferating cell nuclear antigen-coupled Cul4-DDB1Cdt2 pathway during S phase and after UV irradiation". The Journal of Biological Chemistry. 283 (43): 29045–52. doi:10.1074/jbc.M806045200. PMC 2662008. PMID 18703516.
외부 링크
- UCSC 게놈 브라우저의 인간 CUL4A 게놈 위치 및 CUL4A 유전자 세부 정보 페이지.
- PDBe-KB에서 UniProt: Q13619(Cullin-4A)에 대한 PDB의 모든 구조 정보 개요.
추가 읽기
- Osaka F, Kawasaki H, Aida N, Saeki M, Chiba T, Kawashima S, Tanaka K, Kato S (Aug 1998). "A new NEDD8-ligating system for cullin-4A". Genes & Development. 12 (15): 2263–8. doi:10.1101/gad.12.15.2263. PMC 317039. PMID 9694792.
- Chen LC, Manjeshwar S, Lu Y, Moore D, Ljung BM, Kuo WL, Dairkee SH, Wernick M, Collins C, Smith HS (Aug 1998). "The human homologue for the Caenorhabditis elegans cul-4 gene is amplified and overexpressed in primary breast cancers". Cancer Research. 58 (16): 3677–83. PMID 9721878.
- Ohta T, Michel JJ, Schottelius AJ, Xiong Y (Apr 1999). "ROC1, a homolog of APC11, represents a family of cullin partners with an associated ubiquitin ligase activity". Molecular Cell. 3 (4): 535–41. doi:10.1016/S1097-2765(00)80482-7. PMID 10230407.
- Hori T, Osaka F, Chiba T, Miyamoto C, Okabayashi K, Shimbara N, Kato S, Tanaka K (Nov 1999). "Covalent modification of all members of human cullin family proteins by NEDD8". Oncogene. 18 (48): 6829–34. doi:10.1038/sj.onc.1203093. PMID 10597293.
- Lyapina S, Cope G, Shevchenko A, Serino G, Tsuge T, Zhou C, Wolf DA, Wei N, Shevchenko A, Deshaies RJ (May 2001). "Promotion of NEDD-CUL1 conjugate cleavage by COP9 signalosome". Science. 292 (5520): 1382–5. Bibcode:2001Sci...292.1382L. doi:10.1126/science.1059780. PMID 11337588. S2CID 14224920.
- Chen X, Zhang Y, Douglas L, Zhou P (Dec 2001). "UV-damaged DNA-binding proteins are targets of CUL-4A-mediated ubiquitination and degradation". The Journal of Biological Chemistry. 276 (51): 48175–82. doi:10.1074/jbc.M106808200. PMID 11673459.
- Yasui K, Arii S, Zhao C, Imoto I, Ueda M, Nagai H, Emi M, Inazawa J (Jun 2002). "TFDP1, CUL4A, and CDC16 identified as targets for amplification at 13q34 in hepatocellular carcinomas". Hepatology. 35 (6): 1476–84. doi:10.1053/jhep.2002.33683. PMID 12029633.
- Liu J, Furukawa M, Matsumoto T, Xiong Y (Dec 2002). "NEDD8 modification of CUL1 dissociates p120(CAND1), an inhibitor of CUL1-SKP1 binding and SCF ligases". Molecular Cell. 10 (6): 1511–8. doi:10.1016/S1097-2765(02)00783-9. PMID 12504025.
- Min KW, Hwang JW, Lee JS, Park Y, Tamura TA, Yoon JB (May 2003). "TIP120A associates with cullins and modulates ubiquitin ligase activity". The Journal of Biological Chemistry. 278 (18): 15905–10. doi:10.1074/jbc.M213070200. PMID 12609982.
- Groisman R, Polanowska J, Kuraoka I, Sawada J, Saijo M, Drapkin R, Kisselev AF, Tanaka K, Nakatani Y (May 2003). "The ubiquitin ligase activity in the DDB2 and CSA complexes is differentially regulated by the COP9 signalosome in response to DNA damage". Cell. 113 (3): 357–67. doi:10.1016/S0092-8674(03)00316-7. PMID 12732143. S2CID 11639677.
- Higa LA, Mihaylov IS, Banks DP, Zheng J, Zhang H (Nov 2003). "Radiation-mediated proteolysis of CDT1 by CUL4-ROC1 and CSN complexes constitutes a new checkpoint". Nature Cell Biology. 5 (11): 1008–15. doi:10.1038/ncb1061. PMID 14578910. S2CID 26070877.
- Wertz IE, O'Rourke KM, Zhang Z, Dornan D, Arnott D, Deshaies RJ, Dixit VM (Feb 2004). "Human De-etiolated-1 regulates c-Jun by assembling a CUL4A ubiquitin ligase" (PDF). Science. 303 (5662): 1371–4. Bibcode:2004Sci...303.1371W. doi:10.1126/science.1093549. PMID 14739464. S2CID 40501515.
- Obuse C, Yang H, Nozaki N, Goto S, Okazaki T, Yoda K (Feb 2004). "Proteomics analysis of the centromere complex from HeLa interphase cells: UV-damaged DNA binding protein 1 (DDB-1) is a component of the CEN-complex, while BMI-1 is transiently co-localized with the centromeric region in interphase". Genes to Cells. 9 (2): 105–20. doi:10.1111/j.1365-2443.2004.00705.x. PMID 15009096. S2CID 21813024.
- Hu J, McCall CM, Ohta T, Xiong Y (Oct 2004). "Targeted ubiquitination of CDT1 by the DDB1-CUL4A-ROC1 ligase in response to DNA damage". Nature Cell Biology. 6 (10): 1003–9. doi:10.1038/ncb1172. PMID 15448697. S2CID 34194616.
- Nag A, Bagchi S, Raychaudhuri P (Nov 2004). "Cul4A physically associates with MDM2 and participates in the proteolysis of p53". Cancer Research. 64 (22): 8152–5. doi:10.1158/0008-5472.CAN-04-2598. PMID 15548678.
- Matsuda N, Azuma K, Saijo M, Iemura S, Hioki Y, Natsume T, Chiba T, Tanaka K, Tanaka K (May 2005). "DDB2, the xeroderma pigmentosum group E gene product, is directly ubiquitylated by Cullin 4A-based ubiquitin ligase complex". DNA Repair. 4 (5): 537–45. doi:10.1016/j.dnarep.2004.12.012. PMID 15811626.