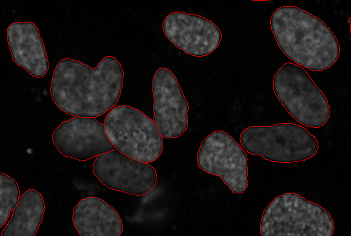
생체 이미지 정보학
Bioimage informatics생물 이미지 정보학은 생물정보학과 컴퓨터 생물학의 하위 분야다.[1] 그것은 특히 세포와 분자 이미지를 대규모로 높은 처리량으로 분석하기 위한 계산 기법의 사용에 초점을 맞추고 있다. 복잡하고 이질적인 이미지와 관련 메타데이터로부터 유용한 지식을 얻는 것이 목표다.
자동화된 현미경은 최소한의 개입으로 많은 수의 이미지를 수집할 수 있다. 이것은 데이터 폭증으로 이어졌고, 이것은 절대적으로 자동 처리를 필요로 한다. 게다가, 그리고 놀랍게도, 이러한 작업들 중 몇몇에 대해, 자동화된 시스템이 인간보다 더 잘 수행할 수 있다는 증거가 있다.[2][3] 또한 자동화된 시스템은 평가가 원하는 결과에 영향을 받을 수 있는 인간 기반 분석과는 달리 편견이 없다.
이러한 데이터 집약적인 문제에서 생물학적 지식을 추출, 비교, 검색 및 관리하기 위한 새로운 이미지 처리, 컴퓨터 비전, 데이터 마이닝, 데이터베이스 및 시각화 기법 개발에 점점 더 초점을 맞추고 있다.[4][5]
데이터 모달성
여러 데이터 수집 시스템과 플랫폼이 사용되는데, 이를 최적으로 처리하기 위해서는 서로 다른 방법이 필요하다.
형광 현미경
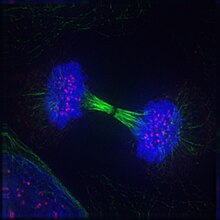
형광 현미경 검사는 살아있는 세포와 고정된 세포 모두에서 세포 이하의 분자를 직접 시각화할 수 있다. 관심 분자는 녹색 형광 단백질, 또 다른 형광 단백질 또는 형광 라벨 항체로 표시된다. 와이드필드, 콘포칼 또는 투포톤 등 여러 종류의 현미경이 정기적으로 사용된다. 대부분의 현미경 시스템은 시계열(영화) 수집도 지원할 것이다.
일반적으로 필터는 각 염료가 별도로 이미징되도록 사용한다(예를 들어, 파란색 필터는 회흐스트를 이미지화한 다음 GFP를 이미지화하기 위해 녹색 필터로 빠르게 전환된다. 소비를 위해 각 채널을 다른 색으로 보여줌으로써 영상이 거짓으로 표시되는 경우가 많지만, 이는 사용된 원래 파장과는 관련이 없을 수도 있다. 어떤 경우에는 원래의 이미지가 보이지 않는 파장으로 획득될 수도 있었다(적외선은 일반적이다).
영상 획득 단계의 선택은 분석에 영향을 미치며 종종 특별한 처리가 필요하다. 콘포칼라 스택은 3D 처리가 필요하며 광야 사이비 스택은 초점 밖의 빛을 제거하기 위해 디지털 디콘볼루션의 혜택을 받는 경우가 많다.
많은 이미지를 자동으로 획득할 수 있는 자동현미경의 등장은 눈으로 분석할 수 없는 이유 중 하나이다(그렇지 않으면 주석이 연구 병목현상이 될 것이다). 자동 현미경을 사용하는 것은 일부 이미지가 초점이 맞지 않을 수 있다는 것을 의미한다(자동 포커스 검색 시스템이 때때로 잘못될 수 있음), 소수의 셀을 포함하거나 파편들로 채워질 수 있다. 따라서 생성된 영상은 올바르게 영상화하고 초점을 맞출 다른 위치를 선택했을 것이므로 운영자가 획득한 영상보다 분석하기가 더 어려울 것이다. 반면에, 운영자는 실험 전에 기대했던 것과 가장 유사한 표현형을 가진 세포만을 선택함으로써 그의 선택에 무의식적인 편견을 도입할 수도 있다.
역사학
조직학은 현미경 아래에서 조직 조각이 얼룩지고 관찰되는 현미경 응용이다(일반적으로 가벼운 현미경, 그러나 전자 현미경도 사용된다).
광현미경을 사용할 때는 형광 이미징의 경우와 달리 일반적으로 표준 컬러 카메라 시스템을 사용하여 이미지를 획득한다. 이는 인간이 흔히 영상을 해석하던 그 분야의 역사를 부분적으로 반영하고 있지만, 또한 표본이 불소포를 자극하기보다는 백색 빛과 모든 채집된 빛으로 조명될 수 있다는 사실 또한 반영하고 있다. 둘 이상의 염료를 사용할 경우, 필요한 사전 처리 단계는 채널을 분리하고 순수 염료 고유 강도의 추정치를 복구하는 것이다.
염색된 단백질의 세포하위 위치는 역사학 영상에서 확인할 수 있는 것으로 나타났다.
만약 의학 진단이 목표라면, 조직학 애플리케이션은 종종 생물 이미지 정보학의 자매 분야인 디지털 병리학이나 자동화된 조직 이미지 분석의 영역에 속하게 될 것이다. 동일한 계산 기법이 적용되는 경우가 많지만, 목표는 연구보다는 의학적으로 적용된다.
중요한 문제
세포하위 위치 분석
세포하위 위치 분석은 이 분야의 초기 문제들 중 하나였다. 감시 모드에서 문제는 영상을 기반으로 주요 세포기관에서 영상을 인식할 수 있는 분류기를 배우는 것이다.
사용되는 방법은 기계 학습을 기반으로 하며 이미지에서 계산된 숫자 형상에 기반한 차별적 분류기를 구축한다. 특징은 할릭 텍스처 특징이나 생물학적 요인을 포착하기 위해 특별히 설계된 특징과 같은 컴퓨터 비전으로부터의 일반적인 특징들(예: 핵 마커를 전형적인 예로 들 수 있는 공동 국지화)이다.
유기농을 식별하는 기본적인 문제에 대해서는 ? 결과보다 우수한 정확도 값을 포함하여 매우 높은 정확도 값을 얻을 수 있다.[2] 이러한 방법들은 기초 세포 생물학 연구에 유용하지만 암세포의 위치가 바뀌는 단백질의 발견에도 적용되어 왔다.[6]
그러나 많은 단백질이 동시에 여러 곳에 국부화되며(혼합된 패턴) 다른 막 결합성분이 아님에도 많은 패턴을 구별할 수 있기 때문에 오르가넬로 분류하는 것은 문제의 제한된 형태다. 이 분야에는 몇 가지 미해결 문제가 있으며 연구가 진행 중이다.
고콘텐츠 선별
자동화된 영상 기술(고내용 선별이라고도 함)을 이용한 높은 처리량 화면은 약물 발견과 기본적인 생물학적 연구 모두를 위한 표준 방법이 되었다. 멀티웰 플레이트, 로보틱스, 자동 현미경 검사를 사용하면, 동일한 검사를 가능한 시약(일반적으로 작은 분자나 RNAi)의 대형 라이브러리에 매우 빠르게 적용할 수 있으며, 단기간에 수천 개의 이미지를 얻을 수 있다. 데이터 생성량이 많아 자동 영상 분석이 필수다.[7]
양수 및 음수 조절을 이용할 수 있을 때, 이 문제는 분류 문제로 접근될 수 있으며, 부세포 위치 분석에 사용되는 것과 동일한 형상 연산 및 분류 기법을 적용할 수 있다.
분할
셀 분할은 아래 많은 분야에서 중요한 하위 문제(실행 가능성 분석에서 셀 카운트만 얻는 것이 목표인 경우 때로는 단독으로 유용하기도 한다) 목표는 다세포 이미지에서 세포의 경계를 식별하는 것이다. 이를 통해 각 셀을 개별적으로 처리하여 파라미터를 측정할 수 있다. 3D 데이터에서는 3D 공간에서 분할을 수행해야 한다.
핵 마커의 이미징은 많은 영상에 걸쳐 공통적이기 때문에 널리 사용되는 프로토콜은 핵을 분할하는 것이다. 이는 핵 측정이 필요한 경우 그 자체로 유용할 수 있으며, 또는 분열을 전체 이미지로 확장하는 분수령을 시드하는 역할을 할 수 있다.
단순 임계값 설정에서 레벨 설정 방법에 이르기까지 모든 주요 분할 방법이 셀 영상에 보고되었다. 여러 가지 이미지 양식과 서로 다른 셀 유형이 있기 때문에 각 셀은 서로 다른 트레이드오프를 의미하므로 이 문제에 대해 허용되는 해결책은 한 가지도 없다.
중요한 절차로서의 세포 영상 분할은 종종 개별 세포의 유전자 발현과 콜로칼레이션 관계 등을 연구하기 위해 사용된다. 이러한 단일 셀 분석의 경우, 셀을 분할하면서 셀의 정체성을 고유하게 결정하는 것이 종종 필요하다. 그러한 인식과제는 종종 계산적으로 비교가 되지 않는다. 세포 라인이 잘 정의된 씨엘레건과 같은 모델 유기체의 경우 이미지 분할과 패턴 인식 방법을 모두 결합해 이미지 분석을 통해 세포 정체성을 명시적으로 인식할 수 있다.[9] 셀의[10] 동시 분할과 인식은 또한 "atlas"나 다른 셀의 사전 정보가 있을 때 이 문제에 대한 보다 정확한 해결책으로 제안되었다. 단세포 분해능에서의 유전자 발현은 이러한 유형의 영상 기반 접근법을 사용하여 얻을 수 있기 때문에, 이러한 방법들을 RNAseq와 같은 다른 단세포 유전자 발현 정량화 방법과 결합하는 것이 가능하다.
추적
추적은 생물 이미지 정보학에서 나타나는 또 다른 전통적인 이미지 처리 문제다. 문제는 영화의 후속 프레임에 등장하는 사물을 연관시키는 것이다. 분할과 마찬가지로 2차원 및 3차원 형태로 문제를 제기할 수 있다.[11]
형광 이미징의 경우, 매우 낮은 대비 영상에 대해 추적을 수행해야 하는 경우가 많다. 시료를 손상시키고 염료를 파괴하는 더 많은 빛을 비추는 방식으로 고대비를 얻으므로 조명은 최소한으로 유지된다. 종종 광자 예산, 즉 샘플의 손상이 너무 커서 더 이상 데이터를 신뢰할 수 없게 되기 전에 이미징에 사용할 수 있는 광자의 수를 생각해 보는 것이 유용하다. 따라서 고대비 영상을 얻으려면 몇 개의 프레임만 사용할 수 있지만 긴 영화의 경우 각 프레임의 대비는 매우 낮을 것이다.
등록
다른 라벨링 방법, 다른 개인, 다른 시점의 샘플 등에 해당하는 것과 같이 다른 성질의 이미지 데이터 샘플을 고려할 때, 더 나은 비교를 위해 이미지를 등록해야 하는 경우가 많다. 한 가지 예는 시간 코스 데이터를 수집할 때 카메라 위치의 사소한 이동을 보정할 수 있도록 후속 프레임의 영상을 등록해야 하는 경우가 많다. 또 다른 예는 모델 동물의 많은 이미지(예: C. 엘레건이나 드로소필라 뇌 또는 마우스 뇌)가 수집될 때, 그들의 패턴을 비교하기 위해 이러한 이미지(예: 동일하거나 다른 뉴런 집단에 해당하는 이미지, 유전자 발현에 공유되거나 다른 이미지 등)를 등록해야 하는 경우가 많다.
의료용 이미지 등록 소프트웨어 패키지는 초기부터 현미경 이미지 등록 어플리케이션에 사용되기 위한 시도였다. 그러나 종종 훨씬 더 큰 이미지 파일 크기와 실험에서 훨씬 더 많은 수의 샘플로 인해 많은 경우 새로운 3D 이미지 등록 소프트웨어를 개발해야 한다. BrainAligner[12]는 신뢰할 수 있는 랜드마크 매칭 전략을 사용하여 3D 변형 및 비선형 등록 프로세스를 자동화하는 데 사용되어 온 소프트웨어다. 그것은 주로 잠자리, 쥐를 포함한 다른 어플리케이션들과 함께 HMI의 Janelia Farm에서 5만개 이상의 3D 표준화된 초파리 뇌 이미지를 생성하는 데 사용되어 왔다.
중요 장소
대학과 연구소의 과학자들로 구성된 컨소시엄은 2005년부터 생물 이미지 정보학에[13] 관한 연례 회의를 조직해 왔다. ISMB 컨퍼런스는 2010년부터 바이오이미징 & 데이터 시각화 트랙을 갖고 있다. 생물정보학 저널은 또한 2012년에 바이오이미지 정보학 트랙을 도입했다. OpenAccess 저널 BMC 생물정보학에는 생물 이미지 분석, 시각화 및 관련 애플리케이션에 대한 섹션이 있다. 다른 계산 생물학 및 생물정보학 저널도 정기적으로 생물 이미지 정보학 작업을 발행한다. 유럽 연합 코스트(European Union Cost) 조치인 NEUBIAS(유럽 바이오 이미지 분석가 네트워크)는 2017년부터 바이오 이미지 분석가 양성 학교와 태그가튼을 비롯해 연례 회의를 조직하고 있다.
소프트웨어
ImageJ, FIJI, CellProfiler, Chunkflow 또는 Bike와 같은 그래픽 사용자 인터페이스를 통해 생체 이미지 정보학 방법을 이용할 수 있도록 하는 몇 가지 패키지가 있다. Vaa3D와 같은 시각화 및 분석 플랫폼은 최근 몇 년 동안 등장했으며 특히 신경과학과 데스크톱 애플리케이션을 위해 대규모 프로젝트에서 모두 사용되어 왔다.
다른 연구자들은 파이썬, C++ 또는 MATLAB와 같은 우수한 컴퓨터 비전 지원을 가진 프로그래밍 언어에 기초하여 그들만의 방법을 개발한다. 파이썬을 위한 마호타스 도서관이 인기 있는 예 중 하나이다. 비록 연구자의 예는 R이 존재하는 것처럼 컴퓨터 비전 지원이 적은 언어 프로그래밍 방법을 개발하였다(예: 트랙뎀).
참고 항목
- Focus stacking 다른 초점 거리를 가진 복수의 영상을 하나로 결합하는 기법.
- 고내용 선별
- 디지털 병리학
- 메디컬 이미징
외부 링크
- Vaa3D: 고성능 다차원 영상 시각화 및 분석
- Bioformats 수십 가지 형식을 지원하는 이미지 파일 IO 엔진
참조
- ^ Peng, H; Bateman A; Valencia A; Wren JD (2012). "Bioimage informatics: a new category in Bioinformatics". Bioinformatics. 28 (8): 1057. doi:10.1093/bioinformatics/bts111. PMC 3324521. PMID 22399678.
- ^ a b Murphy, Robert; Velliste, M.; Porreca, G. (2003). "Robust numerical features for description and classification of subcellular location patterns in fluorescence microscope images". The Journal of VLSI Signal Processing. 35 (3): 311–321. CiteSeerX 10.1.1.186.9521. doi:10.1023/b:vlsi.0000003028.71666.44. S2CID 8134907.
- ^ Nattkemper, Tim; Thorsten Twellmann; Helge Ritter; Walter Schubert (2003). "Human vs. machine: evaluation of fluorescence micrographs". Computers in Biology and Medicine. 33 (1): 31–43. CiteSeerX 10.1.1.324.4664. doi:10.1016/S0010-4825(02)00060-4. PMID 12485628.
- ^ Peng H (September 2008). "Bioimage informatics: a new area of engineering biology". Bioinformatics. 24 (17): 1827–36. doi:10.1093/bioinformatics/btn346. PMC 2519164. PMID 18603566.
- ^ "The quest for quantitative microscopy". Nature Methods. 9 (7): 627. 2012. doi:10.1038/nmeth.2102. PMID 22930824.
- ^ Glory, Estelle; Justin Newberg; Robert F. Murphy (2008). "Automated comparison of protein subcellular location patterns between images of normal and cancerous tissues". Biomedical Imaging: From Nano to Macro, 2008. ISBI 2008. 5th IEEE International Symposium on.
- ^ Shariff, Aabid; Joshua Kangas; Luis Pedro Coelho; Shannon Quinn; Robert F Murphy (2010). "Automated image analysis for high-content screening and analysis". Journal of Biomolecular Screening. 15 (7): 726–734. doi:10.1177/1087057110370894. PMID 20488979.
- ^ Coelho, Luis Pedro; Aabid Shariff; Robert F. Murphy (2009). "Nuclear segmentation in microscope cell images: a hand-segmented dataset and comparison of algorithms". Biomedical Imaging: From Nano to Macro, 2009. ISBI'09. IEEE International Symposium on. IEEE. doi:10.1109/ISBI.2009.5193098. PMC 2901896.
- ^ Long, Fuhui; Peng, H.; Liu, X.; Kim, S.; Myers, E.W (Sep 2009). "A 3D digital atlas of C. elegans and its application to single-cell analyses". Nature Methods. 6 (9): 667–672. doi:10.1038/nmeth.1366. PMC 2882208. PMID 19684595.
- ^ Qu, Lei; Long, F.; Liu, X.; Kim, S.; Myers, E.W.; Peng, H. (2011). "Simultaneous recognition and segmentation of cells: application in C. elegans". Bioinformatics. 27 (20): 2895–2902. doi:10.1093/bioinformatics/btr480. PMC 3187651. PMID 21849395.
- ^ 듀푸, 알렉상드르, 바실리 Shinin, Shahragim Tajbakhsh, 낸시 Guillén-Aghion, J-C.Olivo-Marin, 크리스토프 짐머(2005년)."고 역동적인 3D현미경학에서 더불어 적극적인 표면과 형광 세포를 세분화"(PDF).영상 처리, 14일 IEEETransactions이, 9.를 대신하여 서명함. 1396–1410. doi:10.1109/TIP.2005.852790.2014-03-02.에 있는 원본(PDF)에서 Archived.
- ^ Peng, Hanchuan; Chung, P.; Long, F.; Qu, L.; Jenett, A.; Seeds, A.; Myers, E.W.; Simpson, J.H (2011). "BrainAligner: 3D registration atlases of Drosophila brains". Nature Methods. 8 (6): 493–498. doi:10.1038/nmeth.1602. PMC 3104101. PMID 21532582.
- ^ "Bioimage Informatics Annual Meeting".
- ^ Bruijning, Marjolein; Visser, Marco D.; Hallmann, Caspar A.; Jongejans, Eelke; Golding, Nick (2018). "trackdem: Automated particle tracking to obtain population counts and size distributions from videos in r". Methods in Ecology and Evolution. 9 (4): 965–973. doi:10.1111/2041-210X.12975. ISSN 2041-210X.