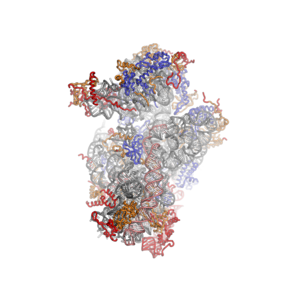
진핵생물 리보솜
Eukaryotic ribosome리보솜은 번역이라고 불리는 단백질의 합성을 촉매하는 크고 복잡한 분자 기계이다.리보솜은 단백질 인코딩 메신저 RNA(mRNA)의 배열에 따라 아미노아실화전달 RNA(tRNA)를 선택하고 아미노산을 폴리펩타이드 사슬에 공유 결합시킨다.모든 유기체의 리보솜은 고도로 보존된 촉매 중심을 공유합니다.하지만, 진핵생물의 리보솜은 원핵생물의 리보솜보다 훨씬 더 크고 더 복잡한 조절과 생물 [1][2]발생 경로를 따릅니다.진핵생물 리보솜은 원핵생물 리보솜보다 침전 속도가 빠르기 때문에 스베드베르크 단위의 침전 계수를 언급하며 80S 리보솜으로도 알려져 있다.진핵생물 리보솜은 침강계수에 따라 지정된 작은 서브유닛(40S)과 큰 서브유닛(60S) 두 개의 불평등한 서브유닛을 가지고 있다.두 서브유닛은 리보솜 RNA(rRNA)로 구성된 골격에 배치된 수십 개의 리보솜 단백질을 포함한다.작은 서브유닛은 tRNA 안티코돈과 mRNA 사이의 상보성을 모니터링하며, 큰 서브유닛은 펩타이드 결합 형성을 촉매한다.
구성.
그들의 원핵생물 호몰로그와 비교하여, 많은 진핵생물 리보솜 단백질은 보존된 코어에 삽입되거나 확장됨으로써 확대됩니다.또한 원핵상동체를 가지지 않는 진핵생물 리보솜의 작고 큰 서브유닛에서 몇 가지 추가 단백질이 발견된다.40S 서브유닛은 원핵생물 16S rRNA와 상동하는 18S 리보솜 RNA(약칭 18S rRNA)를 포함한다.60S 서브유닛은 원핵생물 23S 리보솜 RNA와 상동하는 28S rRNA를 포함한다.또한 23S rRNA의 5' 말단에 해당하는 5.8S rRNA와 짧은 5S rRNA를 포함하고 있으며, 18S와 28S 모두 원핵상대체의 핵심 rRNA 접힘에 여러 개의 삽입을 가지며, 이를 확장 세그먼트라고 한다.고고학 및 박테리아 호몰로그를 포함한 단백질의 자세한 목록은 40S 및 60S 서브유닛에 대한 개별 문서를 참조하십시오.최근의 연구는 리보솜 조성의 이질성, 즉 야생형 효모세포와 배아줄기세포의 핵심 리보솜 단백질 사이의 화학량 측정이 성장 조건과 mRNA당 [3]결합된 리보솜의 수에 좌우된다는 것을 시사한다.
| 진핵생물[4] | 세균[4] | ||
|---|---|---|---|
| 리보솜 | 침강 계수 | 80 S | 70 S |
| 분자량 | 최대 3.2×106 Da | 최대 2.0×106 Da | |
| 직경 | 최대 250 ~ 300 † | 최대 200 † | |
| 대형 서브유닛 | 침강 계수 | 60 S | 50 S |
| 분자량 | 최대 2.0×106 Da | 최대 1.3×106 Da | |
| 단백질 | 46 | 33 | |
| rRNA |
|
| |
| 소형 서브유닛 | 침강 계수 | 40 S | 30 S |
| 분자량 | 최대 1.26×10 Da | ~0.7×106 Da | |
| 단백질 | 33 | 20 | |
| rRNA |
|
|
구조 결정
진핵생물 리보솜의 초기 구조는 전자현미경법에 의해 결정되었다.효모와 포유류의 [6][7]리보솜에 대한[5] 첫 번째 3D 구조는 30-40Ω 분해능에서 얻어졌다.저온전자현미경법에 의한 효모 리보솜의 고분해능 구조는 단백질과 RNA 구조 요소의 [8]식별을 가능하게 했다.최근에는 리보솜 복합체와 [9][10][11]번역에 관여하는 인자에 대해 서브 나노미터 분해능의 구조를 얻었다.1990년대에 원자 분해능으로 최초의 박테리아[12][13][14] 및 고고[15] 리보솜 구조가 결정된 후, 2011년까지 10년이 더 걸렸고, 주로 충분한 [16][17][18]품질의 결정을 얻기 어렵기 때문에 X선 결정학으로 진핵 리보솜의 고해상도 구조가 얻어졌다.Tetrahymena thermophila의 진핵생물 40S 리보솜 구조의 전체 구조와 번역 개시 [16]중 40S 서브유닛과 eIF1의 상호작용에 대한 많은 내용이 발표 및 설명되었다.진핵생물 60S 서브유닛 구조도 eIF6와 [17]복합체 T. 서모필라로부터 결정되었다.효모 사카로미세스 세레비시아에 있는 진핵생물 80S 리보솜의 전체 구조는 3.0A [18]분해능에서 결정학으로 얻어졌다.이러한 구조는 진핵생물 특이적 요소의 정확한 구조, 보편적으로 보존된 핵심과의 상호작용, 그리고 두 개의 리보솜 소단위 사이의 모든 진핵생물 특이적 브릿지를 보여줍니다.
진핵생물 리보솜의 원자 좌표(PDB 파일) 및 구조 인자는 다음과 같은 등록 코드에 따라 단백질 데이터 뱅크(PDB)에 축적되었습니다.
| 복잡한 | 소스 유기체 | 결의안 | PDB 식별자[19] |
|---|---|---|---|
| 80S: Stm1 | 세레비시아에 | 3.0 † | |
| 40S: eIF1 | 테르모필라 | 3.9 † | |
| 60S: eIF6 | 테르모필라 | 3.5 † |
아키텍처
일반적인 기능
리보솜의 일반적인 건축적 특징은 왕국 전체에 [20]보존되어 있습니다.작은 서브유닛의 구조는 머리와 몸통이라는 두 개의 큰 세그먼트로 나눌 수 있습니다.몸의 특징은 왼발과 오른발, 어깨와 플랫폼이다.머리에는 새의 부리를 연상시키는 뾰족한 돌출부가 있다.대형 서브유닛의 특징적인 "크라운뷰"에서 구조용 랜드마크에는 중심돌기, L1-stalk [21][22]및 P-stalk가 포함된다.진핵생물 특이 RNA 및 단백질 요소의 대부분은 40S 및 60S[17] 서브유닛의 용매 노출 측면에서 발견됩니다.서브유닛 계면뿐만 아니라 펩티딜전달효소 중심 및 디코딩 부위와 같은 중요한 기능 영역은 대부분 보존되며, 주변 영역에서는 일부 차이가 관찰된다.주로 RNA와 상호작용하는 원핵생물 리보솜 단백질과는 대조적으로, 진핵생물 특이적 단백질 세그먼트는 다수의 단백질-단백질 상호작용에 관여한다.장거리 상호작용은 진핵생물 특이적 리보솜 단백질의 헬리컬 확장과 여러 진핵생물 리보솜 단백질에 의해 함께 매개되어 단백질 간 베타시트를 형성한다.
리보솜 RNA 코어는 회색 튜브로 표시되고 확장 세그먼트는 빨간색으로 표시됩니다.일반적으로 보존된 단백질은 파란색으로 표시됩니다.이 단백질들은 진핵생물, 고세균 그리고 박테리아에 상동성을 가지고 있다.진핵생물과 고세균 사이에서만 공유되는 단백질은 주황색으로 표시되고 진핵생물에 고유한 단백질은 빨간색으로 표시됩니다.
rRNA와 단백질의 공진화
40S 서브유닛의 구조는 진핵생물 특이단백질(rpS7, rpS10, rpS12 및 RACK1)과 다수의 진핵생물 특이단백질 확장이 [16]작은 서브유닛의 용매 노출 쪽에 위치한다는 것을 밝혀냈다.여기서, 그들은 rRNA 확장 세그먼트의 안정화에 참여한다.또한 rRNA가 단백질 rpS10 및 rpS12로 [16]치환되어 40S 서브유닛의 부리를 개조하였다.40S 서브유닛에서 관찰된 바와 같이 60S 서브유닛(RPL6, RPL22, RPL27, RPL28, RPL29 및 RPL36)의 모든 진핵생물 특이 단백질과 많은 확장이 용매 노출 측에 위치하여 진핵생물 특이 RNA 확장 세그먼트와의 상호작용의 복잡한 네트워크를 형성한다.RPL6, RPL27 및 RPL29는 각각 ES7–ES39, ES31–ES20–ES26 및 ES9–ES12와 RPL28 안정화 확장 세그먼트 ES7A [17]사이의 접점을 중개한다.
유비퀴틴융합단백질
진핵생물에서 작은 서브유닛 단백질 RPS27A(또는 eS31)와 큰 서브유닛 단백질 RPL40(또는 eL40)은 N말단 유비퀴틴 도메인을 가진 융합단백질로서 번역되는 처리 폴리펩타이드이다.두 단백질 모두 리보솜의 중요한 기능 중심 옆에 위치한다. eS31)과 eL40의 삼촌 저장 유비퀴틴 도메인은 각각 디코딩 부위와 번역 인자 결합 부위 근처에 위치할 것이다.이러한 위치들은 단백질 분해 분해 분해가 기능성 [16][17]리보솜의 생산에 필수적인 단계임을 시사한다.실제로, eS31의 핵심과 유비퀴틴 도메인 사이의 링커 돌연변이는 [23]효모에서 치명적이다.
활성 사이트
박테리아, 고고학 및 진핵생물 리보솜 구조를 비교한 결과 활성 부위(일명 펩티딜전달효소 중심(PTC))에서 매우 높은 수준의 보존이 확인되었다.어떤 진핵생물 특이 단백질 요소도 촉매 [17]작용에 직접 참여할 만큼 충분히 가깝지 않다.단, RPL29는 T. thermophila의 활성 부위로부터 18Ω 이내에 투영되며, 진핵생물 특이적 확장은 60S 서브유닛의 [17][21]PTC 부근에서 여러 단백질을 연결하며, 이에 대응하는 50S 단백질은 단일 [15]실체이다.
서브유닛간교량
2개의 리보솜 서브유닛을 가로지르는 컨택은 서브유닛간 브릿지로 알려져 있습니다.진핵생물 리보솜에서는 60S 확장 세그먼트와 [24]단백질에 의해 추가 접촉이 이루어진다.구체적으로는 60S 단백질 RPL19의 C말단 연장은 40S rRNA의 ES6E와 상호작용하고, 60S 단백질 RPL24의 C말단 연장은 40S RPS6 및 RRNA 나선 h10과 상호작용한다.또한 60S 확장 세그먼트 ES31 및 ES41은 각각 40S 서브유닛의 rpS3A(S1) 및 rpS8과 상호작용하고 염기성 25-아미노산 펩타이드 RPL41은 80S 리보솜의 서브유닛 [21][24]계면에 위치하여 양 서브유닛의 RNA 원소와 상호작용한다.
시그널링에 관여하는 리보솜 단백질
두 개의 40S 리보솜 단백질(RACK1 및 RPS6(또는 eS6)은 세포 시그널링에 관련되었다. RACK1은 활성화 단백질 키나제 C(PKC)의 수용체로 처음 기술되었으며,[16] 진핵생물 리보솜의 핵심 구성 요소이며 머리 뒤쪽에 위치한다.신호 전송 패스를 리보솜에 직접 링크할 수도 있지만 관련성이 없는 것으로 보이는 여러 변환 프로세스에서도 역할을 합니다( 참조).리보솜 단백질 eS6는 40S 서브유닛의 오른쪽 발에 위치하며 포유류 목표물인 라파마이신(mTOR)[26] 시그널링에 반응하여 인산화된다.
기능적 측면
번역 개시
단백질 합성은 주로 번역 개시 단계에서 조절된다.진핵생물에서 표준 개시 경로는 적어도 12개의 단백질 개시 인자를 필요로 하며, 그 중 일부는 그 자체로 큰 [27]복합체이다.40S의 구조:eIF1 및 60S: eIF6 복합체는 우선 진핵생물 리보솜과 조절인자 사이의 원자 상호작용에 대한 상세한 통찰력을 제공한다.eIF1은 시작 코돈 선택에 관여하며, eIF6는 입체적으로 서브유닛의 결합을 방해한다.그러나 진핵생물 개시 인자와 리보솜과의 상호작용에 대한 구조적 정보는 제한적이며 주로 호몰로지 모델 또는 저해상도 [28]분석에서 도출된다.원자 수준에서 진핵생물 리보솜과 개시인자 사이의 상호작용에 대한 설명은 조절과정을 기계적으로 이해하는 데 필수적이지만, 개시복합체의 고유한 역학 및 유연성 때문에 중요한 기술적 난제를 나타낸다.포유류의 초기 시작 복합체의 첫 번째 구조는 극저온 전자 현미경법에 [29]의해 이루어졌다.시작 복합체의 다른 구조는 크라이오-EM 기술 [30][31]개선에 의해 곧 뒤따랐다.이러한 구조는 진핵생물에서의 번역 개시 과정을 더 잘 이해하는데 도움을 줄 것이다.
리보솜 단백질의 조절 역할
최근의 유전적 증거는 진핵생물 리보솜의 개별 단백질이 [32][33][34]번역 조절에 직접적으로 기여한다는 것을 시사하는 것으로 해석되었다.그러나, 이러한 해석은 논란의 여지가 있고 일부 연구자들은 리보솜 단백질 유전자의 유전자 변화가 리보솜의 전체 수나 리보솜 생물 생성 [35][36]과정에 간접적으로 영향을 미친다고 제안했다.
단백질 전위 및 표적화
세포에서 그들의 기능을 발휘하기 위해 새로 합성된 단백질은 단백질 표적화 [37]및 전위 시스템에 의해 달성되는 세포 내의 적절한 위치를 목표로 삼아야 한다.성장하는 폴리펩타이드는 큰 서브유닛의 좁은 터널을 통해 리보솜을 떠납니다.60S 서브유닛의 출구터널 주변은 세균성 및 고고성 50S 서브유닛과 매우 유사합니다.추가 요소는 터널 출구 주변의 단백질의 두 번째 계층으로 제한되며, 아마도 전위 [17]기계의 구성 요소와의 보존된 상호작용에 의해 제한된다.진핵생물에서는 [38]표적화 및 전위 장치가 훨씬 더 복잡하다.
리보솜 질환과 암
리보솜병증은 리보솜 단백질이나 rRNA 유전자 또는 리보솜 생물 [39]형성에 관여하는 다른 유전자의 결함으로 인해 발생하는 선천적인 인간 질환이다.예로는 X-연관성 각화증 선천성 각화증(X-DC),[40] 다이아몬드-블랙팬 [41]빈혈(Diamond-Blackfan 빈혈), 트라브 콜린스 증후군(TCS),[41][42] 슈와흐만-보디안-다이아몬드 증후군(SBDS)[39] 등이 있다. SBDS는 GTP1-GPLase에 의한 GTP 가수분해능력에 영향을 미치는 SBTPS 결합 능력에 영향을 미치는 SBS 단백질의 돌연변이에 의해 발생한다.
치료 기회
번역과 신장 주기의 각 단계에서 세균성 번역의 대부분의 임상적으로 관련된 번역 화합물이 억제제[44]방해가 되는 antibacterials지만, 진핵 번역의 억제제 또한 암이나 항진균제 che에 응용 프로그램에 대한 치료 잠재력을 가질 계획에 대한 리보솜 두드러지는 약물 표적.나방에라피.[45] 신장 억제제는 항종양 활성을 '체내'와 '체외'[46][47][48]로 나타낸다.진핵생물 번역 연장의 독성 억제제 중 하나는 글루타리미드 항생물질 시클로헥시미드(CHX)이며, 이는 진핵생물 60S 서브유닛과 공결정화되어 리보솜 E 부위에 결합한다.진핵생물 리보솜의 구조적 특성은 진핵생물 리보솜과 박테리아 리보솜 사이의 차이를 약물의 선택성을 향상시키고 그에 따라 부작용을 줄이기 위해 이용할 수 있는 새로운 항균제 설계를 위한 구조 기반 방법의 사용을 가능하게 할 수 있다.
형성 메커니즘
진핵생물 리보솜은 핵에서 생성되고 조립된다.리보솜 단백질은 핵으로 들어가 4개의 rRNA 가닥과 결합하여 완성된 리보솜을 구성하는 2개의 리보솜 서브유닛(작은 단위와 큰 단위)을 생성한다.리보솜 유닛은 핵공을 통해 핵을 떠나 단백질 합성을 위해 세포질에 한 번 결합합니다.
레퍼런스
- ^ "Difference Between 70S Ribosomes and 80S Ribosomes, RNA, Micromolecules". www.microbiologyprocedure.com. Archived from the original on 2008-09-05. Retrieved 2009-08-06.
- ^ "80S Ribosomes, Eukaryotic Ribosomes, Prokaryotic Ribosomes, Nucleic Acids, Sedimentation Coefficient". www.microbiologyprocedure.com. Archived from the original on 2009-06-23. Retrieved 2009-08-06.
- ^ Slavov, Nikolai; Semrau, Stefan; Airoldi, Edoardo; Budnik, Bogdan; van Oudenaarden, Alexander (2015). "Differential Stoichiometry among Core Ribosomal Proteins". Cell Reports. 13 (5): 865–873. doi:10.1016/j.celrep.2015.09.056. ISSN 2211-1247. PMC 4644233. PMID 26565899.
- ^ a b 값은 Tetrahymena thermophila(PDB: 4V8P) 및 Thermus thermophilus(PDB: 4V5D)의 리보솜에 기초한다.단백질의 정확한 크기, 무게, 수는 유기체마다 다르다.
- ^ Verschoor, A; Warner, JR; Srivastava, S; Grassucci, RA; Frank, J (Jan 1998). "Three-dimensional structure of the yeast ribosome". Nucleic Acids Res. 26 (2): 655–661. doi:10.1093/nar/26.2.655. PMC 147289. PMID 9421530.
- ^ Verschoor, A; Frank, J (Aug 1990). "Three-dimensional structure of the mammalian cytoplasmic ribosome". J Mol Biol. 214 (3): 737–749. doi:10.1016/0022-2836(90)90289-X. PMID 2388265.
- ^ Dube, P; Wieske, M; Stark, H; Schatz, M; Stahl, J; Zemlin, F; Lutsch, G; van Heel, M (Mar 1998). "The 80S rat liver ribosome at 25 A resolution by electron cryomicroscopy and angular reconstitution". Structure. 6 (3): 389–399. doi:10.1016/s0969-2126(98)00040-9. PMID 9551559.
- ^ Spahn, CM; Beckmann, R; Eswar, N; Penczek, PA; Sali, A; Blobel, G; Frank, J (Nov 2001). "Structure of the 80S ribosome from Saccharomyces cerevisiae--tRNA-ribosome and subunit-subunit interactions". Cell. 107 (3): 373–386. doi:10.1016/s0092-8674(01)00539-6. PMID 11701127.
- ^ Halic, M; Gartmann, M; Schlenker, O; Mielke, T; Pool, MR; Sinning, I; Beckmann, R (May 2006). "Signal recognition particle receptor exposes the ribosomal translocon binding site". Science. 312 (5774): 745–747. Bibcode:2006Sci...312..745H. doi:10.1126/science.1124864. hdl:11858/00-001M-0000-0010-842E-9. PMID 16675701. S2CID 7237420.
- ^ Becker, T; Bhushan, S; Jarasch, A; Armache, JP; Funes, S; Jossinet, F; Gumbart, J; Mielke, T; Berninghausen, O; Schulten, K; Westhof, E; Gilmore, R; Mandon, EC; Beckmann, R (Dec 2009). "Structure of monomeric yeast and mammalian Sec61 complexes interacting with the translating ribosome". Science. 326 (5958): 1369–1373. Bibcode:2009Sci...326.1369B. doi:10.1126/science.1178535. PMC 2920595. PMID 19933108.
- ^ Schüler, M; Connell, SR; Lescoute, A; Giesebrecht, J; Dabrowski, M; Schroeer, B; Mielke, T; Penczek, PA; Westhof, E; Spahn, CM (Dec 2006). "Structure of the ribosome-bound cricket paralysis virus IRES RNA". Nat Struct Mol Biol. 13 (12): 1092–1096. doi:10.1038/nsmb1177. hdl:11858/00-001M-0000-0010-8321-7. PMID 17115051. S2CID 8243970.
- ^ Clemons, WM Jr; May, JL; Wimberly, BT; McCutcheon, JP; Capel, MS; Ramakrishnan, V (Aug 1999). "Structure of a bacterial 30S ribosomal subunit at 5.5 A resolution". Nature. 400 (6747): 833–840. doi:10.1038/23631. PMID 10476960. S2CID 14808559.
- ^ Cate, JH; Yusupov, MM; Yusupova, GZ; Earnest, TN; Noller, HF (Sep 1999). "X-ray crystal structures of 70S ribosome functional complexes". Science. 285 (5436): 2095–2104. doi:10.1126/science.285.5436.2095. PMID 10497122.
- ^ Yusupov, MM; Yusupova, GZ; Baucom, A; Lieberman, K; Earnest, TN; Cate, JH; Noller, HF (May 2001). "Crystal structure of the ribosome at 5.5 A resolution". Science. 292 (5518): 883–896. doi:10.1126/science.1060089. PMID 11283358. S2CID 39505192.
- ^ a b Ban, N; Nissen, P; Hansen, J; Moore, PB; Steitz, TA (Aug 2000). "The complete atomic structure of the large ribosomal subunit at 2.4 A resolution". Science. 289 (5481): 905–920. doi:10.1126/science.289.5481.905. PMID 10937989. S2CID 14056415.
- ^ a b c d e f g h i j Rabl, J; Leibundgut, M; Ataide, SF; Haag, A; Ban, N (Feb 2011). "Crystal structure of the eukaryotic 40S ribosomal subunit in complex with initiation factor 1". Science. 331 (6018): 730–736. Bibcode:2011Sci...331..730R. doi:10.1126/science.1198308. hdl:20.500.11850/153130. PMID 21205638. S2CID 24771575.
- ^ a b c d e f g h i j k Klinge, S; Voigts-Hoffmann, F; Leibundgut, M; Arpagaus, S; Ban, N (Nov 2011). "Crystal structure of the eukaryotic 60S ribosomal subunit in complex with initiation factor 6". Science. 334 (6058): 941–948. Bibcode:2011Sci...334..941K. doi:10.1126/science.1211204. PMID 22052974. S2CID 206536444.
- ^ a b Ben-Shem A, Garreau de Loubresse N, Melnikov S, Jenner L, Yusupova G, Yusupov M (February 2011). "The structure of the eukaryotic ribosome at 3.0 Å resolution". Science. 334 (6062): 1524–1529. Bibcode:2011Sci...334.1524B. doi:10.1126/science.1212642. PMID 22096102. S2CID 9099683.
- ^ 크기 제한으로 인해 리보솜 구조는 종종 여러 개의 좌표 파일로 분할됩니다.
- ^ Melnikov, S; Ben-Shem, A; Garreau; de Loubresse, N; Jenner, L; Yusupova, G; Yusupov, M (Jun 2012). "One core, two shells: bacterial and eukaryotic ribosomes". Nat Struct Mol Biol. 19 (6): 560–567. doi:10.1038/nsmb.2313. PMID 22664983. S2CID 6267832.
- ^ a b c Klinge, S; Voigts-Hoffmann, F; Leibundgut, M; Ban, N (May 2012). "Atomic structures of the eukaryotic ribosome". Trends Biochem Sci. 37 (5): 189–198. doi:10.1016/j.tibs.2012.02.007. PMID 22436288.
- ^ Jenner, L; Melnikov, S; de Loubresse, NG; Ben-Shem, A; Iskakova, M; Urzhumtsev, A; Meskauskas, A; Dinman, J; Yusupova, G; Yusupov, M (Dec 2012). "Crystal structure of the 80S yeast ribosome". Curr Opin Struct Biol. 22 (6): 759–767. doi:10.1016/j.sbi.2012.07.013. PMID 22884264.
- ^ Lacombe, T; García-Gómez, JJ; de la Cruz, J; Roser, D; Hurt, E; Linder, P; Kressler, D (Apr 2009). "Linear ubiquitin fusion to Rps31 and its subsequent cleavage are required for the efficient production and functional integrity of 40S ribosomal subunits". Mol Microbiol. 72 (1): 69–84. doi:10.1111/j.1365-2958.2009.06622.x. PMID 19210616. S2CID 33924290.
- ^ a b c Ben-Shem, A; Garreau; de Loubresse, N; Melnikov, S; Jenner, L; Yusupova, G; Yusupov, M (Dec 2011). "The structure of the eukaryotic ribosome at 3.0 Ã resolution". Science. 334 (6062): 1524–1529. doi:10.1126/science.1212642. PMID 22096102. S2CID 9099683.
- ^ Nilsson, J; Sengupta, J; Frank, J; Nissen, P (Dec 2004). "Regulation of eukaryotic translation by the RACK1 protein: a platform for signalling molecules on the ribosome". EMBO Rep. 5 (12): 1137–1141. doi:10.1038/sj.embor.7400291. PMC 1299186. PMID 15577927.
- ^ Palm, L; Andersen, J; Rahbek-Nielsen, H; Hansen, TS; Kristiansen, K; Højrup, P (Mar 1995). "The phosphorylated ribosomal protein S7 in Tetrahymena is homologous with mammalian S4 and the phosphorylated residues are located in the C-terminal region. Structural characterization of proteins separated by two-dimensional polyacrylamide gel electrophoresis". J Biol Chem. 270 (11): 6000–6005. doi:10.1074/jbc.270.11.6000. PMID 7890730.
- ^ Hinnebusch, AG; Lorsch, JR (Oct 2012). "The mechanism of eukaryotic translation initiation: new insights and challenges". Cold Spring Harb Perspect Biol. 4 (10): a011544. doi:10.1101/cshperspect.a011544. PMC 3475172. PMID 22815232.
- ^ Voigts-Hoffmann, F; Klinge, S; Ban, N (Dec 2012). "Structural insights into eukaryotic ribosomes and the initiation of translation". Curr Opin Struct Biol. 22 (6): 768–777. doi:10.1016/j.sbi.2012.07.010. PMID 22889726.
- ^ Hashem, Y.; Georges, A.; Dhote, V.; Langlois, R.; Liao, H. Y.; Grassucci, R. A.; Frank, J. (2013). "Structure of the mammalian ribosomal 43S preinitiation complex bound to the scanning factor DHX29". Cell. 153 (5): 1108–1119. doi:10.1016/j.cell.2013.04.036. PMC 3730827. PMID 23706745.
- ^ Hasshem, Y., De Georges, A., 도테, V., 랑글루아, R., Liao, H. Y., Grassucci, R. A., ... 및 Frank, J. (2013).40S 서브유닛에 접근하기 위해 eIF3를 대체하는 C형 간염 바이러스 형태의 내부 리보솜 진입부위.자연.
- ^ Fernández, I. S.; Bai, X. C.; Hussain, T.; Kelley, A. C.; Lorsch, J. R.; Ramakrishnan, V.; Scheres, S. H. (2013). "Molecular architecture of a eukaryotic translational initiation complex". Science. 342 (6160): 1240585. doi:10.1126/science.1240585. PMC 3836175. PMID 24200810.
- ^ Gilbert, Wendy V. (2011). "Functional specialization of ribosomes?". Trends in Biochemical Sciences. 36 (3): 127–132. doi:10.1016/j.tibs.2010.12.002. ISSN 0968-0004. PMC 3056915. PMID 21242088.
- ^ Topisirovic, I; Sonenberg, N (Apr 2011). "Translational control by the eukaryotic ribosome". Cell. 145 (3): 333–334. doi:10.1016/j.cell.2011.04.006. PMID 21529706.
- ^ Preiss, Thomas (2015). "All Ribosomes Are Created Equal. Really?". Trends in Biochemical Sciences. 41 (2): 121–123. doi:10.1016/j.tibs.2015.11.009. ISSN 0968-0004. PMID 26682497.
- ^ Ferretti, Max B.; Karbstein, Katrin (2019-02-07). "Does functional specialization of ribosomes really exist?". RNA. Cold Spring Harbor Laboratory. 25 (5): 521–538. doi:10.1261/rna.069823.118. ISSN 1355-8382. PMC 6467006. PMID 30733326.
- ^ Farley-Barnes, Katherine I.; Ogawa, Lisa M.; Baserga, Susan J. (2019). "Ribosomopathies: Old Concepts, New Controversies". Trends in Genetics. Elsevier BV. 35 (10): 754–767. doi:10.1016/j.tig.2019.07.004. ISSN 0168-9525. PMC 6852887. PMID 31376929.
- ^ Boehringer, Daniel; Greber, Basil; Ban, Nenad (2011). "Mechanistic insight into co-translational protein processing, folding, targeting, and membrane insertion". Ribosomes: 405–418. doi:10.1007/978-3-7091-0215-2_32. ISBN 978-3-7091-0214-5.
- ^ Bohnsack, Markus T.; Schleiff, Enrico (2010). "The evolution of protein targeting and translocation systems". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1803 (10): 1115–1130. doi:10.1016/j.bbamcr.2010.06.005. PMID 20600359.
- ^ a b Narla, A; Ebert, BL (Apr 2010). "Ribosomopathies: human disorders of ribosome dysfunction". Blood. 115 (16): 3196–3205. doi:10.1182/blood-2009-10-178129. PMC 2858486. PMID 20194897.
- ^ Stumpf, CR; Ruggero, D (Aug 2011). "The cancerous translation apparatus". Curr Opin Genet Dev. 21 (4): 474–483. doi:10.1016/j.gde.2011.03.007. PMC 3481834. PMID 21543223.
- ^ a b Narla, A; Ebert, BL (Oct 2011). "Translational medicine: ribosomopathies". Blood. 118 (16): 4300–1. doi:10.1182/blood-2011-08-372250. PMID 22021450.
- ^ Dauwerse, JG; Dixon, J; Seland, S; Ruivenkamp, CA; van Haeringen, A; Hoefsloot, LH; Peters, DJ; Boers, AC; Daumer-Haas, C; Maiwald, R; Zweier, C; Kerr, B; Cobo, AM; Toral, JF; Hoogeboom, AJ; Lohmann, DR; Hehr, U; Dixon, MJ; Breuning, MH; Wieczorek, D (Jan 2011). "Mutations in genes encoding subunits of RNA polymerases I and III cause Treacher Collins syndrome". Nat Genet. 43 (1): 20–22. doi:10.1038/ng.724. PMID 21131976. S2CID 205357102.
- ^ Finch, AJ; Hilcenko, C; Basse, N; Drynan, LF; Goyenechea, B; Menne, TF; González Fernández, A; Simpson, P; D'Santos, CS; Arends, MJ; Donadieu, J; Bellanné-Chantelot, C; Costanzo, M; Boone, C; McKenzie, AN; Freund, SM; Warren, AJ (May 2011). "Uncoupling of GTP hydrolysis from eIF6 release on the ribosome causes Shwachman-Diamond syndrome". Genes & Development. 25 (9): 917–929. doi:10.1101/gad.623011. PMC 3084026. PMID 21536732.
- ^ Blanchard, SC; Cooperman, BS; Wilson, DN (Jun 2010). "Probing translation with small-molecule inhibitors". Chem. Biol. 17 (6): 633–645. doi:10.1016/j.chembiol.2010.06.003. PMC 2914516. PMID 20609413.
- ^ Pelletier, J.; Peltz, S.W. (2007). "Therapeutic Opportunities in Translation". Cold Spring Harbor Monograph Archive. 48: 855–895.
- ^ Schneider-; Poetsch, T.; Usui, T.; et al. (2010a). "Garbled messages and corrupted translations". Nature Methods. 6 (3): 189–198. doi:10.1038/nchembio.326. PMID 20154667.
- ^ Schneider; Poetsch, T.; Ju, J.; et al. (2010). "2010b. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin". Nat Chem Biol. 6 (3): 209–217. doi:10.1038/nchembio.304. PMC 2831214. PMID 20118940.
- ^ Dang, Y.; et al. (2011). "Inhibition of eukaryotic translation elongation by the antitumor natural product Mycalamide B." RNA. 17 (8): 1578–1588. doi:10.1261/rna.2624511. PMC 3153980. PMID 21693620.
메모들
- "EMDB-1067: Ribosomal 80S-eEF2-sordarin complex from S. cerevisiae - EM Navigator". emnavi.protein.osaka-u.ac.jp. Archived from the original on 2012-12-19. Retrieved 2009-08-06.
- Giavalisco P, Wilson D, Kreitler T, et al. (March 2005). "High heterogeneity within the ribosomal proteins of the Arabidopsis thaliana 80S ribosome". Plant Mol. Biol. 57 (4): 577–591. doi:10.1007/s11103-005-0699-3. hdl:11858/00-001M-0000-0010-86C6-1. PMID 15821981. S2CID 14500573.
- "Ribosomes". www.cs.stedwards.edu. Archived from the original on 2009-03-20. Retrieved 2009-08-06.