투과전자현미경 DNA배열분석
Transmission electron microscopy DNA sequencing투과전자현미경 DNA 염기서열 기술은 투과전자현미경 기술을 이용한 단분자 염기서열 기술이다.이 방법은 1960년대와 70년대에 [1]고안되어 개발되었지만 시료의 손상 정도가 인식되면서 [2]인기를 잃었다.
DNA가 전자현미경으로 선명하게 가시화되기 위해서는 무거운 원자로 표시되어야 한다.또, 라벨 부착 DNA 분자의 촬상에 필요한 분해능을 얻는 데 있어서, 특수한 촬상 기술이나 수차 보정 광학이 유리하다.이론적으로는 투과전자현미경 DNA 시퀀싱이 매우 긴 읽기 길이를 제공할 수 있지만 전자빔 손상 문제는 여전히 남아있을 수 있으며 아직 기술이 상업적으로 개발되지 않았다.
역사
제임스 왓슨과 프란시스 크릭이 DNA의 구조를 추론한 지 몇 년 후, 그리고 프레드릭 생어가 빠른 DNA 염기서열 분석을 위한 첫 번째 방법을 발표하기 거의 20년 전에, 미국인 물리학자 리처드 파인만은 전자 현미경을 언젠가 생물학자들이 DNA 사슬의 염기서열을 볼 수 있도록 하는 도구로 상상했다.파인만은 전자현미경이 충분히 강력해지면 DNA를 포함한 모든 화합물의 원자구조를 시각화할 수 있을 것이라고 믿었다.[3]
1970년 앨버트 크루는 주사 투과 전자현미경으로 고각 고리형 다크필드 이미징(HAADF) 기술을 개발했다.이 기술을 사용하여, 그는 얇은 비정질 탄소막 [4]위에 각각의 무거운 원자를 시각화했다.2010년에 Krivanek와 동료들은 수차 보정 전자 광학 및 저가속 전압의 조합을 포함한 HAADF 방법의 몇 가지 기술적 개선을 보고했다.후자는 빔에 의한 손상을 줄이고 가벼운 원자에 대한 영상 대비를 높일 수 있기 때문에 생물학적 물체를 촬영하는 데 매우 중요합니다.그 결과 질화붕소 단분자층에서의 단일 원자 치환을 [5]촬상할 수 있다.
다수의 화학 및 형광 배열 기술의 발명에도 불구하고, 전자 현미경은 단일 분자 DNA 배열 분석을 수행하는 수단으로 여전히 연구되고 있다.예를 들어, 2012년 하버드 대학, 뉴햄프셔 대학, ZS 제네틱스의 과학자들이 공동으로 이 [6]기술을 사용하여 DNA의 긴 염기서열을 읽을 수 있는 능력을 입증했지만, 전송 전자 현미경 DNA 염기서열 분석 기술은 아직 상업적으로 [7]이용되기에는 멀었다.
원칙
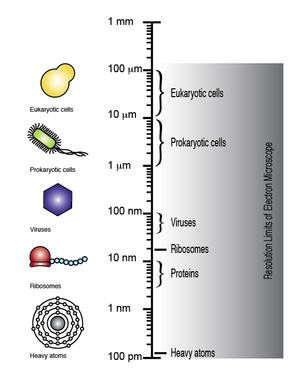
전자현미경은 최대 100pm의 분해능을 얻을 수 있는 능력이 있어 바이러스, 리보솜, 단백질, 지질, 작은 분자, 심지어 단일 원자 같은 미세 생체 분자와 구조를 [8]관찰할 수 있다.
DNA는 전자현미경으로 관찰했을 때 볼 수 있지만, 얻어진 화상의 분해능은 개별 염기의 배열, 즉 DNA 염기서열을 해독할 수 있을 만큼 높지 않다.단, 중원자 또는 금속에 의한 DNA 염기의 차동표기에서는 각각의 염기를 시각화하고 구별할 수 있다.따라서 전자현미경법은 DNA의 [7][9][10][11]염기서열을 결정하기 위해 DNA를 직접 촬영하는 데 사용될 수 있다.
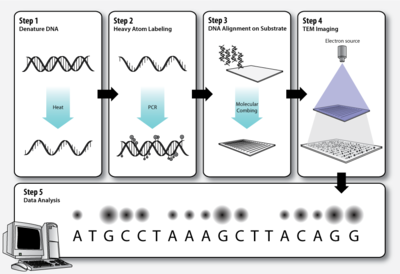
워크플로우
1단계 – DNA 변성
표준중합효소사슬반응(PCR)과 마찬가지로 2가닥 DNA분자를 변성시킨 후 제2가닥을 표식뉴클레오티드로 합성해야 한다.
2단계 – 무거운 원자 라벨링
생물학적 분자(C, H, N, O, P, S)를 구성하는 원소들은 투과전자현미경법으로 개별 원자로 명확하게 가시화되기에는 너무 가볍다(낮은 원자번호, Z).이 문제를 피하기 위해 DNA 베이스에 무거운 원자(높은 Z)로 라벨을 붙일 수 있습니다.각 뉴클레오티드는 투과전자 마이크로그래프로 식별할 수 있도록 특징적인 무거운 라벨을 부착한다.
- ZS Genetics는 브롬(Z=35), 요오드(Z=53) 및 트리클로로메탄(총 Z=63)의 세 가지 무거운 레이블을 사용할 것을 제안합니다.이것들은 마이크로그래프에서 다른 어둡고 밝은 점으로 보일 것이고, 네 번째 DNA 베이스는 라벨이 붙어있지 않을 것이다.
- Halcyon Molecular는 Toste 그룹과 협력하여 퓨린 염기와 피리미딘 염기가 각각 백금 디아민 또는 오스뮴 테트라옥시드 비피리딘으로 기능화할 수 있다고 제안합니다.그런 다음 오스뮴(Z=76), 이리듐(Z=77), 금(Z=79) 또는 우라늄(Z=92)과 같은 중금속 원자는 이러한 작용기와 금속 결합을 형성하여 개별 [12]염기에 라벨을 붙일 수 있습니다.
순서 3 – 기판상의 DNA 정렬
DNA 분자는 얇고 단단한 기판 위에 펼쳐져야만 전자 현미경에서 라벨이 부착된 염기의 순서를 명확하게 볼 수 있습니다.분자 빗질은 DNA 분자를 확장하기 위해 후퇴하는 공기-물 계면의 힘을 이용하는 기술로,[13][14] 한번 건조하면 되돌릴 수 없이 실란 층에 결합됩니다.이것은 고체 기질에서 DNA의 정렬을 달성할 수 있는 하나의 수단이다.
스텝 4 – TEM 이미징
투과전자현미경법(TEM)은 전자빔을 매우 얇은 샘플에 통과시킴으로써 고배율 고해상도 이미지를 생성합니다.원자 분해능은 기존의 TEM에서 입증되었지만 공간 분해능의 추가적인 개선은 현미경 렌즈의 구면 및 색수차를 보정해야 한다.이것은 브라운관과 유사한 방식으로 미세하게 초점을 맞춘 전자 빔으로 물체를 스캔하여 이미지를 얻는 주사 투과 전자 현미경 검사에서만 가능했습니다.그러나, 달성된 분해능 향상은 훨씬 더 높은 빔 강도, 부수적인 샘플 손상 및 관련 이미징 아티팩트에 [15]의한 연구 대상물의 조사와 함께 이루어진다.샘플에 무거운 원자가 포함되어 있는지 가벼운 원자가 포함되어 있는지에 따라 다음과 같은 다양한 이미징 기법이 적용됩니다.
- 고리 모양의 암시장 이미징은 투과 전자 현미경 [5]샘플에서 원자의 핵을 빗나가면서 전자의 산란을 측정합니다.이것은 무거운 원자가 포함된 샘플에 가장 적합합니다. 왜냐하면 그것들은 전자의 산란을 더 많이 일으키기 때문입니다.이 기술은 붕소, 질소, [5]탄소처럼 가벼운 원자를 촬영하는 데 사용되었지만, 그러한 가벼운 원자에 대해서는 신호가 매우 약하다.만약 고리 모양의 암장 현미경을 투과 전자 현미경 DNA 염기서열 분석에 사용한다면, 강력한 신호를 검출할 수 있도록 DNA 염기들에 무거운 원자를 붙일 필요가 있을 것이다.
- 고리형 밝기 필드 이미징은 샘플을 통해 직접 전달되는 전자를 검출하고 원자핵과의 상호작용에 의해 생성되는 파장 간섭을 측정합니다.이 기술은 고리형 다크필드 영상법보다 감도가 높은 광원자를 검출할 수 있다.사실, 결정성 고체의 산소,[16] 질소,[16] 리튬,[17] 그리고[18] 수소는 고리 모양의 밝은 전자장 전자 현미경을 사용하여 이미징되었습니다.따라서, 이론적으로 DNA 사슬에서 원자의 직접적인 이미지를 얻는 것은 가능하지만, DNA의 구조는 결정성 고체보다 훨씬 덜 기하학적이기 때문에, 사전 라벨이 없는 직접적인 영상은 가능하지 않을 수 있다.
스텝 5 – 데이터 분석
전자 현미경상의 어둡고 밝은 점, 다른 라벨이 붙은 DNA 베이스에 대응하는 점은 컴퓨터 소프트웨어에 의해 분석된다.
적용들
투과전자현미경 DNA 염기서열처리는 아직 상업적으로 이용가능하지는 않지만, 이 기술이 언젠가 제공할 긴 판독길이는 다양한 맥락에서 유용하게 될 것입니다.
데노보 게놈 조립체
게놈의 염기서열을 분석할 때는 한 번 읽으면 염기서열을 파악할 수 있을 정도로 짧은 조각으로 분해해야 합니다.그런 다음 이 읽기들은 서로 겹치는 영역을 정렬하여 직소 퍼즐처럼 다시 맞춰야 합니다. 이 과정을 de novo 게놈 어셈블리라고 합니다.시퀀스 플랫폼이 제공하는 읽기 길이가 길수록 겹치는 영역이 길어지고 게놈을 쉽게 조립할 수 있습니다.계산의 관점에서 보면, 마이크로유체 생거 배열은 여전히 참조 게놈 배열이 존재하지 않는 게놈 배열과 조립에 가장 효과적인 방법입니다.읽기 길이가 비교적 길기 때문에 개별 시퀀싱 읽기 간에 상당한 중복이 발생하므로 어셈블리의 통계적 신뢰성이 향상됩니다.또한 긴 Sanger 판독은 반복적인 DNA 배열의 대부분의 영역을 포괄할 수 있으며, 그렇지 않으면 잘못된 정렬을 유발하여 배열 어셈블리를 교란시킬 수 있습니다.그러나 생거 염기서열 분석을 통한 de novo 게놈 조립은 매우 비싸고 시간이 많이 소요됩니다.2세대 염기서열 분석 [19]기술은 비용이 적게 들지만 읽기 길이가 짧기 때문에 일반적으로 de novo 게놈 조립에는 적합하지 않습니다.일반적으로 전송 전자 현미경 DNA 염기서열을 포함한 3세대 [11]염기서열 기술은 낮은 염기서열 비용을 유지하면서 읽기 길이를 개선하는 것을 목표로 한다.따라서 3세대 염기서열 분석 기술이 발전함에 따라 빠르고 저렴한 de novo 게놈 조립이 현실화될 것이다.
풀 하플로타입
하플로타입은 단일 염색체에서 함께 유전되는 일련의 연결된 대립 유전자들이다.DNA 배열은 하플로타입을 구성하는 모든 단일 뉴클레오티드 다형(SNP)을 유전자형으로 만드는데 사용될 수 있다.그러나 짧은 DNA 염기서열 판독은 종종 단계별로 수행될 수 없다. 즉, 헤테로 접합 변형을 올바른 하플로타입에 자신 있게 할당할 수 없다.실제로 짧은 판독 DNA 배열 데이터를 사용한 하플로타이핑은 SNP를 정확하게 식별하기 위해 매우 높은 커버리지(각 DNA 베이스의 평균 50배 이상의 커버리지)를 필요로 하며, 멘델리안 전달을 사용하여 하플로타이핑을 [20]추정할 수 있도록 부모로부터의 추가 시퀀스 데이터를 필요로 한다.전송 전자 현미경 DNA 염기서열 분석 등 긴 판독을 생성하는 염기서열 분석 기술은 한 번의 판독으로 전체 하플로블록을 캡처할 수 있습니다.즉, 하플로타입은 여러 판독치 간에 분리되지 않으며 유전적으로 연결된 대립 유전자가 배열 데이터에 함께 남아 있습니다.그러므로, 긴 독해는 하플로타이핑을 더 쉽고 정확하게 만들어, 집단 유전학 분야에 이롭다.
번호 변형 복사
유전자는 보통 이배체 인간 게놈에 두 개의 복사본으로 존재합니다. 이 표준 복사본 번호에서 벗어난 유전자는 복사본 번호 변형(CNVs)이라고 합니다.카피 번호의 변동은, 양성(통상은 카피 번호 다형이라고 불리는 일반적인 변동) 또는 [21]병원성이 있습니다.CNV는 형광인사이트하이브리제이션(FISH) 또는 비교게놈하이브리제이션(CGH)에 의해 검출된다.결손이 발생하는 특정 중단점을 검출하기 위해 또는 복제 또는 증폭 이벤트에 의해 도입된 게놈 병변을 검출하기 위해 CGH를 타일 어레이(어레이 CGH)를 사용하여 실행할 수도 있고 변이 영역을 시퀀싱할 수도 있다.긴 시퀀싱 판독은 단일 시퀀싱 판독으로 캡처할 경우 증폭된 세그먼트의 방향을 분석할 수 있기 때문에 중복 또는 증폭 분석에 특히 유용합니다.
암
암게노믹스, 즉 종양게노믹스는 높은 처리량의 2세대 DNA 염기서열 분석 기술이 암 게놈 전체의 염기서열에 적용되고 있는 신흥 분야이다.이 짧은 판독 시퀀스 데이터 분석에는 짧은 판독 [22]데이터를 사용하여 de novo 게놈 어셈블리와 관련된 모든 문제가 포함됩니다.게다가, 암 게놈은 종종 이배체이다.[23]기본적으로 대규모 복사 번호 변형인 이러한 수차는 읽기 빈도를 사용하여 복사 [22]번호를 추정하는 2세대 시퀀싱 기술로 분석할 수 있습니다.그러나 판독 시간이 길수록 암 게놈에 존재하는 복사본 번호, 증폭된 영역의 방향 및 SNP를 더 정확하게 파악할 수 있습니다.
마이크로바이옴 배열 분석
마이크로바이옴은 미세환경에 존재하는 미생물의 총집합과 각각의 게놈을 말한다.예를 들어, 100조 개로 추정되는 미생물 세포가 주어진 시간에 [24]인체에 군집한다.인간 마이크로바이옴은 특히 흥미롭다. 왜냐하면 이러한 보상 박테리아는 인간의 건강과 면역에 중요하기 때문이다.지구의 박테리아 게놈의 대부분은 아직 배열이 정해지지 않았다; 마이크로바이옴 염기서열 분석 프로젝트를 진행하려면 광범위한 신규 게놈 조립이 필요하며, 이는 짧은 판독 DNA 염기서열 분석 [25]기술로는 벅찬 전망이다.더 오래 읽으면 새로운 미생물 게놈을 조립하는 데 큰 도움이 될 것이다.
장점과 단점
다른 2세대 및 3세대 DNA 염기서열 기술에 비해 투과전자현미경 DNA 염기서열 기술은 많은 잠재적 강점과 약점을 가지고 있으며, 이는 궁극적으로 미래 DNA 염기서열 기술로서의 유용성과 우수성을 결정할 것이다.
힘
- 더 긴 판독 길이: ZS Genetics는 투과 전자 현미경 DNA 시퀀싱의 잠재적 판독 길이를 [7]1일 17억 염기쌍의 비율로 10,000에서 20,000 염기쌍으로 추정했습니다.이렇게 긴 판독 길이는 다른 [11]응용 프로그램들 중에서 쉽게 de novo 게놈 조립과 하플로타입의 직접 검출을 가능하게 할 것이다.
- 비용 절감:투과 전자 현미경 DNA 염기서열 분석의 비용은 인간 게놈당 5,000달러에서 10,000달러 정도로 추정되며, 이는 고가의 2세대 DNA 염기서열 분석의 대안과 [10]비교된다.
- 디페이징 없음:합성 중 동기성 상실로 인한 DNA 가닥의 디스테이지화는 2세대 염기서열 분석 기술의 주요 문제이다.투과전자현미경 DNA 염기서열결정 및 기타 여러 제3세대 염기서열결정기술에서는 한 [7][11]번에 하나의 분자만 판독되므로 판독치의 동기화가 불필요하다.
- 처리 시간 단축:DNA의 네이티브 단편을 읽는 능력은 전체 게놈 배열의 일반적인 작업 흐름에서 복잡한 템플릿 준비를 불필요한 단계로 만듭니다.그 결과, 턴어라운드 시간을 단축할 [11]수 있습니다.
약점
- 고자본비용: 투과전자현미경 DNA배열분석에 필요한 충분한 분해능을 가진 투과전자현미경은 약 100만달러의 비용이 들기 때문에 이 방법에 의한 DNA배열처리를 추구하려면 [10]상당한 투자가 필요하다.
- 기술적인 어려움:선택적 중원자 라벨링 및 라벨링된 DNA를 기판에 부착 및 고치는 것은 심각한 기술적 [10]과제이다.또한 DNA 시료는 전자현미경의 고진공 및 고에너지 전자의 집속빔에 의한 조사에 대해 안정적이어야 한다.
- 잠재적 PCR 편향 및 아티팩트:PCR은 무거운 원자 또는 금속으로 DNA 가닥을 표시하기 위한 수단으로 전송 전자 현미경 DNA 염기서열 분석에만 사용되고 있지만, 단일 [10]증폭 중에 템플릿 표현에 치우침이나 오류가 발생할 가능성이 있습니다.
다른 시퀀싱 테크놀로지와의 비교
많은 비생저 2세대 및 3세대 DNA 배열 기술은 개인화된 유전의학이 완전히 실현될 수 있도록 스루풋을 높이고 비용을 절감하는 공통의 목적으로 개발되어 왔습니다.
X Prize Foundation(Santa Monica, CA, USA)이 지원하는 1,000만 달러의 Archon X Genomics 상과 National Institute of Health(NIH-NHGRI)의 National Human Genome Research Institute(NIH-NHGRI)가 지원하는 7,000만 달러의 보조금이 모두 급속한 연구 개발을 촉진하고 있습니다.nologies.[7]
다른 접근법, 기술, 전략이 각각의 DNA 염기서열 기술을 정의하는 것이기 때문에, 각각은 각각의 장단점을 가지고 있습니다.다양한 2세대 및 3세대 DNA 배열 기술 간의 중요한 매개변수 비교는 표 1에 제시되어 있다.
| 플랫폼 | 시대 | 읽기 길이(bp) | 정확성. | 인간 게놈당 비용(US$) | 계측기의 비용(US$) | 실행 시간(h/Gbp)[7] |
|---|---|---|---|---|---|---|
| 합성에 의한 대규모 병렬 파이로시퀀싱 | 둘째 | 400–500 | 40개 베이스의 Q20 읽기 길이(400개 베이스의 99%, 이전 베이스의 99% 이상) | 1,000,000 | 500,000 | 75 |
| 합성에 의한 시퀀싱 | 둘째 | 2×75 | 30분기 기준 콜(70% 이상) | 60,000 | 450,000 | 56 |
| 비즈 기반의 대규모 병렬 클론 결합 기반 시퀀스 처리 | 둘째 | 100 | 99.94% | 60,000 | 591,000 | 42 |
| 합성에 의한 대규모 병렬 단일 분자 시퀀싱 | 셋째 | 30–35 | 20×1200 이상에서 99.995 % (원시 오류율: 5 % 이하) | 70,000 | 1,350,000 | ~12 |
| 단일 분자, 합성에 의한 실시간 배열 | 셋째 | 1000–1500 | 15×109로 99.3%(1회 판독 오류율: 15~20%) | – | – | 1 미만 |
| 나노포어 배열법 | 셋째 | 잠재적으로 무제한? | -- | -- | -- | 20을 넘다 |
| 투과전자현미경 단일분자배열법(ZS Genetics, Halcyon Molecular) | 셋째 | 잠재적으로 무제한? | -- | ~10,000 | ~1,000,000 | ~14 |
레퍼런스
- ^ [마이클 비어와 리처드 조벨(1961)]"전자 얼룩 II: 착색된 DNA 분자의 가시성에 관한 전자 현미경 연구" J. Mol. Biol.제3권 1961년 12월 제6호, 717~726페이지, IN3~IN5"]
- ^ [M. Cole et al(1977)] "표지된 폴리뉴클레오티드의 분자 현미경 검사:오스뮴 원자의 안정성" J. Mol. Biol.제117권, 제2호, 1977년 12월 5일, 387~400페이지]
- ^ 파인만 R. (1959)바닥에는 공간이 많이 있다.칼텍 강의.
- ^ Crewe, Albert V; Wall, J.; Langmore, J. (1970). "Visibility of a single atom". Science. 168 (3937): 1338–1340. Bibcode:1970Sci...168.1338C. doi:10.1126/science.168.3937.1338. PMID 17731040. S2CID 31952480.
- ^ a b c Krivanek OL; Chisholm, Matthew F.; Nicolosi, Valeria; Pennycook, Timothy J.; Corbin, George J.; Dellby, Niklas; Murfitt, Matthew F.; Own, Christopher S.; Szilagyi, Zoltan S.; Oxley, Mark P.; Pantelides, Sokrates T.; Pennycook, Stephen J.; et al. (2010). "Atom-by-atom structural and chemical analysis by annular dark-field electron microscopy". Nature. 464 (7288): 571–4. Bibcode:2010Natur.464..571K. doi:10.1038/nature08879. PMID 20336141. S2CID 1331554.
- ^ Bell D, Thomas W, Murtagh K, Dionne C, Grahm A, Anderson J, Glover W (2012). "DNA Base Identification by Electron Microscopy". Microscopy and Microanalysis. 18 (5): 1049–1053. Bibcode:2012MiMic..18.1049B. doi:10.1017/S1431927612012615. PMID 23046798.
- ^ a b c d e f Gupta PK (2008). "Single-molecule DNA sequencing technologies for future genomics research". Trends in Biotechnology. 26 (11): 602–11. doi:10.1016/j.tibtech.2008.07.003. PMID 18722683.
- ^ Campbell NA와 Reice JB. (2002년)생물학 (제6판).샌프란시스코:벤자민 커밍스.ISBN 0-8053-6624-5
- ^ Dear PH (2003). "One by one: Single molecule tools for genomics". Briefings in Functional Genomics and Proteomics. 1 (4): 397–416. doi:10.1093/bfgp/1.4.397. PMID 15239886.
- ^ a b c d e f Xu, M; Fujita, Daisuke; Hanagata, Nobutaka; et al. (2009). "Perspectives and Challenges of Emerging Single-Molecule DNA Sequencing Technologies". Small. 5 (23): 2638–49. doi:10.1002/smll.200900976. PMID 19904762.
- ^ a b c d e Schadt EE; Turner, S.; Kasarskis, A.; et al. (2010). "A window into third-generation sequencing". Human Molecular Genetics. 19 (R2): R227-40. doi:10.1093/hmg/ddq416. PMID 20858600.
- ^ Advanced Sequencing Technology Awards 2010.Genome.gov 를 참조해 주세요.2011-02-25에 취득.
- ^ Bensimon A; Simon, A; Chiffaudel, A; Croquette, V; Heslot, F; Bensimon, D; et al. (1994). "Alignment and sensitive detection of DNA by a moving interface". Science. 265 (5181): 2096–8. Bibcode:1994Sci...265.2096B. doi:10.1126/science.7522347. PMID 7522347.
- ^ Michalet X, et al. (1997). "Dynamic Molecular Combing: Stretching the Whole Human Genome for High-Resolution Studies". Science. 277 (5331): 1518–23. doi:10.1126/science.277.5331.1518. PMID 9278517. S2CID 22699914.
- ^ Haider M; Uhlemann, Stephan; Schwan, Eugen; Rose, Harald; Kabius, Bernd; Urban, Knut; et al. (1998). "Electron microscopy image enhanced". Nature. 392 (6678): 768. Bibcode:1998Natur.392..768H. doi:10.1038/33823. S2CID 205002987.
- ^ a b Okunishi E; Ishikawa, I; Sawada, H; Hosokawa, F; Hori, M; Kondo, Y; et al. (2009). "Visualization of Light Elements at Ultrahigh Resolution by STEM Annular Bright Field Microscopy". Microscopy and Microanalysis. 15 (S2): 164–165. Bibcode:2009MiMic..15S.164O. doi:10.1017/S1431927609093891.
- ^ Oshima Y; Sawada, H.; Hosokawa, F.; Okunishi, E.; Kaneyama, T.; Kondo, Y.; Niitaka, S.; Takagi, H.; Tanishiro, Y.; Takayanagi, K.; et al. (2010). "Direct imaging of lithium atoms in LiV2O4 by spherical aberration-corrected electron microscopy". Journal of Electron Microscopy. 59 (6): 457–61. doi:10.1093/jmicro/dfq017. PMID 20406731.
- ^ Ishikawa R; Okunishi, Eiji; Sawada, Hidetaka; Kondo, Yukihito; Hosokawa, Fumio; Abe, Eiji; et al. (2011). "Direct imaging of hydrogen-atom columns in a crystal by annular bright-field electron microscopy". Nature Materials. 10 (4): 278–281. Bibcode:2011NatMa..10..278I. doi:10.1038/nmat2957. PMID 21317899.
- ^ Shendure J, Ji H (2008). "Next-generation DNA sequencing". Nature Biotechnology. 26 (10): 1135–45. doi:10.1038/nbt1486. PMID 18846087. S2CID 6384349.
- ^ Durbin, Richard M.; Altshuler, David L.; Durbin, Richard M.; Abecasis, Gonçalo R.; Bentley, David R.; Chakravarti, Aravinda; Clark, Andrew G.; Collins, Francis S.; et al. (2010). "A map of human genome variation from population-scale sequencing". Nature. 467 (7319): 1061–73. Bibcode:2010Natur.467.1061T. doi:10.1038/nature09534. PMC 3042601. PMID 20981092.
- ^ Rodruiguez-Revenga L.; Mila, Montserrat; Rosenberg, Carla; Lamb, Allen; Lee, Charles; et al. (2007). "Structural variation in the human genome: the impact of copy number variants on clinical diagnosis". Genetics in Medicine. 9 (9): 600–6. doi:10.1097/GIM.0b013e318149e1e3. PMID 17873648.
- ^ a b Tucker T; Marra, Marco; Friedman, Jan M.; et al. (2009). "Massively Parallel Sequencing: The Next Big Thing in Genetic Medicine". The American Journal of Human Genetics. 85 (2): 142–54. doi:10.1016/j.ajhg.2009.06.022. PMC 2725244. PMID 19679224.
- ^ Torres EM; Williams, B. R.; Amon, A.; et al. (2008). "Aneuploidy: Cells Losing Their Balance". Genetics. 179 (2): 737–46. doi:10.1534/genetics.108.090878. PMC 2429870. PMID 18558649.
- ^ Savage DC (1977). "Microbial Ecology of the Gastrointestinal Tract". Annual Review of Microbiology. 31: 107–33. doi:10.1146/annurev.mi.31.100177.000543. PMID 334036.
- ^ Hamady M, Knight R (2009). "Microbial community profiling for human microbiome projects: Tools, techniques, and challenges". Genome Research. 19 (7): 1141–52. doi:10.1101/gr.085464.108. PMC 3776646. PMID 19383763.