사이트 지향 돌연변이 유발
Site-directed mutagenesis부위 지향적 돌연변이 유발은 유전자 및 유전자 생성물의 DNA 배열에 특정적이고 의도적인 돌연변이를 만드는 데 사용되는 분자 생물학 방법입니다.부위 특이적 돌연변이 유발 또는 올리고뉴클레오티드 지향 돌연변이 유발이라고도 불리는 이것은 DNA, RNA 및 단백질 분자의 구조와 생물학적 활성을 조사하기 위해 그리고 단백질 공학을 위해 사용됩니다.
부위 지향적 돌연변이 유발은 DNA 배열에 돌연변이를 도입함으로써 DNA 라이브러리를 만드는 가장 중요한 실험실 기술 중 하나이다.부위 지향적 돌연변이 유발을 달성하기 위한 수많은 방법이 있지만, 올리고뉴클레오티드 합성 비용이 감소함에 따라 인공 유전자 합성은 부위 지향적 돌연변이 유발의 대안으로 가끔 사용된다.2013년 이후 원핵 바이러스 방어 시스템을 기반으로 한 CRISPR/Cas9 기술의 개발로 게놈 편집도 가능해졌으며, 돌연변이 유발은 비교적 [1]쉽게 생체 내에서 이루어질 수 있다.
역사
방사선 또는 화학적 돌연변이를 사용한 돌연변이 유발의 초기 시도는 부위 특이적이지 않았으며 무작위 [2]돌연변이를 생성했다.뉴클레오티드와 다른 화학물질의 유사체들은 나중에 국부적인 점 [3]돌연변이를 생성하는데 사용되었고, 그러한 화학물질의 예로는 아미노푸린,[4] 니트로소구아니딘,[5] 그리고 [6]비술파이트가 있다.부위 지향적 돌연변이 유발은 1974년 Charles Weissmann의 연구실에서 [7][8]GC의 AT로의 전환을 유도하는 뉴클레오티드4 유사체 N-히드록시시티딘을 사용하여 달성되었다.그러나 이러한 돌연변이 유발 방법은 달성할 수 있는 돌연변이의 종류에 따라 제한되며, 이후의 사이트 지향 돌연변이 유발 방법만큼 구체적이지 않습니다.
1971년 클라이드 허치슨과 마셜 에델은 파지 δX174와 제한핵산가수분해효소의 [9][10]작은 조각으로 돌연변이를 만들 수 있다는 것을 보여주었다.허치슨은 이후 1978년 공동 연구자인 마이클 스미스와 함께 [11]DNA 중합효소와 함께 프라이머 확장법으로 올리고뉴클레오티드를 사용함으로써 부위 지향적 돌연변이 유발에 대한 보다 유연한 접근법을 만들었다.이 과정을 개발한 공로로 마이클 스미스는 나중에 1993년 10월에 캐리 B와 함께 노벨 화학상을 공동 수상했다. 중합효소 연쇄반응을 발명한 멀리스.
기본 메커니즘
기본 절차에서는 짧은 DNA 프라이머를 합성해야 합니다.이 합성 프라이머는 원하는 돌연변이를 포함하고 돌연변이 부위 주변의 템플릿 DNA를 보완하여 관심 유전자의 DNA와 교배할 수 있습니다.변환은 단일 염기 변경(점 변환), 다중 염기 변경, 삭제 또는 삽입일 수 있습니다.단일 가닥 프라이머는 DNA 중합효소를 사용하여 연장되며, DNA 중합효소는 나머지 유전자를 복제합니다.이렇게 복제된 유전자는 돌연변이 부위를 포함하고, 그리고 나서 벡터 안에서 숙주 세포에 도입되어 복제된다.마지막으로 돌연변이가 원하는 돌연변이를 포함하고 있는지 확인하기 위해 DNA 염기서열처리에 의해 선택된다.
접근
단일 프라이머 확장을 사용하는 원래 방법은 돌연변이 수율이 낮기 때문에 비효율적이었다.이 결과물 혼합물은 돌연변이 사슬뿐만 아니라 원래 변이되지 않은 템플릿도 포함하고 돌연변이 자손과 돌연변이하지 않은 자손의 혼합 집단을 생성한다.또, 사용하는 템플릿은 돌연변이 스트랜드가 비메틸화된 상태에서 메틸화되며, 메틸화된 템플릿 DNA에 유리한 미스매치 복구 시스템이 존재하기 때문에 돌연변이 수가 감소한다.그 이후 돌연변이 유발의 효율성을 개선하기 위해 많은 접근법이 개발되어 왔다.
2000년대 초반 이후 대부분 실험실에서 거의 사용되지 않았지만, 많은 방법들이 부위별 돌연변이를 유전자에 도입하는 더 간단하고 쉬운 방법을 가능하게 하기 때문에 부위별 돌연변이 [12]유발에 영향을 미칠 수 있다.
쿤켈법
1985년 토마스 쿤켈은 돌연변이를 [13]선택할 필요성을 줄이는 기술을 도입했다.변이되는 DNA 단편을 M13mp18/19 등의 파지미드에 삽입하여 dUTPase(dut) 및 유라실델글리코시다아제(udg)의 2가지 효소가 결핍된 대장균주로 변환한다.두 효소 모두 dCTP에서 dUTP로의 자발적 탈아미노화에 의해 돌연변이로부터 세균 염색체를 보호하는 DNA 복구 경로의 일부이다.dUTPase 결핍은 dUTP의 분해를 방지하여 세포 내에서 높은 수준의 dUTP를 발생시킵니다.우라실 탈글리코시다아제 결핍은 새로 합성된 DNA에서 우라실이 제거되는 것을 막습니다.따라서 이중 돌연변이 대장균이 파지 DNA를 복제함에 따라 효소기계는 dTTP 대신 dUTP를 잘못 삽입하여 일부 유라실(SSUDNA)을 포함하는 단일 가닥 DNA를 생성할 수 있다.ssUDNA는 배지에 방출된 박테리오파지에서 추출되어 돌연변이 유발용 템플릿으로 사용된다.원하는 돌연변이를 포함한 올리고뉴클레오티드를 프라이머 확장에 이용한다.형성되는 헤테로듀플렉스 DNA는 dUTP를 포함하는 하나의 부모 돌연변이 없는 가닥과 dTTP를 포함하는 돌연변이 가닥으로 구성됩니다.DNA는 야생형 dut 및 udg 유전자를 운반하는 대장균 변종으로 변환됩니다.여기서 우라실을 포함한 부모 DNA 가닥은 분해되어 생성된 DNA의 거의 대부분이 돌연변이 가닥으로 이루어진다.
카세트 돌연변이 발생
다른 방법과는 달리 카세트 돌연변이 유발에는 DNA 중합효소를 사용하는 프라이머 확장이 필요하지 않습니다.이 방법에서는 DNA 조각을 합성하여 플라스미드에 [14]삽입한다.플라스미드 내 부위의 제한효소에 의한 분열과 플라스미드에 대한 관심유전자의 돌연변이를 포함하는 한 쌍의 상보적 올리고뉴클레오티드 결합을 포함한다.보통 플라스미드와 올리고뉴클레오티드를 절단하는 제한효소는 동일하여 플라스미드와 삽입물이 서로 결합할 수 있도록 한다.이 방법은 거의 100%의 효율로 돌연변이를 생성할 수 있지만, 돌연변이를 일으키는 사이트 옆에 적절한 제한 사이트를 사용할 수 있기 때문에 제한됩니다.
PCR 부위 지향 돌연변이 유발
카세트 돌연변이 유발에서 제한 부위의 제한은 올리고뉴클레오티드 "프라이머"와 중합효소 연쇄 반응을 사용하여 극복될 수 있으며, 따라서 두 개의 편리한 제한 부위가 포함된 더 큰 조각이 생성될 수 있다.PCR의 지수적 증폭은 겔 전기영동에 의해 원래의 비변성 플라스미드에서 분리될 수 있는 충분한 양의 원하는 돌연변이를 포함하는 단편을 생성하며, 이는 표준 재조합 분자생물학 기술을 사용하여 원래의 맥락에 삽입될 수 있다.같은 기술에 많은 변형이 있다.가장 간단한 방법은 단편을 생성하기 위해 사용되는 두 개의 올리고뉴클레오티드 중 하나가 돌연변이를 포함하는 단편의 끝 중 하나에 돌연변이 부위를 배치한다.이것은 PCR의 단일 단계를 포함하지만, 매우 긴 프라이머를 사용하지 않는 한 돌연변이 부위 근처에 적절한 제한 부위를 필요로 하는 내재적인 문제를 안고 있다.따라서 다른 변이들은 3-4개의 올리고뉴클레오티드를 사용한다.그 중 2개는 두 개의 편리한 제한 부위를 커버하고 플라스미드에 소화 및 결합할 수 있는 단편을 생성하는 비변형 올리고뉴클레오티드는 그 단편에서 멀리 떨어진 단편을 보완할 수 있다.편리한 제한 사이트.이들 방법에는 연결시킬 최종 프래그먼트가 원하는 돌연변이를 포함할 수 있도록 PCR의 여러 단계가 필요합니다.원하는 변환 및 관련 제한 사이트를 사용하여 fragment를 생성하는 설계 프로세스는 번거로울 수 있습니다.SDM-Assist와[15] 같은 소프트웨어 도구를 사용하면 프로세스를 단순화할 수 있습니다.
전체 플라스미드 돌연변이 유발
플라스미드 조작의 경우, 다른 부위 지향 돌연변이 유발 기술은 주로 매우 효율적이지만 비교적 단순하고 사용하기 쉬우며 키트로 상업적으로 이용 가능한 기술로 대체되었다.이들 기술의 일례는 키크체인지법이다.키크체인지법은 [16]한 쌍의 상보적 돌연변이 유발 프라이머를 사용하여 pfu 중합효소 등의 고충실성 비사슬화 DNA 중합효소를 사용하여 열순환 반응에서 플라스미드 전체를 증폭시킨다.그 반응은 갈라진 원형 DNA를 생성한다.템플릿 DNA는 메틸화 DNA에 특화된 DpnI와 같은 제한 효소에 의한 효소적 소화에 의해 제거되어야 한다.대부분의 대장균 균주에서 생성된 모든 DNA는 메틸화된다. 따라서 대장균에서 생합성되는 템플릿 플라스미드는 소화되는 반면, 체외에서 생성되고 따라서 메틸화되지 않은 돌연변이 플라스미드는 소화되지 않은 상태로 남게 된다.이러한 이중 가닥 플라스미드 돌연변이 유발 방법에서는 열순환 반응을 사용할 수 있지만 PCR에서처럼 DNA를 기하급수적으로 증폭할 필요는 없습니다.대신 증폭은 선형이기 때문에 연쇄반응이 없기 때문에 PCR로 설명하는 것은 부정확합니다.
pfu 중합효소는 높은 확장온도(7070 °C)에서 가닥이 없어질 수 있으므로 권장온도 68 °C에서 확장반응을 수행해야 한다.일부 애플리케이션에서는 이 방법을 통해 여러 개의 프라이머 [17]복사본을 삽입할 수 있습니다.SPRINP라고 불리는 이 방법의 변형은 이 아티팩트를 방지하고 다양한 유형의 사이트 유도 돌연변이 [17]유발에 사용되어 왔습니다.
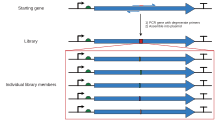
올리고 지시 대상(SMOOT)의 주사 돌연변이 유발과 같은 다른 기술은 플라스미드 돌연변이 [18]유발에서 돌연변이 유발 올리고뉴클레오티드를 반랜덤하게 결합할 수 있다.이 기술은 전체 유전자에 걸쳐 단일 돌연변이에서 포괄적인 코돈 돌연변이까지 플라스미드 돌연변이 유발 라이브러리를 만들 수 있습니다.
생체내 부위 지향 돌연변이 유발 방법
- 델리토 퍼페토[19]
- 트랜스포즈먼트 "팝인 팝업"
- PCR과 1개의 재활용 가능한 마커를 사용한 유전자 직접 삭제 및 부위 특이적 돌연변이 유발
- 긴 상동 영역을 사용한 PCR과 하나의 재활용 가능한 마커에 의한 유전자 직접 삭제 및 부위 특이적 돌연변이 유발
- 합성 올리고뉴클레오티드에[20] 의한 생체 내 부위 지향 돌연변이 유발
크리스퍼
2013년 이후 CRISPR-Cas9 기술의 개발로 다양한 생물체의 게놈에 다양한 돌연변이를 효율적으로 도입할 수 있게 되었다.이 방법은 트랜스포존 삽입 부위가 필요하지 않고 마커를 남기지 않으며, 효율성과 단순성으로 인해 게놈 [21][22]편집에 선호되는 방법이 되었다.
적용들
부위 지향적 돌연변이 유발은 개선되거나 특별한 특성을 가진 합리적으로 설계된 단백질을 생성할 수 있는 돌연변이를 생성하기 위해 사용된다(즉, 단백질 공학).
조사 도구 – DNA의 특정 돌연변이를 통해 DNA 배열 또는 단백질의 기능과 특성을 합리적인 방법으로 조사할 수 있습니다.또한 단백질의 부위 지향 돌연변이 유발에 의한 단일 아미노산 변화는 번역 후 수정의 중요성을 이해하는 데 도움을 줄 수 있다.예를 들어 기질단백질 중 특정 세린(포스포수용체)을 알라닌(포스포비수용체)으로 변경하는 것이 인산기의 부착을 차단하여 인산화 연구를 가능하게 한다.이 접근법은 키나제[23] HIPK2에 의한 단백질 CBP의 인산화 발견에 사용되었습니다.또 다른 포괄적인 접근법은 부위 포화 돌연변이 유발이며, 여기서 하나의 코돈 또는 코돈 세트가 특정 [24]위치에서 모든 가능한 아미노산으로 치환될 수 있습니다.
상업적 용도 – 단백질은 특정 용도에 맞는 돌연변이 형태를 생성하도록 설계될 수 있습니다.예를 들어 일반적으로 사용되는 세탁세제는 표백제에 의해 산화될 수 있는 메티오닌을 가진 야생형인 서브틸리신을 포함할 수 있으며, [25]그 과정에서 단백질의 활성을 현저하게 감소시킨다.이 메티오닌은 알라닌 또는 다른 잔류물로 대체될 수 있으며, 산화에도 내성이 있어 [26]표백제 존재 시 단백질을 활성 상태로 유지할 수 있습니다.
유전자 합성
DNA 올리고뉴클레오티드 합성의 비용이 떨어짐에 따라, 완전한 유전자의 인공 합성은 이제 유전자에 돌연변이를 도입하는 실행 가능한 방법이다.이 방법은 특정 [27]유기체에 최적화하기 위해 유전자의 코돈 사용을 완전히 재설계하는 것을 포함하여 여러 부위에 걸쳐 광범위한 돌연변이 유발을 가능하게 한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Hsu PD, Lander ES, Zhang F (June 2014). "Development and applications of CRISPR-Cas9 for genome engineering". Cell. 157 (6): 1262–78. doi:10.1016/j.cell.2014.05.010. PMC 4343198. PMID 24906146.
- ^ Kilbey, B. J. (1995). "Charlotte Auerbach (1899-1994)". Genetics. 141 (1): 1–5. doi:10.1093/genetics/141.1.1. PMC 1206709. PMID 8536959.
- ^ Shortle, D.; Dimaio, D.; Nathans, D. (1981). "Directed Mutagenesis". Annual Review of Genetics. 15: 265–294. doi:10.1146/annurev.ge.15.120181.001405. PMID 6279018.
- ^ Caras, I. W.; MacInnes, M. A.; Persing, D. H.; Coffino, P.; Martin Jr, D. W. (1982). "Mechanism of 2-aminopurine mutagenesis in mouse T-lymphosarcoma cells". Molecular and Cellular Biology. 2 (9): 1096–1103. doi:10.1128/MCB.2.9.1096. PMC 369902. PMID 6983647.
- ^ McHugh, G. L.; Miller, C. G. (1974). "Isolation and Characterization of Proline Peptidase Mutants of Salmonella typhimurium". Journal of Bacteriology. 120 (1): 364–371. doi:10.1128/JB.120.1.364-371.1974. PMC 245771. PMID 4607625.
- ^ D Shortle & D Nathans (1978). "Local mutagenesis: a method for generating viral mutants with base substitutions in preselected regions of the viral genome". Proceedings of the National Academy of Sciences. 75 (5): 2170–2174. Bibcode:1978PNAS...75.2170S. doi:10.1073/pnas.75.5.2170. PMC 392513. PMID 209457.
- ^ R A Flavell; D L Sabo; E F Bandle & C Weissmann (1975). "Site-directed mutagenesis: effect of an extracistronic mutation on the in vitro propagation of bacteriophage Qbeta RNA". Proc Natl Acad Sci U S A. 72 (1): 367–371. Bibcode:1975PNAS...72..367F. doi:10.1073/pnas.72.1.367. PMC 432306. PMID 47176.
- ^ Willi Müller; Hans Weber; François Meyer; Charles Weissmann (1978). "Site-directed mutagenesis in DNA: Generation of point mutations in cloned β globin complementary DNA at the positions corresponding to amino acids 121 to 123". Journal of Molecular Biology. 124 (2): 343–358. doi:10.1016/0022-2836(78)90303-0. PMID 712841.
- ^ Hutchison Ca, 3.; Edgell, M. H. (1971). "Genetic Assay for Small Fragments of Bacteriophage φX174 Deoxyribonucleic Acid". Journal of Virology. 8 (2): 181–189. doi:10.1128/JVI.8.2.181-189.1971. PMC 356229. PMID 4940243.
{{cite journal}}: CS1 maint: 숫자 이름: 작성자 목록(링크) - ^ Marshall H. Edgell, Clyde A. Hutchison, III, and Morton Sclair (1972). "Specific Endonuclease R Fragments of Bacteriophage X174 Deoxyribonucleic Acid". Journal of Virology. 9 (4): 574–582. doi:10.1128/JVI.9.4.574-582.1972. PMC 356341. PMID 4553678.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Hutchison CA, Phillips S, Edgell MH, Gillam S, Jahnke P, Smith M (September 1978). "Mutagenesis at a specific position in a DNA sequence" (PDF). J. Biol. Chem. 253 (18): 6551–60. doi:10.1016/S0021-9258(19)46967-6. PMID 681366.
- ^ Braman, Jeff, ed. (2002). In Vitro Mutagenesis Protocols. Methods in Molecular Biology. Vol. 182 (2nd ed.). Humana Press. ISBN 978-0896039100.
- ^ Kunkel TA. (1985). "Rapid and efficient site-specific mutagenesis without phenotypic selection". Proceedings of the National Academy of Sciences. 82 (2): 488–92. Bibcode:1985PNAS...82..488K. doi:10.1073/pnas.82.2.488. PMC 397064. PMID 3881765.
- ^ Wells, J. A.; Estell, D. A. (1988). "Subtilisin--an enzyme designed to be engineered". Trends in Biochemical Sciences. 13 (8): 291–297. doi:10.1016/0968-0004(88)90121-1. PMID 3154281.
- ^ Karnik, Abhijit; Karnik, Rucha; Grefen, Christopher (2013). "SDM-Assist software to design site-directed mutagenesis primers introducing "silent" restriction sites". BMC Bioinformatics. 14 (1): 105. doi:10.1186/1471-2105-14-105. ISSN 1471-2105. PMC 3644487. PMID 23522286.
- ^ Papworth, C., Bauer, J. C., Braman, J. and Wright, D. A. (1996). "Site-directed mutagenesis in one day with >80% efficiency". Strategies. 9 (3): 3–4.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ a b Edelheit, O; Hanukoglu, A; Hanukoglu, I (2009). "Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies". BMC Biotechnol. 9: 61. doi:10.1186/1472-6750-9-61. PMC 2711942. PMID 19566935.
- ^ Cerchione, Derek; Loveluck, Katherine; Tillotson, Eric L.; Harbinski, Fred; DaSilva, Jen; Kelley, Chase P.; Keston-Smith, Elise; Fernandez, Cecilia A.; Myer, Vic E.; Jayaram, Hariharan; Steinberg, Barrett E. (16 April 2020). "SMOOT libraries and phage-induced directed evolution of Cas9 to engineer reduced off-target activity". PLOS ONE. 15 (4): e0231716. Bibcode:2020PLoSO..1531716C. doi:10.1371/journal.pone.0231716. ISSN 1932-6203. PMC 7161989. PMID 32298334.
- ^ Storici F.; Resnick MA. (2006). The delitto perfetto approach to in vivo site-directed mutagenesis and chromosome rearrangements with synthetic oligonucleotides in yeast. Methods in Enzymology. Vol. 409. pp. 329–45. doi:10.1016/S0076-6879(05)09019-1. ISBN 9780121828141. PMID 16793410.
- ^ Storici F.; Resnick MA (2003). "Delitto perfetto targeted mutagenesis in yeast with oligonucleotides". Genetic Engineering. 25: 189–207. PMID 15260239.
- ^ Damien Biot-Pelletier and Vincent J. J. Martin (2016). "Seamless site-directed mutagenesis of the Saccharomyces cerevisiae genome using CRISPR-Cas9". Journal of Biological Engineering. 10: 6. doi:10.1186/s13036-016-0028-1. PMC 4850645. PMID 27134651.
{{cite journal}}: CS1 maint: 작성자 파라미터 사용(링크) - ^ Xu S (20 August 2015). "The application of CRISPR-Cas9 genome editing in Caenorhabditis elegans". J Genet Genomics. 42 (8): 413–21. doi:10.1016/j.jgg.2015.06.005. PMC 4560834. PMID 26336798.
- ^ Kovács KA, Steinmann M, Halfon O, Magistretti PJ, Cardinaux JR (November 2015). "Complex regulation of CREB-binding protein by homeodomain-interacting protein kinase 2" (PDF). Cellular Signalling. 27 (11): 2252–60. doi:10.1016/j.cellsig.2015.08.001. PMID 26247811.
- ^ Reetz, M. T.; Carballeira J. D. (2007). "Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes". Nature Protocols. 2 (4): 891–903. doi:10.1038/nprot.2007.72. PMID 17446890. S2CID 37361631.
- ^ Stauffer CE, Etson D (October 10, 1969). "The effect on subtilisin activity of oxidizing a methionine residue". Journal of Biological Chemistry. 244 (19): 5333–8. doi:10.1016/S0021-9258(18)63664-6. PMID 5344139.
- ^ Estell DA, Graycar TP, Wells JA (10 June 1985). "Engineering an enzyme by site-directed mutagenesis to be resistant to chemical oxidation". Journal of Biological Chemistry. 260 (11): 6518–21. doi:10.1016/S0021-9258(18)88811-1. PMID 3922976.
- ^ Yury E. Khudyakov, Howard A. Fields, ed. (25 September 2002). Artificial DNA: Methods and Applications. CRC Press. p. 13. ISBN 9781420040166.
외부 링크
| 라이브러리 리소스 정보 사이트 지향 돌연변이 유발 |