하플로그룹 R-M269
Haplogroup R-M269| 하플로그룹 R-M269 | |
|---|---|
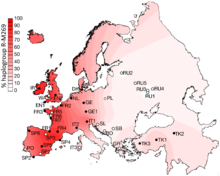 유럽에서 Haplogroup R-M269에 대한 예상 공간 주파수 분포.[1] | |
| 가능한 출발 시간 | 4000[2][3]–10,000 BP |
| 가능한 원산지 | 인도-유럽 이주[4] 관련 동유럽 |
| 조상 | R1b1a1a(R-P297) |
| 후손 | L23; L51/M412, L151/P310; Z2103 |
| 돌연변이 정의 | M269 |
Happlogroup R-M269는 SNP 마커 M269에 의해 정의된 인간 Y-크로모솜 Happlogroup R1b의 하위 표지다. ISOG 2020에 따르면 그것은 유전학적으로 R1b1a1b로 분류된다. 강도 높은 연구를 거쳤으며, 이전에는 R1b1a2(2003~2005년), R1b1c(2005~2008년), R1b1b2(2008~2011년), R1b1a1a2(2011~2020년)로 분류되었다.[5]
R-M269는 특히 서유럽의 유전역사에 관심이 있어 유럽의 가장 흔한 집단이다. 동서로 경사로에서 빈도가 증가한다(폴란드는 22.7%로 추산됨, 웨일스는 92.3%로 추정됨). 약 1억 1천만 명의 유럽 남성(2010년 추정)이 운반하고 있다.[3] 돌연변이 M269의 나이는 대략 4,000년에서 1만년 전으로 추정된다.[6][3]
기원
R-M269는 이전에는 상위 구석기 시대까지 거슬러 올라갔으나,[7] 2010년경에는 약 1만년 전 신석기 혁명이 시작될 무렵에 형성된 것으로 생각되었다.[8][9][10] 그러나 2015년 이후 더욱 최근의 고고학 연구는 유럽 동부에서 온 고대 사냥꾼들 사이에서 유래를 강하게 시사하고 있다.[4][11]
발라레스케 외 연구진(2010년)은 Y-STR 다양성의 패턴을 바탕으로 신석기 혁명에 아나톨리아를 거쳐 근동의 단일 출처와 유럽에 대한 소개를 주장했다. 이 시나리오에서 유럽의 중석기 수렵채집가들은 새로 들어오는 농부들에 의해 거의 대체되었을 것이다. 이와는 대조적으로, 버스비 외 연구진(2012년)은 발라레스케 외 연구진(2010년)의 결과를 확인할 수 없었고, Y-STR 다양성에 근거한 R-M269의 연령을 신뢰할 수 있는 추정치를 낼 수 없었다.[3][12] 게다가, 더 최근의 연구는 초기 유럽 농부들의 Y-DNA가 전형적으로 집단 G2a라는 것을 발견했다.[13]
2015년 연구에 따르면,[4] 하플로그룹 R1b1(*)에 속한 사마라(기일 5640-5555Cal BC)의 수렵채집가는 하플로그룹 R-M269와 R-M478의 조상이었다. 저자들에 따르면, 동유럽 수렵-채집자들에서 R1b의 기저 형태 발생은 집단 R-M269에 "지리적 타당성 있는 출처"를 제공한다. R-M269의 하위 표지는, R-Z2103과 같은, 야마나야 문화 및 관련 집단과 관련된 개인에서 발견된 고대의 DNA에 만연되어 있는 것으로 밝혀졌으며,[4][14] 이 하플로그군의 분산은 소위 "계파 조상"과 적어도 인도-유럽 언어의 일부의 확산과 관련이 있다.[4][15]
하위 표지의 R-P311은 현대 인구에서 실질적으로 서유럽에 국한되어 있다. R-P311은 서유럽에서 발견된 신석기 시대의 고대 DNA에 빠져 있어 현재 분포가 신석기 말기 이후 유럽 내 인구 이동 때문이라는 점을 강하게 시사하고 있다. P311의 3대 서브클라드는 U106(S21), L21(M529, S145), U152(S28)이다. 이들은 각각 로우 국가, 영국 섬, 알프스 산맥에 있는 중심지로 서유럽 내에서 명확한 관절을 보여준다.[16] 이러한 선은 벨 비커 문화의 이베리아 스텝 관련 비이버 스텝 관련 그룹들과 연관되어 있으며, "BC 2500년 이후 서유럽에 스테프 조상이 도래하는 것과 관련된 주요 혈통"인 스텝 관련 조상과 R1b-M269 하위 표지의 관계를 보여준다.[17]
분배
이 글의 사실적 정확성은 논쟁의 여지가 있다. (2020년 8월)(이과 시기 |
유럽 R1b는 R-M269가 지배하고 있다. 그동안 유라시아 중부 전역에서는 전반적으로 낮은 주파수에서 발견됐지만,[18] 퍼머 지역의 바시키르(84.0%), 베이막스키 구(81.0%)[19] 중 상대적으로 높은 주파수를 보였다. 이 마커는 중국과 인도에서 1% 미만의 주파수로 존재한다. 아래 표에는 아시아, 유럽, 아프리카의 지역에서 M269의 주파수가 더 자세히 나와 있다.
빈도는 웨일스 약 92%, 아일랜드 82%, 스코틀랜드 70%, 스페인 68%, 프랑스 60%(노르망디 약 76%) 포르투갈 약 60%,[20] 동부 잉글랜드 약 45%, 독일 50%, 네덜란드 42%, 덴마크 43%, 이탈리아 39% 등이다. 그것은 아일랜드의 일부 지역에서 95%만큼 높다. 또한 알제리 일부 지역에서는 주파수가 10%로 최고조에 달하는 북아프리카의 일부 지역에서도 발견된다.[21] 나미비아의 헤레로 8%에서도 M269가 관측되었다.[22] R-M269 하위 표지는 10세기(~44%)[23]의 카나리아 제도 엘 히에로 푼타 아줄에서 발굴된 고대 관체(빔베프) 화석에서 발견됐다. 서아시아에서는 이란 출신 아시리아 남성의 29.2%에서 R-M269가 보고됐다.[24] Happlogroup R1b1과 그것의 아시아 서브클라드.[25] M269*(xL23)은 발칸 중부를 중심으로 코소보 7.9%,[20] 북마케도니아 5.1%, 세르비아 4.4%로 가장 빈도가 높은 것으로 나타났다. 코소보는 폴란드 2.4%, 바슈코르토스탄 남동부의 바슈키르족 2.4%, 32.2%를 제외하면 대부분 M269*와 L23*의 비중이 큰 지역과 달리 후손 L23* 또는 L23(xM412)의 비율이 11.4%로 높은 점이 눈에 띈다.[20] 특히 이 바슈키르 인구 역시 M269 자매지점 M73의 비율이 23.4%[20]로 높다. 아르메니아 아라라트 계곡에서 실험한 110명 중 5명은 R1b1a2*에 속했고 36~L23*에 속했으며, 알려진 L23의 하위층에는 아무도 속하지 않았다.[26] Trofimova et al. (2015) found a surprising high frequency of R1b-L23 (Z2105/2103) among the peoples of the Idel-Ural. 21 out of 58 (36.2%) of Burzyansky District Bashkirs, 11 out of 52 (21.2%) of Udmurts, 4 out of 50 (8%) of Komi, 4 out of 59 (6.8%) of Mordvins, 2 out of 53 (3.8%) of Besermyan and 1 out of 43 (2.3%) of Chuvash were R1b-L23 (Z2105/2103),[27] 최근 분석한 야마나 유골에서 발견된 R1b의 유형으로 사마라 주와 오렌부르크 주 등이 있다.[28]
특히 서유럽 R1b는 R-M269의 특정 서브클레이드(Sardinia와[20][29] 같은 지역에서 일부 소량의 다른 유형들이 발견됨)에 의해 지배되고 있다. 유럽 내에서 R-M269는 R-M412로, R-L51로도 알려져 있는데, 미레스 외 연구진(2010)에 따르면, "근동, 코카서스, 서아시아에서 사실상 결석"이라고 한다. 이 서유럽 인구는 더욱 R-P312/S116과 R-U106/S21로 나뉘는데, 이는 각각 서부 라인 강 유역과 동부 라인 강 유역에서 확산되는 것으로 보인다. Myres 외 연구진은 R-L23*에서 가장 가까운 친척에 대해 이들이 종종 코카서스, 터키, 그리고 일부 남동유럽 및 회랑지역 인구의 10% 이상이라는 것이 "강제적"이라는 점에 주목한다.
서유럽에서는 "스위스 어퍼 론 계곡의 27%의 예"[20]와 별개로 그것은 존재하지만 일반적으로 훨씬 낮은 수준에 있다. In addition, the sub-clade distribution map, Figure 1h titled "L11(xU106,S116)", in Myres et al. shows that R-P310/L11* (or as yet undefined subclades of R-P310/L11) occurs only in frequencies greater than 10% in Central England with surrounding areas of England and Wales having lower frequencies.[20] 이 R-P310/L11*은 서부 및 남부 발트 해와 (동덴마크에서 10%, 폴란드 북부에서 6%에 이르는) 동스위스 및 주변을 둘러싸고 있는 해안 지역을 제외하고 유라시아 및 북아프리카의 나머지 지역에서는 거의 존재하지 않는다.[20]
| M269([30]R1b1a1a2) |
| ||||||||||||||||||||||||||||||||||||||||||
2009년 670년경 에르고딩(독일 바이에른주 바이에른주)의 초기 중세의 매장지에서 6개의 골격의 대퇴골에서 추출한 DNA는 다음과 같은 결과를 낳았다: 독일, 아일랜드,[31] 미국의 현대 인구에서 가장 가까운 일치점을 가진 하플로그룹 R1b인 반면 2개는 하플로그룹 G2a에 있었다.
M269에 대해 실험하는 인구 연구는 최근 몇 년 동안 더 흔해졌고, 반면 초기 연구에서 이 집단의 남성들은 가능성 있는 것을 추론함으로써 데이터에서만 볼 수 있다. 다음은 M269를 위해 특별히 시험한 대부분의 연구 결과를 요약하여 유럽, 북아프리카, 중동 및 중앙아시아의 분포(전체 인구의 비율)를 중국, 네팔까지 보여준다.
| 나라 | 샘플링 | 견본을 뜨다 | R-M269 | 출처 |
|---|---|---|---|---|
| 웨일스 | 내셔널 | 65 | 92.3% | 발라레스케 외 (2009)[3] |
| 스페인 | 바스크 | 116 | 87.1% | 발라레스케 외 (2009)[3] |
| 아일랜드 | 내셔널 | 796 | 85.4% | 무어 외 (2006)[32] |
| 스페인 | 카탈로니아 | 80 | 81.3% | 발라레스케 외 (2009)[3] |
| 프랑스. | 일레에빌레인 | 82 | 80.5% | 발라레스케 외 (2009)[3] |
| 프랑스. | 오뜨가론 | 57 | 78.9% | 발라레스케 외 (2009)[3] |
| 잉글랜드 | 콘월 | 64 | 78.1% | 발라레스케 외 (2009)[3] |
| 프랑스. | 루아르아틀란티크 | 48 | 77.1% | 발라레스케 외 (2009)[3] |
| 이탈리아 | 투스카니 | 42 | 76.2% | 디 자코모 외 (2003)[33] |
| 프랑스. | 피니스테르 | 75 | 76.0% | 발라레스케 외 (2009)[3] |
| 프랑스. | 바스크 | 61 | 75.4% | 발라레스케 외 (2009)[3] |
| 이탈리아 | 북동부 | 30 | 73.5% | 디 자코모 외 (2003)[33] |
| 스페인 | 동안달루치아 | 95 | 72.0% | 발라레스케 외 (2009)[3] |
| 스페인 | 카스티야 라 만차 | 63 | 72.0% | 발라레스케 외 (2009)[3] |
| 프랑스. | 벤데 | 50 | 68.0% | 발라레스케 외 (2009)[3] |
| 도미니카 공화국 | 내셔널 | 26 | 65.4% | 브라이크 외 (2010)[34] |
| 프랑스. | 배에 드솜메 | 43 | 62.8% | 발라레스케 외 (2009)[3] |
| 잉글랜드 | 레스터셔 주 | 43 | 62.0% | 발라레스케 외 (2009)[3] |
| 이탈리아 | 북동부(라딘) | 79 | 60.8% | 발라레스케 외 (2009)[3] |
| 포르투갈 | 내셔널 | 657 | 59.9% | 벨레자 외 (2006)[35] |
| 이탈리아 | 롬바르디 | 80 | 59.0% | 보티니 외 (2009)[36] |
| 스페인 | 갈리시아 | 88 | 58.0% | 발라레스케 외 (2009)[3] |
| 스페인 | 서안달루치아 | 72 | 55.0% | 발라레스케 외 (2009)[3] |
| 포르투갈 | 남쪽 | 78 | 46.2% | 발라레스케 외 (2009)[3] |
| 덴마크 | 내셔널 | 56 | 42.9% | 발라레스케 외 (2009)[3] |
| 네덜란드 | 내셔널 | 84 | 42.0% | 발라레스케 외 (2009)[3] |
| 아르메니아/터키 | 아라라트 계곡 | 41 | 37.3% | 헤레라 외 연구진(2012년)[26] |
| 러시아 | 바시키르스 | 471 | 34.40% | 로보프(2009)[19] |
| 이탈리아 | 시칠리아 동구 | 246 | 34.14% | 토파넬리 외 (2015년)[37] |
| 이탈리아 | 시칠리아 서 | 68 | 33.0% | 토파넬리 외 (2015년)[37] |
| 독일. | 바이에른 주 | 80 | 32.3% | 발라레스케 외 (2009)[3] |
| 터키 | 반 호 | 33 | 32.0% | 헤레라 외 연구진(2012년) |
| 아르메니아 | 가드만 | 30 | 31.3% | 헤레라 외 연구진(2012년) |
| 이란 | 아시리아인 | 48 | 29.2% | 그루그니, 비올라 외 (2012)[24] |
| 폴란드 | 내셔널 | 110 | 22.7% | 미레스 외 (2007)[38] |
| 슬로베니아 | 내셔널 | 75 | 21.3% | Battaglia. (2008)[39] |
| 코소보 알바니아인 | 내셔널 | 114 | 21.1% | 페리치[40] 2005 |
| 슬로베니아 | 내셔널 | 70 | 20.6% | 발라레스케 외 (2009)[3] |
| 터키 | 중앙 | 152 | 19.1% | 신니오슬루 외 (2004)[41] |
| 북마케도니아의 알바니아인 | 내셔널 | 64 | 18.8% | Battaglia. (2008)[39] |
| 알바니아인 | 내셔널 | 55 | 18.2% | Battaglia. (2008)[39] |
| 크레타 | 내셔널 | 193 | 17.0% | 킹 외 (2008)[42] |
| 이탈리아 | 사르디니아 | 930 | 17.0% | 콘투 외 (2008)[43] |
| 터키 | 사순 | 16 | 15.4% | 헤레라 외 연구진(2012년) |
| 이란 | 북쪽 | 33 | 15.2% | 레게이로 외 (2006)[44] |
| 몰도바 | 268 | 14.6% | 바르자리(2006)[45] | |
| 그리스 | 내셔널 | 171 | 13.5% | 킹 외 (2008)[42] |
| 터키 | 서쪽 | 163 | 13.5% | 신니오슬루 외 (2004)[41] |
| 루마니아 | 내셔널 | 54 | 13.0% | 바르자리(2006)[45] |
| 크로아티아 | 내셔널 | 89 | 12.4% | Battaglia. (2008)[39] |
| 터키 | 동쪽 | 208 | 12.0% | 신니오슬루 외 (2004)[41] |
| 알제리 | 북서(오란 지역) | 102 | 11.8% | 로비노 외 (2008)[46] |
| 러시아 | 로슬라블 (스몰렌스크 주) | 107 | 11.2% | 발라놉스키 외 (2008)[47] |
| 이라크 | 내셔널 | 139 | 10.8% | 알자헤리 외 (2003)[48] |
| 네팔 | 뉴어 | 66 | 10.6% | 게이든 외 (2007)[49] |
| 불가리아 | 내셔널 | 808 | 10.5% | 카라차낙 외(2013년)[50] |
| 세르비아 | 내셔널 | 100 | 10.0% | 벨라레스케 외 (2009)[3] |
| 레바논 | 내셔널 | 914 | 7.3% | 잘루아 외 (2008)[51] |
| 튀니지 | 내셔널 | 601 | 0.3% | 베카다 외 (2013년)[52] |
| 튀니지 | 튀니지 | 139 | 7.2% | 애덤스 외 (2008)[53] |
| 모로코 | 내셔널 | 760 | 3.5% | 베카다 외 (2013년)[52] |
| 리비아 | 내셔널 | 83 | 0.0% | 베카다 외 (2013년)[52] |
| 이집트 | 내셔널 | 360 | 2.9% | 베카다 외 (2013년)[52] |
| 알제리 | 내셔널 | 156 | 7.0% | 베카다 외 (2013년)[52] |
| 알제리 | 알제르스, 티지 오우조우 | 46 | 6.5% | 애덤스 외 (2008)[53] |
| 보스니아헤르체고비나 | 세르비아인 | 81 | 6.2% | 마르자노비치 외 (2005)[54] |
| 이란 | 남쪽 | 117 | 6.0% | 레게이로 외 (2006)[44] |
| 러시아 | 레피예프카 (보로네즈 주) | 96 | 5.2% | 발라놉스키 외 (2008)[47] |
| UAE | 164 | 3.7% | 카데나스 외 (2007)[55] | |
| 보스니아헤르체고비나 | 보스니아어 | 85 | 3.5% | 마르자노비치 외 (2005)[54] |
| 파키스탄 | 176 | 2.8% | 센굽타 외 (2006)[56] | |
| 러시아 | 벨고로드 | 143 | 2.8% | 발라놉스키 외 (2008)[47] |
| 러시아 | 오스트로프 (프스코프 주) | 75 | 2.7% | 발라놉스키 외 (2008)[47] |
| 러시아 | 프리스텐 (쿠르스크 주) | 45 | 2.2% | 발라놉스키 외 (2008)[47] |
| 보스니아헤르체고비나 | 크로아츠 | 90 | 2.2% | 마르자노비치 외 (2005)[54] |
| 카타르 | 72 | 1.4% | 카데나스 외 (2007)[55] | |
| 중국 | 128 | 0.8% | 센굽타 외 (2006)[56] | |
| 인도 | 여러 가지 | 728 | 0.5% | 센굽타 외 (2006)[56] |
| 크로아티아 | 오시젝 | 29 | 0.0% | Battaglia. (2008)[39] |
| 예멘 | 62 | 0.0% | 카데나스 외 (2007)[55] | |
| 티베트 | 156 | 0.0% | 게이든 외 (2007)[49] | |
| 네팔 | 타망 | 45 | 0.0% | 게이든 외 (2007)[49] |
| 네팔 | 카트만두 | 77 | 0.0% | 게이든 외 (2007)[49] |
| 일본. | 23 | 0.0% | 센굽타 외 (2006)[56] |
서브클레이드
이 글의 사실적 정확성은 논쟁의 여지가 있다. (2020년 8월)(이과 시기 |
R1b1a1a2a(R-L23)
R-L23* (R1b1a1a2a*)는 현재 유럽에서 가장 흔하게 발견되고 있다. 아나톨리아, 코카서스.
R1b1a1a2a1(R-L51)
R-L51* (R1b1a1a2a1*)은 현재 프랑스 남부와 이탈리아 북부를 중심으로 한 지리적 클러스터에 집중되어 있다.
R1b1a1a2a1a(R-L151)
R-L151 (L151/PF6542, CTS7650/FGC44/PF6544/S1164, L11, L52/PF6541, P310/PF6546/S129, P311/PF6545/S128) also known as R1b1a1a2a1, and its subclades, include most males with R1b in Western Europe.
R1b1a1a1a2a1a1a1(R-U106)
이 하위 표지는 S21과 M405로도 알려진 SNP U106의 존재에 의해 정의된다.[8][57] 그것은 유럽에서 R1b의 25% 이상을 차지하는 것으로 보인다.[8] 전체 인구의 비율을 따져보면 진원지는 인구의 44%를 차지하는 튀김랜드다.[58] 총 인구수로 보면, 그것의 진앙지는 R1을 합친 60%로 구성된 중부 유럽이다.[58] Happlogroup R-Z18 참조
| U106/S21/M405 |
| |||||||||||||||||||||||||||
R1b의 이 하위 표는 유전적 족보학자들 사이에서 자주 논의되지만, 다음 표는 지금까지 2007년 미레스 외 연구진 및 심스 외 연구진에서 발표된 동료 검토 결과를 나타낸다.[38][57]
| 인구 | 표본크기 | R-M269 | R-U106 | R-U106-1 |
|---|---|---|---|---|
| 오스트리아 | 22 | 27% | 23% | 0.0% |
| 중남미 | 33 | 0.0% | 0.0% | 0.0% |
| 체코 | 36 | 28% | 14% | 0.0% |
| 덴마크 | 113 | 34% | 17% | 0.9% |
| 동유럽[38] | 44 | 5% | 0.0% | 0.0% |
| 잉글랜드[38] | 138 | 57% | 20% | 1.4% |
| 프랑스[38] | 56 | 52% | 7% | 0.0% |
| 독일[38] | 332 | 43% | 19% | 1.8% |
| 아일랜드[38] | 102 | 80% | 6% | 0.0% |
| 이탈리아[20] | 34 | 53% | 6% | 0.0% |
| 조던[38] | 76 | 0.0% | 0.0% | 0.0% |
| 중동[38] | 43 | 0.0% | 0.0% | 0.0% |
| 네덜란드[38] | 94 | 54% | 35% | 2.1% |
| 오세아니아[38] | 43 | 0.0% | 0.0% | 0.0% |
| 오만[38] | 29 | 0.0% | 0.0% | 0.0% |
| 파키스탄[38] | 177 | 3% | 0.0% | 0.0% |
| 팔레스타인[38] | 47 | 0.0% | 0.0% | 0.0% |
| 폴란드[38] | 110 | 23% | 8% | 0.0% |
| 러시아[38] | 56 | 21% | 5.4% | 1.8% |
| 슬로베니아[38] | 105 | 17% | 4% | 0.0% |
| 스위스[38] | 90 | 58% | 13% | 0.0% |
| 터키[38] | 523 | 14% | 0.4% | 0.0% |
| 우크라이나[38] | 32 | 25% | 9% | 0.0% |
| 미국[38] | 58 | 5% | 5% | 0.0% |
| 미국(유럽) | 125 | 46% | 15% | 0.8% |
| 미국(아프리카) | 118 | 14% | 2.5% | 0.8% |
R-P312
R-P312(또는 R-S116)로 더 잘 알려진 R1b1a1a2a2a2는 R-U106과 함께 유럽에서 가장 흔한 R-M269 유형 중 하나이다. 미레스 외는 그것을 라인 유역의 서쪽에서 발원하고 퍼지는 것으로 묘사했다.[20]
R-P312는 그것의 복잡한 내부 구조에 관한 중요하고 지속적인 연구의 대상이었다.
| P312 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||
R-DF27
R-M153
R-M153은 주로 바스크와 가스콘스에서 발견된 R-DF27의 하위 표지로, 이 중 Y-DNA 풀의 상당 부분을 대표하지만,[53][59] 일반적으로 이베리아인들 사이에서도 가끔 발견된다. 처음 위치했을 때(Bosch 2001[60]) H102로 설명되었으며, 7개의 바스크와 1개의 안달루시아인이 포함되어 있었다.
R-M167은 M167 마커의 존재에 의해 정의된 R-DF27의 하위 표지다. 이 마커(현재의 happlogroup 명명법이 존재하기 훨씬 이전에)를 시험한 첫 번째 작가는 1999년 허레스였는데, 그는 다양한 모집단에서 1158명의 남성들을 시험했다.[61] 그는 바스크 (13/117:11%)와 카탈루냐 (7/32:22%) 사이에서 비교적 흔하다는 것을 발견했다. 다른 프랑스, 영국, 스페인, 베아나, 독일에서도 발견되었다.
2000년에는 다양한 populations[62]또한 동일한 마커를 시험하고, haplogroup Hg22의 이름을 짓고 다시 바스크인(19%)주로 중, 프랑스(5%)들의 낮은 주파수에서 발견되었다 3616 남자들에 대한 연구에서 루마니아인, Slovenians, D중 로서(알.,, Bavarians(3%), 스페인 사람,(2%)남부 포르투갈어(2%), 단일 사건들에utch, 벨기에인 및 영어.::2001년에 보쉬는 이 마커를 바스크 5개와 카탈로니아 5개로 H103이라고 표현했다.[60] 추가 지역 연구들은 바스크들 사이에서뿐만 아니라 아스투리아스, 칸타브리아, 갈리시아에서도 상당한 양을 발견했다.[60] 아조레스에서 발생한 사건들이 보고되었다.[citation needed] 2008년 로페스-파라와[59] 아담스의 [53]두 연구 논문은 각각 피레네족과 동부 이베리아족의 전부 또는 대부분과 강한 연관성을 확인했다.
2006년 포르투갈에 대한 대규모 연구에서 657명의 남성들이 테스트를 받은 베레자 외 연구진은 1.5%~3.5%에서 [35]모든 주요 지역에서 유사한 낮은 수준을 확인했다.
R-L165
이 하위 표지는 L165라고도 하는 S68 마커의 존재에 의해 정의된다. 영국, 스칸디나비아, 스코틀랜드에서 발견된다(이 나라에서는 주로 북섬과 외딴섬에서 발견된다). 따라서 바이킹과 함께 영국 섬에 도착했다는 설이 제기되었다.[63]
R-U152
R-U152는 S28이라고도 하는 U152 마커의 존재에 의해 정의된다.[8] 그것의 발견은 EthnoAncestry에[64] 의해 2005년에 발표되었고 이후 Sims 등이 독립적으로 확인했다. (2007).[57] 미레스 외는 이 격언이 "스위스, 이탈리아, 프랑스, 서폴란드에서 가장 빈번(20~44%)하며 영국과 독일의 일부 지역에서는 추가 사례가 15%를 넘는다"고 보도했다.[38] 마찬가지로 크로시아니 외 연구진(2010년)[65]은 북부, 중부 이탈리아 및 프랑스에서 주파수 피크를 보고하였다. 오스트리아 티롤에서 135명의 남성 표본 중 9명이 U152/S28 양성반응을 보였다.[66] 이러한 명백한 핵심 영역과는 거리가 먼 미레스 외 연구진은 또한 북 바슈코르토스탄의 하위 인구 집단을 언급하고 있는데, 이 지역에서 실험 대상인 70명의 남성 중 71%가 R-U152에 속한다. 그들은 이것을 고립된 설립자 효과의 결과라고 제안한다.[20] 킹 외 연구진(2014년)은 남성 라인에서 제5대 보포트 공작 헨리 서머셋의 4명의 살아있는 후손이 U-152 양성반응을 보였다고 보고했다.[67] 유럽 중부 벨 비커, 할슈타트, 투물루스 문화권으로부터의 고대 표본들이 이 하위 표지에 속했다.[68][69][70]
R-L21
R-L21은 R-M529와 R-S145로도 알려져 있다.[8] 미레스 등은 아일랜드, 스코틀랜드, 웨일즈(전체 남성 인구의 25~50%)에서 가장 흔하다고 보고한다.[20]
R-L159.2 R-L21 내의 이 하위 표지는 마커 L159의 존재에 의해 정의되며, Haplogroup I2a1(L159.1) 내부에 존재하는 병렬 돌연변이 때문에 L159.2로 알려져 있다. L159.2는 라이긴 왕과 디아마리트 맥 무르차다 왕과 관련이 있는 것으로 보인다. 맨 섬과 헤브리데스를 비롯한 아일랜드 해안을 비롯해 노르웨이, 서부 및 남부 스코틀랜드, 북부 및 남부 잉글랜드, 북서 프랑스, 덴마크 북부 등지에서 볼 수 있다.[71]
R-L193 R-L21 내의 이 하위 표지는 마커 L193의 존재에 의해 정의된다. 이 표식을 가진 많은 성들은 지리적으로 스코틀랜드의 서부 "경계 지역"과 연관되어 있다. 몇몇 다른 성들은 하이랜드 연대를 가지고 있다. R-L193은 지난 2000년 안에 태어날 가능성이 있는 비교적 젊은 부속 건물이다.
R-L226 R-L21 내의 이 하위 표지는 S168이라고도 하는 마커 L226의 존재에 의해 정의된다. 흔히 아일랜드식 3종이라고 불리는 이것은 중부 서부 아일랜드에 집중되어 있으며 달 gCais와 연관되어 있다.[72]
R-DF21 R-L21 내의 이 하위 표지는 S192라는 마커 DF21의 존재에 의해 정의된다. 전체 L21 남성의 약 10%를 차지하고 있으며, c.3000살이다.[73]
R-L371 R-L21 내의 이 하위 표지는 웨일스 모달이라 불리며 고대 웨일스 왕과 왕자와 관련된 표식자 L371의 존재에 의해 정의된다.[74][75][76]
참고 항목
참조
- ^ Valaresque 외 연구진(2010), 그림 1B: "보간 공간 주파수 표면으로 표시된 hgR1b1b2의 happlogroup 주파수 지리 분포. 채워진 원은 마이크로 위성 데이터와 TMRCA 추정치가 이용 가능한 모집단을 나타낸다. 채워지지 않은 원은 R1b1b2 주파수만을 나타내기 위해 포함된 모집단을 나타낸다. 모집단 코드는 표 1에 정의되어 있다."
- ^ "개별 모집단에 대한 평균 추정치는 다양하지만(표 2) 가장 오래된 값은 중앙 터키(7,989y [95% 신뢰 구간(CI: 5,661–11,014]), 최연소(5,460y [3,764–7,777])이다. 전체 데이터 세트의 평균 추정치는 6,512y(95% CI: 4,577–9,063년)이며, 성장률은 1.95%(1.02%–3.30%)이다. 따라서 신석기 시대 이전에는 시작할 수 없었던 급속한 팽창의 명확한 증거를 보게 된다." 발라레스케 외(2010년).
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa Balaresque P, Bowden GR, Adams SM, Leung HY, King TE, Rosser ZH, et al. (January 2010). Penny D (ed.). "A predominantly neolithic origin for European paternal lineages". PLOS Biology. 8 (1): e1000285. doi:10.1371/journal.pbio.1000285. PMC 2799514. PMID 20087410.
- ^ a b c d e Haak W, Lazaridis I, Patterson N, Rohland N, Mallick S, Llamas B, et al. (June 2015). "Massive migration from the steppe was a source for Indo-European languages in Europe". Nature. 522 (7555): 207–11. arXiv:1502.02783. Bibcode:2015Natur.522..207H. doi:10.1038/nature14317. PMC 5048219. PMID 25731166.
- ^ 각 연도의 ISOGG 트리에 따른 날짜.
- ^ "개별 모집단에 대한 평균 추정치는 다양하지만(표 2) 가장 오래된 값은 중앙 터키(7,989y [95% 신뢰 구간(CI: 5,661–11,014]), 최연소(5,460y [3,764–7,777])이다. 전체 데이터 세트의 평균 추정치는 6,512y(95% CI: 4,577–9,063년)이며, 성장률은 1.95%(1.02%–3.30%)이다. 따라서 신석기 시대 이전에는 시작할 수 없었던 급속한 팽창의 명확한 증거를 보게 된다." 발라레스케 외(2010년).
- ^ Semino O, Passarino G, Oefner PJ, Lin AA, Arbuzova S, Beckman LE, et al. (November 2000). "The genetic legacy of Paleolithic Homo sapiens sapiens in extant Europeans: a Y chromosome perspective". Science. 290 (5494): 1155–9. Bibcode:2000Sci...290.1155S. doi:10.1126/science.290.5494.1155. PMID 11073453.
- ^ a b c d e 국제유전계보학회(ISOGG) – Y-DNA Happlogroup R과 그 하위클라데스
- ^ Arredi B, Poloni ES, Tyler-Smith C (2007). "The peopling of Europe". In Crawford, Michael H. (ed.). Anthropological genetics: theory, methods and applications. Cambridge, UK: Cambridge University Press. p. 394. ISBN 978-0-521-54697-3.
- ^ Cruciani F, Trombetta B, Antonelli C, Pascone R, Valesini G, Scalzi V, et al. (June 2011). "Strong intra- and inter-continental differentiation revealed by Y chromosome SNPs M269, U106 and U152". Forensic Science International. Genetics. 5 (3): e49–52. doi:10.1016/j.fsigen.2010.07.006. PMID 20732840.
- ^ Allentoft, Morten E.; Sikora, Martin; Sjögren, Karl-Göran; Rasmussen, Simon; Rasmussen, Morten; Stenderup, Jesper; Damgaard, Peter B.; Schroeder, Hannes; Ahlström, Torbjörn; Vinner, Lasse; Malaspinas, Anna-Sapfo (June 2015). "Population genomics of Bronze Age Eurasia". Nature. 522 (7555): 167–172. Bibcode:2015Natur.522..167A. doi:10.1038/nature14507. ISSN 1476-4687. PMID 26062507. S2CID 4399103.
- ^ Busby GB, Brisighelli F, Sánchez-Diz P, Ramos-Luis E, Martinez-Cadenas C, Thomas MG, et al. (March 2012). "The peopling of Europe and the cautionary tale of Y chromosome lineage R-M269". Proceedings. Biological Sciences. 279 (1730): 884–92. doi:10.1098/rspb.2011.1044. PMC 3259916. PMID 21865258.
- ^ Mathieson, Iain; Lazaridis, Iosif; Rohland, Nadin; Mallick, Swapan; Patterson, Nick; Roodenberg, Songül Alpaslan; Harney, Eadaoin; Stewardson, Kristin; Fernandes, Daniel; Novak, Mario; Sirak, Kendra (December 2015). "Genome-wide patterns of selection in 230 ancient Eurasians". Nature. 528 (7583): 499–503. Bibcode:2015Natur.528..499M. doi:10.1038/nature16152. ISSN 1476-4687. PMC 4918750. PMID 26595274.
- ^ a b Olalde I, Brace S, Allentoft ME, Armit I, Kristiansen K, Booth T, et al. (March 2018). "The Beaker phenomenon and the genomic transformation of northwest Europe". Nature. 555 (7695): 190–196. Bibcode:2018Natur.555..190O. doi:10.1038/nature25738. PMC 5973796. PMID 29466337.
- ^ Anthony DW (2019-12-06). "Ancient DNA, Mating Networks, and the Anatolian Split". In Serangeli M, Olander T (eds.). Dispersals and Diversification. BRILL. pp. 21–53. doi:10.1163/9789004416192_003. ISBN 978-90-04-41619-2.
- ^ Hammer M (2013). Origins of R-M269 Diversity in Europe. FamilyTreeDNA 9th Annual Conference.
- ^ Sjögren, Karl-Göran; Olalde, Inigo; Carver, Sophie; Allentoft, Morten E.; Knowles, Tim; Kroonen, Guus; Pike, Alistair W.G.; Schröter, Peter; Brown, Keri A. (2019-12-11). "Kinship and social organization in Copper Age Europe. A cross-disciplinary analysis of archaeology, DNA, isotopes, and anthropology from two Bell Beaker cemeteries". bioRxiv: 863944. doi:10.1101/863944. hdl:10261/236801. S2CID 212833639. Retrieved 2021-08-09.
- ^ Underhill PA, Shen P, Lin AA, Jin L, Passarino G, Yang WH, et al. (November 2000). "Y chromosome sequence variation and the history of human populations". Nature Genetics. 26 (3): 358–61. doi:10.1038/81685. PMID 11062480. S2CID 12893406.
- ^ a b Lobov AS, et al. (2009). "Structure of the Gene Pool of Bashkir Subpopulations" (PDF) (in Russian). Archived from the original (PDF) on 2011-08-16.
- ^ a b c d e f g h i j k l Myres NM, Rootsi S, Lin AA, Järve M, King RJ, Kutuev I, et al. (January 2011). "A major Y-chromosome haplogroup R1b Holocene era founder effect in Central and Western Europe". European Journal of Human Genetics. 19 (1): 95–101. doi:10.1038/ejhg.2010.146. PMC 3039512. PMID 20736979.
- ^ Robino C, Crobu F, Di Gaetano C, Bekada A, Benhamamouch S, Cerutti N, Piazza A, Inturri S, Torre C (May 2008). "Analysis of Y-chromosomal SNP haplogroups and STR haplotypes in an Algerian population sample". International Journal of Legal Medicine. 122 (3): 251–5. doi:10.1007/s00414-007-0203-5. PMID 17909833. S2CID 11556974.
- ^ Wood ET, Stover DA, Ehret C, Destro-Bisol G, Spedini G, McLeod H, et al. (July 2005). "Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes". European Journal of Human Genetics. 13 (7): 867–76. doi:10.1038/sj.ejhg.5201408. PMID 15856073. S2CID 20279122.
- ^ Ordóñez AC, Fregel R, Trujillo-Mederos A, Hervella M, de-la-Rúa C, Arnay-de-la-Rosa M (2017). "Genetic studies on the prehispanic population buried in Punta Azul cave (El Hierro, Canary Islands)". Journal of Archaeological Science. 78: 20–28. doi:10.1016/j.jas.2016.11.004.
- ^ a b Grugni V, Battaglia V, Hooshiar Kashani B, Parolo S, Al-Zahery N, Achilli A, et al. (2012). "Ancient migratory events in the Middle East: new clues from the Y-chromosome variation of modern Iranians". PLOS ONE. 7 (7): e41252. Bibcode:2012PLoSO...741252G. doi:10.1371/journal.pone.0041252. PMC 3399854. PMID 22815981.
- ^ 러시아 DNA 계보학회의 의사진행, 3, 1676–1695 (러시아어).
- ^ a b c d e Herrera KJ, Lowery RK, Hadden L, Calderon S, Chiou C, Yepiskoposyan L, Regueiro M, Underhill PA, Herrera RJ (March 2012). "Neolithic patrilineal signals indicate that the Armenian plateau was repopulated by agriculturalists". European Journal of Human Genetics. 20 (3): 313–20. doi:10.1038/ejhg.2011.192. PMC 3286660. PMID 22085901.
- ^ Трофимова Натал'я Вадимовна (Feb. 2015), "Изменчивость Митохондриальной ДНК и Y-Хромосомы в Популяциях Волго-Уральского Региона" Archived 2017-04-02 at the Wayback Machine ("Mitochondrial DNA variation and the Y-chromosome in the population of the Volga-Ural Region"). Автореферат. диссертации на соискание ученой степени кандидата биологических наук. Уфа – 2015.
- ^ Haak W, Lazaridis I, Patterson N, Rohland N, Mallick S, Llamas B, et al. (June 2015). "Massive migration from the steppe was a source for Indo-European languages in Europe". Nature. 522 (7555): 207–11. arXiv:1502.02783. Bibcode:2015Natur.522..207H. bioRxiv 10.1101/013433. doi:10.1038/nature14317. PMC 5048219. PMID 25731166.
- ^ Morelli L, Contu D, Santoni F, Whalen MB, Francalacci P, Cucca F (April 2010). Lalueza-Fox C (ed.). "A comparison of Y-chromosome variation in Sardinia and Anatolia is more consistent with cultural rather than demic diffusion of agriculture". PLOS ONE. 5 (4): e10419. Bibcode:2010PLoSO...510419M. doi:10.1371/journal.pone.0010419. PMC 2861676. PMID 20454687.
- ^ 2017년 기준 ISOGG 트리(isogg.org)
- ^ Vanek D, Saskova L, Koch H (June 2009). "Kinship and Y-chromosome analysis of 7th century human remains: novel DNA extraction and typing procedure for ancient material". Croatian Medical Journal. 3. 50 (3): 286–95. doi:10.3325/cmj.2009.50.286. PMC 2702742. PMID 19480023.
- ^ Moore LT, McEvoy B, Cape E, Simms K, Bradley DG (February 2006). "A Y-chromosome signature of hegemony in Gaelic Ireland". American Journal of Human Genetics. 78 (2): 334–8. doi:10.1086/500055. PMC 1380239. PMID 16358217.
- ^ a b "Clinal patterns of human Y chromosomal diversity in continental Italy and Greece are dominated by drift and founder effects, Di Giacomo et al. (2003) (PDF)". 2002. CiteSeerX 10.1.1.553.5711. Cite 저널은 필요로 한다.
journal=(도움말) - ^ Bryc K, Velez C, Karafet T, Moreno-Estrada A, Reynolds A, Auton A, et al. (May 2010). "Colloquium paper: genome-wide patterns of population structure and admixture among Hispanic/Latino populations". Proceedings of the National Academy of Sciences of the United States of America. 107 Suppl 2: 8954–61. Bibcode:2010PNAS..107.8954B. doi:10.1073/pnas.0914618107. PMC 3024022. PMID 20445096.
- ^ a b Beleza S, Gusmão L, Lopes A, Alves C, Gomes I, Giouzeli M, et al. (March 2006). "Micro-phylogeographic and demographic history of Portuguese male lineages". Annals of Human Genetics. 70 (Pt 2): 181–94. doi:10.1111/j.1529-8817.2005.00221.x. PMID 16626329. S2CID 4652154.
395/657
- ^ Boattini A, Martinez-Cruz B, Sarno S, Harmant C, Useli A, Sanz P, Yang-Yao D, Manry J, Ciani G, Luiselli D, Quintana-Murci L, Comas D, Pettener D (2013). "Uniparental markers in Italy reveal a sex-biased genetic structure and different historical strata". PLOS ONE. 8 (5): e65441. Bibcode:2013PLoSO...865441B. doi:10.1371/journal.pone.0065441. PMC 3666984. PMID 23734255.
- ^ a b Tofanelli S, Brisighelli F, Anagnostou P, Busby GB, Ferri G, Thomas MG, Taglioli L, Rudan I, Zemunik T, Hayward C, Bolnick D, Romano V, Cali F, Luiselli D, Shepherd GB, Tusa S, Facella A, Capelli C (March 2016). "The Greeks in the West: genetic signatures of the Hellenic colonisation in southern Italy and Sicily". European Journal of Human Genetics. 24 (3): 429–36. doi:10.1038/ejhg.2015.124. PMC 4757772. PMID 26173964.
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z Myres NM, Ekins JE, Lin AA, Cavalli-Sforza LL, Woodward SR, Underhill PA (August 2007). "Y-chromosome short tandem repeat DYS458.2 non-consensus alleles occur independently in both binary haplogroups J1-M267 and R1b3-M405". Croatian Medical Journal. 48 (4): 450–9. PMC 2080563. PMID 17696299.
- ^ a b c d e Battaglia V, Fornarino S, Al-Zahery N, Olivieri A, Pala M, Myres NM, et al. (June 2009). "Y-chromosomal evidence of the cultural diffusion of agriculture in Southeast Europe". European Journal of Human Genetics. 17 (6): 820–30. doi:10.1038/ejhg.2008.249. PMC 2947100. PMID 19107149.
- ^ Pericić M, Lauc LB, Klarić IM, Rootsi S, Janićijevic B, Rudan I, et al. (October 2005). "High-resolution phylogenetic analysis of southeastern Europe traces major episodes of paternal gene flow among Slavic populations". Molecular Biology and Evolution. 22 (10): 1964–75. doi:10.1093/molbev/msi185. PMID 15944443.
- ^ a b c Cinnioğlu C, King R, Kivisild T, Kalfoğlu E, Atasoy S, Cavalleri GL, et al. (January 2004). "Excavating Y-chromosome haplotype strata in Anatolia" (PDF). Human Genetics. 114 (2): 127–48. doi:10.1007/s00439-003-1031-4. PMID 14586639. S2CID 10763736. Archived from the original (PDF) on 2006-06-19.
- ^ a b King RJ, Ozcan SS, Carter T, Kalfoğlu E, Atasoy S, Triantaphyllidis C, et al. (March 2008). "Differential Y-chromosome Anatolian influences on the Greek and Cretan Neolithic". Annals of Human Genetics. 72 (Pt 2): 205–14. doi:10.1111/j.1469-1809.2007.00414.x. PMID 18269686. S2CID 22406638.
- ^ Contu D, Morelli L, Santoni F, Foster JW, Francalacci P, Cucca F (January 2008). Hawks J (ed.). "Y-chromosome based evidence for pre-neolithic origin of the genetically homogeneous but diverse Sardinian population: inference for association scans". PLOS ONE. 3 (1): e1430. Bibcode:2008PLoSO...3.1430C. doi:10.1371/journal.pone.0001430. PMC 2174525. PMID 18183308.
174/930
- ^ a b Regueiro M, Cadenas AM, Gayden T, Underhill PA, Herrera RJ (2006). "Iran: tricontinental nexus for Y-chromosome driven migration". Human Heredity. 61 (3): 132–43. doi:10.1159/000093774. PMID 16770078. S2CID 7017701.
- ^ a b Varzari A (2006). Population History of the Dniester-Carpathians: Evidence from Alu Insertion and Y-Chromosome Polymorphisms (PDF) (Dissertation). Ludwig-Maximilians-Universität München.
- ^ Robino C, Crobu F, Di Gaetano C, Bekada A, Benhamamouch S, Cerutti N, et al. (May 2008). "Analysis of Y-chromosomal SNP haplogroups and STR haplotypes in an Algerian population sample". International Journal of Legal Medicine. 122 (3): 251–5. doi:10.1007/s00414-007-0203-5. PMID 17909833. S2CID 11556974.
- ^ a b c d e Balanovsky O, Rootsi S, Pshenichnov A, Kivisild T, Churnosov M, Evseeva I, et al. (January 2008). "Two sources of the Russian patrilineal heritage in their Eurasian context". American Journal of Human Genetics. 82 (1): 236–50. doi:10.1016/j.ajhg.2007.09.019. PMC 2253976. PMID 18179905.
- ^ Al-Zahery N, Semino O, Benuzzi G, Magri C, Passarino G, Torroni A, Santachiara-Benerecetti AS (September 2003). "Y-chromosome and mtDNA polymorphisms in Iraq, a crossroad of the early human dispersal and of post-Neolithic migrations" (PDF). Molecular Phylogenetics and Evolution. 28 (3): 458–72. doi:10.1016/S1055-7903(03)00039-3. PMID 12927131.
16/139
- ^ a b c d Gayden T, Cadenas AM, Regueiro M, Singh NB, Zhivotovsky LA, Underhill PA, Cavalli-Sforza LL, Herrera RJ (May 2007). "The Himalayas as a directional barrier to gene flow". American Journal of Human Genetics. 80 (5): 884–94. doi:10.1086/516757. PMC 1852741. PMID 17436243.
- ^ Karachanak S, Grugni V, Fornarino S, Nesheva D, Al-Zahery N, Battaglia V, Carossa V, Yordanov Y, Torroni A, Galabov AS, Toncheva D, Semino O (2013). "Y-chromosome diversity in modern Bulgarians: new clues about their ancestry". PLOS ONE. 8 (3): e56779. Bibcode:2013PLoSO...856779K. doi:10.1371/journal.pone.0056779. PMC 3590186. PMID 23483890.
- ^ Zalloua PA, Xue Y, Khalife J, Makhoul N, Debiane L, Platt DE, Royyuru AK, Herrera RJ, Hernanz DF, Blue-Smith J, Wells RS, Comas D, Bertranpetit J, Tyler-Smith C (April 2008). "Y-chromosomal diversity in Lebanon is structured by recent historical events". American Journal of Human Genetics. 82 (4): 873–82. doi:10.1016/j.ajhg.2008.01.020. PMC 2427286. PMID 18374297.
- ^ a b c d e Bekada A, Fregel R, Cabrera VM, Larruga JM, Pestano J, Benhamamouch S, González AM (2013-02-19). "Introducing the Algerian mitochondrial DNA and Y-chromosome profiles into the North African landscape". PLOS ONE. 8 (2): e56775. Bibcode:2013PLoSO...856775B. doi:10.1371/journal.pone.0056775. PMC 3576335. PMID 23431392.
- ^ a b c d Adams SM, Bosch E, Balaresque PL, Ballereau SJ, Lee AC, Arroyo E, et al. (December 2008). "The genetic legacy of religious diversity and intolerance: paternal lineages of Christians, Jews, and Muslims in the Iberian Peninsula". American Journal of Human Genetics. 83 (6): 725–36. doi:10.1016/j.ajhg.2008.11.007. PMC 2668061. PMID 19061982.
- ^ a b c Marjanovic D, Fornarino S, Montagna S, Primorac D, Hadziselimovic R, Vidovic S, Pojskic N, Battaglia V, Achilli A, Drobnic K, Andjelinovic S, Torroni A, Santachiara-Benerecetti AS, Semino O (November 2005). "The peopling of modern Bosnia-Herzegovina: Y-chromosome haplogroups in the three main ethnic groups". Annals of Human Genetics. 69 (Pt 6): 757–63. doi:10.1111/j.1529-8817.2005.00190.x. PMID 16266413. S2CID 36632274.
- ^ a b c Cadenas AM, Zhivotovsky LA, Cavalli-Sforza LL, Underhill PA, Herrera RJ (March 2008). "Y-chromosome diversity characterizes the Gulf of Oman". European Journal of Human Genetics. 16 (3): 374–86. doi:10.1038/sj.ejhg.5201934. PMID 17928816.
- ^ a b c d Sengupta S, Zhivotovsky LA, King R, Mehdi SQ, Edmonds CA, Chow CE, et al. (February 2006). "Polarity and temporality of high-resolution y-chromosome distributions in India identify both indigenous and exogenous expansions and reveal minor genetic influence of Central Asian pastoralists". American Journal of Human Genetics. 78 (2): 202–21. doi:10.1086/499411. PMC 1380230. PMID 16400607.
8/176 R-M73 and 5/176 R-M269 for a total of 13/176 R1b in Pakistan and 4/728 R-M269 in India
- ^ a b c Sims LM, Garvey D, Ballantyne J (January 2007). "Sub-populations within the major European and African derived haplogroups R1b3 and E3a are differentiated by previously phylogenetically undefined Y-SNPs". Human Mutation. 28 (1): 97. doi:10.1002/humu.9469. PMID 17154278. S2CID 34556775.
- ^ a b "Origins of R-M269 Diversity in Europe" (PDF).
- ^ a b López-Parra AM, Gusmão L, Tavares L, Baeza C, Amorim A, Mesa MS, Prata MJ, Arroyo-Pardo E (January 2009). "In search of the pre- and post-neolithic genetic substrates in Iberia: evidence from Y-chromosome in Pyrenean populations". Annals of Human Genetics. 73 (1): 42–53. doi:10.1111/j.1469-1809.2008.00478.x. PMID 18803634. S2CID 43273988.
- ^ a b c Bosch E, Calafell F, Comas D, Oefner PJ, Underhill PA, Bertranpetit J (April 2001). "High-resolution analysis of human Y-chromosome variation shows a sharp discontinuity and limited gene flow between northwestern Africa and the Iberian Peninsula". American Journal of Human Genetics. 68 (4): 1019–29. doi:10.1086/319521. PMC 1275654. PMID 11254456.
- ^ Hurles ME, Veitia R, Arroyo E, Armenteros M, Bertranpetit J, Pérez-Lezaun A, et al. (November 1999). "Recent male-mediated gene flow over a linguistic barrier in Iberia, suggested by analysis of a Y-chromosomal DNA polymorphism". American Journal of Human Genetics. 65 (5): 1437–48. doi:10.1086/302617. PMC 1288297. PMID 10521311.
- ^ Rosser ZH, Zerjal T, Hurles ME, Adojaan M, Alavantic D, Amorim A, et al. (December 2000). "Y-chromosomal diversity in Europe is clinal and influenced primarily by geography, rather than by language". American Journal of Human Genetics. 67 (6): 1526–43. doi:10.1086/316890. PMC 1287948. PMID 11078479.
- ^ Moffat A, Wilson JF (2011). The Scots: a genetic journey. Birlinn. pp. 181–182, 192. ISBN 978-0-85790-020-3.
- ^ http://ethnoancestry.com/R1b.html[출처?]
- ^ Cruciani F, Trombetta B, Antonelli C, Pascone R, Valesini G, Scalzi V, Vona G, Melegh B, Zagradisnik B, Assum G, Efremov GD, Sellitto D, Scozzari R (June 2011). "Strong intra- and inter-continental differentiation revealed by Y chromosome SNPs M269, U106 and U152". Forensic Science International. Genetics. 5 (3): e49–52. doi:10.1016/j.fsigen.2010.07.006. PMID 20732840.
- ^ Niederstätter H, Berger B, Erhart D, Parson W (August 2008). "Recently introduced Y-SNPs improve the resolution within Y-chromosome haplogroup R1b in a central European population sample (Tyrol, Austria)". Forensic Science International: Genetics Supplement Series. 1: 226–227. doi:10.1016/j.fsigss.2007.10.158.
- ^ 헨리 서머셋은 차례로 에드워드 3세 (1312–1377)의 아들인 존 오브 가운트 (1340–1399)로부터 침엽수를 물려받았다. 하플로그룹 G2에 속하는 것으로 밝혀진 리차드 3세의 유해를 분석하는 맥락에서 에드워드 3세와 헨리 서머셋 사이에 가장 가능성이 높은 거짓 패트리엇 사건의 가능성이 논의되었다; 아마도 건트의 존이 불법이라는 취지의 소문을 확인했을 것이다(조나단 섬프션, 분할 하우스: 100년 전쟁 3차, 2009년, 페이지 274). 왕 TE, Fortes 때 GG, Balaresque P, 토마스 MG볼딩 국장 D, Maisano Delser P, 노이만 R, 파슨 W, 냅. M, 월시 S, Tonasso L, 홀트 J-Kayser는 M, 준 애플비 J-포스터 P, Ekserdjian D, Hofreiter M, Schürer K(12월 2014년)."왕 리처드 3세의 유해를 식별".네이쳐 커뮤니케이션. 5(5631):5631.Bibcode:2014NatCo...5.5631K. doi:10.1038/ncomms6631.PMC4268703.PMID 25463651."male-line 친척들과 이 유골에서 Y염색체 haplotypes false-paternity 사건은 그 사이에 있는 세대에서 발생하는에게 부여될 수 있일치하지 않습니다.".
- ^ Olalde I, Brace S, Allentoft ME, Armit I, Kristiansen K, Booth T, et al. (March 2018). "The Beaker phenomenon and the genomic transformation of northwest Europe". Nature. 555 (7695): 190–196. Bibcode:2018Natur.555..190O. doi:10.1038/nature25738. PMC 5973796. PMID 29466337.
- ^ Allentoft ME, Sikora M, Sjögren KG, Rasmussen S, Rasmussen M, Stenderup J, et al. (June 2015). "Population genomics of Bronze Age Eurasia". Nature. 522 (7555): 167–72. Bibcode:2015Natur.522..167A. doi:10.1038/nature14507. PMID 26062507. S2CID 4399103.
- ^ Damgaard PB, Marchi N, Rasmussen S, Peyrot M, Renaud G, Korneliussen T, et al. (May 2018). "137 ancient human genomes from across the Eurasian steppes". Nature. 557 (7705): 369–374. Bibcode:2018Natur.557..369D. doi:10.1038/s41586-018-0094-2. PMID 29743675. S2CID 13670282.
- ^ "R-L159 Project Goals".
- ^ Wright DM (2009). "A Set of Distinctive Marker Values Defines a Y-STR Signature for Gaelic Dalcassian Families". Journal of Genetic Genealogy.
- ^ "R-DF21 and Subclades Project".
- ^ Bevan N (2014-09-25). "Dafydd Iwan's rare genetic roots unveiled in new project". walesonline. Retrieved 2018-04-05.
- ^ Bodden T (2014-09-26). "Dafydd Iwan 'descended from Welsh kings' who ruled in England". northwales. Retrieved 2018-04-05.
- ^ "Family Tree DNA - My FamilyTree DNA Project Website Title". www.familytreedna.com. Retrieved 2018-04-05.

