SOGA2
SOGA2| MTCL1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| 에일리어스 | MTCL1, CCDC165, KIAA0802, SOGA2, 마이크로튜브 가교 계수 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| 외부 ID | OMIM: 615766 MGI: 1915867 HomoloGene: 41017 GeneCard: MTCL1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| 위키데이터 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
SOGA2는 포도당 자가포도증 억제제 2 또는 CCDC165로도 알려져 있으며, 사람에게서 SOGA2 [5][6]유전자에 의해 암호화되는 단백질이다.SOGA2에는 SOGA1과 SOGA3라는 [7][8]두 개의 인간 패럴로그가 있습니다.인간의 유전자 코드 배열은 길이 151,349개의 염기쌍이며, mRNA는 6092개의 염기쌍, 단백질 배열은 1586개의 아미노산이다.SOGA2 유전자는 고릴라, 개코원숭이, 갈라고, 쥐, 쥐, 고양이 등에서 보존된다.얼룩말 핀치나 [9]아놀과 같은 유기체에서 볼 수 있는 원거리 보존이 있다.SOGA2는 특히 뇌(특히 소뇌와 해마), 대장, 뇌하수체, 소장, 척수, 고환, 태아 [10]뇌에서 높은 발현을 보이며 사람에게서 널리 발현된다.
진
궤적
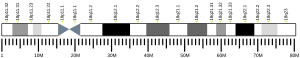
SOGA2 유전자는 염색체 18의 짧은 팔에 8717369 - 8832775(18p11.22)[11]에 위치한다.
호몰로지와 진화
패럴로그
SOGA2에는 인간 단백질 SOGA1과 인간 단백질 SOGA3의 두 가지 주요 [9]패럴로그가 있다. SOGA1은 자가 파지에 의한 포도당 [12]억제에 관여하는 것으로 나타났다.SOGA2 인간으로부터 직교체가 분기하는 속도(% 정체성으로 측정)는 SOGA2에서 SOGA1의 대략적인 중복 이벤트를 ~254.1MYA로, SOGA2에서 SOGA3의 중복 이벤트를 ~329.1MYA로 설정한다.
| 단백질명 | 가입 번호 | 시퀀스 길이(aa) | 인간 단백질에 대한 배열 동일성 | 메모들 |
| SOGA3 | NP_001012279.1 | 947 | 58% | 최대 500 N 단자 aa로 저장 |
| SOGA1 isoform 2 | NP_954650.2 | 1016 aa | 65% | 최초 900 aaa까지 보존. |
| SOGA1 isoform 1 | NP_542194.2 | 1661 | 41% | ~180-1150을 제외하고 시퀀스의 길이에 걸쳐 보존됩니다. |
맞춤법
많은 맞춤법들이 [9]진핵생물에서 확인되었다.
| 통칭 | 단백질명 | 인간 혈통과의 차이(MYA) | 가입 번호 | 시퀀스 길이(aa) | 인간 단백질에 대한 배열 동일성 | 단백질 도메인 차이 |
|---|---|---|---|---|---|---|
| 고릴라 | 단백질 SOGA2 | 8.8 | XP_004059220.1 | 1586 | 99% | |
| 개코원숭이 | 단백질 SOGA2 | 29 | XP_003914218 | 1587 | 98% | |
| 갈라고 | 단백질 SOGA3 | 74 | XP_003801047.1 | 1583 | 88% | DUF4201이 없다. |
| 쥐. | CCDC165 | 92.3 | XP_237548.6 | 2060 | 81% | DUF4201이 없다. |
| 마우스 | SOGA2 | 92.3 | NP_001107570.1 | 1893 | 80% | |
| 집고양이 | 단백질 SOGA2 | 94.2 | XP_003995077.1 | 1700 | 84% | DUF4201이 없다. |
| 소. | CCDC166 | 94.2 | XP_581047.5 | 1525 | 74% | DUF4201이 없다. |
| 아프리카 코끼리 | CCDC167과 같은 | 98.7 | XP_003406836.1 | 1544 | 73% | |
| 얼룩말 핀치 | 단백질 SOGA2 | 296 | XP_002193121.1 | 1598 | 69% | DUF4201이 없다. |
| 레드 정글풀 | CCDC165 | 296 | XP_423729.3 | 1600 | 70% | DUF4201이 없다. |
| 캐롤라이나 아놀 | 미특징단백질KIAA0802양 | 296 | XP_003225723.1 | 1839 | 67% | DUF4201이 없다. |
원거리 호몰로그
| 통칭 | 단백질명 | 인간 혈통과의 차이(MYA) | 가입 번호 | 시퀀스 길이(aa) | 인간 단백질에 대한 배열 동일성 | 단백질 도메인 차이 |
|---|---|---|---|---|---|---|
| 열대발톱개구리 | 비특징단백질 C20orf117양 | 371.2 | XP_002942331.1 | 1584 | 39% | |
| 자줏빛 성게 | 미특정단백질 LOC578090 | 742.9 | XP_783370.2 | 1587 | 47% | DUF4201이 없다. |
| 보디루 | 중심체 단백질 E, 추정치 | 782.7 | XP_002429877.1 | 2086 | 30% | 공유 도메인 없음 |
| 남쪽집모기 | 보존가정단백질 | 782.7 | XP_001843754.1 | 1878 | 32% | 공유 도메인 없음 |
| 돼지벌레 | 표면항원반복단백질 | 937.5 | XP_003380263.1 | 2030 | 36% | 공유 도메인 없음 |
상동 도메인
SOGA2는 N단말기 영역에서 가장 먼 곳에 보존되며,[13] 이 영역에는 알 수 없는 기능의 3개의 도메인이 포함되어 있습니다.
단백질
단백질 내부 조성물
SOGA2는 글리신(사람의 평균 단백질에 대한 SOGA2 구성의 r은 1.723), 글루탐산염(r = 1.647), 아르기닌(r = 1.357)이 풍부하다.또한 티로신(r = 0.16), 이소류신(r = 0.4430), 페닐알라닌(r = 0.5808) 및 발린(r = 0.6161)[14][15]의 성분도 평소보다 낮다.
프라이머리 구조 및 아이소폼
SOGA2에는 Q9Y4B5-1, Q9Y4B5-2, Q9Y4B5-3, Q9Y4B5-4의 [16]4개의 ISO 형식이 있습니다.
도메인과 모티브
SOGA2에는 aa 16-235의 알 수 없는 기능 도메인 4201(DUF4201)이 포함되어 있습니다.이 도메인은 진핵생물에서 [17]단백질 패밀리를 포함하는 코일 도메인에 특이합니다.또한 알 수 없는 기능의 도메인 3166(DUF3166)의 2개의 복사본이 포함되어 있습니다.하나는 aa 140-235에서, 다른 하나는 aa 269-364에서.[11]
번역 후 수정
SOGA2는 번역 후 많은 수정이 예상됩니다.맞춤법에 의해 공유되는 인간 SOGA2의 변형은 다음과 같다.

이차 구조
예측 소프트웨어인 [21]FLEE, [22]GOR4, SOSUICoil은 SOGA2의 2차 구조가 랜덤 코일의 영역이 산재한 알파 헬리크로 지배된다는 데 의견이 일치한다.GOR4는 SOGA2가 알파 헬리에 의해 지배된다는 것을 나타내며, 47.79%의 알파 나선과 46.6%의 랜덤 코일에 비해 확장된 가닥(병렬 또는 역평행 베타 시트) 구성에서 5.61%의 잔류물만 예측했다.
3차 구조
SOGA2는 보존성이 높은N 터미널 영역에서 시퀀스 기능을 공유합니다.이 호몰로지는 Phyre2[24] [25]및 NCBI 구조를 통해 공개된 3D 구조에 대한 호몰로지를 기반으로 3차 구조를 예측할 수 있도록 한다.
 Phyre2가 [24]예측한 SOGA2의 3D 구조.구조는 7앙스트롬 분해능에서 트로포미오신의 결정 구조에 기초하며, 12%의 동일성을 가진다.N 말단 영역을 포함하는 CCDC에서 283개의 잔류물이 일치한다. |  1I84 S, 탈인산화 상태 3D 구조에서 조절 경쇄가 있는 닭 전어 평활근 마이신의 무거운 메로미오신 서브프래그먼트.강조 표시된 영역은 SOGA2로 [25]보존됩니다. |
유전자 발현
주최자
인간 SOGA2의 프로모터는 다음과 같습니다.
유전자 발현 데이터
EST 프로파일은 인간의 경우 SOGA2가 뼈, 뇌,[26] 귀, 눈, 그리고 많은 다른 것들을 포함한 몸 전체의 많은 부위에서 많이 발현된다는 것을 보여준다.간암 검체에는 많은 수의 전사물이 있다.인간의 미세 배열 데이터는 SOGA2가 적당히 발현되며, 특히 뇌(특히 소뇌와 해마), 대장, 뇌하수체, 소장, 척수, 고환 및 태아 [10]뇌에서 높은 발현을 보인다.뇌 조직 특이적 미세 배열 데이터에 따르면 SOGA2는 소뇌 반구의 후엽과 쥐 뇌의 해충 후엽 전체에 걸쳐 높은 발현을 보인다.뇌의 [27]대부분의 다른 부분에서는 발현이 낮다.
트랜스크립트 바리안트
인간의 경우, SOGA2 유전자는 17개의 다른 전사를 생성하며, 그 중 8개는 단백질 생성물을 형성한다.인간의 주요 성명은 성문 ID ENST00000359865 또는 SOGA2-001이다.[28]
기능.
가능한 전사 계수
인간 SOGA2의 가능한 전사 인자는 다음과 같습니다.[29]
- 변조기 인식 계수 2
- cAMP반응성원소결합단백질1
- FOXP1의 대체 스플라이싱 변종
- MDS1/EVI1양유전자1
- Ikaros 2, 림프구 분화의 가능한 조절제
상호 작용
단백질 복합체 공동면역침강(Co-IP) 실험은 세포사 조절제, ATP 결합 카세트(ABC) 운반체 및 단백질 키나제 A 결합 [30]단백질과 같은 상호작용하는 단백질을 밝혀냈다.
540개의 상호작용 단백질은 ABCF1, ACTB, ACTL6A, BCLAF1, BCLAF1, CHEK1,[30] MAGE2를 포함한다.
Wolf pSort에 의한 K-근접 이웃 분석 결과 인간의 경우 SOGA2는 주로 핵, 세포질, 세포핵 공간에 집중되어 있는 것으로 나타났다.골지에 [31]국산화되었을 가능성은 거의 없습니다.
또한 STRING 데이터베이스를 통해 MARK2, MARK4, PPP2R2B를 포함한 다수의 단백질 상호작용 물질이 확인되었다.
임상적 의의
SOGA2는 현재 알려진 질병 연관성이나 돌연변이가 없다.
레퍼런스
- ^ a b c GRCh38: 앙상블 릴리즈 89: ENSG00000168502 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리즈 89: ENSMUSG000052105 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Nagase T; Ishikawa K; Suyama M; Kikuno R; Miyajima N; Tanaka A; Kotani H; Nomura N; Ohara O (April 1999). "Prediction of the coding sequences of unidentified human genes. XI. The complete sequences of 100 new cDNA clones from brain which code for large proteins in vitro". DNA Res. 5 (5): 277–86. doi:10.1093/dnares/5.5.277. PMID 9872452.
- ^ "Entrez Gene: SOGA2".
- ^ "SOGA1". NCBI. Retrieved April 27, 2013.
- ^ "SOGA3". NCBI. Retrieved April 27, 2013.
- ^ a b c "BLAST". NCBI BLAST. Retrieved April 27, 2013.
- ^ a b "GEO Profile 10132039". NCBI GEO. Retrieved April 27, 2013.
- ^ a b "NCBI". National Center for Biotechnology Information. Retrieved 12 May 2013.
- ^ Cowherd RB, Cowerd RB, Asmar MM, et al. (October 2010). "Adiponectin lowers glucose production by increasing SOGA". Am. J. Pathol. 177 (4): 1936–45. doi:10.2353/ajpath.2010.100363. PMC 2947288. PMID 20813965.
- ^ "CLUSTALW". SDSC Biology Workbench. Retrieved April 27, 2013.[영구 데드링크]
- ^ "CLC Sequence Viewer". Retrieved 12 May 2011.[영구 데드링크]
- ^ Nagase T; Ishikawa K; Suyama M; Kikuno R; Miyajima N; Tanaka A; Kotani H; Nomura N; Ohara O (Jan 2011). "Computational analysis of amino acid composition in human proteins". Bioinformatics Trends. 6 (1&2): 39–43.
- ^ "GeneCards". Retrieved 9 May 2011.
- ^ "NCBI Conserved Domains". National Center for Biotechnology Information. Retrieved 12 May 2013.
- ^ "SumoPlot". ABGENT. Retrieved April 27, 2013.
- ^ "Sulfinator". expasy. Retrieved April 27, 2013.
- ^ Blom N; Gammeltoft S; Brunak S (December 1999). "Sequence and structure-based prediction of eukaryotic protein phosphorylation sites". J. Mol. Biol. 294 (5): 1351–62. doi:10.1006/jmbi.1999.3310. PMID 10600390.
- ^ "PELE". SDSC Biology Workbench. Retrieved 27 April 2013.[영구 데드링크]
- ^ "GOR4". npsa-pbil. Retrieved 27 April 2013.[영구 데드링크]
- ^ "SOSUICoil". bp.nuap.nagoya-u.ac.jp. Archived from the original on 2011-07-22. Retrieved 27 April 2013.
- ^ a b Kelley LA; Sternberg MJ (2009). "Protein structure prediction on the Web: a case study using the Phyre server" (PDF). Nat Protoc. 4 (3): 363–71. doi:10.1038/nprot.2009.2. hdl:10044/1/18157. PMID 19247286. S2CID 12497300.
- ^ a b "NCBI Structure". NCBI. Retrieved May 13, 2013.
- ^ "Unigene". National Center for Biotechnology Information. Retrieved April 27, 2013.
- ^ "Allen Brain Atlas, SOGA2 microarray experiments". Allen Brain Atlas. Retrieved April 27, 2013.
- ^ "Ensemble: gene SOGA2". Ensembl. Retrieved April 27, 2013.
- ^ "El Dorado". Genomatix. Retrieved May 8, 2013.[영구 데드링크]
- ^ a b "Molecular Interaction Database - MINT". Archived from the original on 6 May 2006. Retrieved 9 May 2011.
- ^ Horton P, Park KJ, Obayashi T, et al. (July 2007). "WoLF PSORT: protein localization predictor". Nucleic Acids Res. 35 (Web Server issue): W585–7. doi:10.1093/nar/gkm259. PMC 1933216. PMID 17517783.
추가 정보
- Strausberg RL, Feingold EA, Grouse LH, et al. (2003). "Generation and initial analysis of more than 15,000 full-length human and mouse cDNA sequences". Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16899–903. Bibcode:2002PNAS...9916899M. doi:10.1073/pnas.242603899. PMC 139241. PMID 12477932.
- Brajenovic M, Joberty G, Küster B, et al. (2004). "Comprehensive proteomic analysis of human Par protein complexes reveals an interconnected protein network". J. Biol. Chem. 279 (13): 12804–11. doi:10.1074/jbc.M312171200. PMID 14676191.
- Ota T, Suzuki Y, Nishikawa T, et al. (2004). "Complete sequencing and characterization of 21,243 full-length human cDNAs". Nat. Genet. 36 (1): 40–5. doi:10.1038/ng1285. PMID 14702039.
- Gerhard DS, Wagner L, Feingold EA, et al. (2004). "The Status, Quality, and Expansion of the NIH Full-Length cDNA Project: The Mammalian Gene Collection (MGC)". Genome Res. 14 (10B): 2121–7. doi:10.1101/gr.2596504. PMC 528928. PMID 15489334.
- Nusbaum C, Zody MC, Borowsky ML, et al. (2005). "DNA sequence and analysis of human chromosome 18". Nature. 437 (7058): 551–5. Bibcode:2005Natur.437..551N. doi:10.1038/nature03983. PMID 16177791.
- Nousiainen M, Silljé HH, Sauer G, et al. (2006). "Phosphoproteome analysis of the human mitotic spindle". Proc. Natl. Acad. Sci. U.S.A. 103 (14): 5391–6. Bibcode:2006PNAS..103.5391N. doi:10.1073/pnas.0507066103. PMC 1459365. PMID 16565220.
- Beausoleil SA, Villén J, Gerber SA, et al. (2006). "A probability-based approach for high-throughput protein phosphorylation analysis and site localization". Nat. Biotechnol. 24 (10): 1285–92. doi:10.1038/nbt1240. PMID 16964243. S2CID 14294292.
- Olsen JV, Blagoev B, Gnad F, et al. (2006). "Global, in vivo, and site-specific phosphorylation dynamics in signaling networks". Cell. 127 (3): 635–48. doi:10.1016/j.cell.2006.09.026. PMID 17081983. S2CID 7827573.