법의학적 DNA 분석
Forensic DNA analysis| 다음에 대한 시리즈 일부 |
| 법의학 |
|---|
 |
DNA 프로파일링은 법적, 수사적 목적을 위한 DNA 프로파일을 결정하는 것이다.기술이 발전하고 적은 시작 재료로 더 많은 정보를 결정할 수 있게 되면서 DNA 분석 방법은 수년에 걸쳐 수없이 바뀌었다.현대 DNA 분석은 모집단 내에서 생성된 프로파일의 희귀성에 대한 통계적 계산에 기초한다.
법의학 조사의 도구로 가장 잘 알려져 있지만, DNA 프로파일링은 또한 비예측적인 목적으로 사용될 수 있다.친자확인 검사, 그리고 인간 계보 연구는 DNA 프로파일링의 비예측적 사용의 두 가지에 불과하다.
역사
DNA 프로파일을 만드는 방법은 1984년 알렉 제프리스와 그의 팀에 의해 개발되었다.[1]
방법들
폐기된 방법
RFLP 분석
DNA 프로파일링의 첫 번째 진정한 방법은 제한된 조각 길이 다형성 분석이었다.법의학 사례 연구에서 RFLP 분석을 처음 사용한 것은 1985년 영국에서였다.[2]이러한 유형의 분석은 개인을 구별하기 위해 가변 수 탠덤 반복측정(VNTR)을 사용했다.VNTR은 게놈 전체에 걸쳐 흔하며 동일한 DNA 서열을 반복하여 구성된다.[3]다른 개인은 게놈의 특정 위치에서 다른 횟수의 반복을 가질 수 있다.[2]예를 들어, A는 4개, B는 5개의 반복을 가질 수 있다.그 차이점들은 젤 전기영동증이라고 불리는 과정을 통해 시각화되었다.작은 조각들은 그것들을 분리하는 큰 조각들보다 젤을 통해 더 멀리 이동하게 될 것이다.[4]이러한 차이는 개인을 구별하기 위해 사용되었으며 여러 VNTR 사이트를 함께 실행할 때 RFLP 분석은 개인화 능력이 높다.[5]
RFLP 분석 과정은 시간이 매우 많이 소요되었고 사용된 반복측정기의 길이 때문에 9개에서 100개의 염기쌍 사이에 중합효소 연쇄반응과 같은 증폭 방법을 사용할 수 없었다.[3][6]이 RFLP는 이미 더 많은 양의 DNA를 사용할 수 있고 분해된 샘플에서 잘 수행되지 않는 샘플로 제한되었다.[7]RFLP 분석은 마침내 은퇴하고 새로운 방법으로 대체되기 전에 대부분의 법의학 실험실에서 수행된 주요 분석 유형이었다.2000년 연방수사국(FBI)에 의해 완전히 버림받아 STR 분석으로 대체됐다.[8]
DQ 알파 검정
1991년에 개발된 DQ 알파 테스트는 중합효소 연쇄반응을 이용한 최초의 법의학적 DNA 기법이었다.[8][9]이 기법은 RFLP 분석보다 훨씬 적은 수의 세포를 사용할 수 있게 해 이전에는 필요했던 많은 양의 DNA 물질을 가지고 있지 않았던 범죄 현장에 더 유용하게 쓰였다.[10]DQ 알파 1 로쿠스(또는 위치)는 또한 다형성이었고, 그러한 결과를 산출할 수 있었던 개인의 풀을 제한하고 배제의 확률을 높이는 데 사용될 수 있는 여러 가지 다른 알레르기를 가지고 있었다.[11]
DQ 알파 로커스는 1993년 폴리마커라는 상업적으로 구할 수 있는 키트에서 다른 로키와 결합되었다.[12]폴리마커는 현대 멀티플렉싱 키트의 전구체로서 하나의 제품으로 여러 개의 서로 다른 로키를 검사할 수 있도록 했다.RFLP 분석보다 민감하지만 폴리마커는 기존 RFLP 테스트와 동일한 차별적 힘을 포함하지 않았다.[12]1995년까지 과학자들은 증폭된 파편 길이 다형성(AmpflP)이라고 불리는 PCR 기술과 결합된 VNTR 기반 분석으로 돌아가려고 시도했다.[8]
앰프FLP

AmpFLP는 법의학적 사건 조사를 위해 VNTR 분석을 PCR과 결합하려는 첫 번째 시도였다.이 방법은 8~16개의 기본 쌍 사이에 RFLP 분석보다 짧은 VNTR을 사용했다.AmpFLP의 짧은 기본 쌍 크기는 PCR의 증폭 프로세스에서 더 잘 작동하도록 설계되었다.[6]이 기법을 통해 RFLP 분석의 식별력을 얻을 수 있을 것으로 기대되며, DNA가 덜 변형되었거나 분해된 샘플을 처리할 수 있을 것으로 기대된다.그러나 법의학 실험실이 다른 기법으로 빠르게 이동함에 따라 법의학 샘플에 대한 차별화 능력이 제한되어 AmpFLP 분석에 사용할 법의학 애플리케이션에 대한 몇 개의 로키만 검증되었다.[13]
이 기술은 새로운 방법에 비해 비용이 저렴하고 설정이 단순하여 아직 소규모 국가에서 사용되고 있지만 궁극적으로 널리 사용되지 않았다.[14][15]1990년대 후반에 이르러 연구소는 STR 분석을 포함한 새로운 방법으로 전환하기 시작했다.이것들은 심지어 DNA의 더 짧은 조각을 사용했고, 이전 방법들의 차별적 힘을 유지하고 개선하면서 PCR을 사용하여 더 신뢰성 있게 증폭될 수 있었다.[8]
현재 방법
STR 분석

짧은 탠덤 반복(STR) 분석은 현대의 DNA 실험실에서 행해지는 법의학적 DNA 분석의 일차적인 유형이다.STR 분석은 반복 장치의 크기를 2~6개의 기본 쌍으로 축소하고 여러 개의 서로 다른 로키를 하나의 PCR 반응에 결합함으로써 과거에 사용된 RFLP와 AmpFLP를 기반으로 한다.이러한 멀티플렉스 검사 키트는 게놈 전체에 걸쳐 수십 개의 서로 다른 로키에 대한 알레르기의 값을 생성할 수 있으며, 동시에 완전한 개별화 프로파일을 얻는 데 걸리는 시간을 제한할 수 있다.STR 분석은 DNA 프로파일링의 금본위제가 되었고 법의학 응용에 광범위하게 사용된다.
또한 STR 분석은 Y 염색체만으로 제한될 수 있다.Y-STR 분석은 Y 염색체가 부계선 아래 동일하기 때문에(변이가 발생한 경우는 제외) 친자 관계가 있거나 가족 탐색 시 사용할 수 있다.특정 멀티플렉싱 키트는 자동 및 Y-STR 로키를 모두 하나의 키트에 결합하여 대량의 데이터를 얻는 데 걸리는 시간을 더욱 단축시킨다.
현재 STR 분석에는 전체 DNA 프로파일을 생성하기 위해 여러 개의 세포가 필요하다.그러나 과학은 단일 세포에 대한 STR 분석을 이용하여 완전한 DNA 프로파일을 만드는 데 점점 가까워지고 있다.[16]
mtDNA 시퀀싱
미토콘드리아 DNA 염기서열은 대부분의 세포에 존재하는 별도의 미토콘드리아 DNA를 사용하는 전문 기술이다.이 DNA는 모계 선으로 전해져 내려오고 있으며 개인들 사이에 독특한 것은 아니다.그러나 세포에 존재하는 미토콘드리아의 수 때문에, MTDNA 분석은 STR 분석이 유용할 만큼 충분한 데이터를 생성하지 못하는 고도로 분해된 샘플이나 샘플에 사용될 수 있다. mtDNA는 또한 머리카락의 축과 같이 자가 DNA가 없는 위치에도 존재한다.
mtDNA를 다룰 때 오염 가능성이 높아지기 때문에 미토콘드리아 샘플을 처리하는 실험실은 거의 없다.교차 오염을 방지하기 위해 서로 다른 샘플을 추가로 분리하는 전문 프로토콜을 가지고 있는 이들.
래피드 DNA
래피드 DNA는 전체 DNA 추출, 증폭, 분석 과정을 완벽하게 자동화하는 '스왑 인프로파일 아웃(swab in-profile out.빠른 DNA 기기는 면봉에서 DNA 프로파일로 90분 이내에 이동할 수 있으며, 훈련을 받은 과학자가 이 과정을 수행할 필요가 없다.이 기구들은 경찰관이 체포 대상자의 DNA 프로파일을 입수할 수 있도록 범법자 예약 과정에 사용하기 위해 검토되고 있다.
최근, 2017년 신속 DNA법이 미국에서 통과되어, FBI가 이 기술을 전국적으로 이행하기 위한 프로토콜을 만들도록 지시하였다.현재 이들 기기에서 얻은 DNA는 표준 임계값을 만족시킬 만큼 충분한 loci를 분석하지 않기 때문에 국가 DNA 데이터베이스에 업로드할 수 없다.그러나 이미 여러 경찰기관이 라피드 DNA 도구를 사용해 해당 지역에서 체포된 사람들로부터 샘플을 채취하고 있다.이 지역 DNA 데이터베이스는 연방이나 주 정부의 규제를 받지 않는다.
대규모 병렬 시퀀싱
차세대 시퀀싱이라고도 알려진 MPS(Massive Parallel Sequence)는 로키의 직접 시퀀싱을 도입하여 STR 분석을 기반으로 구축된다.각 위치에 존재하는 반복 횟수 대신 MPS는 과학자에게 실제 기본 쌍 시퀀스를 제공할 것이다.이론적으로 MPS는 기존의 STR 분석으로는 포착되지 않는 반복 세그먼트 내에서 무작위 지점 돌연변이를 볼 수 있기 때문에 일란성 쌍둥이를 구별할 수 있는 능력을 가지고 있다.
프로필 희귀성
DNA 프로파일을 입증적 방법으로 사용할 경우 모집단 내에서 프로필이 얼마나 희귀한지를 설명하는 일치 통계가 제공된다.특히, 이 통계량은 모집단에서 무작위로 추출한 사람이 특정 DNA 프로파일을 가질 확률이다.프로파일이 누군가를 "매치"할 확률은 아니다.이 통계를 결정하는 방법에는 여러 가지가 있으며, 각각의 방법은 경험과 선호도에 따라 다양한 실험실에서 사용된다.그러나, 우도비 계산은 가장 일반적으로 사용되는 다른 두 가지 방법, 즉 배제되지 않은 무작위 인간과 결합한 포함 확률보다 선호되는 방법이 되고 있다.일치 통계는 DNA 프로파일에 한 명 이상의 기여자가 있는 혼합물 해석에서 특히 중요하다.이러한 통계가 법정 환경이나 실험실 보고서에서 제공될 때, 그것들은 대개 특정 지역에서 가장 일반적인 세 가지 인종에 대해 주어진다.이것은 다른 위치에서의 알레 주파수가 개인의 조상에 따라 변화했기 때문이다.https://strbase.nist.gov/training/6_Mixture-Statistics.pdf
무작위남 제외되지 않음
이 방법으로 생성된 확률은 분석된 데이터에서 무작위로 모집단을 추출할 수 없는 확률이다.이런 유형의 매치 통계는 과학적 배경이 없는 개인에게 법정에서 설명하기 쉽지만 피의자의 유전자형을 고려하지 않아 변별력이 많이 떨어진다.이 접근방식은 표본이 분해되거나 단일 프로파일을 결정할 수 없을 정도로 많은 기여자를 포함할 때 일반적으로 사용된다.통계를 얻는 방법이 간단하기 때문에 일반인에게 설명하는 데도 유용하다.단, 한정된 차별력 때문에 RMNE는 다른 방법을 사용할 수 없는 한 일반적으로 수행되지 않는다.혼합물이 있음을 나타내는 데이터에는 RMNE를 사용하지 않는 것이 좋다.
포함/제외 가능성 결합
포함 또는 제외의 결합 확률은 관련성이 없는 무작위 개인이 DNA 프로파일 또는 DNA 혼합물의 기여자가 될 확률을 계산한다.이 방법에서, 각 개별 로커에 대한 통계는 모집단 통계량을 사용하여 결정되고, 그 다음 결합하여 총 CPI 또는 CPE를 얻는다.이러한 계산은 사용 가능한 모든 데이터와 함께 사용 가능한 모든 로키에 대해 반복되며, 그 다음 각 값을 함께 곱하여 포함 또는 제외의 총 결합 확률을 구한다.값을 함께 곱하기 때문에 CPI를 사용하여 극소수를 달성할 수 있다.혼합물이 표시될 때 CPI 또는 CPE는 허용 가능한 통계적 계산으로 간주된다.https://www.promega.com/-/media/files/resources/conference-proceedings/ishi-15/parentage-and-mixture-statistics-workshop/generalpopulationstats.pdf?la=en
단일 소스 프로파일에 대한 계산 예제
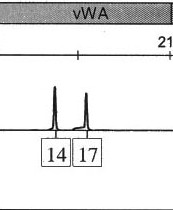
vWA = .10204에서 백인이 16 알레르기를 가질 확률
vWA = .26276에서 백인의 알레르기가 17일 확률
백인이 14와 17 알레르(P)를 모두 가질 확률 = .10204 + .26276 = .3648
다른 모든 알레르기가 존재할 확률(Q) = 1 - P 또는 1 - .3648 = .6352
vWA 제외 확률 = Q + 2Q2(1-Q) 또는 .63522 + 2(.6352)(1 - .6352) = .86692096 96 86.69%
vWA = 1 - CPE 또는 1 - .86692096 = .13307904 ≈ 13.31%
혼합물 종단에 대한 계산 예제
vWA = .10204에서 백인이 14개의 알레르기를 가질 확률
vWA = .11224에서 백인이 15 알레르기를 가질 확률
vWA = .5153에서 백인이 16 알레르기를 가질 확률
vWA = .08418에서 백인이 19 알레르기를 가질 확률
백인이 14, 15, 16 또는 19 알레르기를 가질 확률(P) = .10204 + .11224 + .1153 + .08418 = .4999
다른 모든 알레르기가 존재할 확률(Q) = 1 - P 또는 1 - .499999 = .50001
vWA에 대한 제외 확률 = Q2 + 2Q(1-Q) 또는 .500012 + 2(.50001)(1 - .50001) = .75000999999 ≈ 75%
vWA = 1 - CPE 또는 1 - .75000999999 = .249001 001 25%
우도비
우도비(LR)는 어느 것이 더 가능성이 높은지를 결정하기 위해 두 개의 다른 확률을 비교한 것이다.LR은 재판과 관련된 경우 검찰의 주장과 변호인의 주장 중 시작 가정을 고려할 때 변론의 확률을 비교한 것이다.이 시나리오에서, 검찰은 그들이 적임자를 가지고 있다고 절대 확신하지 않는 한(100%) 기소하지 않을 것이라고 가정하기 때문에, 검찰의 확률은 종종 1과 같다.가능성 비율은 여러 기여자를 나타내는 데이터에 대한 통계를 제시하는 유용성 및 주어진 데이터 집합에서 가장 가능성이 높은 알레르기의 조합을 예측하는 확률론적 유전자형 소프트웨어에서의 사용으로 인해 실험실에서 더 흔해지고 있다.
우도비 사용의 단점은 분석가들이 어떻게 특정한 값에 도달했는지 이해하기 매우 어렵고, 더 많은 데이터가 방정식에 도입될수록 관련된 수학은 매우 복잡해진다는 것이다.법정 환경에서 이러한 문제와 싸우기 위해, 일부 실험실은 우도비의 실제 숫자 값을 대체하는 "언어적 척도"를 설정했다.
참조
- ^ McKie, Robin (May 23, 2009). "Eureka moment that led to the discovery of DNA fingerprinting". The Guardian. Retrieved October 29, 2017.
- ^ a b "DNA Typing by RFLP Analysis". National Forensic Science Technology Center. 2005. Archived from the original on January 3, 2015. Retrieved November 10, 2017.
- ^ a b Griffiths, Anthony J.F.; Lewontin, Richard C.; Gelbart, William M.; Miller, Jeffrey H. Modern Genetic Analysis: Integrating Genes and Genomes (Second ed.). W. H. Freeman and Company. p. 274. Retrieved November 10, 2017.
- ^ Fisher, Barry A. J. (2005). Techniques of Crime Scene Investigation (Seventh ed.). CRC Press. p. 240. Retrieved November 11, 2017.
- ^ Rudin, Norah; Inman, Keith (2002). An Introduction to Forensic DNA Analysis (Second ed.). CRC Press. p. 41. Retrieved November 11, 2017.
- ^ a b James, Stuart H.; Nordby, Jon J., eds. (2005). Forensic Science: An Introduction to Scientific and Investigative Techniques (Second ed.). Taylor & Francis. p. 286. Retrieved November 10, 2017.
- ^ "Disadvantages". National Forensic Science Technology Center. 2005. Archived from the original on January 2, 2015. Retrieved November 10, 2017.
- ^ a b c d Tilstone, William J.; Savage, Kathleen A.; Clark, Leigh A. (2006). Forensic Science: An Encyclopedia of History, Methods, and Techniques. ABC CLIO. p. 49. Retrieved October 30, 2017.
- ^ Riley, Donald E. (April 6, 2005). "DNA Testing: An Introduction For Non-Scientists An Illustrated Explanation". Retrieved October 29, 2017.
- ^ McClintock, J. Thomas (2008). Forensic DNA Analysis: A Laboratory Manual. CRC Press. p. 64. Retrieved October 29, 2017.
- ^ Blake, E; Mihalovich, J; Hiquchi, R; Walsh, PS; Erlich, H (May 1992). "Polymerase chain reaction (PCR) amplification and human leukocyte antigen (HLA)-DQ alpha oligonucleotide typing on biological evidence samples: casework experience". Journal of Forensic Sciences. 37 (3): 700–726. PMID 1629670.
- ^ a b "DQ-Alpha". National Forensic Science Technology Center. 2005. Archived from the original on November 10, 2014. Retrieved October 30, 2017.
- ^ Bär, W.; Fiori, A.; Rossi, U., eds. (October 1993). Advances in Forensic Haemogenetics. 15th Congress of the International Society for Forensic Haemogenetics. p. 255. Retrieved November 10, 2017.
- ^ "AmpFLPs". National Forensic Science Technology Center. 2005. Archived from the original on November 21, 2014. Retrieved November 5, 2017.
- ^ "DNA Fingerprinting Methods". Fingerprinting.com. Retrieved November 5, 2017.
- ^ Ostojic, Lana; O’Connor, Craig; Wurmbach, Elisa (1 March 2021). "Micromanipulation of single cells and fingerprints for forensic identification". Forensic Science International: Genetics. 51. doi:10.1016/j.fsigen.2020.102430.