글루타민 프로테아제
Glutamic protease| 스키탈리도펩신 B | |||||||
|---|---|---|---|---|---|---|---|
 | |||||||
| 식별자 | |||||||
| 유기체 | |||||||
| 기호 | 해당 없음 | ||||||
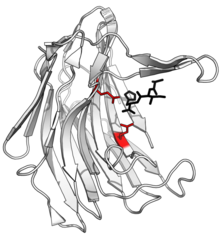
| PDB | 1S2K | ||||||
| 유니프로트 | P15369 | ||||||
| 기타자료 | |||||||
| EC 번호 | 3.4.23.32 | ||||||
| |||||||
글루탐 프로테아제는 활성 부위 내에 글루탐산 잔류물을 함유한 프로테롤리틱 효소 그룹이다.이러한 유형의 프로테아제는 2004년에 처음 설명되었고 프로테아제의 여섯 번째 촉매 유형이 되었다.[1]이 프로테아제 집단의 구성원들은 이전에는 아스파라테아제라고 가정했지만, 구조적인 결단은 그것이 새로운 프로테아제 가족에 속한다는 것을 보여주었다.이 프로테아제 그룹의 첫 번째 구조는 스키탈리도글루타민펩티다아제였으며, 활성 부위는 촉매 디아드, 글루타민산(E), 글루타민(Q)을 함유하고 있어 에콜린이라는 이름이 생겨났다.이 보호제군은 주로 식물과 인간에게 영향을 미치는 병원성 곰팡이에서 발견된다.[2]
분포 및 유형

글루타믹 프로테아제(G1과 G2)의 독립된 두 가족이 있으며, 분포가 제한되어 있다.그것들은 원래 주로 아스코미코타 망울에 있는 필라멘트성 곰팡이에 한정되어 있다고 생각되었다.[3]그러나 이후 글루타민 단백질은 박테리아와 고고학에서 확인되었다.[4]
글루타민 단백질의 첫 번째 슈퍼 패밀리는 균사체 스키탈리듐 리니콜라와 아스페르길루스 니제르바. 마크로스포루스에서 확인되었으며, 이로부터 스키탈리도글루타민 펩티다아제(에콜리신)와 아스페르길로글루타민 펩티다아제가 각각 파생된다.이 두 개의 프로테아제는 활성 사이트 Glu와 Gln 잔여물을 포함하고 있으며 MEROPS 제품군 G1에 따라 그룹화된다.[5][6]
융합적으로 진화한 글루탐 펩타미아제(pluaramic peptidase, phi-29)는 목 전첨판 단백질(pre-necutage phi-29)으로 활성현장에서 글루와 아스프 디아드를 사용하며, MEROPS 계열 G2로 분류된다.[7]
특성.
이러한 효소는 산성 단백질이다. 예를 들어 에콜리신은 카제인을 기질로 사용할 때 pH 2.0에서 가장 활발하다.[2]에콜로신은 P1 부위의 부피가 큰 아미노산 잔류물과 P1 부위의 작은 아미노산 잔류물을 선호한다.프로테아제의 특징은 펩스타틴과 S-PI(아세틸 펩스타틴)에 대한 불감증으로, 이전에 "펩타틴에 민감하지 않은 카복시 단백질아제"[8]로 분류되었다.다른 "페프스타틴 민감성 카르복실 단백질아제"는 2001년에 발견된 세린 프로테아제, 세린-카복실 프로테아제(Sedolisin) 하위 계열에 속한다.[2]또한 이러한 보호제는 DAN(diazoacetyl-DL-norleucine methylster)(7)에 의해 억제되지 않지만 EPNP(1,2-epoxy-3-(p-nitrophenoxy) 프로판(propane)에 의해 억제될 수 있다.[9][10]
활성 부위 및 촉매제
에콜로신의 활성 부위는 기질 결합과 카탈루션에 관여하는 독특한 글루탐산과 글루타민 촉매 디아드를 포함한다.이러한 잔류물은 핵소체의 역할을 하며, 글루탐산이 반응의 첫 단계에서 일반산 역할을 하며, 기질의 펩타이드 결합에 있는 카보닐 산소에 양성자를 기증한다.하나 또는 두 개의 물 분자가 히드록실 그룹을 공급하는 반응에 관여할 수 있으며, 글루탐산은 양성자를 아미드 질소에 추가로 기증하여 펩타이드 결합이 깨지게 된다.글루타민은 글루타민산을 초기 상태로 되돌린다.[11]
참고 항목
참조
- ^ Fujinaga M, Cherney MM, Oyama H, Oda K, James MN (Mar 2004). "The molecular structure and catalytic mechanism of a novel carboxyl peptidase from Scytalidium lignicolum". Proceedings of the National Academy of Sciences of the United States of America. 101 (10): 3364–9. Bibcode:2004PNAS..101.3364F. doi:10.1073/pnas.0400246101. PMC 373467. PMID 14993599.
- ^ a b c Oda K (Jan 2012). "New families of carboxyl peptidases: serine-carboxyl peptidases and glutamic peptidases". Journal of Biochemistry. 151 (1): 13–25. doi:10.1093/jb/mvr129. PMID 22016395.
- ^ Sims AH, Dunn-Coleman NS, Robson GD, Oliver SG (Oct 2004). "Glutamic protease distribution is limited to filamentous fungi". FEMS Microbiology Letters. 239 (1): 95–101. doi:10.1016/j.femsle.2004.08.023. PMID 15451106.
- ^ Jensen K, Østergaard PR, Wilting R, Lassen SF (2010). "Identification and characterization of a bacterial glutamic peptidase". BMC Biochemistry. 11 (47): 47. doi:10.1186/1471-2091-11-47. PMC 3009609. PMID 21122090.
- ^ Sasaki H, Kubota K, Lee WC, Ohtsuka J, Kojima M, Iwata S, Nakagawa A, Takahashi K, Tanokura M (Jul 2012). "The crystal structure of an intermediate dimer of aspergilloglutamic peptidase that mimics the enzyme-activation product complex produced upon autoproteolysis". Journal of Biochemistry. 152 (1): 45–52. doi:10.1093/jb/mvs050. PMID 22569035.
- ^ Takahashi K (2013). "Structure and function studies on enzymes with a catalytic carboxyl group(s): from ribonuclease T1 to carboxyl peptidases". Proceedings of the Japan Academy, Series B. 89 (6): 201–25. Bibcode:2013PJAB...89..201T. doi:10.2183/pjab.89.201. PMC 3749792. PMID 23759941.
- ^ "Family G2". MEROPS.
- ^ "Family G1". MEROPS.
- ^ Murao S, Oda K, Matsushita Y (1973). "Isolation and identification of a microorganism which produces non Streptomyces pepsin inhibitor and N-diazoacetyl-DL-norleucine methylester sensitive acid proteases". Agric. Biol. Chem. 37 (6): 1417–1421. doi:10.1271/bbb1961.37.1417.
- ^ Morihara K, Tsuzuki H, Murao S, Oda K (Mar 1979). "Pepstatin-insenstive acid proteases from Scytalidium lignicolum. Kinetic study with synthetic peptides". Journal of Biochemistry. 85 (3): 661–8. PMID 34596.
- ^ Moselio Schaechter, ed. (2009). Encyclopedia of Microbiology (3rd ed.). Academic Press. p. 499. ISBN 978-0123739391.


