박테리오플랑크톤 계수법
Bacterioplankton counting methods박테리오플랑크톤 카운팅은 특정 수역 내 박테리오플랑크톤의 풍부함을 추정하는 것으로 해양 미생물학자들에게 유용한 정보가 된다. 관찰되고 있는 물 속에 존재하는 수를 결정하기 위해 여러 해 동안 다양한 계수 방법론이 개발되었다. 박테리오플랑크톤 계산에 사용되는 방법으로는 에피플루오렌스 현미경 검사, 유동 세포측정법, 세포분할 빈도수를 통한 생산성 측정법(FDC), 티미딘 결합법, 류신 결합법 등이 있다.
염분, 온도, 위도, 다양한 영양소 수준, 물의 움직임 및 다른 유기체의 존재와 같은 요인들은 박테리오플랑크톤 열거에 영향을 미칠 수 있다.[1][2][3][4][5] 이러한 요인의 변화는 박테리오플랑크톤 카운트에 영향을 미치며, 물의 몸체, 위치, 해안으로부터의 거리 및 계절에 따라 달라진다.[6][7][8]
박테리오플랑크톤 카운트는 보통 ml(세포 ml)당 세포로 표현된다−1.
사용하다
해양 미생물학과 수생 생태계를 이해하는 데 있어 박테리오플랑크톤 수는 유용할 수 있다. 박테리오플랑크톤 수의 관찰은 다음에서 더 많은 정보를 제공할 수 있다.
- 수생 시스템에서[9][10] 다양한 영양소 순환에 관련된 프로세스
- 수생 생산성을 위해
- 환경 변화, 특히 극한 환경 변화 결정용
- 수생생태계의 영양성분 구성
- 다른 수생생물(즉, 새우)의 풍부함과 상태
에피플루오렌스 현미경 검사
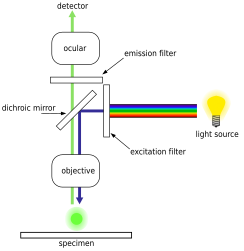
에피플루오렌스 현미경 검사는 형광 염료를 사용하여 특정 생물학적 표지에 결합한 후 렌즈를 통해 식별되는 독특한 방출 스펙트럼을 방출하는 첨단 광학 현미경 기법이다. 형광 염료에는 DAPI, Acridine Orange, SYBR Green 1, YO-PRO-1 등이 있는데, 모두 박테리아와 바이러스 등 생물 검체에서 DNA와 RNA 구조를 모두 염색이 가능하다.[18][19][20][21] 그러나 DNA 얼룩은 주로 박테리아 세포 식별에 사용된다. 현대적인 경구현미경으로, 박테리아 세포의 양을 추정하고 세는 산업 표준은 DAPI 얼룩을 사용하는 것이다.[22] 이 기법은 토양과 퇴적물뿐만 아니라 바닷물, 다양한 담수원과 같은 광범위한 환경 및 위치에서 채취한 표본을 대상으로 수행할 수 있다.[22]
열거기법
표준 실험에서, 준비된 박테리아 샘플은 세는 슬라이드에 올려진 후 후 경구 현미경으로 관찰된다. 배율은 카운팅 슬라이드의 0.1 X 0.1 mm 제곱 단위가 선명하게 보이는 수준으로 설정한다.[23] 이 박테리아를 정량화하기 위해, 세포는 5-30 무작위 제곱 단위 시야로 계산하고, 분야당 평균 박테리아 수를 표로 표시한다.[22] 그런 다음 슬라이드 증착 면적의 총 시야 수를 결정하고 이를 계수 단위당 평균 박테리아 카운트에 곱하여 mL당 총 박테리아 세포 수를 추정하기 위해 이 값을 외삽한다.[23]
신뢰성.
박테리아 세포의 양을 열거하기 위해, 표본 내 박테리아의 작은 부분만 물류상의 이유로 물리적으로 계산되며, 그 결과 총유산은 외삽에 의해 추정된다. 그런 다음 표본 간의 비교에 평균값을 사용한다. 그러나 총 풍부량을 추정하기 위해 작은 부분 집합의 표만을 사용하는 이 기술의 정확성은 문제시 되었다.[22] 주로 미끄럼틀을 세는 과정에서 세균세포의 분포가 불균일하고 일관성이 없는 것으로 나타났다.[22] 또한 이 기법을 이용하여 세균 수를 합법적으로 추정하기 위해서는 20개 시야에서 350개 이상의 개별 세포를 측정해야 한다는 제안이 있었다.[22] 이것은 시간이 오래 걸릴 뿐만 아니라 특정 샘플에서는 달성하기 어려울 수 있다.
플로우 시토메트리

흐름 계량 분석(또는 흐름 계량법)은 많은 임상 애플리케이션에서 일반적인 절차다. 그러나 30여 년 전 발견되었음에도 불구하고 박테리오플랑크톤을 열거한 수생 미생물 생태계의 채택은 상대적으로 더디기만 했다.[24] 그것의 사용은 아직 인식 현미경을 능가하는 것이다.[25] 두 가지 풍요로움 추정 기법이 모두 비교적 정확함에도 불구하고 흐름 세포계는 인간의 실수에 덜 취약하고, 보다 정밀하며, 분해능이 높아 수 만 개의 세포를 단 몇 분 만에 검사할 수 있다.[24] 플로우 시토메트리는 또한 세포의 풍부함 외에도 세포의 크기, 활동성, 형태학에 관한 정보를 제공할 수 있다.[26]
유동 세포측정법은 광합성과 비광합성 박테리오플랑크톤을 구별하고 수량화하기 위해 사용될 수 있다.[26] 시아노박테리아와 피코우카리아 해조류 같은 광합성 원핵생물의 계량화는 광합성 색소의 형광화 능력에 의해 가능하다.[27] 예를 들어, 두 개의 주요 광합성 원핵생물인 프로클로로코쿠스와 신네코코쿠스에서 광합성 색소의 다른 형성은 그 구별을 가능하게 한다.[28][29][30] 프로클로로코쿠스에는 푸른 빛이나 자외선에 의한 흥분 하에서 오직 붉은 형광만을 보이는 디비닐-염색소 a와 b가 들어 있으며, 신네초코쿠스는 주황색과 적색 형광, 피코빌린에서 주황색, 엽록소에서 붉은색을 모두 방출한다. 형광 외에도 프로클로로코쿠스와 시네초코쿠스는 크기가 현저히 다르기 때문에 흐름 세포분석에 따라 다른 산란 신호를 전달한다. 이것은 그들의 차별화에 더욱 도움이 된다.[31] 프로클로로코쿠스 정량화는 그동안 흐름 세포측정을 통해서만 거의 가능했기 때문에 주요 돌파구로 꼽힌다. 이는 프로클로로코쿠스에 존재하는 낮은 엽록소 자폐증을 검출할 수 있는 에피플루오르센스 현미경이 없기 때문이다.[26]
광합성 박테리아 소플랑크톤 외에도 비광합성 박테리아 소플랑크톤은 흐름 세포측정법으로 열거할 수 있다. 이것은 DNA나 음식 바쿠올 얼룩을 통해 이루어진다.[27] 유량 세포측정법은 특히 프로클로로코쿠스와 이질성 박테리아를 구별하는데 성공했는데, 이 박테리아는 비슷한 크기 때문에 초기에 계수가 혼동되었다.
많은 미생물 생태 연구소에서 유동 세포측정법보다 경구 현미경을 사용하는 것은 많은 경제적, 실용적인 요인들에 의해 비난 받을 수 있다. 첫째로, 상업용 유동 세포계를 사용하려면 엄격하게 훈련된 기술자의 전문지식이 필요하다. 둘째, 유량 세포계는 epi-fluorscence 현미경 장비에 비해 상당히 비싸다. 셋째, 많은 흐름 세포계는 혈액 세포를 검사하도록 설계되어 있다; 해양 박테리아는 상대적으로 작아서 많은 상업 흐름 세포계에서 분해능의 한계에 접근한다.[32]
열거 프로세스
박테리오플랭크톤의 유동 세포계 정량화에는 고정, 얼룩, 데이터 처리, 데이터 해석의 네 단계가 포함된다.
고정
고정은 시료를 보존할 뿐만 아니라 세포의 얼룩에 대한 투과성을 증가시키기 위해 행해진다.[24] 그러나 대부분의 일반적인 고정제는 크기, 빛이 어떻게 산란되는가, 자기유동성, 핵산 등과 같은 특정 측면을 변화시킴으로써 세포를 변화시킬 수 있는 능력을 가지고 있다. 세포의 유동 세포계 구분이 이러한 품질에 의존하기 때문에 이것은 문제가 있다. 어떤 고정제는 세포의 완전한 상실을 초래하기도 한다.[24] 현재, 고정 과정에 사용되는 일부 제제는 포름알데히드(포름알데히드와 파라포름알데히드), 에탄올 70%, 글루타알데히드, TCA의 2가지 변형을 포함한다.[33] 단백질과 핵산의 가장 좋은 고정제는 세포에 신속하게 침투할 수 있는 능력 때문에 파라포름알데히드로 추정된다.[24]
얼룩
흐름 세포측정학에서 얼룩은 박테리오플랑크톤과 비박테리아 입자를 구별하는 것을 가능하게 한다. 자외선 방출 염료(DAPI 및 Hoechst 33342)와 청광 방출 핵산 염료(TO-PRO-1, TOTO-1, SYBR Green I)와 같은 광범위한 형광 투시 색소에 시료를 배양하는 것을 포함한다.[31] 오랫동안 유량 세포계는 자외선을 방출하는 염료를 이용하여 감도가 제한된 저비용 유량 세포계에 사용될 수 있는 박테리아 또는 이질성 박테리아와 자생물을 구별하는데 필요한 높은 감도를 가진 값비싼 유량 세포계에 사용될 수 있는 박테리아를 검사했다. SYBR Green I과 같은 푸른색 계열의 염료가 도입되어 저비용 고감도 흐름도계에 박테리오플랭크톤의 고품질 흐름도계 분석이 가능해졌다.[31]
최적의 얼룩을 위한 배양 시간은 화합물마다 다르다. 자외선 차단 염료는 1시간 이상 소요될 수 있고, 청색 광선 방출 염료는 15분만 소요될 수 있다.[24]
얼룩은 세포가 얼룩에 더 잘 스며들도록 만드는 트리톤 X-100과 같은 완충제를 동반할 수 있다. 그것들은 특히 TO-PRO-1과 같은 세포 내 중성 염료에 사용된다. 또한 피코그린, YO-PRO-1, YOYO-1과 같이 이온 강도에 민감한 염료를 희석하는 데도 버퍼를 사용한다. 그러나 트리톤-X-100과 같은 버퍼는 엽록소 형광을 소멸시킬 뿐만 아니라 원치 않는 배경 형광을 만들 수 있기 때문에 버퍼의 사용은 세포에 해로울 수 있다. 이것은 이질성 박테리아와 자생성 원핵생물을 구별하는 어려움을 증가시킬 수 있다.[24]
계산

흐름 세포 분석에서 200개 이상의 세포가 레이저 빔이나 수은 램프 앞을 한 번에 한 번씩 통과한다. 광전자 증배기는 각 입자가 산란하는 빛의 양과 배설 시 방출되는 형광을 모은다. 그런 다음 이 정보는 내실화되고 시스템에 의해 하나의 사건으로 해석된다. 그러나, 아주 적은 노력으로 세포를 셀 수 있는 흐름 세포계의 능력에도 불구하고, 대부분은 세포의 실제 농도를 결정할 방법이 없다. 이는 양이 미리 결정된 참조 비드의 사용(도움말 대 비드 박테리아의 비율을 결정하는 도움), 실험 전후의 무게 측정 및 흐름의 일일 교정을 포함한 다양한 방법을 통해 결정될 수 있다.[24]
유동 세포계의 큰 장점은 박테리오플랭크톤의 다른 개체군을 식별하는 능력이다. 이러한 차별은 옅은 산란, 녹색 형광, 청색 형광, 적색 형광의 네 가지 요인에 대한 분석을 통해 이루어진다. 빛 산란 분석만으로는 불충분하며, 여러 가지 이유로 형광과 함께 종종 조사된다; 첫째, 바닷물은 박테리아처럼 빛을 산란시키는 많은 입자를 포함한다. 둘째, 많은 해양세균의 크기가 해상도 한계에 접근한다. 세포에 의해 산란되는 빛의 양은 세포의 크기뿐만 아니라 내부 구조, 굴절률, 입자의 모양, 방향 등에 의해 결정된다. 산란 광선은 전방 산란(FSC) 또는 측면 산란(SSC)으로 분류된다. 전자는 세포의 부피와 질량과 관련이 있는 반면 후자는 세포의 굴절, 내용, 세분화 지수와 연관되어 있다.
셀 농도가 ml당 2.5 × 106 cells보다 높을 경우, 셀이 근접하게 한 번 이상 지나 단일 이벤트로 기록될 가능성이 커진다. 이것은 우연이라고 알려져 있으며, 샘플을 손으로[31] 희석하면 쉽게 피할 수 있다.
생산성 측정
세포분할 빈도
분열세포의 빈도(FDC)는 수생 이질성 박테리아 공동체의 평균 성장률을 예측하기 위해 사용되는 방법이다.[34] 그 방법은 세포분열, 특히 중분열 형성을 성장률을 대신하는 것으로 사용한다.[34] 후두경 현미경 검사에서 개별 세포 사이의 공극(흡입)이 관찰될 때 세포는 분할된 것으로 간주된다.[34] FDC는 현재 분리한 세포의 비율과 박테리아 공동체의 성장률 사이에 관계가 있다는 가정에 근거한다.[35]
티미딘 법인화
티미딘 통합은 박테리아의 성장을 추정하기 위해 가장 광범위하게 사용되는 방법 중 하나이다.[36] 티미딘은 DNA의 전구체로서, DNA 합성은 삼중수소 티미딘을 핵산으로 통합하여 측정할 수 있다.[37] 티미딘 통합은 성장 중인 세포만이 방사성 티미딘을 통합하여 DNA를 합성할 수 있다는 가정을 이용하여 DNA 합성 속도를 바탕으로 성장을 측정한다.[38]
이 절차의 약점에는 삼중수소 티미딘이 샘플에 첨가되었을 때 DNA 이외의 다른 분자에 대한 라벨링이 포함된다.[36] 탄소가 제한되는 경우 티미딘은 DNA 전구체가 아닌 탄소원으로도 사용될 수 있다.[36] 티미딘 통합 실험의 결과는 다른 분자에 비해 티미딘이 DNA에 통합된 비율을 알 수 없을 때 오해의 소지가 있을 수 있다.[36]
류신 법인화
수생 박테리아 집단에서 단백질 합성의 척도로 루신 결합이 사용된다.[39] 방사성 라벨이 부착된 류신을 샘플에 첨가하고, 그 샘플이 단백질로 축적되면서 세포의 뜨거운 삼염화산(CA)-불용성 부분이 결정된다.[39] 그런 다음 샘플은 멤브레인 필터에 수집된다.[39] 류신 단백질은 수생 세균 개체수의 50% 이상이 차지하며, 류신 결합을 통해 박테리아 공동체의 질소 이용률을 추정할 수 있다.[39]
해양 계절적 계승 역학
박테리아 개체군은 고유한 대사 및 자원 선호도를 가지고 있기 때문에 박테리아 성분의 고해상도 시계열 분석을 사용하면 계절적 박테리아 계승에서 패턴을 식별할 수 있다.[40] 박테리아 공동체 구성의 차이는 광합성 플랑크톤, 원생식 경작기, 페이징과의 종간 박테리아 상호작용의 특정한 순열을 발생시켜 계절성 역학관계에 영향을 준다. 모집단 역학 및 구성의 패턴을 검증하기 위해 사용되는 통계적 방법은 몇 년 동안 반복 가능한 것으로 입증되며, 환경적 요인은 이러한 시간적 패턴의 예측 변수 역할을 했다.[41]
온대지방의 계절적 계승
식물성 플랑크톤 개체군의 계절적 성공이 일정한 반복 패턴을 따르기 때문에 박테리아 역학과 식물성 플랑크톤 계승은 상관관계가 있을 수 있다.[40] 일반적으로 박테리아 성분의 계절적 변화는 온도와 엽록소의 변화에 따르는 반면, 영양소의 가용성은 박테리아 소플랑크톤 증가율을 제한한다.[42][43][44][45][6][46] 늦가을/겨울에 물기둥을 섞는 동안, 표면의 킥으로 가져온 영양소는 뚜렷한 규조류 봄꽃을 피우기 시작하고 디노플라겔라게이트가 그 뒤를 잇는다.[40] 봄철 개화 후에는 식물성 플랑크톤 붕괴로부터 용해된 유기물(DOM)의 방출로 세균 생산과 성장이 높아진다.[47][48] 이러한 초기 계승 단계에서 플라보박테리아(박테로이드제) 계급의 구성원들은 전형적으로 박테리아 집단의 지배적인 구성 요소들이다.[49][50] 게놈 분석과 메타트랜스펙토믹스는 DOM의 분해와 동화를 촉진하는 복수의 가수 분해 효소를 함유한 박테리아의 존재를 밝혀냈다.[51][52][53][54] 봄꽃 피는 동안 로소박터클레이드(알파프로테오박테리아)와 일부 감마프로테오박테리아(감마프로테오박테리아)의 일부 멤버는 대개 DOM 분해와 관련이 있다.[48][49] 기온이 상승하고 봄꽃의 영양분이 고갈되면서 현재 과두농도 해역에서는 작은 식물성 플랑크톤과 시아노박테리아가 자란다.[40]
여름철 물이 층화되면서 로소박터, SAR86(감마프로테오박테리아), SAR11(알파프로테오박테리아) 세균군이 풍부하게 쌓인다.[55][56] 자주 관찰되는 가을 규조류와 디노플라겔레이트 꽃은 발트해의 보충 영양소 투입 및 고주파 샘플링과 상관관계가 있으며, 가을에는 일반적으로 액티노박테리아가 증가하며, 그 다음으로 다른 가을 특유의 플라보박테리아, SAR11, Planctomycetes가 뒤따른다는 것을 발견했다.[49]
지중해에서는 깊은 겨울 혼합을 통해 SAR11의 구성원들이 여름 성층화 기간 동안 지배했던 과점 인구가 서서히 소멸함에 따라 다양성을 증가시킬 수 있다.[57] 지중해의 고고학자 중에서는 타우마카게오타 마린그룹Ⅰ(MGI)과 에우리아카게오타 마린그룹Ⅱ(MGIII)가 있다.B) 겨울철 인구가 우세해졌다.[58] 발트해에 있는 동안, 겨울의 혼합은 엡실론-단백질균과 고고인구를 그들의 깊은 서식지에서 표면으로 데려온다.[49]
참조
- ^ Long RA, Azam F (2001-12-05). "Microscale patchiness of bacterioplankton assemblage richness in seawater". Aquatic Microbial Ecology. 26 (2): 103–113. doi:10.3354/ame026103.
- ^ He J, Zhang F, Lin L, Ma Y, Chen J (2012). "Bacterioplankton and picophytoplankton abundance, biomass, and distribution in the Western Canada Basin during summer 2008". Deep Sea Research Part II: Topical Studies in Oceanography. 81–84: 36–45. Bibcode:2012DSRII..81...36H. doi:10.1016/j.dsr2.2012.08.018.
- ^ Wei C, Bao S, Zhu X, Huang X (2008). "Spatio-temporal variations of the bacterioplankton community composition in Chaohu Lake, China". Progress in Natural Science. 18 (9): 1115–1122. doi:10.1016/j.pnsc.2008.04.005.
- ^ López-Flores R, Boix D, Badosa A, Brucet S, Quintana XD (2009). "Environmental factors affecting bacterioplankton and phytoplankton dynamics in confined Mediterranean salt marshes (NE Spain)". Journal of Experimental Marine Biology and Ecology. 369 (2): 118–126. doi:10.1016/j.jembe.2008.11.003.
- ^ Medvinsky, Alexander B.; Adamovich, Boris V.; Aliev, Rubin R.; Chakraborty, Amit; Lukyanova, Elena V.; Mikheyeva, Tamara M.; Nikitina, Ljudmila V.; Nurieva, Nailya I.; Rusakov, Alexey V. (2017). "Temperature as a factor affecting fluctuations and predictability of the abundance of lake bacterioplankton". Ecological Complexity. 32: 90–98. doi:10.1016/j.ecocom.2017.10.002.
- ^ a b Andersson AF, Riemann L, Bertilsson S (February 2010). "Pyrosequencing reveals contrasting seasonal dynamics of taxa within Baltic Sea bacterioplankton communities". The ISME Journal. 4 (2): 171–81. doi:10.1038/ismej.2009.108. PMID 19829318.
- ^ Ghiglione JF, Murray AE (March 2012). "Pronounced summer to winter differences and higher wintertime richness in coastal Antarctic marine bacterioplankton". Environmental Microbiology. 14 (3): 617–29. doi:10.1111/j.1462-2920.2011.02601.x. PMID 22003839.
- ^ Straza TR, Ducklow HW, Murray AE, Kirchman DL (2010-11-01). "Abundance and single-cell activity of bacterial groups in Antarctic coastal waters". Limnology and Oceanography. 55 (6): 2526–2536. Bibcode:2010LimOc..55.2526S. doi:10.4319/lo.2010.55.6.2526.
- ^ Currie DJ, Kalff J (March 1984). "The relative importance of bacterioplankton and phytoplankton in phosphorus uptake in freshwater1". Limnology and Oceanography. 29 (2): 311–321. Bibcode:1984LimOc..29..311C. doi:10.4319/lo.1984.29.2.0311.
- ^ Lindström ES (December 2001). "Investigating Influential Factors on Bacterioplankton Community Composition: Results from a Field Study of Five Mesotrophic Lakes". Microbial Ecology. 42 (4): 598–605. doi:10.1007/s00248-001-0031-y. PMID 12024242. S2CID 22656746.
- ^ Cotner JB, Biddanda BA (2002-03-01). "Small Players, Large Role: Microbial Influence on Biogeochemical Processes in Pelagic Aquatic Ecosystems". Ecosystems. 5 (2): 105–121. CiteSeerX 10.1.1.484.7337. doi:10.1007/s10021-001-0059-3. S2CID 39074312.
- ^ Harnisz M (March 2013). "Total resistance of native bacteria as an indicator of changes in the water environment". Environmental Pollution. 174: 85–92. doi:10.1016/j.envpol.2012.11.005. PMID 23246751.
- ^ Chen, Xinxin; Wang, Kai; Guo, Annan; Dong, Zhiying; Zhao, Qunfen; Qian, Jie; Zhang, Demin (2016). "Excess phosphate loading shifts bacterioplankton community composition in oligotrophic coastal water microcosms over time". Journal of Experimental Marine Biology and Ecology. 483: 139–146. doi:10.1016/j.jembe.2016.07.009.
- ^ Dai, Wenfang; Zhang, Jinjie; Tu, Qichao; Deng, Ye; Qiu, Qiongfen; Xiong, Jinbo (2017). "Bacterioplankton assembly and interspecies interaction indicating increasing coastal eutrophication". Chemosphere. 177: 317–325. Bibcode:2017Chmsp.177..317D. doi:10.1016/j.chemosphere.2017.03.034. PMID 28319885.
- ^ Urakawa, Hidetoshi; Bernhard, Anne E. (2017). "Wetland management using microbial indicators". Ecological Engineering. 108: 456–476. doi:10.1016/j.ecoleng.2017.07.022.
- ^ Haukka K, Kolmonen E, Hyder R, Hietala J, Vakkilainen K, Kairesalo T, Haario H, Sivonen K (February 2006). "Effect of nutrient loading on bacterioplankton community composition in lake mesocosms". Microbial Ecology. 51 (2): 137–46. doi:10.1007/s00248-005-0049-7. PMID 16435168. S2CID 35399139.
- ^ Zhang D, Wang X, Xiong J, Zhu J, Wang Y, Zhao Q, Chen H, Guo A, Wu J (2014). "Bacterioplankton assemblages as biological indicators of shrimp health status". Ecological Indicators. 38: 218–224. doi:10.1016/j.ecolind.2013.11.002.
- ^ Tanious FA, Veal JM, Buczak H, Ratmeyer LS, Wilson WD (1992-03-31). "DAPI (4',6-diamidino-2-phenylindole) binds differently to DNA and RNA: minor-groove binding at AT sites and intercalation at AU sites". Biochemistry. 31 (12): 3103–3112. doi:10.1021/bi00127a010. PMID 1372825.
- ^ Gonzalez K, Mcvey S, Cunnick J, Udovichenko IP, Takemoto DJ (1995). "Acridine orange differential staining of total DNA and RNA in normal and galactosemic lens epithelial cells in culture using flow cytometry". Current Eye Research. 14 (4): 269–273. doi:10.3109/02713689509033525. PMID 7541739.
- ^ Noble RT, Fuhrman JA (1998-02-13). "Use of SYBR Green I for rapid epifluorescence counts of marine viruses and bacteria". Aquatic Microbial Ecology. 14 (2): 113–118. doi:10.3354/ame014113.
- ^ Marie D, Vaulot D, Partensky F (May 1996). "Application of the novel nucleic acid dyes YOYO-1, YO-PRO-1, and PicoGreen for flow cytometric analysis of marine prokaryotes". Applied and Environmental Microbiology. 62 (5): 1649–55. Bibcode:1996ApEnM..62.1649M. doi:10.1128/AEM.62.5.1649-1655.1996. PMC 167939. PMID 8633863.
- ^ a b c d e f Muthukrishnan T, Govender A, Dobretsov S, Abed RM (2017-01-08). "Evaluating the Reliability of Counting Bacteria Using Epifluorescence Microscopy". Journal of Marine Science and Engineering. 5 (1): 4. doi:10.3390/jmse5010004.
- ^ a b O'Connor JT, O'Connor T, Twait R (2009). Water Treatment Plant Performance Evaluations and Operations. John Wiley & Sons, Inc. pp. 193–198. doi:10.1002/9780470431474.app1. ISBN 9780470431474.
- ^ a b c d e f g h i Gasol, Josep M.; Giorgio, Paul A. del (2000-06-30). "Using flow cytometry for counting natural planktonic bacteria and understanding the structure of planktonic bacterial communities". Scientia Marina. 64 (2): 197–224. doi:10.3989/scimar.2000.64n2197. ISSN 1886-8134.
- ^ Giorgio, Paul A. del; Bird, David F.; Prairie, Yves T.; Planas, Dolors (1996-06-01). "Flow cytometric determination of bacterial abundance in lake plankton with the green nucleic acid stain SYTO 13". Limnology and Oceanography. 41 (4): 783–789. Bibcode:1996LimOc..41..783G. doi:10.4319/lo.1996.41.4.0783. ISSN 1939-5590.
- ^ a b c Sieracki, Michael E.; Haugen, Elin M.; Cucci, Terry L. (1995-08-01). "Overestimation of heterotrophic bacteria in the Sargasso Sea: direct evidence by flow and imaging cytometry". Deep Sea Research Part I: Oceanographic Research Papers. 42 (8): 1399–1409. Bibcode:1995DSRI...42.1399S. doi:10.1016/0967-0637(95)00055-B. ISSN 0967-0637.
- ^ a b Zubkov MV, Burkill PH, Topping JN (2007-01-01). "Flow cytometric enumeration of DNA-stained oceanic planktonic protists". Journal of Plankton Research. 29 (1): 79–86. doi:10.1093/plankt/fbl059.
- ^ WATERBURY, JOHN B.; WATSON, STANLEY W.; GUILLARD, ROBERT R. L.; BRAND, LARRY E. (January 1979). "Widespread occurrence of a unicellular, marine, planktonic, cyanobacterium". Nature. 277 (5694): 293–294. Bibcode:1979Natur.277..293W. doi:10.1038/277293a0. ISSN 1476-4687. S2CID 4270426.
- ^ Chisholm, Sallie W.; Frankel, Sheila L.; Goericke, Ralf; Olson, Robert J.; Palenik, Brian; Waterbury, John B.; West-Johnsrud, Lisa; Zettler, Erik R. (1992-02-01). "Prochlorococcus marinus nov. gen. nov. sp.: an oxyphototrophic marine prokaryote containing divinyl chlorophyll a and b". Archives of Microbiology. 157 (3): 297–300. doi:10.1007/BF00245165. ISSN 0302-8933. S2CID 32682912.
- ^ Chisholm, Sallie W.; Olson, Robert J.; Zettler, Erik R.; Goericke, Ralf; Waterbury, John B.; Welschmeyer, Nicholas A. (July 1988). "A novel free-living prochlorophyte abundant in the oceanic euphotic zone". Nature. 334 (6180): 340–343. Bibcode:1988Natur.334..340C. doi:10.1038/334340a0. ISSN 1476-4687. S2CID 4373102.
- ^ a b c d Marie D, Partensky F, Jacquet S, Vaulot D (January 1997). "Enumeration and Cell Cycle Analysis of Natural Populations of Marine Picoplankton by Flow Cytometry Using the Nucleic Acid Stain SYBR Green I". Applied and Environmental Microbiology. 63 (1): 186–93. Bibcode:1997ApEnM..63..186M. doi:10.1128/AEM.63.1.186-193.1997. PMC 1389098. PMID 16535483.
- ^ Giorgio PA, Bird DF, Prairie YT, Planas D (June 1996). "Flow cytometric determination of bacterial abundance in lake plankton with the green nucleic acid stain SYTO 13". Limnology and Oceanography. 41 (4): 783–789. Bibcode:1996LimOc..41..783G. doi:10.4319/lo.1996.41.4.0783.
- ^ Rice J, Sleigh MA, Burkill PH, Tarran GA, O'connor CD, Zubkov MV (March 1997). "Flow Cytometric Analysis of Characteristics of Hybridization of Species-Specific Fluorescent Oligonucleotide Probes to rRNA of Marine Nanoflagellates". Applied and Environmental Microbiology. 63 (3): 938–44. Bibcode:1997ApEnM..63..938R. doi:10.1128/AEM.63.3.938-944.1997. PMC 1389123. PMID 16535558.
- ^ a b c Hagström A, Larsson U, Hörstedt P, Normark S (May 1979). "Frequency of dividing cells, a new approach to the determination of bacterial growth rates in aquatic environments". Applied and Environmental Microbiology. 37 (5): 805–12. Bibcode:1979ApEnM..37..805H. doi:10.1128/AEM.37.5.805-812.1979. PMC 243306. PMID 16345378.
- ^ Newell SY, Christian RR (July 1981). "Frequency of dividing cells as an estimator of bacterial productivity". Applied and Environmental Microbiology. 42 (1): 23–31. Bibcode:1981ApEnM..42...23N. doi:10.1128/AEM.42.1.23-31.1981. PMC 243955. PMID 16345812.
- ^ a b c d Servais P, Martinez J, Billen G, Vives-Rego J (August 1987). "Determining [H]Thymidine Incorporation into Bacterioplankton DNA: Improvement of the Method by DNase Treatment". Applied and Environmental Microbiology. 53 (8): 1977–9. Bibcode:1987ApEnM..53.1977S. doi:10.1128/AEM.53.8.1977-1979.1987. PMC 204039. PMID 16347424.
- ^ Bell R, Ahlgren G, Ahlgren I (June 1983). "Estimating Bacterioplankton Production by Measuring [3H]thymidine Incorporation in a Eutrophic Swedish Lake". Applied and Environmental Microbiology. 45 (6): 1709–1721. Bibcode:1983ApEnM..45.1709B. doi:10.1128/AEM.45.6.1709-1721.1983. PMC 242528. PMID 16346304.
- ^ Fuhrman J, Azam F (July 1980). "Bacterioplankton secondary production estimates for coastal waters of British Columbia, Canada, Antarctica, and California, USA". Applied and Environmental Microbiology. 39 (6): 1085–1095. doi:10.1128/AEM.39.6.1085-1095.1980. PMC 291487. PMID 16345577.
- ^ a b c d Kirchman D, K'nees E, Hodson R (March 1985). "Leucine incorporation and its potential as a measure of protein synthesis by bacteria in natural aquatic systems". Applied and Environmental Microbiology. 49 (3): 599–607. Bibcode:1985ApEnM..49..599K. doi:10.1128/AEM.49.3.599-607.1985. PMC 373556. PMID 3994368.
- ^ a b c d Bunse C, Pinhassi J (June 2017). "Marine Bacterioplankton Seasonal Succession Dynamics". Trends in Microbiology. 25 (6): 494–505. doi:10.1016/j.tim.2016.12.013. PMID 28108182.
- ^ Fuhrman JA, Hewson I, Schwalbach MS, Steele JA, Brown MV, Naeem S (August 2006). "Annually reoccurring bacterial communities are predictable from ocean conditions". Proceedings of the National Academy of Sciences of the United States of America. 103 (35): 13104–9. Bibcode:2006PNAS..10313104F. doi:10.1073/pnas.0602399103. PMC 1559760. PMID 16938845.
- ^ Pinhassi J, Hagström Å (2000-06-15). "Seasonal succession in marine bacterioplankton". Aquatic Microbial Ecology. 21 (3): 245–256. doi:10.3354/ame021245.
- ^ Pinhassi J, Gómez-Consarnau L, Alonso-Sáez L, Sala MM, Vidal M, Pedrós-Alió C, Gasol JM (2006-10-10). "Seasonal changes in bacterioplankton nutrient limitation and their effects on bacterial community composition in the NW Mediterranean Sea". Aquatic Microbial Ecology. 44 (3): 241–252. doi:10.3354/ame044241.
- ^ Sapp M, Wichels A, Wiltshire KH, Gerdts G (March 2007). "Bacterial community dynamics during the winter-spring transition in the North Sea". FEMS Microbiology Ecology. 59 (3): 622–37. doi:10.1111/j.1574-6941.2006.00238.x. PMID 17381518.
- ^ Gilbert JA, Field D, Swift P, Newbold L, Oliver A, Smyth T, Somerfield PJ, Huse S, Joint I (December 2009). "The seasonal structure of microbial communities in the Western English Channel" (PDF). Environmental Microbiology. 11 (12): 3132–9. doi:10.1111/j.1462-2920.2009.02017.x. hdl:1912/3133. PMID 19659500.
- ^ Gilbert JA, Steele JA, Caporaso JG, Steinbrück L, Reeder J, Temperton B, Huse S, McHardy AC, Knight R, Joint I, Somerfield P, Fuhrman JA, Field D (February 2012). "Defining seasonal marine microbial community dynamics". The ISME Journal. 6 (2): 298–308. doi:10.1038/ismej.2011.107. PMC 3260500. PMID 21850055.
- ^ Riemann L, Steward GF, Azam F (February 2000). "Dynamics of bacterial community composition and activity during a mesocosm diatom bloom". Applied and Environmental Microbiology. 66 (2): 578–87. Bibcode:2000ApEnM..66..578R. doi:10.1128/AEM.66.2.578-587.2000. PMC 91866. PMID 10653721.
- ^ a b Buchan A, LeCleir GR, Gulvik CA, González JM (October 2014). "Master recyclers: features and functions of bacteria associated with phytoplankton blooms". Nature Reviews. Microbiology. 12 (10): 686–98. doi:10.1038/nrmicro3326. PMID 25134618. S2CID 26684717.
- ^ a b c d Lindh MV, Sjöstedt J, Andersson AF, Baltar F, Hugerth LW, Lundin D, Muthusamy S, Legrand C, Pinhassi J (July 2015). "Disentangling seasonal bacterioplankton population dynamics by high-frequency sampling". Environmental Microbiology. 17 (7): 2459–76. doi:10.1111/1462-2920.12720. PMID 25403576.
- ^ Alderkamp A, Sintes E, Herndl GJ (2006-12-21). "Abundance and activity of major groups of prokaryotic plankton in the coastal North Sea during spring and summer". Aquatic Microbial Ecology. 45 (3): 237–246. doi:10.3354/ame045237.
- ^ Fernández-Gómez B, Richter M, Schüler M, Pinhassi J, Acinas SG, González JM, Pedrós-Alió C (May 2013). "Ecology of marine Bacteroidetes: a comparative genomics approach". The ISME Journal. 7 (5): 1026–37. doi:10.1038/ismej.2012.169. PMC 3635232. PMID 23303374.
- ^ Teeling H, Fuchs BM, Becher D, Klockow C, Gardebrecht A, Bennke CM, Kassabgy M, Huang S, Mann AJ, Waldmann J, Weber M, Klindworth A, Otto A, Lange J, Bernhardt J, Reinsch C, Hecker M, Peplies J, Bockelmann FD, Callies U, Gerdts G, Wichels A, Wiltshire KH, Glöckner FO, Schweder T, Amann R (May 2012). "Substrate-controlled succession of marine bacterioplankton populations induced by a phytoplankton bloom". Science. 336 (6081): 608–11. Bibcode:2012Sci...336..608T. doi:10.1126/science.1218344. PMID 22556258. S2CID 29249533.
- ^ Teeling H, Fuchs BM, Bennke CM, Krüger K, Chafee M, Kappelmann L, Reintjes G, Waldmann J, Quast C, Glöckner FO, Lucas J, Wichels A, Gerdts G, Wiltshire KH, Amann RI (April 2016). "Recurring patterns in bacterioplankton dynamics during coastal spring algae blooms". eLife. 5: e11888. doi:10.7554/eLife.11888. PMC 4829426. PMID 27054497.
- ^ Taylor JD, Cottingham SD, Billinge J, Cunliffe M (January 2014). "Seasonal microbial community dynamics correlate with phytoplankton-derived polysaccharides in surface coastal waters". The ISME Journal. 8 (1): 245–8. doi:10.1038/ismej.2013.178. PMC 3869024. PMID 24132076.
- ^ Agawin NS, Duarte CM, Agustí S (1998-09-03). "Growth and abundance of Synechococcus sp. in a Mediterranean Bay: seasonality and relationship with temperature". Marine Ecology Progress Series. 170: 45–53. Bibcode:1998MEPS..170...45A. doi:10.3354/meps170045.
- ^ Alonso-Sáez L, Balagué V, Sà EL, Sánchez O, González JM, Pinhassi J, Massana R, Pernthaler J, Pedrós-Alió C, Gasol JM (April 2007). "Seasonality in bacterial diversity in north-west Mediterranean coastal waters: assessment through clone libraries, fingerprinting and FISH". FEMS Microbiology Ecology. 60 (1): 98–112. doi:10.1111/j.1574-6941.2006.00276.x. PMID 17250750.
- ^ Salter I, Galand PE, Fagervold SK, Lebaron P, Obernosterer I, Oliver MJ, Suzuki MT, Tricoire C (February 2015). "Seasonal dynamics of active SAR11 ecotypes in the oligotrophic Northwest Mediterranean Sea". The ISME Journal. 9 (2): 347–60. doi:10.1038/ismej.2014.129. PMC 4303628. PMID 25238399.
- ^ Hugoni M, Taib N, Debroas D, Domaizon I, Jouan Dufournel I, Bronner G, Salter I, Agogué H, Mary I, Galand PE (April 2013). "Structure of the rare archaeal biosphere and seasonal dynamics of active ecotypes in surface coastal waters". Proceedings of the National Academy of Sciences of the United States of America. 110 (15): 6004–9. Bibcode:2013PNAS..110.6004H. doi:10.1073/pnas.1216863110. PMC 3625260. PMID 23536290.


