MA 플롯
MA plot이 글은 주제를 잘 모르는 사람들에게 불충분한 맥락을 제공한다.더하여 할 수 . (2009년 10월)(이 템플리트 |
MA 플롯은 유전체 데이터의 시각적 표현을 위해 Bland-Altman 플롯을 적용한 것이다.그래프는 데이터를 M(로그 비율) 척도와 A(평균 평균) 척도로 변환한 다음 이러한 값을 표시하여 두 표본에서 측정한 측정값 간의 차이를 시각화한다.원래 두 채널 DNA 마이크로어레이 유전자 발현 데이터의 맥락에서 적용되었지만, MA 플롯은 고투과 시퀀싱 분석을 시각화하는 데도 사용된다.[1][2]null
설명
마이크로 어레이 데이터는 염료 결합과 혼합 효율성의 체계적 편향뿐만 아니라 어레이를 식별하는 데 사용되는 DNA 탐침과 인쇄 팁의 다른 기술적 편향을 제어하기 위해 어레이 내에서 정규화되는 경우가 많다.[3]이러한 체계적인 변화를 최소화함으로써 진정한 생물학적 차이를 발견할 수 있다.정상화가 필요한지 판단하려면 Cy3(G) 강도에 대해 Cy5(R) 강도를 표시하고 선의 기울기가 1 정도인지 확인할 수 있다.기본적으로 R 대 G 그림의 45도 회전인 개선된 방법은 MA-플롯이다.[4]MA-플롯은 평균 강도('A')로 표시된 적색/녹색 강도 비율('M')의 분포도를 나타낸 것이다.M과 A는 다음과 같은 방정식으로 정의된다.null
따라서 M은 강도 비율(또는 로그 강도 간의 차이)의 이항 로그이며 A는 그림에서 점의 평균 로그 강도다.그런 다음 MA 그림을 사용하여 원시 마이크로 어레이 데이터의 강도 의존적 비율을 시각화한다(마이크로레이는 일반적으로 여기서 편향을 나타내며, 그 결과 A가 높을수록 즉, 점이 밝을수록 표본과 대조군 사이의 관측된 차이 가능성이 더 높음).MA 그림은 변수 M을 y축에, A를 x축에 배치하며, 데이터의 분포에 대한 간략한 개요를 제공한다.null
많은 마이크로 어레이 유전자 표현 실험에서, 기본적인 가정은 대부분의 유전자가 그들의 표현에 어떤 변화도 보지 못할 것이라는 것이다. 따라서, 로그(1)가 0이기 때문에 y축(M)의 점의 대부분은 0에 위치할 것이다.그렇지 않다면 통계분석 전 데이터에 LOESS와 같은 정상화 방법을 적용해야 한다.(아래 도표에서 정상화 전 영점 이하를 운행하는 적색선을 참조한다)직선적이지 않기 때문에 데이터를 정상화해야 한다.정상화된 후, 빨간색 선이 0선에 직선으로 나타나며 분홍/검은색으로 표시된다.)null
패키지
R 소프트웨어에 대한 몇 가지 바이오콘덕터 패키지는 MA 플롯을 생성할 수 있는 기능을 제공한다.여기에는 아피(ma.plot, mva.pares), 림마(plotMA), 마레이(maPlot) 및 에지R(maPlot)이 포함된다.
caroline CRAN R 패키지의 raPlot 함수를 사용하여 유사한 "RA" 그림을 생성할 수 있다.null
M, A, p-값으로 유전자를 필터링하고, 이름이나 라소로 검색하고, 선택된 유전자를 저장하는 인터랙티브 MA 플롯을 R-Shiny 코드 Enhanced-MA-Plot로 이용할 수 있다.null
R 프로그래밍 언어의 예
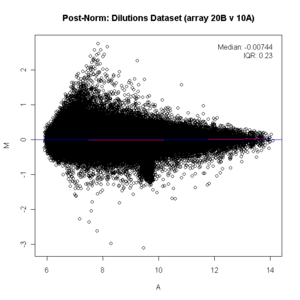
도서관(아첨하다) 만일(요구하다(첨부 자료)) { 자료(희석) } y <- (소비자들(희석)[, c("20B", "10A")]) x11() ma.plot( 로어맨스(로그2(y)), 로그2(y[, 1])-로그2(y[, 2]), 열매를 맺다=1 ) 칭호를 붙이다("Dilutions Dataset(어레이 20B v 10A)") 도서관(preprocessCore) #정량적 정상화를 하다. x <- 정상화하다4분위수(y) x11() ma.plot( 로어맨스(로그2(x)), 로그2(x[, 1])-로그2(x[, 2]), 열매를 맺다=1 ) 칭호를 붙이다("Post Norm: Dilutions Dataset (array 20B v 10A)") 참고 항목
참조
- ^ Robinson, M. D.; McCarthy, D. J.; Smyth, G. K. (11 November 2009). "edgeR: a Bioconductor package for differential expression analysis of digital gene expression data". Bioinformatics. 26 (1): 139–140. doi:10.1093/bioinformatics/btp616. PMC 2796818. PMID 19910308.
- ^ Love, Michael I; Huber, Wolfgang; Anders, Simon (5 December 2014). "Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2". Genome Biology. 15 (12): 550. doi:10.1186/s13059-014-0550-8. PMC 4302049. PMID 25516281.
- ^ YH Yang, S Dudoit, P Luu, DM Lin, V Peng, J Ngai, TP Speed.(2002).cDNA 마이크로 어레이 데이터의 정규화: 단일 슬라이드 및 다중 슬라이드 시스템 변동을 다루는 강력한 합성 방법.핵산 연구 vol. 30 (4) 페이지 e15.
- ^ Dudoit, S, Yang, YH, Callow, MJ, Speed, TP(2002년).복제된 cDNA 마이크로 어레이 실험에서 차등 표현된 유전자를 식별하기 위한 통계적 방법.통계분석 12:1 111–139