빠른 병렬 프로톨리시스
Fast parallel proteolysis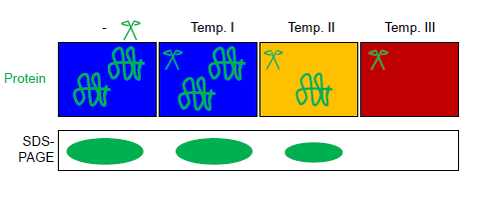
고속 병렬 단백질 분해(Fast parallel proteolyis, FASTpp)는 단백질의 어느 부분이 빠른 단백질 분해 소화에 저항하는지 측정하여 단백질의 온도조절성을 결정하는 방법이다.[1]
역사와 배경
단백질 분해는 단백질 구조를 조사하기 위해 생화학이나 세포 생물학에서 널리 사용된다.[2][3]"제한된 트립신 프로톨리시스"에서 적은 양의 프로테아제는 접힌 단백질과 펴진 단백질 모두를 소화하지만 대체로 다른 비율로: 구조화되지 않은 단백질이 더 빨리 절단되는 반면, 구조화된 단백질은 더 느린 속도로(때로는 규모의 순서로) 절단된다.최근 단백질 분해에 기초한 단백질 안정성의 다른 분석들이 제안되어, 단백질을 분해하기 위해 고유성이 높은 다른 단백질을 이용했다.여기에는 펄스 프로톨리시스,[4] 프로톨리틱스 스캐닝 칼로리메트리 및 FASTpp가 포함된다.
작동 방식
FASTpp는 다양한 조건에서 소화를 억제하는 단백질의 양을 측정한다.이를 위해, 온도 조절 가능한 프로테아제가 사용되는데, 이 프로테아제는 노출된 소수성 잔류물에서 특히 갈라진다.FASTpp 분석은 열전개, 열전열성 프로테아제의 특수성과 SDS-PAGE의 분리력을 결합한 것이다.[6]이러한 조합으로 인해 FASTpp는 최대 85 °C, pH 6-9, 전체 프로테오메의 존재 또는 부재를 포함한 광범위한 물리적 화학적 조건에 걸쳐 접힌 분수의 변화를 감지할 수 있다.적용 범위는 생명공학에서 포인트 돌연변이와 리간드 결합 검사에 이르기까지 다양하다.
적용들
FASTpp는 [1]프로브에 사용됨:
- 리사이트가 단백질 안정성에 미치는 영향
- 열 프로테오메 안정성
- 결합 폴딩 및 바인딩
- 리간드가 접힌 부분과 안정성에 미치는 영향
- 분절 및 안정성에 대한 돌연변이의 영향(예: 점 돌연변이/분실 변이[9][10])
- 운동단백질안정성
기술
첫째, 세포들 유리 구슬, 압력 균질화 또는 화학적 또는 관심의 protein(s)을 가지지 않는다 물리적 용해 방법을 이기고. 생성되(인쇄에 표적 분석)관심이 단백질 이들에서 친화력 방법 본질적으로 어수선한 태그[12]또는 기타 적합한 purificatio에 따라 정화된다.nst여러 직교 크로마토그래피 단계를 수반하는 비율
이 (총 또는 정제된) 단백질 용액은 PCR 스트립의 여러 튜브로 분해된다.모든 지혈대는 열경사 PCR 사이클러에서 온도조절 단백질 분해효소 열성분이 있는 상태에서 다른 최대 온도에 병렬로 노출된다(그림 참조).자동화된 온도 제어는 열 경사로 사이클러(PCR에 일반적으로 사용됨)에서 달성된다.반응 제품은 SDS-PAGE 또는 Western Block으로 분리할 수 있다.[6]프로테아제 열폴리신은 EDTA에 의해 완전히 비활성화될 수 있다.이러한 열성신의 특징은 FASTpp가 질량 분광 분석과 같은 후속 트립신 소화와 호환되도록 한다.[13][14][7]
참조
- ^ a b Minde, D. P.; Maurice, M. M.; Rüdiger, S. G. D. (2012). Uversky, Vladimir N (ed.). "Determining Biophysical Protein Stability in Lysates by a Fast Proteolysis Assay, FASTpp". PLOS ONE. 7 (10): e46147. Bibcode:2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
- ^ Johnson, D. E.; Xue, B.; Sickmeier, M. D.; Meng, J.; Cortese, M. S.; Oldfield, C. J.; Le Gall, T.; Dunker, A. K.; Uversky, V. N. (2012). "High-throughput characterization of intrinsic disorder in proteins from the Protein Structure Initiative". Journal of Structural Biology. 180 (1): 201–215. doi:10.1016/j.jsb.2012.05.013. PMC 3578346. PMID 22651963.
- ^ Hoelen, H.; Kleizen, B.; Schmidt, A.; Richardson, J.; Charitou, P.; Thomas, P. J.; Braakman, I. (2010). Uversky, Vladimir N (ed.). "The Primary Folding Defect and Rescue of ΔF508 CFTR Emerge during Translation of the Mutant Domain". PLOS ONE. 5 (11): e15458. doi:10.1371/journal.pone.0015458. PMC 2994901. PMID 21152102.
- ^ Park, C.; Marqusee, S. (2005). "Pulse proteolysis: A simple method for quantitative determination of protein stability and ligand binding". Nature Methods. 2 (3): 207–212. doi:10.1038/nmeth740. PMID 15782190. S2CID 21364478.
- ^ Tur-Arlandis, G.; Rodriguez-Larrea, D.; Ibarra-Molero, B.; Sanchez-Ruiz, J. M. (2010). "Proteolytic Scanning Calorimetry: A Novel Methodology that Probes the Fundamental Features of Protein Kinetic Stability". Biophysical Journal. 98 (6): L12–L14. Bibcode:2010BpJ....98L..12T. doi:10.1016/j.bpj.2009.11.028. PMC 2849053. PMID 20303845.
- ^ a b Laemmli, U. K. (1970). "Cleavage of structural proteins during the assembly of the head of bacteriophage T4". Nature. 227 (5259): 680–685. Bibcode:1970Natur.227..680L. doi:10.1038/227680a0. PMID 5432063. S2CID 3105149.
- ^ a b Leuenberger, P; Ganscha, S; Kahraman, A (2017). "Cell-wide analysis of protein thermal unfolding reveals determinants of thermostability". Science. 355 (6327): eaai7825. doi:10.1126/science.aai7825. PMID 28232526. S2CID 8432125.
- ^ Demarest, S. J.; Martinez-Yamout, M.; Chung, J.; Chen, H.; Xu, W.; Dyson, H. J.; Evans, R. M.; Wright, P. E. (2002). "Mutual synergistic folding in recruitment of CBP/p300 by p160 nuclear receptor coactivators". Nature. 415 (6871): 549–553. doi:10.1038/415549a. PMID 11823864. S2CID 4423920.
- ^ a b Robaszkiewicz, K.; Ostrowska, Z.; Cyranka-Czaja, A.; Moraczewska, J. (2015). "Impaired tropomyosin–troponin interactions reduce activation of the actin thin filament". Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1854 (5): 381–390. doi:10.1016/j.bbapap.2015.01.004. PMID 25603119.
- ^ Minde, D. P.; Anvarian, Z.; Rüdiger, S. G.; Maurice, M. M. (2011). "Messing up disorder: How do missense mutations in the tumor suppressor protein APC lead to cancer?". Molecular Cancer. 10: 101. doi:10.1186/1476-4598-10-101. PMC 3170638. PMID 21859464.
- ^ Tur-Arlandis, G.; Rodriguez-Larrea, D.; Ibarra-Molero, B.; Sanchez-Ruiz, J. M. (2010). "Proteolytic Scanning Calorimetry: A Novel Methodology that Probes the Fundamental Features of Protein Kinetic Stability". Biophysical Journal. 98 (6): L12–L14. Bibcode:2010BpJ....98L..12T. doi:10.1016/j.bpj.2009.11.028. PMC 2849053. PMID 20303845.
- ^ Minde, DP; Halff, EF; Tans, SJ (2013). "Designing disorder: Tales of the unexpected tails". Intrinsically Disordered Proteins. 1 (1): e26790. doi:10.4161/idp.26790. PMC 5424805. PMID 28516025.
- ^ Chang, Y; Schlebach, JP; Verheul, RA; Park, C (2012). "Simplified proteomics approach to discover protein-ligand interactions". Protein Science. 21 (9): 1280–7. doi:10.1002/pro.2112. PMC 3631357. PMID 22733688.
- ^ Park, C; Marqusee, S (2005). "Pulse proteolysis: A simple method for quantitative determination of protein stability and ligand binding". Nature Methods. 2 (3): 207–12. doi:10.1038/nmeth740. PMID 15782190. S2CID 21364478.


