돌연변이 유발(분자생물학 기술)
Mutagenesis (molecular biology technique)분자생물학에서, 돌연변이 유발은 DNA 돌연변이가 돌연변이 유전자, 단백질, 박테리아 변종, 또는 다른 유전자 변형 유기체의 라이브러리를 생성하기 위해 의도적으로 조작되는 중요한 실험실 기술이다.유전자의 다양한 구성 요소와 조절 요소 및 유전자 생성물은 유전자 궤적, 과정 또는 생성물의 기능을 상세하게 조사할 수 있도록 돌연변이될 수 있다.이 돌연변이는 상업적으로 사용될 수 있는 흥미로운 특성이나 강화된 또는 새로운 기능을 가진 돌연변이 단백질을 생성할 수 있다.또한 실용적 응용을 가지거나 특정 세포 기능의 분자적 기초를 조사할 수 있는 돌연변이 균주가 생성될 수 있다.
오늘날에는 많은 돌연변이 유발 방법이 존재한다.처음에 실험실에서 인위적으로 유도된 돌연변이의 종류는 UV 조사와 같은 메커니즘을 사용하여 완전히 무작위였다.무작위 돌연변이 유발은 게놈의 특정 영역이나 염기서열을 대상으로 할 수 없지만, 부위 지향 돌연변이 유발의 발달로 보다 구체적인 변화가 이루어질 수 있다.2013년부터 원핵 바이러스 방어 시스템을 기반으로 한 CRISPR/Cas9 기술의 개발로 생체 [1]내 게놈 편집 또는 돌연변이 발생이 가능해졌다.사이트 지향 돌연변이 유발은 무작위 돌연변이 유발이 아닌 상황에서 유용하다는 것이 입증되었습니다.돌연변이 유발의 다른 기술로는 조합형 돌연변이 유발과 삽입형 돌연변이 유발이 있다.무작위가 아닌 돌연변이 유발은 DNA [2]복제, 돌연변이 [3]효과 조사, 단백질 [4]공학을 위해 사용될 수 있다.또한 면역 결핍 환자 지원, HIV와 암을 포함한 질병의 연구와 치료, 베타 시상혈증과 [5]같은 질병의 치료와 같은 의학적 응용 프로그램도 있습니다.
무작위 돌연변이 유발
돌연변이 유발에 대한 초기 접근법은 전적으로 무작위 돌연변이를 생성하는 방법에 의존했다.이러한 방법으로 세포 또는 유기체를 자외선이나 돌연변이 유발 화학물질 등의 돌연변이에 피폭시킨 후 원하는 특성을 가진 돌연변이를 선택한다.헤르만 뮬러는 1927년 엑스레이가 [6]초파리에게 유전적 돌연변이를 일으킬 수 있다는 것을 발견하고 유전학 [7]연구를 위해 자신이 만든 돌연변이를 계속 사용했다.대장균의 경우 돌연변이를 먼저 자외선에 노출시켜 선택한 후 한천배지에 도금할 수 있다.형성된 군락은 복제 도금되고, 하나는 풍부한 배지에, 다른 하나는 최소 배지에, 그리고 다른 하나는 특정 영양 요구 사항을 가진 돌연변이는 최소 배지에서 자랄 수 없는 것으로 확인될 수 있습니다.다른 유형의 세포와 다른 매체에서 선택을 위해 유사한 절차를 반복할 수 있습니다.
특정 단백질에서 무작위 돌연변이를 생성하기 위한 많은 방법들이 나중에 흥미롭고 개선된 특성을 가진 돌연변이를 선별하기 위해 개발되었다.이러한 방법들은 올리고뉴클레오티드 합성에 도프된 뉴클레오티드의 사용 또는 복제의 충실도를 낮추거나 뉴클레오티드 [8]유사체를 사용함으로써 뉴클레오티드의 오인입을 강화하는 조건에서 PCR 반응을 수행할 수 있다.유전자에 비바이어스 돌연변이를 통합하기 위한 본 방법의 변형은 배열 포화 돌연변이 [9]유발이다.다음으로 돌연변이를 포함한 PCR 생성물을 발현 벡터로 복제하고 생성된 돌연변이 단백질을 특성화할 수 있다.
동물 연구에서 돌연변이 쥐를 [10][11]생성하기 위해 N-에틸-N-니트로소요소(ENU)와 같은 알킬화제가 사용되어 왔다.에틸메탄술폰산염(EMS)은 동물,[12][13][14] 식물 및 바이러스 돌연변이 생성에도 자주 사용됩니다.
유럽 연합법(2001/18년 지침)에서는 이러한 종류의 돌연변이 유발을 GMO 생산에 사용할 수 있지만, 제품은 규제에서 면제됩니다. 즉, 라벨 부착이나 [15]평가 없음입니다.
사이트 지향 돌연변이 유발
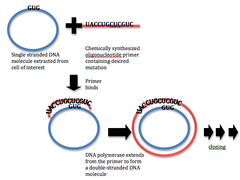
개발 현장 지향 돌연변이 유발 기술 이전에는 모든 돌연변이가 무작위였으며, 과학자들은 원하는 돌연변이를 찾기 위해 원하는 표현형에 대한 선택을 사용해야 했다.무작위 돌연변이 유발 기술은 얼마나 많은 돌연변이가 생성될 수 있는지에 있어 장점이 있다; 그러나 무작위 돌연변이 유발은 단일 뉴클레오티드의 변화를 일으킬 수 있지만, 어떤 뉴클레오티드가 [5]변화하고 있는지에 대해서는 많은 제어를 제공하지 않는다.따라서 많은 연구자들은 DNA에 정확한 부위별 방식으로 선택된 변화를 도입하려고 한다.초기 시도는 국소적인 점 [16]돌연변이를 생성하기 위해 뉴클레오티드와 다른 화학 물질의 유사체를 사용했다.이러한 화학물질에는 AT에서 GC로의 [17]전환을 유도하는 아미노푸린이 포함되며, 니트로소구아니딘,[18] 비황산비소 [19]및 N-히드록시사이티딘은4 GC에서 AT로의 [20][21]전환을 유도할 수 있다.이러한 기술은 특정 돌연변이를 단백질로 조작할 수 있게 한다.그러나 생성된 돌연변이의 종류에 관해서는 유연하지 않으며, 부위 지향적인 돌연변이 유발의 최신 방법만큼 구체적이지 않기 때문에 어느 정도 무작위성이 있다.염색체의 특정 부위에 있는 DNA의 분열, 새로운 뉴클레오티드의 추가, 그리고 염기쌍의 교환과 같은 다른 기술들은 이제 돌연변이가 어디로 [11][8]갈 수 있는지를 결정하는 것이 가능하다.
부위 특이적 돌연변이를 위한 현재의 기법은 1978년에 개발된 프라이머 확장 기법에서 비롯되었다.이러한 기술은 일반적으로 DNA 중합효소와의 프라이머 확장 반응에서 사전 제작된 돌연변이 유발 올리고뉴클레오티드를 사용하는 것을 포함한다.이 방법을 사용하면 특정 부위에 점 돌연변이 또는 작은 DNA의 삭제 또는 삽입이 가능합니다.방법론의 발전으로 이러한 돌연변이 발생은 이제 비교적 단순하고 효율적인 [3]과정이 되었다.
보다 새롭고 효율적인 사이트 지향적 돌연변이 발생 방법이 지속적으로 개발되고 있습니다.예를 들어, "심리스 결찰 복제 추출물" (또는 줄여서 SLiCE)이라고 불리는 기술은 게놈 내의 특정 DNA 배열의 복제를 허용하고, 한 번에 [2]두 개 이상의 DNA 조각을 게놈에 삽입할 수 있습니다.
부위 지향 돌연변이 유발을 통해 특정 돌연변이의 효과를 조사할 수 있습니다.예를 들어, 특정 종이 실험실에서 자주 사용되는 화학 물질에 얼마나 민감한지 결정하는 데 사용되어 왔습니다.실험은 특정 화학 물질의 예상되는 돌연변이를 모방하기 위해 돌연변이를 유도하는 것을 사용했다.돌연변이는 특정 아미노산의 변화를 초래했고, 이 돌연변이의 영향을 [3]분석하였다.
부위 지향적 접근은 단백질의 [22]구조 또는 기능에 중요한 잔류물을 식별하기 위해 알라닌으로 체계적으로 변이되는 알라닌 주사 돌연변이 발생과 같은 기술로 체계적으로 이루어질 수 있다.또 하나의 포괄적인 접근방식은 부위 포화 돌연변이 유발이며, 여기서 하나의 코돈 또는 일련의 코돈이 특정 위치에서 [23][24]가능한 모든 아미노산으로 치환될 수 있다.
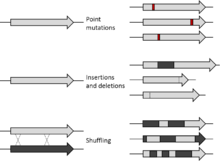
조합 돌연변이 유발
조합 돌연변이 유발은 첨가물 개별 [25]돌연변이의 효과 분석에 기초하여 단백질의 여러 돌연변이를 동시에 조작할 수 있는 부위 지향 단백질 공학 기술이다.단백질 [26]기능에 대한 다수의 돌연변이의 조합 효과를 평가하는 유용한 방법을 제공합니다.다수의 돌연변이는 조합 [25]분석에 의해 특정 특성을 선별할 수 있다.본 기술에서는 돌연변이 [25]단백질의 포괄적인 라이브러리를 얻기 위해 DNA 가닥을 따라 여러 위치 또는 짧은 배열을 완전히 수정할 수 있다.유익한 변이체의 발생률은 돌연변이 유발 라이브러리를 구성하는 다른 방법에 의해 개선될 수 있다.이 기술에 대한 한 가지 접근법은 원하는 돌연변이 부위에서 모든 가능한 조합을 포함하는 배열 라이브러리로 DNA 배열의 일부를 추출하고 대체하는 것이다.삽입된 세그먼트의 내용에는 구조적 중요성, 면역원성 특성 또는 효소 기능의 시퀀스가 포함될 수 있습니다.단백질의 [25]특정 부분의 구조적 또는 기능적 의의를 평가하기 위해 세그먼트를 유전자에 랜덤으로 삽입할 수도 있다.
삽입형 돌연변이 유발
DNA 돌연변이를 일으키는 하나 이상의 염기쌍의 삽입은 삽입형 돌연변이 [27]유발이라고도 한다.이와 같은 공학적 돌연변이는 암 연구에 중요한 정보를 제공할 수 있습니다. 예를 들어 질병의 발달에 대한 기계적 통찰력입니다.레트로바이러스와 트랜스포존은 삽입형 돌연변이 유발의 주요 도구이다.쥐 유방 종양 바이러스나 쥐 백혈병 바이러스와 같은 레트로바이러스는 발암과 관련된 유전자를 식별하고 특정 [28]암의 생물학적 경로를 이해하는 데 사용될 수 있다.전이가 가능한 염색체 세그먼트인 트랜스포존은 암 유전자 [28]발견의 도구로서 삽입형 돌연변이 유발에 적용되고 설계될 수 있다.이러한 염색체 세그먼트는 DNA 염기서열 [28]분석에서 보다 포괄적이고 편향되지 않은 깊이를 허용하는 동시에 삽입형 돌연변이 유발을 거의 모든 조직에 적용할 수 있게 한다.
연구자들은 인간에게 사용될 수 있는 삽입형 돌연변이 유발의 네 가지 메커니즘을 발견했다.첫 번째 메커니즘은 인핸서 삽입이라고 불립니다.인핸서는 특정 유전자의 프로모터와 상호작용함으로써 특정 유전자의 전사를 촉진한다.이 특별한 메커니즘은 내가 골수에 필요로 하는 심각한 면역 결핍 환자들을 돕기 위해 처음 사용되었다.그리고 나서 강화제를 실은 감마레브로바이러스를 환자에게 삽입했다.두 번째 메커니즘은 프로모터 삽입이라고 합니다.프로모터는 델의 셀에 번역을 시작하는 데 필요한 특정 시퀀스를 제공합니다.프로모터 삽입은 연구자들이 HIV 바이러스에 대해 더 많은 것을 알 수 있도록 도와왔다.세 번째 메커니즘은 유전자 불활성화이다.유전자 불활성화의 예로는 백혈병 환자의 T세포의 게놈을 교란시키는 레트로바이러스를 삽입하여 T세포가 암세포를 표적화하도록 하는 CAR라는 특정 항원을 제공하는 것이 있다.마지막 메커니즘은 mRNA 3' 말단 치환이라고 한다.우리의 유전자는 때때로 적혈구 기능을 방해하는 베타 시상혈증을 일으키는 점 돌연변이를 겪는다.이 문제를 해결하기 위해 적혈구를 위한 올바른 유전자 배열이 도입되고 대체가 이루어진다.[5]
상동재조합
상동 재조합은 유기체의 특정 돌연변이를 생성하기 위해 사용될 수 있다.수정되는 유전자와 유사한 DNA 배열을 포함한 벡터가 세포에 도입되어 재조합 과정에 의해 염색체 내의 표적 유전자를 치환한다.이 방법은 예를 들어 녹아웃 생쥐의 생산에 사용되는 것과 같이 돌연변이를 도입하거나 유전자를 녹아웃하는 [29]데 사용될 수 있다.
크리스퍼
2013년 이후, CRISPR-Cas9 기술의 개발로 다양한 종류의 돌연변이를 다양한 생물의 게놈에 효율적으로 도입할 수 있게 되었다.이 방법은 트랜스포존 삽입 부위가 필요하지 않고 마커를 남기지 않으며, 효율성과 단순성으로 인해 게놈 [30][31]편집에 선호되는 방법이 되었다.
유전자 합성
DNA 올리고뉴클레오티드 합성 비용이 떨어짐에 따라, 완전한 유전자의 인공 합성은 이제 유전자에 돌연변이를 도입하는 실행 가능한 방법이다.이 방법은 특정 [32]유기체에 최적화하기 위해 유전자의 코돈 사용을 완전히 재설계하는 것을 포함하여 여러 부위에서 광범위한 돌연변이를 허용합니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Hsu PD, Lander ES, Zhang F (June 2014). "Development and applications of CRISPR-Cas9 for genome engineering". Cell. 157 (6): 1262–78. doi:10.1016/j.cell.2014.05.010. PMC 4343198. PMID 24906146.
- ^ a b Motohashi K (June 2015). "A simple and efficient seamless DNA cloning method using SLiCE from Escherichia coli laboratory strains and its application to SLiP site-directed mutagenesis". BMC Biotechnology. 15: 47. doi:10.1186/s12896-015-0162-8. PMC 4453199. PMID 26037246.
- ^ a b c Doering JA, Lee S, Kristiansen K, Evenseth L, Barron MG, Sylte I, LaLone CA (November 2018). "In Silico Site-Directed Mutagenesis Informs Species-Specific Predictions of Chemical Susceptibility Derived From the Sequence Alignment to Predict Across Species Susceptibility (SeqAPASS) Tool". Toxicological Sciences. 166 (1): 131–145. doi:10.1093/toxsci/kfy186. PMC 6390969. PMID 30060110.
- ^ Choi GC, Zhou P, Yuen CT, Chan BK, Xu F, Bao S, et al. (August 2019). "Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9". Nature Methods. 16 (8): 722–730. doi:10.1038/s41592-019-0473-0. PMID 31308554. S2CID 196811756.
- ^ a b c Bushman FD (February 2020). "Retroviral Insertional Mutagenesis in Humans: Evidence for Four Genetic Mechanisms Promoting Expansion of Cell Clones". Molecular Therapy. 28 (2): 352–356. doi:10.1016/j.ymthe.2019.12.009. PMC 7001082. PMID 31951833.
- ^ Muller HJ (July 1927). "Artificial Transmutation of the Gene" (PDF). Science. 66 (1699): 84–7. Bibcode:1927Sci....66...84M. doi:10.1126/science.66.1699.84. PMID 17802387.
- ^ Crow JF, Abrahamson S (December 1997). "Seventy years ago: mutation becomes experimental". Genetics. 147 (4): 1491–6. doi:10.1093/genetics/147.4.1491. PMC 1208325. PMID 9409815.
- ^ a b Blackburn GM, ed. (2006). Nucleic Acids in Chemistry and Biology (3rd ed.). Royal Society of Chemistry. pp. 191–192. ISBN 978-0854046546.
- ^ Wong TS, Tee KL, Hauer B, Schwaneberg U (February 2004). "Sequence saturation mutagenesis (SeSaM): a novel method for directed evolution". Nucleic Acids Research. 32 (3): 26e–26. doi:10.1093/nar/gnh028. PMC 373423. PMID 14872057.
- ^ Justice MJ, Noveroske JK, Weber JS, Zheng B, Bradley A (1999). "Mouse ENU mutagenesis". Human Molecular Genetics. 8 (10): 1955–63. doi:10.1093/hmg/8.10.1955. PMID 10469849.
- ^ a b Hrabé de Angelis M, Balling R (May 1998). "Large scale ENU screens in the mouse: genetics meets genomics". Mutation Research. 400 (1–2): 25–32. doi:10.1016/s0027-5107(98)00061-x. PMID 9685575.
- ^ Flibotte S, Edgley ML, Chaudhry I, Taylor J, Neil SE, Rogula A, et al. (June 2010). "Whole-genome profiling of mutagenesis in Caenorhabditis elegans". Genetics. 185 (2): 431–41. doi:10.1534/genetics.110.116616. PMC 2881127. PMID 20439774.
- ^ Bökel C (2008). EMS screens : from mutagenesis to screening and mapping. Methods in Molecular Biology. Vol. 420. pp. 119–38. doi:10.1007/978-1-59745-583-1_7. PMID 18641944.
- ^ Favor AH, Llanos CD, Youngblut MD, Bardales JA (2020). "Optimizing bacteriophage engineering through an accelerated evolution platform". Scientific Reports. 10 (1): 13981. doi:10.1038/s41598-020-70841-1. PMC 7438504. PMID 32814789.
- ^ Krinke C (March 2018). "GMO directive : the origins of the mutagenesis exemption". Inf'OGM.
- ^ Shortle D, DiMaio D, Nathans D (1981). "Directed mutagenesis". Annual Review of Genetics. 15: 265–94. doi:10.1146/annurev.ge.15.120181.001405. PMID 6279018.
- ^ Caras IW, MacInnes MA, Persing DH, Coffino P, Martin DW (September 1982). "Mechanism of 2-aminopurine mutagenesis in mouse T-lymphosarcoma cells". Molecular and Cellular Biology. 2 (9): 1096–103. doi:10.1128/mcb.2.9.1096. PMC 369902. PMID 6983647.
- ^ McHugh GL, Miller CG (October 1974). "Isolation and characterization of proline peptidase mutants of Salmonella typhimurium". Journal of Bacteriology. 120 (1): 364–71. doi:10.1128/JB.120.1.364-371.1974. PMC 245771. PMID 4607625.
- ^ Shortle D, Nathans D (May 1978). "Local mutagenesis: a method for generating viral mutants with base substitutions in preselected regions of the viral genome". Proceedings of the National Academy of Sciences of the United States of America. 75 (5): 2170–4. Bibcode:1978PNAS...75.2170S. doi:10.1073/pnas.75.5.2170. PMC 392513. PMID 209457.
- ^ Flavell RA, Sabo DL, Bandle EF, Weissmann C (January 1975). "Site-directed mutagenesis: effect of an extracistronic mutation on the in vitro propagation of bacteriophage Qbeta RNA". Proceedings of the National Academy of Sciences of the United States of America. 72 (1): 367–71. Bibcode:1975PNAS...72..367F. doi:10.1073/pnas.72.1.367. PMC 432306. PMID 47176.
- ^ Müller W, Weber H, Meyer F, Weissmann C (September 1978). "Site-directed mutagenesis in DNA: generation of point mutations in cloned beta globin complementary dna at the positions corresponding to amino acids 121 to 123". Journal of Molecular Biology. 124 (2): 343–58. doi:10.1016/0022-2836(78)90303-0. PMID 712841.
- ^ Vanessa E. Gray; Ronald J. Hause; Douglas M. Fowler (September 1, 2017). "Analysis of Large-Scale Mutagenesis Data To Assess the Impact of Single Amino Acid Substitutions". Genetics. 207 (1): 53–61. doi:10.1534/genetics.117.300064. PMC 5586385. PMID 28751422.
- ^ Reetz, M. T.; Carballeira J. D. (2007). "Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes". Nature Protocols. 2 (4): 891–903. doi:10.1038/nprot.2007.72. PMID 17446890. S2CID 37361631.
- ^ Cerchione, Derek; Loveluck, Katherine; Tillotson, Eric L.; Harbinski, Fred; DaSilva, Jen; Kelley, Chase P.; Keston-Smith, Elise; Fernandez, Cecilia A.; Myer, Vic E.; Jayaram, Hariharan; Steinberg, Barrett E.; Xu, Shuang-yong (16 April 2020). "SMOOT libraries and phage-induced directed evolution of Cas9 to engineer reduced off-target activity". PLOS ONE. 15 (4): e0231716. Bibcode:2020PLoSO..1531716C. doi:10.1371/journal.pone.0231716. PMC 7161989. PMID 32298334.
- ^ a b c d Parker AS, Griswold KE, Bailey-Kellogg C (November 2011). "Optimization of combinatorial mutagenesis". Journal of Computational Biology. 18 (11): 1743–56. Bibcode:2011LNCS.6577..321P. doi:10.1089/cmb.2011.0152. PMC 5220575. PMID 21923411.
- ^ Choi GC, Zhou P, Yuen CT, Chan BK, Xu F, Bao S, Chu HY, Thean D, Tan K, Wong KH, Zheng Z, Wong AS (August 2019). "Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9". Nature Methods. 16 (8): 722–730. doi:10.1038/s41592-019-0473-0. PMID 31308554. S2CID 196811756.
- ^ Uren AG, Kool J, Berns A, van Lohuizen M (November 2005). "Retroviral insertional mutagenesis: past, present and future". Oncogene. 24 (52): 7656–72. doi:10.1038/sj.onc.1209043. PMID 16299527.
- ^ a b c Vassiliou G, Rad R, Bradley A (2010-01-01). Wassarman PM, Soriano PM (eds.). "The use of DNA transposons for cancer gene discovery in mice". Methods in Enzymology. Guide to Techniques in Mouse Development, Part B: Mouse Molecular Genetics (2nd ed.). Academic Press. 477: 91–106. doi:10.1016/s0076-6879(10)77006-3. ISBN 9780123848802. PMID 20699138.
- ^ "Homologous Recombination Method (and Knockout Mouse)". Davidson College.
- ^ Damien Biot-Pelletier; Vincent J. J. Martin (2016). "Seamless site-directed mutagenesis of the Saccharomyces cerevisiae genome using CRISPR-Cas9". Journal of Biological Engineering. 10: 6. doi:10.1186/s13036-016-0028-1. PMC 4850645. PMID 27134651.
- ^ Xu S (20 August 2015). "The application of CRISPR-Cas9 genome editing in Caenorhabditis elegans". J Genet Genomics. 42 (8): 413–21. doi:10.1016/j.jgg.2015.06.005. PMC 4560834. PMID 26336798.
- ^ Khudyakov YE, Fields HA, eds. (25 September 2002). Artificial DNA: Methods and Applications. CRC Press. p. 13. ISBN 9781420040166.
외부 링크
| 라이브러리 리소스 정보 돌연변이 발생 |