엑솜 시퀀스
Exome sequencing전체 엑솜 배열 배열법(WES)으로도 알려진 엑솜 배열법은 게놈(엑솜으로 알려진)에서 유전자의 모든 단백질 코드 영역은 엑솜으로 알려져 있다.그것은 두 단계로 구성되어 있다: 첫 번째 단계는 단백질을 암호화하는 DNA의 하위 집합만을 선택하는 것이다.이 지역들은 엑손이라고 알려져 있다.인간은 인간 게놈의 약 1% 또는 약 3000만 개의 염기쌍을 구성하는 약 18만 개의 엑손이 있다.두 번째 단계는 처리량이 높은 DNA 배열 [1]기술을 사용하여 엑소닉 DNA의 배열을 결정하는 것입니다.
이 접근법의 목표는 단백질 서열을 바꾸는 유전자 변형을 식별하고, 이것을 전체 유전자 서열을 바꾸는 것보다 훨씬 낮은 비용으로 하는 것이다.이러한 변종들은 멘델병과 알츠하이머병과 같은 일반적인 다유전성 질환 모두에 영향을 미칠 수 있기 때문에, 전체 엑솜 배열은 학술 연구 및 임상 진단에 모두 적용되어 왔다.
동기 부여 및 다른 접근법과의 비교
엑솜 배열은 특히 멘델병 연구에 효과적이다.왜냐하면 엑솜 배열은 개인의 모든 유전자에 있는 유전적 변형을 확인하는 효과적인 방법이기 때문이다.이러한 질병들은 매우 드문 유전적 변이들에 의해 종종 발생한다;[2] 대조적으로, SNP 배열과 같은 기술들은 더 넓은 집단의 [3]많은 개인들에게 공통적인 공통적인 유전 변이들을 발견할 수 있을 뿐이다.게다가 심각한 질병을 일으키는 변종들은 단백질 코드[citation needed] 배열에 있을 가능성이 훨씬 높기 때문에(단, 배타적인 것은 아니다), 이 1%의 비용에 초점을 맞추고 있지만 여전히 관련된 변종들의 높은 산출량을 검출한다.
과거에 임상 유전자 테스트는 환자의 임상 증상(즉, 특정 증후군과 관련된 것으로 알려진 하나의 유전자 또는 소수의 유전자에 초점을 맞춘 것)을 기반으로 선택되거나 특정 유형의 변형만을 조사했지만(예: 비교 유전자 교배) 전체 패턴의 절반 미만으로 최종 유전자 진단을 제공했다.exome [4]시퀀싱은 이러한 다른 테스트들을 보완하기 위해 점점 더 많이 사용되고 있다: 질병을 일으키는 것으로 이미 알려진 유전자의 돌연변이를 찾는 것뿐만 아니라 비슷한 [citation needed]특징을 가진 환자의 exome을 비교함으로써 새로운 유전자를 확인하는 것.
기술적 방법론
1단계: 목표 강화 전략
표적 증강 방법은 염기서열 분석 전에 DNA 샘플에서 관심 있는 게놈 영역을 선택적으로 포착할 수 있도록 한다.2005년 [5]직접 게놈 선택(DGS) 방법에 대한 최초 기술 이후 몇 가지 표적 증강 전략이 개발되었다.
표적 포획을 위해 많은 기술이 설명되었지만, 이 중 일부 기술만 전체 [6]엑소시움 포획으로 확장되었습니다.전체 엑솜 시퀀싱에 적용된 첫 번째 타깃 강화 전략은 2007년 어레이 기반 하이브리드 캡처 방식이었지만 최근에는 솔루션 내 캡처가 인기를 끌고 있습니다.
어레이 기반 캡처
마이크로어레이는 표면에 고정된 관심 영역을 타일링하기 위해 인간 게놈의 배열과 함께 단가닥 올리고뉴클레오티드를 포함한다.게놈 DNA는 이중가닥 단편을 형성하기 위해 절단된다.파편들은 끝이 뭉툭한 부분을 만들기 위해 최종 수복을 거치고 범용 프라이밍 시퀀스를 가진 어댑터가 추가된다.이 조각들은 마이크로 어레이의 올리고에 교배됩니다.하이브리드화되지 않은 단편은 씻겨나가고 원하는 단편은 용출된다.그런 다음 PCR을 [7][8]사용하여 fragment를 증폭합니다.
Roche NimbleGen은 최초의 DGS 기술을[5] 채택하여 차세대 시퀀싱에 적용하였습니다.Sequence Capture Human Exome 2.1M Array를 개발하여 최대 180,000개의 코딩 엑손을 [9]캡처했습니다.이 방법은 PCR 기반 방식에 비해 시간 절약과 비용 효율이 모두 뛰어납니다.Agilent Capture Array와 비교 게놈 하이브리드 배열은 표적 시퀀스의 하이브리드 캡처에 사용할 수 있는 다른 방법입니다.이 기술에는 고가의 하드웨어와 비교적 많은 [10]양의 DNA가 필요합니다.
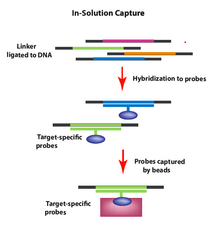
솔루션 내 캡처
용액 내 포획을 사용하여 관심 게놈 영역을 포착하기 위해 맞춤형 올리고뉴클레오티드(프로브) 풀이 단편화된 게놈 DNA 샘플의 용액으로 합성 및 교배된다.프로브(구슬로 라벨링됨)는 관심 있는 게놈 영역에 선택적으로 교배되며, 그 후 비즈(현재 관심 있는 DNA 조각 포함)를 끌어내리고 세척하여 여분의 물질을 제거할 수 있습니다.그런 다음 비즈를 제거하고 게놈 조각의 염기서열을 분석하여 관심 있는 게놈 영역(예: 엑손)의 선택적 DNA 염기서열을 확인할 수 있습니다.
이 방법은 잡종 포획 목표 증강 방법을 개선하기 위해 개발되었습니다.(하이브리드 캡처와는 달리) 솔루션 캡처에서는 필요한 [10]템플릿의 양을 초과하는 관심 영역을 대상으로 하는 프로브가 있습니다.최적의 타깃 사이즈는 약 3.5 메가베이스이며 타깃 영역의 우수한 시퀀스 커버리지를 제공합니다.바람직한 방법은 관심 영역의 기본 쌍 수, 타깃에 대한 읽기 요구, 사내 장비 [11]등 여러 요소에 따라 달라집니다.
순서 2: 순서 지정
기존 Sanger 시퀀싱 방법론 이후 많은 차세대 시퀀싱 플랫폼을 사용할 수 있습니다.다른 플랫폼에는 Roche 454 시퀀서와 Life Technologies SOLiD 시스템, Life Technologies Ion Torrent와 Illumina의 Illlumina Genome Analyzer II(구형) 및 후속 Ilumina MiSeq, HiSeq 및 NovaSeq 시리즈 계측기가 있으며, 이 모든 계측기는 대규모 병렬 시퀀싱에 사용할 수 있습니다.이러한 '짧은 읽기' NGS 시스템은 특히 인간 엑손에서 발견되는 많은 비교적 짧은 DNA 염기서열을 분석하는 데 적합하다.
다른 테크놀로지와의 비교
유전자 변형을 식별하는 여러 가지 기술이 있습니다.각 테크놀로지는 기술적, 재정적 요인 측면에서 장점과 단점을 가지고 있습니다.그러한 두 가지 기술은 마이크로어레이와 전체 유전자 염기서열 분석이다.
마이크로 어레이 기반의 유전자형식
마이크로어레이는 알려진 DNA 배열의 유병률을 테스트하기 위해 잡종화 프로브를 사용합니다. 따라서 그것들은 예기치 않은 [10]유전자 변화를 확인하는 데 사용될 수 없습니다.이와는 대조적으로, 엑솜 배열에 사용되는 높은 처리량 배열 기술은 테스트된 [12]수천 개의 엑소닉 궤적에서 DNA의 뉴클레오티드 배열을 직접 제공한다.따라서 WES는 하이브리드제너타이핑 어레이의 현재 제한사항 중 일부에 대처하고 있습니다.
엑솜 염기서열 분석은 샘플 단위로 하이브리드 기반 기술보다 비용이 많이 들지만, 전체 게놈 염기서열 [citation needed]분석의 비용 감소와 처리량 증가로 인해 비용이 감소하고 있습니다.
전체 유전자 염기서열 분석
엑솜 배열은 단백질 기능에 영향을 미치는 유전자의 코드화 영역에서 발견되는 변종만을 식별할 수 있다.전체 게놈 배열 [1]분석과 같은 다른 방법을 사용하여 찾을 수 있는 질병과 관련된 구조 및 비코드 변형을 식별할 수 없습니다.인간 게놈의 99%가 엑솜 염기서열 분석을 사용하지 않고 있으며 엑솜 염기서열 분석을 통해 전체 게놈 염기서열 [1]분석과 비교하여 최소 20배 이상의 샘플에서 게놈 부분의 염기서열을 분석할 수 있습니다.확인된 희귀 변종을 클리닉으로 변환하기 위해 샘플 크기와 임상 진단을 제공하기 위해 결과를 해석할 수 있는 능력은 유전학의 현재 지식으로 진단을 [9]보조하기 위해 사용되는 Exome Sequencing에 대한 보고가 있음을 나타냅니다.엑솜 배열의 비용은 일반적으로 전체 게놈 [13]배열보다 낮다.
데이터 분석
시퀀싱 접근법에서 생성된 대량의 데이터를 통계적으로 분석하는 것은 어려운 일이다.개인의 엑섬을 배열하는 것만으로 대량의 데이터 및 시퀀스 정보가 생성되므로 상당한 양의 데이터 분석이 필요하다.이 데이터의 분석과 관련된 과제로는 시퀀스 [10]읽기를 정렬하고 조립하는 데 사용되는 프로그램의 변경이 있습니다.다양한 시퀀싱 테크놀로지는 에러율도 다르고 다양한 읽기 길이를 생성하기 때문에 다른 시퀀싱 플랫폼의 결과를 비교하는 데 어려움이 있을 수 있습니다.
잘못된 양성 소견과 잘못된 음성 소견은 게놈 재시퀀싱 접근법과 관련이 있으며 중요한 문제입니다.다음과 같은 외부 데이터의 품질을 개선하기 위한 몇 가지 전략이 개발되었습니다.
- 배열과 배열 기반 유전자형식[1] 사이에서 식별된 유전자 변형 비교
- 코드화된 SNP를 전체 게놈 배열된 장애인과 비교[1]
- HapMap 개인의[1] Sanger 시퀀싱과 SNP 코딩 비교
드문 열성 질환은 dbSNP와 같은 공용 데이터베이스에 단일 뉴클레오티드 다형(SNP)이 없을 것이다.보다 일반적인 열성 표현형은 dbSNP에서 보고된 질병을 유발하는 변종을 가질 수 있다.예를 들어, 가장 일반적인 낭포성 섬유증 변종은 대부분의 모집단에서 약 3%의 대립 유전자 빈도를 가진다.그러한 변이를 걸러내는 것은 그러한 유전자를 고려 대상에서 잘못 제외할 수 있다.열성 질환의 유전자는 보통 우성 질환보다 식별하기가 더 쉽다. 왜냐하면 그 유전자들이 하나 이상의 희귀 익명이 아닌 [1]변종을 가질 가능성이 적기 때문이다.일반적인 유전자 변형을 선별하는 시스템은 대립 유전자의 변이에 대한 정확한 정보를 가지고 있지 않을 수 있는 dbSNP에 의존합니다.연구 엑솜 또는 게놈 전체 염기서열에서 공통되는 변이 목록을 사용하는 것이 더 신뢰할 수 있을 것이다.이 접근법의 과제는 배열된 엑솜의 수가 증가함에 따라 dbSNP도 비범한 변종 수가 증가할 것이라는 것이다.질병 표현형과 [12]관련이 없을 것 같은 일반적인 변형을 정의하기 위해 임계값을 개발할 필요가 있을 것이다.
유전자 이질성과 인구 민족성 또한 후보 유전자의 식별을 더욱 어렵게 만드는 잘못된 양성 및 잘못된 음성 발견의 수를 증가시킬 수 있기 때문에 주요한 한계이다.물론 이질성과 민족성이 존재하는 경우 문턱값의 엄격성을 줄일 수 있지만, 이는 변형을 탐지하는 힘도 감소시킬 것이다.후보 유전자를 식별하기 위해 유전자형 우선 접근법을 사용하는 것 또한 이러한 한계를 극복하기 위한 해결책을 제공할 수 있다.
윤리적 영향
유전체학의 새로운 기술은 연구자들이 기초 연구와 번역 연구에 접근하는 방식을 변화시켰습니다.exome sequencing과 같은 접근방식을 통해 방대한 양의 정보를 다루는 방법에 대한 일련의 의문을 제기하는 개별 게놈에서 생성된 데이터를 대폭 강화할 수 있다.이러한 연구의 개인은 시퀀스 정보에 액세스할 수 있어야 합니까?이 정보를 보험회사와 공유해야 합니까?이 데이터는 예상치 못한 발견으로 이어질 수 있으며 임상 효용과 환자 편익을 복잡하게 만들 수 있습니다.유전체학의 이 영역은 여전히 도전 과제이며, 연구자들은 이 [12]질문들을 어떻게 해결할지를 모색하고 있다.
Exome Sequencing 응용 프로그램
고정 비용 연구는 엑솜 염기서열 분석을 사용함으로써 전체 게놈 염기서열 분석에서 얻을 수 있는 것보다 훨씬 더 높은 깊이까지 샘플을 염기서열화할 수 있습니다.이 깊이를 더하면 신뢰할 수 있는 배리언트콜이 필요한 여러 응용 프로그램에 매우 적합합니다.ome 시퀀싱이 됩니다.
복잡한 장애에서 희귀한 변형 매핑
현재의 연관성 연구는 게놈 전체의 공통적인 변이에 초점을 맞추고 있다. 왜냐하면 이것들은 우리의 현재 분석에서 가장 쉽게 확인될 수 있기 때문이다.그러나 큰 효과를 보이는 질병을 일으키는 변종들은 후보 유전자 연구에서 엑솜 안에 있는 것으로 밝혀졌으며, 음성 선택으로 인해 훨씬 낮은 대립 유전자 빈도에서 발견되며, 현재의 표준 유전자형 분석에서 유형화되지 않은 채로 남아 있을 수 있다.전체 게놈 시퀀싱은 게놈 전체에 걸쳐 새로운 변종을 분석할 수 있는 잠재적인 방법입니다.하지만, 복잡한 장애에서는, 많은 유전자들이 질병 [14]위험과 관련이 있는 것으로 생각됩니다.이러한 근본적인 위험의 이질성은 유전자 발견을 위해 매우 큰 표본 크기가 요구된다는 것을 의미하며, 따라서 전체 게놈 배열은 특별히 비용 효율적이지 않다.이 샘플 크기 문제는 유전자 변이가 변이 [14]수준에서 드물지만 질병 유전자를 효과적으로 매핑하는 새로운 고급 분석 방법의 개발로 완화된다.또한 코딩 영역의 변형은 훨씬 더 광범위하게 연구되었으며, 그 기능적 함의를 도출하기가 훨씬 더 쉬우며, 따라서 대상 엑소메 영역 내의 변형에 대한 실용적인 적용에 더 즉시 접근할 수 있다.
희귀한 변종 유전자 발견에서의 엑솜 배열은 여전히 매우 활발하고 지속적인 연구 분야로 남아 있습니다: 지금까지 발견된 관련 유전자는 거의 없지만, 유전자 세트 전체에 걸쳐 상당한 위험 부담이 관찰된다는 증거가 증가하고 있습니다.
멘델리아 장애의 발견
이 섹션은 어떠한 출처도 인용하지 않습니다.(2016년 12월 (이 및 ) |
큰 효과의 멘델 장애에서, 지금까지의 연구 결과는 코딩 유전자 내의 하나 또는 극소수의 변종이 전체 상태의 기초가 된다는 것을 시사한다.이러한 장애의 심각성 때문에, 소수의 원인 변이는 모집단에서 극히 드물거나 새로운 것으로 추정되며, 표준 유전자형 분석에서는 누락될 수 있다.Exome 시퀀싱은 코딩 영역 전체에 걸쳐 높은 커버리지바리안트 콜을 제공합니다.이 콜은 노이즈에서 진정한 바리안트를 분리하기 위해 필요합니다.멘델 유전자 발견의 성공적인 모델은 부모와 프로반드가 유전자형인 트리오 시퀀싱을 사용하여 de novo 변형을 발견하는 것을 포함한다.
도입 사례
2009년 9월에 발표된 연구는 엑솜 염기서열 분석을 사용하여 인과적 유전자 변형을 식별할 수 있는지 여부를 결정하기 위한 개념 증명 실험에 대해 논의했다.Freeman과 함께 4명의 개인 시퀀스를 작성했습니다.셸던 증후군([1]FSS)(OMIM 193700)은 유전자 MYH3의 돌연변이에 의해 발생하는 것으로 알려진 희귀한 상염색체 우성 질환이다.또한 8명의 HapMap 개인은 금감원의 원인 유전자를 식별하기 위해 공통 변형을 제거하기 위해 염기서열을 분석하였고, 저자들은 공통 변형을 제외한 후 MYH3를 확인할 수 있었으며, 이는 엑솜 염기서열을 사용하여 희귀 [1]장애의 원인 변형을 식별할 수 있음을 확인하였다.이것은 희귀한 멘델 장애의 알려지지 않은 원인 유전자를 식별하기 위한 접근법으로 엑솜 염기서열 분석을 사용한 최초의 연구였다.
그 후, 다른 그룹은 터키계 [9]바터 증후군 의심환자에 대한 임상 진단에 성공했다고 보고했다.바터 증후군은 염분을 소모하는 신장 질환이다.엑솜 염기서열 분석 결과 선천성 염화물 설사(CLD)와 관련된 SLC26A3라고 불리는 유전자에서 예기치 않게 잘 보존된 열성 돌연변이가 나타났다.CLD의 분자 진단은 담당 임상의에 의해 확인되었다.이 예는 진단되지 않은 유전자 질환 환자를 평가할 때 임상 도구로서 전체 배열을 사용하는 개념의 증거를 제공했다.이 보고서는 환자의 분자 진단을 위한 차세대 염기서열 분석 기술의 첫 번째 응용으로 평가된다.
두 번째 보고서는 상염색체 열성 유전의 드문 질환인 밀러 증후군(MIM#263750)으로 알려진 멘델 증후군을 가진 개인의 엑솜 염기서열처리에 대해 수행되었다.밀러 증후군을 가진 두 형제자매와 두 명의 혈연관계가 없는 개인이 연구되었다.그들은 비동명 돌연변이, 스플라이스 수용체 및 기증자 부위, 짧은 코드 삽입 또는 [2]결실과 같은 병원성 가능성이 있는 변형을 조사했다.밀러 증후군은 드문 질환이기 때문에, 원인 변이가 아직 확인되지 않은 것으로 예상된다.공공 SNP 데이터베이스의 일반적인 단일 뉴클레오티드 다형(SNP)에 대한 이전의 엑솜 배열 연구는 후보 유전자를 추가로 제외하기 위해 사용되었다.이들 유전자를 제외한 후 저자들은 밀러증후군을 가진 사람들 사이에서 공유되는 DHDH의 돌연변이를 발견했다.밀러 증후군을 가진 각 개체는 DHDH 돌연변이에 대한 복합 헤테로 접합체였으며,[2] 해당 개체의 각 부모가 매개체로 확인됨에 따라 유전되었다.
이것은 희귀한 멘델병의 원인이 되는 새로운 유전자를 식별하는 엑솜 배열이 처음으로 나타난 것이다.이 흥미로운 발견은 엑솜 염기서열분열이 기존 방법의 한계로 인해 불가능했던 복잡한 질병에서 원인 유전자를 찾을 수 있는 잠재력을 가지고 있다는 것을 보여준다.표적 포획과 대규모 병렬 시퀀싱은 개별 인간 게놈에서 단백질 코드 변화를 일으키는 변형을 검출하기 위한 높은 민감도와 특수성을 가진 비용 효율적이고 재현 가능하며 강력한 전략을 나타낸다.
임상 진단
엑솜 시퀀싱은 환자의 질병의 유전적 원인을 진단하는 데 사용될 수 있다.기초 질환 유전자 돌연변이의 식별은 진단 및 치료 접근법에 주요 영향을 미칠 수 있으며, 질병 자연사의 예측을 안내할 수 있으며, 위험에 처한 가족 [1][2][9][15][16][17]구성원을 검사할 수 있다.단일 유전자 분석보다 엑솜 염기서열 분석을 우수하게 만드는 요인은 여러 가지가 있는데, 여기에는 비정형 임상[17] 증상으로 인해 테스트되지 않은 유전자의 돌연변이를 식별하는 능력이나 다른 유전자의 돌연변이가 같은 환자의 [2]다른 표현형에 기여하는 임상 사례를 식별하는 능력 등이 포함된다.
질병의 유전적 원인을 진단한 후, 이 정보는 적절한 치료법의 선택을 안내할 수 있습니다.이 전략이 클리닉에서 처음으로 성공적으로 수행된 것은 염증성 장질환이 있는 [16][18]유아의 치료였다.이전에 여러 가지 기존 진단법을 사용했지만 결과는 유아의 증상을 설명할 수 없었다.엑솜 배열 데이터의 분석에서 XIAP 유전자의 돌연변이가 확인되었다.이 유전자의 기능에 대한 지식은 아기의 치료를 안내했고,[16] 아이의 병을 고치는 골수 이식을 이끌었다.
연구자들은 바터 증후군과 선천성 염화물 [9]설사를 가진 환자의 근본적인 돌연변이를 확인하기 위해 엑솜 시퀀싱을 사용해 왔다.Bilgular의 그룹은 또한 엑솜 염기서열 분석을 사용하고 심각한 뇌 기형을 가진 환자의 근본적인 돌연변이를 확인하면서, "이 발견들은" 전통적인 방법들이 어려운 환경에서 질병 위치를 식별하기 위해 전체 엑솜 염기서열 분석을 사용하는 것을 강조한다...우리의 결과는 이 기술이 진단 분류의 경계에 대한 궤적 이질성과 불확실성으로 인해 매핑이 혼란스러워진 상황에서 유전자 발견에 특히 유용할 것이라는 것을 보여주며, 의학에 대한 광범위한 적용에 대한 밝은 미래를 가리키고 있다."[15]
남아프리카 케이프타운 대학의 연구원들은 심장병과 심장 마비의 위험을 높이는 부정맥성 우심실 심근증으로 알려진 유전 질환의 근본적인 원인으로 CDH2의 유전자 변이를 발견하기 위해 엑솜 염기서열 분석을 사용했다.[1]
상업적 비용
여러 회사가 소비자에게 exome sequencing을 제공했습니다.Knome은 수천 [19]달러의 비용을 들여 소비자에게[when?] 엑섬 시퀀싱 서비스를 제공한 최초의 회사였다.그 후 23andMe는 2011년 9월에 발표된 파일럿 WES 프로그램을 2012년에 중단했습니다.소비자들은 999달러의 비용으로 외부 데이터를 얻을 수 있었다.회사는 원시 데이터를 제공했고 분석을 [19][20][21]제공하지 않았습니다.
2012년 11월, Gene by Gene의 사업부인 DNADTC는 80배 커버리지와 [22]695달러의 초기 가격으로 엑솜을 제공하기 시작했다.DNADTC 웹사이트당 가격은 현재 895달러입니다.2013년 10월, BGI는 50X 커버리지에서 [23]499달러에 퍼스널 엑섬 시퀀싱 프로모션을 발표했습니다.2016년 6월 Genos는 CLIA 인증을 받은 75배 소비자용 엑솜을 [24][25][26]타액으로 배열하여 399달러의 더 낮은 가격을 달성할 수 있었습니다.
36개 연구에 대한 2018년 리뷰에서 Exome Sequencing 비용은 555달러에서 5,169달러이며, 진단 수율은 환자 [13]그룹에 따라 3%에서 79%에 이르는 것으로 나타났다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c d e f g h i j Ng SB, Turner EH, Robertson PD, Flygare SD, Bigham AW, Lee C, Shaffer T, Wong M, Bhattacharjee A, Eichler EE, Bamshad M, Nickerson DA, Shendure J (10 September 2009). "Targeted capture and massively parallel sequencing of 12 human exomes". Nature. 461 (7261): 272–276. Bibcode:2009Natur.461..272N. doi:10.1038/nature08250. PMC 2844771. PMID 19684571.
- ^ a b c d e Sarah B Ng; Kati J Buckingham; Choli Lee; Abigail W Bigham; Holly K Tabor; Karin M Dent; Chad D Huff; Paul T Shannon; Ethylin Wang Jabs; Deborah A Nickerson; Jay Shendure; Michael J Bamshad (2010). "Exome sequencing identifies the cause of a mendelian disorder". Nature Genetics. 42 (1): 30–35. doi:10.1038/ng.499. PMC 2847889. PMID 19915526.
- ^ Wang, D. G.; Fan, J. B.; Siao, C. J.; Berno, A.; Young, P.; Sapolsky, R.; Ghandour, G.; Perkins, N.; Winchester, E. (1998-05-15). "Large-scale identification, mapping, and genotyping of single-nucleotide polymorphisms in the human genome". Science. 280 (5366): 1077–1082. Bibcode:1998Sci...280.1077W. doi:10.1126/science.280.5366.1077. ISSN 0036-8075. PMID 9582121.
- ^ Rauch, A; Hoyer, J; Guth, S; Zweier, C; Kraus, C; Becker, C; Zenker, M; Hüffmeier, U; Thiel, C; Rüschendorf, F; Nürnberg, P; Reis, A; Trautmann, U (Oct 1, 2006). "Diagnostic yield of various genetic approaches in patients with unexplained developmental delay or mental retardation". American Journal of Medical Genetics Part A. 140 (19): 2063–74. doi:10.1002/ajmg.a.31416. PMID 16917849. S2CID 24570999.
- ^ a b Stavros Basiardes; Rose Veile; Cindy Helms; Elaine R. Mardis; Anne M. Bowcock; Michael Lovett (2005). "Direct Genomic Selection". Nature Methods. 1 (2): 63–69. doi:10.1038/nmeth0105-63. PMID 16152676. S2CID 667227.
- ^ Teer, J. K.; Mullikin, J. C. (12 August 2010). "Exome sequencing: the sweet spot before whole genomes". Human Molecular Genetics. 19 (R2): R145–R151. doi:10.1093/hmg/ddq333. PMC 2953745. PMID 20705737.
- ^ Emily H. Turner; Sarah B. Ng; Deborah A. Nickerson; Jay Shendure (2009). "Methods for Genomic Partitioning". Annu Rev Genom Hum Genet. 10 (1): 30–35. doi:10.1146/annurev-genom-082908-150112. PMID 19630561.
- ^ Mertes F, Elsharawy A, Sauer S, van Helvoort JM, van der Zaag PJ, Franke A, Nilsson M, Lehrach H, Brookes AJ (2011). "Targeted enrichment of genomic DNA regions for next-generation sequencing" (PDF). Brief. Funct. Genomics. 10 (6): 374–386. doi:10.1093/bfgp/elr033. PMC 3245553. PMID 22121152.
- ^ a b c d e Choi M, Scholl UI, Ji W, Liu T, Tikhonova IR, Zumbo P, Nayir A, Bakkaloğlu A, Ozen S, Sanjad S, Nelson-Williams C, Farhi A, Mane S, Lifton RP (10 November 2009). "Genetic diagnosis by whole exome capture and massively parallel DNA sequencing". Proc Natl Acad Sci U S A. 106 (45): 19096–19101. Bibcode:2009PNAS..10619096C. doi:10.1073/pnas.0910672106. PMC 2768590. PMID 19861545.
- ^ a b c d Kahvejian A, Quackenbush J, Thompson JF (2008). "What would you do if you could sequence everything?". Nature Biotechnology. 26 (10): 1125–1133. doi:10.1038/nbt1494. PMC 4153598. PMID 18846086.
- ^ Lira Mamanova; Coffey, Alison J; Scott, Carol E; Kozarewa, Iwanka; Turner, Emily H; Kumar, Akash; Howard, Eleanor; Shendure, Jay; Turner, Daniel J; et al. (February 2010). "Target-enrichment strategies for nextgeneration sequencing". Nature Methods. 7 (2): 111–118. doi:10.1038/nmeth.1419. PMID 20111037. S2CID 21410733.
- ^ a b c Biesecker LG (Jan 2010). "Exome sequencing makes medical genomics a reality". Nat. Genet. 42 (1): 13–14. doi:10.1038/ng0110-13. PMID 20037612.
- ^ a b Schwarze, K; Buchanan, J; Taylor, JC; Wordsworth, S (May 2018). "Are whole Exome and whole Genome Sequencing Approaches Cost-Effective? A Systematic Review of the Literature". Value in Health. 21: S100. doi:10.1016/j.jval.2018.04.677. ISSN 1098-3015.
- ^ a b Weijun Luo; Chaolin Zhang; Yong-hui Jiang; Cory R. Brouwer (2018). "Systematic reconstruction of autism biology from massive genetic mutation profiles". Science Advances. 4 (4): e1701799. Bibcode:2018SciA....4.1799L. doi:10.1126/sciadv.1701799. PMC 5895441. PMID 29651456.
- ^ a b Bilgüvar K, Oztürk AK, Louvi A, Kwan KY, Choi M, Tatli B, Yalnizoğlu D, Tüysüz B, Cağlayan AO, Gökben S, Kaymakçalan H, Barak T, Bakircioğlu M, Yasuno K, Ho W, Sanders S, Zhu Y, Yilmaz S, Dinçer A, Johnson MH, Bronen RA, Koçer N, Per H, Mane S, Pamir MN, Yalçinkaya C, Kumandaş S, Topçu M, Ozmen M, Sestan N, Lifton RP, State MW, Günel M (9 September 2010). "Whole-exome sequencing identifies recessive WDR62 mutations in severe brain malformations". Nature. 467 (7312): 207–210. Bibcode:2010Natur.467..207B. doi:10.1038/nature09327. PMC 3129007. PMID 20729831.
{{cite journal}}: CS1 maint: 작성자 파라미터 사용(링크) - ^ a b c Worthey EA, Mayer AN, Syverson GD, Helbling D, Bonacci BB, Decker B, Serpe JM, Dasu T, Tschannen MR, Veith RL, Basehore MJ, Broeckel U, Tomita-Mitchell A, Arca MJ, Casper JT, Margolis DA, Bick DP, Hessner MJ, Routes JM, Verbsky JW, Jacob HJ, Dimmock DP (Mar 2011). "Making a definitive diagnosis: successful clinical application of whole exome sequencing in a child with intractable inflammatory bowel disease". Genet. Med. 13 (3): 255–262. doi:10.1097/GIM.0b013e3182088158. PMID 21173700.
- ^ a b Raffan E, Hurst LA, Turki SA, Carpenter G, Scott C, Daly A, Coffey A, Bhaskar S, Howard E, Khan N, Kingston H, Palotie A, Savage DB, O'Driscoll M, Smith C, O'Rahilly S, Barroso I, Semple RK (2011). "Early Diagnosis of Werner's Syndrome Using Exome-Wide Sequencing in a Single, Atypical Patient". Front Endocrinol (Lausanne). 2 (8): 8. doi:10.3389/fendo.2011.00008. PMC 3356119. PMID 22654791.
- ^ Warr, A.; Robert, C.; Hume, D.; Archibald, A.; Deeb, N.; Watson, M. (2 July 2015). "Exome Sequencing: Current and Future Perspectives". G3. 5 (8): 1543–1550. doi:10.1534/g3.115.018564. PMC 4528311. PMID 26139844.
- ^ a b Herper, Matthew (27 September 2011). "The Future Is Now: 23andMe Now Offers All Your Genes For $999". Forbes. Retrieved 11 December 2011.
- ^ "23andMe Launches Pilot Program for Direct-to-Consumer Exome Sequencing". GenomeWeb. GenomeWeb LLC. 28 September 2011. Retrieved 11 December 2011.
- ^ "Personal Genome Sequencing in Ostensibly Healthy Individuals and the PeopleSeq Consortium" (PDF). Genomes2People. 14 June 2016.
- ^ Vorhaus, Dan (29 November 2012). "DNA DTC: The Return of Direct to Consumer Whole Genome Sequencing". Genomics Law Report. Retrieved 30 May 2013.
- ^ "Ultimate Exome Promotion". BGI Americas. 18 October 2013. Archived from the original on 10 November 2013. Retrieved 17 November 2013.
- ^ "Owning Your Data: The Genos Model". Bio-IT World. 6 July 2016.
- ^ "Consumer Genomics Startup Genos Research Plans to Let Customers Explore, Share Their Data". GenomeWeb LLC. 13 June 2016.
- ^ "Genos - Own your DNA, Learn about Yourself, Drive Research". www.genosresearch.com. Retrieved 2016-10-18.