Enhancer-FACS-seq
Enhancer-FACS-seqEnhancer-FACS-seq(eFS)[1][2]는 브리검 여성병원 및 하버드 [4]의과대학 Bulyk[3] 연구소에서 개발한 매우 병렬적인 인핸서 분석으로 전체 Drosophila melanogaster 배아의 맥락에서 활성, 조직 특이적 전사 인핸서의 식별을 목표로 합니다.이 기술은 조직 특이적 인핸서를 선별하기 위한 현미경 검사를 전체 배아에서 분리된 세포의 형광 활성 세포 분류(FACS)와 높은 처리량 Illumina 시퀀싱에 의한 식별으로 대체한다.
서론
전사 규제
메타조아에서, 환경 스트레스에 반응하고, 적절하게 분화하며, 세포 순환을 통해 정상적으로 진행하기 위해, 진핵 세포는 수천 개의 [5]유전자의 고도로 조절된 전사를 포함하는 특이하고 조정된 유전자 발현 프로그램을 필요로 합니다.이 유전자 조절은 대부분 조직 고유의 방식으로 CRM([6]cis-regulatory module)이라고 불리는 비코드 게놈 영역에 전사 인자가 결합함으로써 제어된다.염색질의 구조를 조절함으로써 유전자 발현을 활성화하거나 억제하고, 따라서 전사 조절에 긍정적이거나 부정적인 영향을 미치는 것.유전자 발현을 활성화하는 CRM은 종종 전사 증강제라고 불리는 반면, 유전자 발현을 억제하는 CRM은 전사 소음제라고 불린다.
드로소필라 멜라노가스터의 인핸서 검출
생물학에 대한 강력한 모델 유기체와 전사 증강제 연구에도 불구하고, 드로소필라 멜라노가스터에서 추정된 50,000개의 전사 증강제 중 5% 미만의 조직 특이 활성이 발견되었습니다.[7][8]지난 10년 동안, 드로소필라 멜라노가스터에서 조직 또는 세포형 특이 활성을 검출하기 위한 주요 방법은 처리량이 낮고 비용이 많이 드는 전통적인 리포터 분석을 [9][10]통해 후보 강화제를 테스트하는 것이었다.지난 몇 년 동안, 인핸서 발견이 개선되고 다른 병렬 리포터 분석이 [11][12][13][14][15][16]개발되었지만, 지금까지 어느 것도 전체 배아의 세포 유형 관심의 게놈 맥락에서 인핸서 활동을 직접적으로 식별할 수 있도록 허락하지 않았다.
방법론
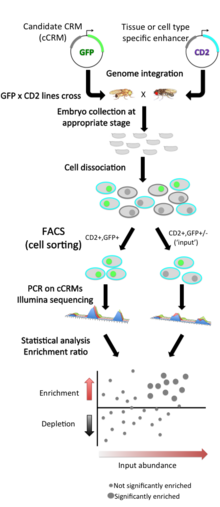
각 후보 CRM(cCRM)은 리포터 유전자의 업스트림에 복제된다.기존의 리포터 분석과 비교하여, 주요 혁신은 조직 특이적 강화제를 선별하기 위해 현미경 검사 대신 분리된 세포의 형광 활성 세포 분류(FACS)를 사용하는 것이다.이 접근방식은 2마커 시스템을 이용한다.각 배아에서 하나의 마커(여기서는 쥐 CD2 세포 표면 단백질)는 FACS에 의해 분류되는 특정 조직의 세포를 라벨링하기 위해 사용되며, 다른 마커(여기서는 녹색 형광 단백질 GFP)는 CRM 활동의 리포터로 사용된다.
세포는 조직형에 따라 분류된 후 GFP 형광에 의해 분류되며, 이중양성으로 분류된 세포 및 전체 입력 세포에서 PCR에 의해 cCRM을 회수한다.두 모집단의 높은 처리량 시퀀싱을 통해 입력 및 분류된 모집단에서 각 cCRM의 상대적 풍요를 측정할 수 있다. 그런 다음 테스트 중인 CD2 양성 세포 유형에서 입력과 비교하여 이중 양성 세포에서 각 cCRM의 농축 또는 고갈을 평가할 수 있다.
중요한 결과
이 [17]방법에 대한 최초 보고서에서는 다양한 게놈 데이터 소스(예: TF 결합 영역, 공동활성제 결합 영역, DNase I 과민성 부위, Bulyk lab의 PhylCRM 알고리즘)에서 약 500개의 cCRM 라이브러리를 게놈 DNA에서 PCR로 추출했다.그리고 나서 배아 중배엽과 특정 중배엽 세포 유형에서 활성을 검사했다.결과는 eFS에 의해 테스트된 68개의 cCRM에 대해 드로소필라 멜라노가스터 배아에서 전통적인 리포터 유전자 분석에 의해 검증되었다.유의하게 농축된 cCRM에서 eFS의 특이성은 우수했으며, CD2 양성 세포 대부분이 GFP를 발현하는 민감도는 양호했다.중배엽에서 활성화된 것으로 확인된 강화제 중에서 알려진 강화제 관련 크로마틴 마크 H3K27ac, H3K4me1, Pol II가 유의하게 농축된 것으로 확인되었다.
장점과 미래의 응용 프로그램
eFS의 장점
- 전체 배아의 활성, 조직 특이적 전사 증강제의 고도로 병렬 식별
- 후보자는 게놈 맥락에서 분석된 활동을 증강시킨다.
- 검출된 인핸서의 높은 특이성
장래의 응용 프로그램
eFS 검사를 사용하여 다른 세포 또는 조직 유형을 분석할 수 있습니다.GFP 발현 CD2-음성 세포와 CD2-양성 세포의 농도를 평가하고, 리포터 형질전환 수컷 파리의 공통 풀을 다른 세포 타입의 CD2 발현 암컷과 교차시킴으로써 특이성과 활성을 측정할 수 있다.드로소필라에서 규제 게놈의 주석을 가속화하면 원칙적으로 전사 규제의 네트워크 특성을 탐색할 수 있는 대규모 규제 상호작용 데이터가 생성되어야 한다.
레퍼런스
- ^ "Highly parallel enhancer assays". Archived from the original on 2014-12-16. Retrieved 2014-12-15.
- ^ Gisselbrecht SS; et al. (2013). "Highly parallel assays of tissue-specific enhancers in whole Drosophila embryos" (PDF). Nat Methods. 10 (8): 774–80. doi:10.1038/nmeth.2558. PMC 3733245. PMID 23852450.
- ^ "Bulyk Lab". Archived from the original on 2014-12-16. Retrieved 2014-12-15.
- ^ "Bulyk Lab". Archived from the original on 2014-12-16. Retrieved 2014-12-15.
- ^ Lockhart DJ and Winzeler EA (2000). "Genomics, gene expression and DNA arrays". Nature. 405 (6788): 827–36. doi:10.1038/35015701. PMID 10866209. S2CID 2242529.
- ^ Bulyk ML (2003). "Computational prediction of transcription-factor binding site locations". Genome Biol. 5 (1): 201. doi:10.1186/gb-2003-5-1-201. PMC 395725. PMID 14709165.
- ^ Pfeiffer BD; et al. (2008). "Tools for neuroanatomy and neurogenetics in Drosophila". Proc Natl Acad Sci U S A. 105 (28): 9715–20. Bibcode:2008PNAS..105.9715P. doi:10.1073/pnas.0803697105. PMC 2447866. PMID 18621688.
- ^ Halfon MS; et al. (2007). "REDfly 2.0: an integrated database of cis-regulatory modules and transcription factor binding sites in Drosophila". Nucleic Acids Res. 36 (Database issue): D594-8. doi:10.1093/nar/gkm876. PMC 2238825. PMID 18039705.
- ^ Sandmann T; et al. (2007). "A core transcriptional network for early mesoderm development in Drosophila melanogaster". Genes Dev. 21 (4): 436–49. doi:10.1101/gad.1509007. PMC 1804332. PMID 17322403.
- ^ Zinzen RP; et al. (2009). "Combinatorial binding predicts spatio-temporal cis-regulatory activity". Nature. 462 (7269): 65–70. Bibcode:2009Natur.462...65Z. doi:10.1038/nature08531. PMID 19890324. S2CID 4320071.
- ^ Gertz J; et al. (2009). "Analysis of combinatorial cis-regulation in synthetic and genomic promoters". Nature. 457 (7226): 215–8. Bibcode:2009Natur.457..215G. doi:10.1038/nature07521. PMC 2677908. PMID 19029883.
- ^ Sharon E; et al. (2012). "Inferring gene regulatory logic from high-throughput measurements of thousands of systematically designed promoters". Nat Biotechnol. 30 (6): 521–30. doi:10.1038/nbt.2205. PMC 3374032. PMID 22609971.
- ^ Nam J; et al. (2010). "Functional cis-regulatory genomics for systems biology". Proc. Natl. Acad. Sci. U.S.A. 107 (8): 3930–5. Bibcode:2010PNAS..107.3930N. doi:10.1073/pnas.1000147107. PMC 2840491. PMID 20142491.
- ^ Melnikov A; et al. (2012). "Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay". Nat Biotechnol. 30 (3): 271–7. doi:10.1038/nbt.2137. PMC 3297981. PMID 22371084.
- ^ Patwardhan RP; et al. (2012). "Massively parallel functional dissection of mammalian enhancers in vivo". Nat Biotechnol. 30 (3): 265–70. doi:10.1038/nbt.2136. PMC 3402344. PMID 22371081.
- ^ Arnold CD; et al. (2014). "Quantitative genome-wide enhancer activity maps for five Drosophila species show functional enhancer conservation and turnover during cis-regulatory evolution". Nat Genet. 46 (7): 685–92. doi:10.1038/ng.3009. PMC 4250274. PMID 24908250.
- ^ Gisselbrecht SS; et al. (2013). "Highly parallel assays of tissue-specific enhancers in whole Drosophila embryos" (PDF). Nat Methods. 10 (8): 774–80. doi:10.1038/nmeth.2558. PMC 3733245. PMID 23852450.
- ^ "PhylCRM software package homepage". Archived from the original on 2016-03-04. Retrieved 2014-12-15.