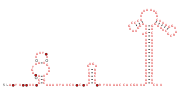
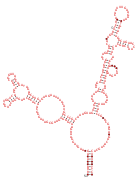
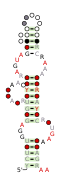
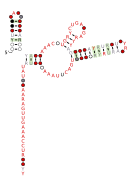
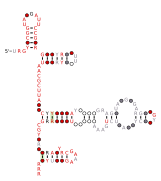
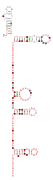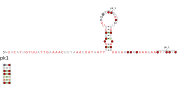RAGATH RNA 모티브
RAGATH RNA motifs| RAGATH-8 | |
|---|---|
 | |
| 식별자 | |
| 기호. | RAGATH-8 |
| Rfam | RF02687 |
| 기타 데이터 | |
| RNA형 | 진; sRNA |
| 가세요 | GO:0003824 |
| 그렇게 | SO:0000370 |
| PDB 구조 | PDBe |
트위스터와 해머헤드 리보자임(RAGATH)과 관련된 유전자와 관련된 RNA는 [1]박테리아에서 자가 소거 리보자임을 찾기 위해 고안된 생체 정보학 전략을 말한다.또한 이 전략을 사용하여 발견된 후보 RNA 또는 RAGATH RNA 모티브를 참조합니다.
트위스터 [2]리보자임의 발견으로, 박테리아의 많은 유전적 요소들이 리보자임을 트위스터 리보자임과 이전에 발견된 해머헤드 리보자임 [2]근처에 종종 위치한다는 것이 인식되었다.이러한 유전자 요소에는 여러 가지 유전자 클래스가 포함되어 있으며, 이들 중 다수는 뮤 유사 화지의 특징입니다.주변 요소에는 트위스터와 해머헤드 리보자임도 포함됩니다.즉, 트위스터 및 해머헤드 리보자임은 종종 다른 트위스터 또는 해머헤드 리보자임 근처의 박테리아에 위치합니다.
이러한 관찰을 감안할 때, 연구자들은 다른 종류의 자기 결절 리보자임도 이러한 유전적 요소와 연관될 것이라고 가설을 세웠다.따라서, 기존에 확립된 [3]방법을 사용하여 보존된 RNA 구조를 찾기 위해 관련 유전자 요소 근처의 비부호화 영역에 대한 검색을 수행하였다.그러한 RNA 구조는 자가 분해 리보자임으로 후보될 수 있다.
이 방법을 사용하여 이전에는 알려지지 않았던 자가 절단 리보자임 클래스가 발견되었다: 트위스터 자매, 권총 및 도끼 리보자임.해머헤드와 HDV 리보자임의 특이한 예도 발견되었다.추가로 보존된 12개의 RNA 구조는 리보자임으로 기능하지 않는 것으로 보이며, 이러한 RNA의 생물학적 및 생화학적인 기능은 알려지지 않았다.보존된 모든 RNA는 "RAGATH RNA 모티브"로 명명되었고, 미해결 RNA는 RAGATH-4에서 RAGATH-15까지 번호가 매겨졌다.체외에서 자가 삭제되지 않은 추가적인 RAGATH 모티브도 나중에 발표되었다.[4]
RAGATH-18 RNA는 예측된 [5]꼬임회전을 포함한다.이 킨크 턴의 특정 예는 킨크 턴 구조가 그 시퀀스와 어떻게 관련되어 있는지를 더 잘 이해하기 위해 연구되었다.
레퍼런스
- ^ Weinberg Z, Kim PB, Chen TH, Li S, Harris KA, Lünse CE, Breaker RR (2015). "New classes of self-cleaving ribozymes revealed by comparative genomics analysis". Nat. Chem. Biol. 11 (8): 606–10. doi:10.1038/nchembio.1846. PMC 4509812. PMID 26167874.
- ^ a b Roth A, Weinberg Z, Chen AG, Kim PB, Ames TD, Breaker RR (2014). "A widespread self-cleaving ribozyme class is revealed by bioinformatics". Nat. Chem. Biol. 10 (1): 56–60. doi:10.1038/nchembio.1386. PMC 3867598. PMID 24240507.
- ^ Weinberg Z, Wang JX, Bogue J, Yang J, Corbino K, Moy RH, Breaker RR (2010). "Comparative genomics reveals 104 candidate structured RNAs from bacteria, archaea, and their metagenomes". Genome Biol. 11 (3): R31. doi:10.1186/gb-2010-11-3-r31. PMC 2864571. PMID 20230605.
- ^ Weinberg Z, Lünse CE, Corbino KA, Ames TD, Nelson JW, Roth A, Perkins KR, Sherlock ME, Breaker RR (October 2017). "Detection of 224 candidate structured RNAs by comparative analysis of specific subsets of intergenic regions". Nucleic Acids Res. 45 (18): 10811–10823. doi:10.1093/nar/gkx699. PMC 5737381. PMID 28977401.
- ^ Huang L, Liao X, Li M, Wang J, Peng X, Wilson TJ, Lilley DM (June 2021). "Structure and folding of four putative kink turns identified in structured RNA species in a test of structural prediction rules". Nucleic Acids Res. 49 (10): 5916–5924. doi:10.1093/nar/gkab333. PMC 8191799. PMID 33978763.