피크
PEAKS| 개발자 | 바이오 인포매틱스 솔루션 주식회사 |
|---|---|
| 안정된 릴리스 | PIKS 8.5 / 2017년 10월 12일 |
| 운영 체제 | 창문들 |
| 유형 | 대량사양 단백질 식별, De Novo Sequencing, 데이터베이스 검색 및 정량화 |
| 면허증. | 독자 사양 상용 소프트웨어 |
| 웹 사이트 | http://www.bioinfor.com/ |
PIKS는 펩타이드 배열 분석, 단백질 식별 및 정량화를 위해 설계된 탠덤 질량 분석용 단백질학 소프트웨어 프로그램입니다.
묘사
PIKS는 일반적으로 de novo 펩타이드 시퀀싱 보조 검색 엔진 [1]데이터베이스 검색을 통해 펩타이드 식별(단백질 ID)에 사용됩니다.PIKS는 또한 자동 펩타이드 배열 태그 기반 검색(SPIDER)[2]과 PTM [3]식별을 통해 PTM과 돌연변이 특성을 통합했습니다.
PIKS는 각 펩타이드에 대한 완전한 배열, 개별 아미노산 할당에 대한 신뢰도 점수, 높은 처리량 분석을 위한 간단한 보고 등을 제공한다.
이 소프트웨어는 여러 검색 엔진의 결과를 비교할 수 있습니다.PIKS inChorus는 Sequest, OMSSA, X!Tandem, 마스코트와 같은 다른 단백질 ID 검색 엔진과 자동으로 교차 검사합니다.이 접근법은 잘못된 양성 펩타이드 할당을 방지합니다.
PIKS Q는 단백질 정량화, 지원 라벨(ICAT, iTRAQ, SILAC, TMT, 018 등) 및 라벨 프리 기술을 위한 추가 도구입니다.
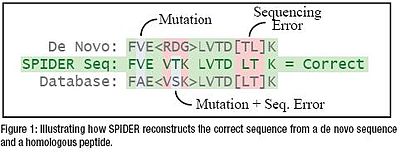
SPIDER는 PIKS 내의 시퀀스 태그 기반 검색 툴로, de novo 시퀀스 오류와 호몰로지 돌연변이 사이의 오버랩 가능성을 처리합니다.de novo 배열 태그와 호몰로그를 모두 자동적이고 [2]효율적으로 결합하여 실제 펩타이드 배열을 재구성합니다.
PIKS 소프트웨어에서 사용되는 알고리즘 모음이 자동 모노클로널 항체 [4]시퀀싱의 첫 번째 방법임이 입증된 전문 프로젝트인 PIKS AB로 조정 및 구성되었습니다.
메모들
- ^ Zhang, J.; Xin, L.; Shan, B.; Chen, W.; Xie, M.; Yuen, D.; Zhang, W.; Zhang, Z.; Lajoie, G.; Ma, B. (2011). "PEAKS DB: De Novo Sequencing Assisted Database Search for Sensitive and Accurate Peptide Identification". Molecular & Cellular Proteomics. 11 (4): M111.010587. doi:10.1074/mcp.M111.010587. PMC 3322562. PMID 22186715.
- ^ a b 마비, 존슨.De Novo 배열 및 호몰로지 검색 분자 및 세포 프로테오믹스. 10.1074/mcp.O111.014902 (2011)
- ^ Han, X.; He, L.; Xin, L.; Shan, B.; Ma, B. (2011). "PEAKS PTM: Mass Spectrometry Based Identification of Peptides with Unspecified Modifications". Journal of Proteome Research. 10 (7): 2930–2936. doi:10.1021/pr200153k. PMID 21609001.
- ^ Tran, Ngoc Hieu; Rahman, M. Ziaur; He, Lin; Xin, Lei; Shan, Baozhen; Li, Ming (26 August 2016). "Complete De Novo Assembly of Monoclonal Antibody Sequences". Scientific Reports. 6: 31730. doi:10.1038/srep31730. ISSN 2045-2322. PMC 4999880. PMID 27562653.