살모넬라
Salmonella| 살모넬라 | |
|---|---|
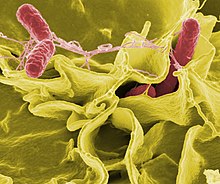 | |
| 살모넬라 티피무륨(붉은 색)이 배양된 인간 세포를 침범하는 것을 보여주는 컬러 강화 주사 전자 현미경 사진 | |
| 과학적 분류 | |
| 도메인: | 박테리아 |
| 문: | 슈도모나도타 |
| 클래스: | 감마프로테오박테리아 |
| 순서: | 세균 입력 |
| 패밀리: | 엔테로박테리아과 |
| 속: | 살모넬라 리그니에르, 1900 |
| 종 및 아종[1] | |
| |
살모넬라균(Salmonella)은 장대균과에 속하는 그람 음성 박테리아의 한 속입니다.살모넬라의 알려진 두 종은 살모넬라 장생균과 살모넬라 봉고리입니다.장티푸스는 2,600개 이상의 [4]혈청형을 포함하는 6개의[2][3] 아종으로 더 나뉩니다.살모넬라는 미국의 수의사 다니엘 엘머 살몬 (1850–1914)의 이름을 따서 지어졌습니다.
살모넬라 종들은 포자를 형성하지 않고 주로 운동성 장세균으로, 세포 직경이 약 0.7~1.5 μm, 길이가 2~5 μm,[5] 그리고 세포 본체 주변에 있는 주변 편모세포를 가지고 있습니다.그것들은 화학영양생물로, 유기적인 공급원을 사용하여 산화와 환원 반응으로부터 에너지를 얻습니다.또한 산소가 있을 때 산소와 함께 아데노신 삼인산을 생성하거나 산소가 [5]없을 때 다른 전자 수용체 또는 발효를 사용할 수 있는 혐기성 동물입니다.
살모넬라종은 세포 내 [6]병원체이며, 그 중 특정 혈청형은 질병을 유발합니다.대부분의 감염은 대변에 오염된 음식의 섭취로 인한 것입니다.장티푸스 살모넬라 혈청형은 사람들 사이에서만 옮겨질 수 있고 장티푸스와 부종성 장티푸스뿐만 아니라 음식물 매개 질병을 일으킬 수 있습니다.장티푸스는 장티푸스 살모넬라균이 혈류를 침범하는 것은 물론 온몸으로 퍼져 장기에 침입해 내독소(중풍 형태)를 분비함으로써 발생합니다.이는 생명을 위협하는 저혈량 쇼크와 패혈성 쇼크로 이어질 수 있으며 항생제를 포함한 집중 치료가 필요합니다.
비형성 살모넬라 혈청형은 동물성이며 동물로부터 그리고 사람들 사이에서 옮겨질 수 있습니다.그들은 보통 위장관에만 침입하여 항생제 없이도 증상을 해결할 수 있는 살모넬라증을 일으킵니다.그러나 사하라 이남 아프리카에서는 비형성 살모넬라균이 침습적일 수 있고 부형성 발열을 일으킬 수 있어 즉각적인 항생제 [7]치료가 필요합니다.
분류학
살모넬라속은 엔테로박테리아과의 일부입니다.그것의 분류법은 수정되었고 혼동할 수 있는 가능성이 있습니다.이 속은 두 종으로 구성되어 있는데, 두 종 중 후자는 6종의 아종으로 나뉘어져 있습니다: 장티카속, 장티카속, 살라미속, 아리조나속, 장티카속, 장티카속,[8][9] 장티카속.분류군에는 체세포 O(지질다당류)와 편모 H 항원(코프만-)을 기반으로 정의된 2500개 이상의 혈청형(세로바)이 포함되어 있습니다.흰색 분류).혈청형의 전체 이름은 예를 들어 살모넬라 장티카 서브스프로 지정됩니다.장형 Tifimurium, 그러나 Salmonella Tifimurium으로 축약될 수 있습니다.임상 및 역학 조사를 지원하기 위한 균주의 추가 분화는 항생제 민감도 테스트 및 펄스 필드 젤 전기영동, 다초점 시퀀스 타이핑 및 점점 더 전체 게놈 시퀀싱과 같은 다른 분자 생물학 기술에 의해 달성될 수 있습니다.역사적으로 살모넬라균은 임상적으로 숙주 선호도와 인간의 [10]질병 징후에 따라 침습성(형질) 또는 비침습성(비형질 살모넬라균)으로 분류되었습니다.
역사
살모넬라균은 1880년 칼 에버스에 의해 장티푸스 [11]환자들의 패치와 비장에서 처음으로 시각화되었습니다.4년 후, Georg Theodor Gaffky는 순수한 [12]배양에서 병원체를 배양할 수 있었습니다.그로부터 1년 후, 의학 연구자인 Theobald Smith는 나중에 살모넬라 장티카로 알려지게 될 것을 발견했습니다.콜레라).그 당시, 스미스는 미국 농무부 수의과에서 연구실 조수로 일하고 있었습니다.그 부서는 동물 병리학자인 [13]Daniel Elmer Salmon의 관리하에 있었습니다.처음에, 살모넬라 콜레라는 돼지 콜레라의 원인 물질로 여겨졌기 때문에, 살모넬라 콜레라와 스미스는 그것을 "호그 콜레라 바실러스"라고 이름 지었습니다.살모넬라라는 이름은 1900년 조셉 레온 리그니에르가 살모넬라의 이름을 따서 [14]: 16 살모넬라라고 부르자고 제안했을 때까지 사용되지 않았습니다.
1930년대 후반, 오스트레일리아의 세균학자 낸시 앳킨슨은 애들레이드에 있는 사우스 오스트레일리아의 병리학 및 세균학 정부 연구소(나중에 의학 및 수의학 연구소)에 살모넬라 타이핑 연구소를 설립했습니다.앳킨슨이 1943년에 분리된 살모넬라 애들레이드를 포함한 여러 새로운 살모넬라 균주를 설명한 곳이 바로 여기였습니다.앳킨슨은 1957년에 [15]살모넬라균에 대한 그녀의 연구를 출판했습니다.
혈청형
혈청형은 특정 항원에 대한 항체와 세포를 혼합함으로써 이루어집니다.그것은 위험에 대한 개념을 제공할 수 있습니다.2014년의 한 연구는 S. 판독이 어린 칠면조 샘플들 사이에서 매우 흔하지만,[16] 인간 살모넬라증의 중요한 기여자는 아니라는 것을 보여주었습니다.혈청형 검사는 [17]의심되는 감염원의 혈청형과 사람의 혈청형을 일치시킴으로써 오염원을 식별하는 데 도움이 될 수 있습니다.혈청형의 [18]알려진 항생제 내성에서 적절한 예방 치료법을 식별할 수 있습니다.
xMAP 살모넬라 혈청형 검사와 전통적인 혈청형 검사 및 전체 게놈 시퀀싱에 의한 불일치 해결을 비교한 결과, 살모넬라 혈청형의 빠른 [19]방법으로 분자 혈청형 검사가 유망하다는 연구 결과가 나왔습니다.
장티푸스 살모넬라와 비티푸스 살모넬라의 구별을 위한 실시간 PCR 분석도 이용할 [20]수 있습니다.
탐지, 배양 및 성장 조건

대부분의 살모넬라 아종은 [21]황화수소를 생산하는데, 이는 삼중 당철 시험에 사용되는 것과 같이 황산철이 함유된 배지에서 배양함으로써 쉽게 검출될 수 있습니다.대부분의 절연체는 운동 단계와 비운동 단계의 두 단계로 존재합니다.일차 배양에 운동성이 없는 배양은 크레이지 튜브 또는 [22]도랑판을 사용하여 운동성 단계로 전환될 수 있습니다.RVS 육수는 임상 [23]샘플에서 검출하기 위해 살모넬라 종을 농축하는 데 사용될 수 있습니다.
또한 추출된 살모넬라 DNA에서 다중 또는 실시간 중합효소 연쇄반응(qPCR)[25]을[24] 사용하여 살모넬라를 감지하고 하위 유형화할 수 있습니다.
살모넬라 성장 속도론의 수학적 모델은 닭고기, 돼지고기, 토마토, 그리고 [26][27][28][29][30]멜론을 위해 개발되었습니다.살모넬라균은 40분의 [14][16][17][18]세포분열 간격으로 무성생식을 합니다.
살모넬라 종들은 주로 숙주와 관련된 생활 방식을 주도하지만, 박테리아는 오염 후 몇 주 동안 욕실 환경에서 지속될 수 있고, 박테리아 저장소 역할을 하며 숙주 [31]간의 전염을 용이하게 할 수 있는 수원으로부터 자주 격리되는 것으로 밝혀졌습니다.살모넬라균은 건조함에서 살아남는 능력으로 악명 높고 건조한 환경과 음식에서 [32]수년 동안 지속될 수 있습니다.
박테리아는 얼어서 [33][34]파괴되는 것이 아니라 자외선과 열로 인해 파괴가 가속화됩니다.55 °C (131 °F)에서 90분 동안 또는 60 °C (140 °[35]F)에서 12분 동안 가열한 후 소멸하지만 땅콩 버터와 같은 고지방, 고액체 물질에 접종하면 내열성을 얻고 30분 [36]동안 90 °C (194 °F)까지 생존할 수 있습니다.살모넬라균 감염을 방지하기 위해 식품을 내부 온도 75°C(167°F)로 가열하는 것이 좋습니다.[37][38]
살모넬라 종들은 인간과 동물, 특히 파충류의 소화관에서 발견될 수 있습니다.파충류나 양서류의 피부에 있는 살모넬라균은 [39]동물을 다루는 사람들에게 전달될 수 있습니다.음식과 물은 감염된 사람이나 [40]동물의 배설물과 접촉하면 박테리아에 오염될 수 있습니다.
명명법
처음에 각 살모넬라 "종"은 임상적 고려에 따라 이름이 붙여졌습니다. 예를 들어 살모넬라 티푸스(쥐티푸스), 콜레라 수이스(S. cholerae-suis).많은 종에 대해 숙주 특이성이 존재하지 않는다는 것이 인식된 후, 새로운 변종은 새로운 변종이 [41]분리된 위치에 따라 종 이름을 받았습니다.
1987년, Le Minor와 Popoff는 살모넬라균이 이전의 "종" 이름을 혈청형으로 [42]바꾸면서 오직 한 종인 S. enterica로만 구성되어 있다고 주장하기 위해 분자 발견을 사용했습니다.1989년 리브스 등은 혈청형 V가 자신의 종으로 남아 있어야 한다고 제안하여 S. bongori라는 [43]이름을 부활시켰습니다.따라서 현재(2005년까지) 명명법이 구체화되어 장티카(serotype I), 살라마에(serotype II), 아리조나에(IIIa), 디아리존과(IIIb), 후테네(IV), 인디카(VI)[3][44][45][46] 등 6종의 아종이 인정되었습니다.감염증 전문가들이 새로운 명명법에 익숙하지 않기 때문에 전통적인 명명법은 여전히 일반적입니다.
혈청형 또는 혈청형은 유기체가 제시하는 항원에 기초한 살모넬라의 분류입니다.카우프만-화이트 분류 체계는 혈청학적 다양성을 서로 구별합니다.혈청형은 보통 속과 종 다음으로 아종 그룹으로 분류되며, 혈청형/세로바는 대문자로 표시되지만 이탤릭체로 표시되지는 않습니다.살모넬라 장티케라세로바 티피무륨이 그 예입니다.살모넬라를 유형화하고 하위 유형화하기 위한 보다 현대적인 접근법에는 펄스 필드 겔 전기영동, 다중 위치 VNTR 분석, 다중 위치 시퀀스 유형화 및 다중 PCR 기반 [47][48]방법과 같은 DNA 기반 방법이 포함됩니다.
2005년에 세 번째 종인 살모넬라 서브테라네아가 제안되었지만, 세계보건기구에 따르면 보고된 박테리아는 [49]살모넬라속에 속하지 않습니다.2016년에 S. subteranea를 [50]Atlantibacter subteranea에 할당하는 것이 제안되었지만, LPSN은 IJSB와 [51]IJSEM의 외부에서 만들어졌기 때문에 이를 무효 출판물로 거부합니다. GTB와 NCBI는 2016년 [52][53]재할당에 동의합니다.
GTB RS202는 S. arizonae, S. diarizonae, S. houtenae가 그들 자신의 [54]종이어야 한다고 보고합니다.
병원성
살모넬라종은 세포내 [6]병원균입니다.살모넬라균은 상피 세포, M 세포, 대식세포,[55] 수지상 세포를 포함한 다양한 세포 유형을 침범할 수 있습니다.적합한 혐기성 유기체로서, 살모넬라는 산소를 사용하여 호기성 환경(즉, 산소가 이용 가능할 때)에서 아데노신 삼인산(ATP)을 만듭니다.그러나 혐기성 환경(즉, 산소를 이용할 수 없을 때)에서 살모넬라는 발효에 의해 ATP를 생성합니다; 전자 전달 사슬의 끝에 산소보다 덜 효율적인 네 개의 전자 수용체 중 하나 이상을 치환함으로써: 황산염, 질산염, 황 또는 푸마레이트.[56]
대부분의 감염은 동물의 배설물에 의해 오염된 음식물의 섭취 또는 상업 식당의 식품 서비스 직원에 의한 것과 같은 사람의 배설물에 의한 것입니다.살모넬라 혈청형은 두 가지 주요 그룹, 즉 유형 및 비 유형으로 나눌 수 있습니다.장티푸스 혈청형으로는 살모넬라 티피와 살모넬라 파라티피 A가 있는데, 이들은 사람에게 적응하고 다른 동물에서는 발생하지 않습니다.비형성 혈청형이 더 흔하며, 보통 자기 제한적인 위장병을 유발합니다.그들은 다양한 동물들을 감염시킬 수 있고, 인간과 다른 [57][citation needed]동물들 사이에 옮겨질 수 있다는 것을 의미하는 동물원성입니다.
살모넬라 병원성과 숙주 상호작용은 지난 몇 년간 광범위하게 연구되어 왔습니다.살모넬라의 대부분의 중요한 독성 유전자는 5개의 병원성 섬, 이른바 살모넬라 병원성 섬(SPI)에 암호화되어 있습니다.이것들은 염색체로 암호화되어 있으며 세균-숙주 상호작용에 상당한 기여를 합니다.플라스미드, 편모 또는 바이오필름 관련 단백질과 같은 더 많은 특성이 감염에 기여할 수 있습니다.SPI는 적절한 환경 [58]스트레스가 있는 경우에만 유전자 발현을 허용하는 복잡하고 미세하게 조정된 규제 네트워크에 의해 규제되는 특징이 있습니다.
살모넬라 티피무륨 병원성에 대한 추정 쿼럼 센서인 SdiA 호모로그의 분자 모델링 및 활성 부위 분석은 AHL 전사 [59]조절기의 특정 결합 패턴을 나타냅니다.또한 살모넬라 플라스미드 독성 유전자 spvB가 오토파지를 [60]억제하여 세균 독성을 강화하는 것으로 알려져 있습니다.
장티푸스 살모넬라
장티푸스는 인간이나 고등 영장류에 엄격하게 적응한 살모넬라 혈청형에 의해 발생합니다 - 살모넬라 티피, 파라티피 A, 파라티피 B, 그리고 파라티피 C를 포함합니다.질병의 체계적인 형태에서 살모넬라균은 장의 림프계를 통과하여 환자의 혈액(형질 형태)으로 이동하고 다양한 장기(간, 비장, 신장)로 옮겨져 2차 초점(중증 형태)을 형성합니다.내독소는 먼저 혈관과 신경 기관에 작용하여 투과성을 높이고 혈관의 톤을 감소시키며, 열 조절에 이상을 일으키고 구토와 설사를 일으킵니다.심각한 형태의 질병에서는 물-소금 대사를 혼란시키고, 순환 혈액량과 동맥압을 감소시키며, 저혈압 쇼크를 유발하기에 충분한 액체와 전해질이 손실됩니다.패혈성 쇼크도 발생할 수 있습니다.혼합성 쇼크(저혈량 및 패혈성 쇼크의 징후 모두 포함)는 심각한 살모넬로시스에서 더 흔합니다.심한 경우 저산소증과 [citation needed]독소혈증으로 인한 신부전의 결과로 과뇨와 조혈증이 발생할 수 있습니다.
비형성 살모넬라
비침습적
살모넬라균의 비형성 혈청형에 감염되면 일반적으로 식중독이 발생합니다.감염은 보통 높은 농도의[clarification needed] 박테리아가 포함된 음식을 섭취할 때 발생합니다.유아와 어린 아이들은 적은[clarification needed] 수의 박테리아를 섭취함으로써 쉽게 얻을 수 있는 감염에 훨씬 더 취약합니다.유아의 경우 세균이 많은 먼지를 흡입하여 감염될 [citation needed]수 있습니다.
그 유기체들은 소화관을 통해 들어가고 건강한 성인들에게 질병을 일으키기 위해 많은 양을 섭취해야 합니다.감염은 살아있는 살모넬라균(살모넬라에서 생성된 독소뿐만 아니라)이 위장관에 도달한 후에만 시작될 수 있습니다.일부 미생물은 위에서 죽고, 나머지 미생물은 소장으로 들어가 조직에서 증식합니다.위산도는 섭취된 박테리아의 대부분을 파괴하는 원인이 되지만, 살모넬라균은 섭취된 박테리아의 일부가 [61]생존할 수 있도록 산성 환경에 대한 내성을 어느 정도 발전시켰습니다.세균 군집은 식도에서 생성된 점액에 갇힐 수도 있습니다.배양 기간이 끝날 때까지, 근처의 숙주 세포는 죽은 살모넬라균에서 방출된 내독소에 의해 독이 됩니다.내독소에 대한 국소 반응은 장염과 위장 [citation needed]장애입니다.
약 2,000개의 비형성 살모넬라 혈청형이 알려져 있는데, 이것은 매년 미국에서 140만 개의 질병을 유발할 수 있습니다.심각한 질병에 걸릴 위험이 있는 사람들은 유아, 노인, 장기 이식 수혜자,[40] 그리고 면역력이 손상된 사람들을 포함합니다.
침습적
선진국에서는 비형성 혈청형이 주로 위장병으로 나타나는 반면, 사하라 사막 이남의 아프리카에서는 이러한 혈청형이 혈류 감염에 큰 문제를 일으킬 수 있으며, 발열을 보이는 사람들의 혈액에서 가장 일반적으로 분리된 세균입니다.아프리카에서 비형성 살모넬라균에 의한 혈류 감염은 2012년에 사례 치사율이 20-25%인 것으로 보고되었습니다.침습성 비형성 살모넬라 감염의 대부분의 경우 살모넬라 장티푸스 또는 살모넬라 장티푸스에 의해 발생합니다.75년 전 아프리카 대륙 동남부에서 새로운 형태의 살모넬라 티피무륨(ST313)이 출현했고, 18년 뒤 아프리카 중부에서 두 번째 파도가 밀려왔습니다.iNT의 두 번째 물결은 아마도 콩고 분지에서 시작되었을 것이고, 그 사건 초기에 항생제 클로람페니콜에 내성을 갖게 하는 유전자를 얻었습니다.이것은 치료를 어렵게 만들면서, 매우 가난한 아프리카 지역에서 값비싼 항균제를 사용할 필요성을 만들었습니다.사하라 이남 아프리카에서 iNT의 유병률이 다른 지역에 비해 증가한 것은 특히 어린이의 HIV, 말라리아, 영양실조 등의 부담으로 어느 정도 면역억제나 장애를 가진 아프리카 인구의 비율이 높기 때문인 것으로 생각됩니다.iNTS의 유전자 구성은 더 장티푸스와 같은 박테리아로 진화하고 있으며, 인체에 효율적으로 퍼질 수 있습니다.증상은 발열, 간경화, 호흡기 증상 등 다양하며 위장 증상이 [62]없는 경우가 많습니다.
역학
산발적인 것으로 간주되기 때문에 살모넬라균 감염 사례의 60%에서 80%가 진단되지 않습니다.[63]2010년 3월, 100,000명-년 당 1140명의 발병률을 추정하기 위한 데이터 분석이 완료되었습니다.같은 분석에서, 9,380만 명의 위장염이 살모넬라균 감염에 의한 것이었습니다.5번째 백분위수에서 추정된 금액은 6,180만 건이었고 95번째 백분위수에서 추정된 금액은 1억 3,160만 건이었습니다.살모넬라균에 의한 사망자는 대략 155,000명이었습니다.[64]2014년 불가리아와 포르투갈과 같은 나라에서는 4세 미만의 어린이들이 살모넬라균에 [65]감염될 확률이 각각 32배와 82배 높았습니다.감염에 가장 취약한 사람들은 어린이, 임산부, 노인,[66] 그리고 면역력이 부족한 사람들입니다.
살모넬라균 감염의 위험 요소에는 다양한 음식이 포함됩니다.닭고기와 돼지고기와 같은 고기는 오염될 가능성이 있습니다.다양한 채소와 새싹에도 살모넬라균이 있을 수 있습니다.마지막으로, 치킨너겟과 감자 파이와 같은 다양한 가공 식품들도 이 [67]박테리아를 포함할 수 있습니다.
성공적인 예방 방법은 FDA, 미국 농무부, 식품 안전 검사국과 같은 기존 기관에서 나옵니다.이 모든 조직은 미국의 공공 안전을 보장하기 위해 기준과 검사를 만듭니다. 예를 들어, USDA와 협력하는 FSIS 기관은 살모넬라 행동 계획을 시행하고 있습니다.최근, 그것은 2016년 2월에 2년 계획 업데이트를 받았습니다.살모넬라균 감염을 줄이기 위한 그들의 성과와 전략이 [68]계획에 제시되어 있습니다.질병통제예방센터는 또한 날것 음식을 안전하게 다루는 방법, 그리고 이러한 제품들을 보관하는 올바른 방법과 같은 예방 관리에 대한 귀중한 정보를 제공합니다.유럽 연합에서는 유럽 식품 안전 당국이 위험 관리 및 위험 평가를 통해 예방 조치를 만들었습니다.2005년부터 2009년까지 EFSA는 살모넬라에 대한 노출을 줄이기 위한 접근법을 마련했습니다.그들의 접근 방식에는 가금류의 위험 평가 및 위험 관리가 포함되어 감염 사례가 절반으로 [69]감소했습니다.라틴 아메리카에서 박사에 의해 개발된 가금류의 살모넬라에 대한 경구 투여 백신.세리 레이튼은 박테리아가 [70]새들을 오염시키는 것을 방지하기 위해 도입되었습니다.
최근의 살모넬라 티피무륨 발병은 초콜릿과 [71]관련이 있습니다.
글로벌 모니터링
독일에서는 식품 매개 감염이 [72]보고되어야 합니다.1990년부터 2016년까지, 공식적으로 기록된 사례는 약 20만 건에서 약 13,000 [73]건으로 감소했습니다.미국에서는 매년 [74]약 120만 건의 살모넬라균 감염 사례가 발생하는 것으로 추정됩니다.세계보건기구의 한 연구는 2000년에 21,650,974건의 장티푸스가 발생했고, 그 중 216,510건이 사망했으며, 5,412,744건의 부티푸스가 [75]발생했다고 추정했습니다.
감염의 분자 메커니즘
감염 메커니즘은 신체의 다른 대상과 그들이 유발하는 다른 증상 때문에 장티푸스 혈청형과 비형성 혈청형 사이에서 다릅니다.두 그룹 모두 장세포벽이 만들어내는 장벽을 넘어 들어가야 하지만 일단 이 장벽을 지나면 [citation needed]다른 전략을 사용해 감염을 유발합니다.
독성으로 전환
위장관의 목표 조직으로 이동하는 동안, 살모넬라는 위산, 장에서 담즙의 세제와 같은 활동, 산소 공급 감소, 경쟁하는 정상적인 장내 세균총, 그리고 마지막으로 장벽에 있는 세포 표면에 존재하는 항균 펩타이드에 노출됩니다.이 모든 형태는 살모넬라균이 감지하고 반응할 수 있다는 것을 강조합니다. 그리고 그것들은 독성 인자를 형성하고 장에서 정상적인 성장에서 [76]독성으로의 전환을 조절합니다.
바이러스성으로 전환하면 호스트 내부의 복제 틈새(예: 인간)에 액세스할 수 있으며, 다음과 같은 여러 [citation needed]단계로 요약할 수 있습니다.
- 접근, 그들이 장막막을 통해 또는 편모를 통해 능동적인 수영을 통해 숙주 세포를 향해 이동하고, 점액 장벽을 통과하고, 장에 있는 상피에 가까이 위치하는 접근,
- 박테리아 접착제를 사용하여 숙주 세포에 부착하는 접착제 및 3분비 시스템,
- 살모넬라균이 숙주 세포로 침입하는 침입(아래 변형 메커니즘 참조),
- 숙주 세포 안에서 박테리아가 번식할 수 있는 복제,
- 확산, 박테리아가 혈액의 세포를 통해 다른 장기로 퍼질 수 있습니다(면역 방어를 피하는 데 성공한 경우).그 대신에, 박테리아는 장내로 돌아가서 장내 인구의 씨를 다시 뿌릴 수 있습니다.
- 재침습(2차 감염, 현재 전신 부위에 있는 경우) 및 추가 복제.
진입 메커니즘
비형성 혈청형은 장내 염증 및 설사와 관련된 과정인 세균 매개 내분비에 의해 장벽의 M 세포에 우선적으로 들어갑니다.그것들은 또한 장벽의 세포들 사이의 긴밀한 결합을 방해할 수 있고, 이온, 물, 그리고 장 밖으로 들어오고 나가는 면역 세포들의 흐름을 막는 세포의 능력을 손상시킬 수 있습니다.세균 매개 내분비에 의한 염증과 긴밀한 접합의 중단이 설사를 [77]유발하는 데 크게 기여하는 것으로 생각됩니다.
살모넬라균은 또한 식세포증과 CD18 양성 면역 세포에 의한 밀매를 통해 장 장벽을 뚫을 수 있는데, 이는 장티푸스 살모넬라균 감염의 메커니즘일 수 있습니다.이것은 장 장벽을 통과하는 더 은밀한 방법으로 생각되며, 따라서 비형성 살모넬라보다 더 적은 수의 장티푸스 살모넬라가 [77]감염에 필요하다는 사실에 기여할 수 있습니다.살모넬라 세포는 대식세포증을 [78]통해 대식세포로 들어갈 수 있습니다.장티푸스 혈청형은 면역 세포를 포함하고 [77]있고 몸 전체에 면역 체계와 관련된 조직을 둘러싸고 있는 결합 조직의 네트워크인 단핵 식세포 시스템을 통해 몸 전체에 전파를 달성하기 위해 이것을 사용할 수 있습니다.
감염을 일으키는 살모넬라균의 성공의 대부분은 감염 동안 서로 다른 시간에 발현되는 두 가지 유형의 III 분비 시스템(T3SS)에 기인합니다.T3SS-1은 숙주 세포질 내에 박테리아 이펙터의 주입을 가능하게 합니다.이러한 T3SS-1 효과기는 비식세포에 의한 살모넬라의 흡수를 허용하는 막 주름의 형성을 자극합니다.살모넬라는 또한 살모넬라 함유 바쿠올(SCV)이라고 불리는 막으로 둘러싸인 구획 내에 존재합니다.SCV의 산성화는 T3SS-2의 발현으로 이어집니다.살모넬라에 의한 T3SS-2 이펙터의 분비는 숙주 세포질에서의 효율적인 생존과 전신 [77]질환의 확립을 위해 필요합니다.게다가, 두 T3SS는 장의 식민화, 장 염증 반응의 유도 및 설사에 관여합니다.이러한 시스템은 감염을 [citation needed]달성하기 위해 협력해야 하는 많은 유전자를 포함하고 있습니다.
S의 SPI1 type III 분비 시스템에 의해 주입된 AvrA 독소.티피무륨은 세린/트레오닌 아세틸전달효소 활성으로 인해 선천 면역계를 억제하는 작용을 하며, 진핵생물 표적 세포 피티산(IP6)[79]에 결합해야 합니다.이로 인해 호스트는 [citation needed]감염에 더 취약해집니다.
임상 증상
살모넬라증은 허리 통증이나 척추 질환을 일으킬 수 있는 것으로 알려져 있습니다.그것은 5가지 임상 패턴으로 나타날 수 있습니다: 위장 감염, 장열, 세균혈증, 국소 감염, 그리고 만성 저장고 상태.초기 증상은 발열, 허약, 근육통 등입니다.균혈증 상태에서, 그것은 신체의 어느 부분으로도 퍼질 수 있고 이것은 국소적인 감염을 유발하거나 종기를 형성합니다.국소 살모넬라 감염의 형태는 관절염, 요로 감염, 중추신경계 감염, 뼈 감염, 연조직 감염 [80]등입니다.감염은 잠복 형태로 오래 남아있을 수 있으며, 망상내피세포의 기능이 저하되면 활성화되어 급성 살모넬라증 [80]후 수개월 또는 수년 후에 2차적으로 뼈에 감염을 확산시킬 수 있습니다.
2018년 임페리얼 칼리지 런던 연구는 또한 살모넬라균이 아연 금속단백질가수분해효소 이펙터 계열을 사용하여 면역계의 특정 팔(예: 5개의 NF-카파B 단백질 중 3개)을 어떻게 방해하고 다른 팔은 [81]건드리지 않는지 보여줍니다.살모넬라 갑상선 농양 또한 [82]보고되었습니다.
산화 버스트에 대한 저항
살모넬라 병인의 특징은 식세포 내에서 생존하고 증식하는 박테리아의 능력입니다.식세포는 병원체에 대한 방어책으로 일산화질소와 산소 라디칼과 같은 DNA 손상제를 생산합니다.따라서, 살모넬라 종들은 게놈 무결성에 도전하는 분자들의 공격에 직면해야 합니다.부크마이어 [83]외 연구진은 RecA 또는 RecBC 단백질 기능이 부족한 S. 엔테리카 돌연변이체가 대식세포에 의해 합성된 산화성 화합물에 매우 민감하다는 것을 보여주었고, 더 나아가 이러한 발견은 S. 엔테리카에 의한 성공적인 전신 감염이 RecA- 및 RecBC 매개 DNA [83][84]손상의 재조합 복구를 필요로 한다는 것을 나타냅니다.
호스트 적응
장티푸스와 엔테리티디스와 같은 몇몇 혈청형을 통해, 장티푸스와 같은 다른 혈청형은 몇몇 다른 포유류 숙주 종들을 감염시킬 수 있는 능력의 징후를 보여주는 반면, 장티푸스와 같은 다른 혈청형은 소수의 [85]숙주들에게만 제한되는 것으로 보입니다.살모넬라 혈청형이 숙주에게 적응한 방법 중 일부는 유전 물질의 손실과 돌연변이를 포함합니다.더 복잡한 포유류 종에서, 병원체 특이적 면역 반응을 포함하는 면역 체계는 편모세포와 같은 구조에 대한 항체의 결합을 통해 살모넬라의 혈청을 목표로 합니다.편모가 형성되도록 코드하는 유전 물질의 손실을 통해, 살모넬라는 숙주의 면역 [86]체계를 피할 수 있습니다. 박테리아 독성 유전자 (mgtCBR operon)의 mgtC 리더 RNA는 편모를 코드하는 flJB 유전자의 mRNA와 직접 염기쌍을 형성하여 감염 동안 편모넬라 생성을 감소시키고 [87]분해를 촉진합니다.키셀라 외 연구진의 연구에서, S. 엔테리카의 더 많은 병원성 혈청은 수렴 [88]진화에서 발전한 특정 접착제를 공통적으로 가지고 있는 것으로 밝혀졌습니다.이것은 이러한 살모넬라 균주가 면역 시스템과 같은 유사한 조건에 노출됨에 따라 유사한 구조가 숙주에서 유사하고 더 발전된 방어를 무효화하기 위해 별도로 진화했다는 것을 의미합니다.여전히, 살모넬라균이 매우 다양한 유형으로 진화한 방법에 대해 많은 의문이 남아 있지만, 살모넬라균은 여러 단계를 거쳐 진화했을 수 있습니다.Baumler 등으로서.살모넬라균은 수평적인 유전자 전달, 추가적인 병원성 섬들로 인한 새로운 혈청의 형성,[89] 그리고 그것의 조상에 대한 근사치를 통해 진화했을 가능성이 가장 높다고 제안했습니다.그래서, 살모넬라균은 다른 병원성 박테리아로부터 유전 정보를 얻음으로써 많은 다른 혈청형으로 진화할 수 있었습니다.서로 다른 혈청형의 게놈에 여러 병원성 섬이 있다는 것은 이 [89]이론에 신빙성을 주었습니다.
살모넬라 sv.뉴포트는 식물의 식민지화 생활 방식에 적응하는 징후가 있으며, 이는 생산과 관련된 식인성 질병과의 불균형적인 연관성에 역할을 할 수 있습니다.sv 중에 선택된 다양한 기능.토마토의 뉴포트 지속성은 insv에 대해 선택된 것과 유사한 것으로 보고되었습니다.동물 [90]숙주에게서 온 티피무륨.sv 고유의 papA 유전자.뉴포트는 토마토에서 균주의 적합성에 기여하고 식물과 동물 [90]숙주를 식민지화할 수 있는 다른 엔테로박테리아과의 게놈에 상동체를 가지고 있습니다.
조사.
병원체로서의 중요성 외에도, 장티푸스 종의 상동체로서 장티푸스와 같은 비형성 살모넬라 종이 일반적으로 사용됩니다.많은 연구 결과를 이전할 수 있으며 오염 시 연구자의 위험을 감소시키지만 제한적이기도 합니다.예를 들어, 이 [91]모델을 사용하여 특정 장티푸스 독소를 연구할 수 없습니다.그러나 일반적으로 사용되는 마우스 장 위장염 모델과 같은 강력한 연구 도구는 살모넬라 티피무륨의 [92]사용을 기반으로 합니다.
유전학은 S.티피무륨은 근본적인 박테리아 생리학에 대한 이해를 이끈 유전적 도구의 개발에 중요한 역할을 해왔습니다.이러한 개발은 S에서 최초의 일반화된 변환 단계 P22의[93] 발견에 의해 가능해졌습니다.빠르고 쉬운 유전자 편집을 가능하게 한 티피무륨.결국, 이것은 미세한 구조 유전자 분석을 가능하게 했습니다.돌연변이의 많은 수가 [94]박테리아에 대한 유전적 명명법의 개정으로 이어졌습니다.트랜스포손 전달, 돌연변이 유발, 염색체 재배열 구성을 포함한 유전자 도구로서의 트랜스포손의 많은 사용 또한 S에서 개발되었습니다.티피무륨.이 유전자 도구들은 또한 발암물질에 대한 간단한 테스트, 에임스 [95]테스트로 이어졌습니다.
전통적인 항균제에 대한 자연적인 대안으로, 파지는 살모넬라균과 다른 식원성 [96]박테리아에 매우 효과적인 통제제로 인식되고 있습니다.
고대 DNA
서유라시아 전역에 걸쳐 최대 6,500년 된 인간 유골에서 장티푸스 유전체가 재구성되었으며, 이는 선사 시대 동안 전신 장티푸스에 지리적으로 광범위하게 감염된 증거와 숙주 [97][98]적응의 진화에서 신석기화 과정의 가능한 역할을 제공합니다.멕시코 식민지에서 추가로 재구성된 게놈은 16세기 뉴스페인에서 유행한 [99]코콜리츨리의 원인으로 S. 엔테리카를 제시합니다.
참고 항목
- 1984년 라즈니쉬 생물학 테러
- 1998년 호르멜 대재앙
- 2008년 미국 살모넬라증 발병
- 미국 공중보건협회 대 버츠 사건
- 아황산 비스무트 한천
- 식품 테스트 스트립
- 호스트-병원체 인터페이스
- 식중독 발생 목록
- 2008~2009년 땅콩 매개 살모넬라증
- 라이트 카운티 에그
- XLD 한천
레퍼런스
- ^ "Salmonella". NCBI taxonomy. Bethesda, MD: National Center for Biotechnology Information. Retrieved 27 January 2019.
- ^ Su LH, Chiu CH (2007). "Salmonella: clinical importance and evolution of nomenclature". Chang Gung Medical Journal. 30 (3): 210–219. PMID 17760271.
- ^ a b Ryan MP, O'Dwyer J, Adley CC (2017). "Evaluation of the Complex Nomenclature of the Clinically and Veterinary Significant Pathogen Salmonella". BioMed Research International. 2017: 3782182. doi:10.1155/2017/3782182. PMC 5429938. PMID 28540296.
- ^ Gal-Mor O, Boyle EC, Grassl GA (2014). "Same species, different diseases: how and why typhoidal and non-typhoidal Salmonella enterica serovars differ". Frontiers in Microbiology. 5: 391. doi:10.3389/fmicb.2014.00391. PMC 4120697. PMID 25136336.
- ^ a b Fàbrega A, Vila J (April 2013). "Salmonella enterica serovar Typhimurium skills to succeed in the host: virulence and regulation". Clinical Microbiology Reviews. 26 (2): 308–341. doi:10.1128/CMR.00066-12. PMC 3623383. PMID 23554419.
- ^ a b Jantsch J, Chikkaballi D, Hensel M (March 2011). "Cellular aspects of immunity to intracellular Salmonella enterica". Immunological Reviews. 240 (1): 185–195. doi:10.1111/j.1600-065X.2010.00981.x. PMID 21349094. S2CID 19344119.
- ^ Ryan I KJ, Ray CG, eds. (2004). Sherris Medical Microbiology (4th ed.). McGraw Hill. pp. 362–8. ISBN 978-0-8385-8529-0.
- ^ Brenner FW, Villar RG, Angulo FJ, Tauxe R, Swaminathan B (July 2000). "Salmonella nomenclature". Journal of Clinical Microbiology. 38 (7): 2465–2467. doi:10.1128/JCM.38.7.2465-2467.2000. PMC 86943. PMID 10878026.
- ^ Gillespie, Stephen H., Hawkey, Peter M., eds. (2006). Principles and practice of clinical bacteriology (2nd ed.). Hoboken, NJ: John Wiley & Sons. ISBN 9780470017968.
- ^ Okoro CK, Kingsley RA, Connor TR, Harris SR, Parry CM, Al-Mashhadani MN, et al. (November 2012). "Intracontinental spread of human invasive Salmonella Typhimurium pathovariants in sub-Saharan Africa". Nature Genetics. 44 (11): 1215–1221. doi:10.1038/ng.2423. PMC 3491877. PMID 23023330.
- ^ Eberth CJ (1880-07-01). "Die Organismen in den Organen bei Typhus abdominalis". Archiv für Pathologische Anatomie und Physiologie und für Klinische Medicin (in German). 81 (1): 58–74. doi:10.1007/BF01995472. S2CID 6979333.
- ^ Hardy A (August 1999). "Food, hygiene, and the laboratory. A short history of food poisoning in Britain, circa 1850-1950". Social History of Medicine. 12 (2): 293–311. doi:10.1093/shm/12.2.293. PMID 11623930.
- ^ "FDA/CFSAN—Food Safety A to Z Reference Guide—Salmonella". FDA–Center for Food Safety and Applied Nutrition. 2008-07-03. Archived from the original on 2009-03-02. Retrieved 2009-02-14.
- ^ a b Heymann DA, Alcamo IE, Heymann DL (2006). Salmonella. Philadelphia: Chelsea House Publishers. ISBN 978-0-7910-8500-4. Retrieved 31 July 2015.
- ^ McEwin E, "Atkinson, Nancy (1910–1999)", Australian Dictionary of Biography, Canberra: National Centre of Biography, Australian National University, retrieved 2022-10-27
- ^ a b "Serotypes Profile of Salmonella Isolates from Meat and Poultry Products, January 1998 through December 2014".
- ^ a b "Steps in a Foodborne Outbreak Investigation". 2018-11-09.
- ^ a b Yoon KB, Song BJ, Shin MY, Lim HC, Yoon YH, Jeon DY, et al. (June 2017). "Antibiotic Resistance Patterns and Serotypes of Salmonella spp. Isolated at Jeollanam-do in Korea". Osong Public Health and Research Perspectives. 8 (3): 211–219. doi:10.24171/j.phrp.2017.8.3.08. PMC 5525558. PMID 28781944.
- ^ Luo Y, Huang C, Ye J, Octavia S, Wang H, Dunbar SA, et al. (2020-09-07). "Comparison of xMAP Salmonella Serotyping Assay With Traditional Serotyping and Discordance Resolution by Whole Genome Sequencing". Frontiers in Cellular and Infection Microbiology. 10: 452. doi:10.3389/fcimb.2020.00452. PMC 7504902. PMID 33014887.
- ^ Nair S, Patel V, Hickey T, Maguire C, Greig DR, Lee W, et al. (August 2019). Ledeboer NA (ed.). "Real-Time PCR Assay for Differentiation of Typhoidal and Nontyphoidal Salmonella". Journal of Clinical Microbiology. 57 (8): e00167–19. doi:10.1128/JCM.00167-19. PMC 6663909. PMID 31167843.
- ^ Clark MA, Barrett EL (June 1987). "The phs gene and hydrogen sulfide production by Salmonella typhimurium". Journal of Bacteriology. 169 (6): 2391–2397. doi:10.1128/jb.169.6.2391-2397.1987. PMC 212072. PMID 3108233.
- ^ "UK Standards for Microbiology Investigations: Changing the Phase of Salmonella" (PDF). UK Standards for Microbiology Investigations: 8–10. 8 January 2015. Archived (PDF) from the original on 2022-10-09. Retrieved 2 August 2015.
- ^ Snyder JW, Atlas RM (2006). Handbook of Media for Clinical Microbiology. CRC Press. p. 374. ISBN 978-0849337956.
- ^ Alvarez J, Sota M, Vivanco AB, Perales I, Cisterna R, Rementeria A, Garaizar J (April 2004). "Development of a multiplex PCR technique for detection and epidemiological typing of salmonella in human clinical samples". Journal of Clinical Microbiology. 42 (4): 1734–1738. doi:10.1128/JCM.42.4.1734-1738.2004. PMC 387595. PMID 15071035.
- ^ Hoorfar J, Ahrens P, Rådström P (September 2000). "Automated 5' nuclease PCR assay for identification of Salmonella enterica". Journal of Clinical Microbiology. 38 (9): 3429–3435. doi:10.1128/JCM.38.9.3429-3435.2000. PMC 87399. PMID 10970396.
- ^ Dominguez SA, Schaffner DW (December 2008). "Modeling the growth of Salmonella in raw poultry stored under aerobic conditions". Journal of Food Protection. 71 (12): 2429–2435. doi:10.4315/0362-028x-71.12.2429. PMID 19244895.
- ^ Pin C, Avendaño-Perez G, Cosciani-Cunico E, Gómez N, Gounadakic A, Nychas GJ, et al. (March 2011). "Modelling Salmonella concentration throughout the pork supply chain by considering growth and survival in fluctuating conditions of temperature, pH and a(w)". International Journal of Food Microbiology. 145 (Suppl 1): S96-102. doi:10.1016/j.ijfoodmicro.2010.09.025. PMID 20951457.
- ^ Pan W, Schaffner DW (August 2010). "Modeling the growth of Salmonella in cut red round tomatoes as a function of temperature". Journal of Food Protection. 73 (8): 1502–1505. doi:10.4315/0362-028X-73.8.1502. PMID 20819361.
- ^ Li D, Friedrich LM, Danyluk MD, Harris LJ, Schaffner DW (June 2013). "Development and validation of a mathematical model for growth of pathogens in cut melons". Journal of Food Protection. 76 (6): 953–958. doi:10.4315/0362-028X.JFP-12-398. PMID 23726189.
- ^ Li D. "Development and validation of a mathematical model for growth of salmonella in cantaloupe". Archived from the original on November 20, 2013.
- ^ Winfield MD, Groisman EA (July 2003). "Role of nonhost environments in the lifestyles of Salmonella and Escherichia coli". Applied and Environmental Microbiology. 69 (7): 3687–3694. Bibcode:2003ApEnM..69.3687W. doi:10.1128/aem.69.7.3687-3694.2003. PMC 165204. PMID 12839733.
- ^ Mandal RK, Kwon YM (8 September 2017). "Global Screening of Salmonella enterica Serovar Typhimurium Genes for Desiccation Survival". Frontiers in Microbiology. 8 (1723): 1723. doi:10.3389/fmicb.2017.01723. PMC 5596212. PMID 28943871.
- ^ Sorrells KM, Speck ML, Warren JA (January 1970). "Pathogenicity of Salmonella gallinarum after metabolic injury by freezing". Applied Microbiology. 19 (1): 39–43. doi:10.1128/AEM.19.1.39-43.1970. PMC 376605. PMID 5461164.
Mortality differences between wholly uninjured and predominantly injured populations were small and consistent (5% level) with a hypothesis of no difference.
- ^ Beuchat LR, Heaton EK (June 1975). "Salmonella survival on pecans as influenced by processing and storage conditions". Applied Microbiology. 29 (6): 795–801. doi:10.1128/AEM.29.6.795-801.1975. PMC 187082. PMID 1098573.
Little decrease in viable population of the three species was noted on inoculated pecan halves stored at -18, -7, and 5°C for 32 weeks.
- ^ Goodfellow SJ, Brown WL (August 1978). "Fate of Salmonella Inoculated into Beef for Cooking". Journal of Food Protection. 41 (8): 598–605. doi:10.4315/0362-028x-41.8.598. PMID 30795117.
- ^ Ma L, Zhang G, Gerner-Smidt P, Mantripragada V, Ezeoke I, Doyle MP (August 2009). "Thermal inactivation of Salmonella in peanut butter". Journal of Food Protection. 72 (8): 1596–1601. doi:10.4315/0362-028x-72.8.1596. PMID 19722389.
- ^ 식품 안전 교육을 위한 파트너십 (PFSE) Fight BAC! Wayback Machine에서 2013-08-31 기본 브로셔 보관.
- ^ 웨이백 머신에서 2012-05-03 USDA 내부 조리 온도 차트 보관.USDA는 Wayback Machine 팩트시트 페이지의 Safe Food Handling Archived 2013-06-05에서 다른 리소스를 이용할 수 있습니다.국립 가정식량보존센터도 참고하세요.
- ^ "Reptiles, Amphibians, and Salmonella". Centers for Disease Control and Prevention. U.S. Department of Health & Human Services. 25 November 2013. Retrieved 3 August 2013.
- ^ a b Goldrick BA (March 2003). "Foodborne diseases". The American Journal of Nursing. 103 (3): 105–106. doi:10.1097/00000446-200303000-00043. PMID 12635640.
- ^ F. 카우프만:살모넬라-그룹의 다이 박테리올로지에.몽크스가드, 코펜하겐, 1941
- ^ Le Minor L, Popoff MY (1987). "Request for an Opinion: Designation of Salmonella enterica sp. nov., nom. rev., as the type and only species of the genus Salmonella". Int. J. Syst. Bacteriol. 37 (4): 465–468. doi:10.1099/00207713-37-4-465.
- ^ Reeves MW, Evins GM, Heiba AA, Plikaytis BD, Farmer JJ (February 1989). "Clonal nature of Salmonella typhi and its genetic relatedness to other salmonellae as shown by multilocus enzyme electrophoresis, and proposal of Salmonella bongori comb. nov". Journal of Clinical Microbiology. 27 (2): 313–320. doi:10.1128/JCM.27.2.313-320.1989. PMC 267299. PMID 2915026.
- ^ Janda JM, Abbott SL (2006)."장세균", ASM 출판사.
- ^ Judicial Commission Of The International Committee On Systematics Of Prokaryotes (January 2005). "The type species of the genus Salmonella Lignieres 1900 is Salmonella enterica (ex Kauffmann and Edwards 1952) Le Minor and Popoff 1987, with the type strain LT2T, and conservation of the epithet enterica in Salmonella enterica over all earlier epithets that may be applied to this species. Opinion 80". International Journal of Systematic and Evolutionary Microbiology. 55 (Pt 1): 519–520. doi:10.1099/ijs.0.63579-0. PMID 15653929.
- ^ Tindall BJ, Grimont PA, Garrity GM, Euzéby JP (January 2005). "Nomenclature and taxonomy of the genus Salmonella". International Journal of Systematic and Evolutionary Microbiology. 55 (Pt 1): 521–524. doi:10.1099/ijs.0.63580-0. PMID 15653930.
- ^ Porwollik S, ed. (2011). Salmonella: From Genome to Function. Caister Academic Press. ISBN 978-1-904455-73-8.
- ^ Achtman M, Wain J, Weill FX, Nair S, Zhou Z, Sangal V, et al. (2012). "Multilocus sequence typing as a replacement for serotyping in Salmonella enterica". PLOS Pathogens. 8 (6): e1002776. doi:10.1371/journal.ppat.1002776. PMC 3380943. PMID 22737074.

- ^ Grimont PA, Xavier Weill F (2007). Antigenic Formulae of the Salmonella Serovars (PDF) (9th ed.). Institut Pasteur, Paris, France: WHO Collaborating Centre for Reference and Research on Salmonella. p. 7. Archived (PDF) from the original on 2022-10-09. Retrieved 26 August 2015.
- ^ Hata H, Natori T, Mizuno T, Kanazawa I, Eldesouky I, Hayashi M, et al. (May 2016). "Phylogenetics of family Enterobacteriaceae and proposal to reclassify Escherichia hermannii and Salmonella subterranea as Atlantibacter hermannii and Atlantibacter subterranea gen. nov., comb. nov". Microbiology and Immunology. 60 (5): 303–311. doi:10.1111/1348-0421.12374. PMID 26970508. S2CID 32594451.
- ^ "Species: Atlantibacter subterranea". lpsn.dsmz.de.
- ^ "GTDB - Tree at g__Atlantibacter". gtdb.ecogenomic.org.
- ^ "Taxonomy browser (Atlantibacter)". www.ncbi.nlm.nih.gov.
- ^ "GTDB - Tree at g__Salmonella". gtdb.ecogenomic.org.
- ^ LaRock DL, Chaudhary A, Miller SI (April 2015). "Salmonellae interactions with host processes". Nature Reviews. Microbiology. 13 (4): 191–205. doi:10.1038/nrmicro3420. PMC 5074537. PMID 25749450.
- ^ Garai P, Gnanadhas DP, Chakravortty D (July 2012). "Salmonella enterica serovars Typhimurium and Typhi as model organisms: revealing paradigm of host-pathogen interactions". Virulence. 3 (4): 377–388. doi:10.4161/viru.21087. PMC 3478240. PMID 22722237.
- ^ "What is the difference between nontyphoidal salmonellae and S typhi or S paratyphi?". www.medscape.com. Retrieved 2021-09-28.
- ^ Fàbrega A, Vila J (April 2013). "Salmonella enterica serovar Typhimurium skills to succeed in the host: virulence and regulation". Clinical Microbiology Reviews. 26 (2): 308–341. doi:10.1128/CMR.00066-12. PMC 3623383. PMID 23554419.
- ^ Gnanendra S, Anusuya S, Natarajan J (October 2012). "Molecular modeling and active site analysis of SdiA homolog, a putative quorum sensor for Salmonella typhimurium pathogenecity reveals specific binding patterns of AHL transcriptional regulators". Journal of Molecular Modeling. 18 (10): 4709–4719. doi:10.1007/s00894-012-1469-1. PMID 22660944. S2CID 25177221.
- ^ Li YY, Wang T, Gao S, Xu GM, Niu H, Huang R, Wu SY (February 2016). "Salmonella plasmid virulence gene spvB enhances bacterial virulence by inhibiting autophagy in a zebrafish infection model". Fish & Shellfish Immunology. 49: 252–259. doi:10.1016/j.fsi.2015.12.033. PMID 26723267.
- ^ Garcia-del Portillo F, Foster JW, Finlay BB (October 1993). "Role of acid tolerance response genes in Salmonella typhimurium virulence". Infection and Immunity. 61 (10): 4489–4492. doi:10.1128/IAI.61.10.4489-4492.1993. PMC 281185. PMID 8406841.
- ^ Feasey NA, Dougan G, Kingsley RA, Heyderman RS, Gordon MA (June 2012). "Invasive non-typhoidal salmonella disease: an emerging and neglected tropical disease in Africa". Lancet. 379 (9835): 2489–2499. doi:10.1016/S0140-6736(11)61752-2. PMC 3402672. PMID 22587967.
- ^ "Salmonella (non-typhoidal)". www.who.int. Retrieved 2019-12-05.
- ^ Majowicz SE, Musto J, Scallan E, Angulo FJ, Kirk M, O'Brien SJ, et al. (March 2010). "The global burden of nontyphoidal Salmonella gastroenteritis". Clinical Infectious Diseases. 50 (6): 882–889. doi:10.1086/650733. PMID 20158401.
- ^ "Salmonellosis - Annual Epidemiological Report 2016 [2014 data]". European Centre for Disease Prevention and Control. 2016-01-31. Retrieved 2019-12-05.
- ^ "Get the Facts about Salmonella!". Center for Veterinary Medicine, FDA. 2019-06-06.
- ^ "Prevention General Information Salmonella CDC". www.cdc.gov. 2019-03-06. Retrieved 2019-12-05.
- ^ "Salmonella". fsis.usda.gov.
- ^ "Salmonella". European Food Safety Authority. Retrieved 2019-12-05.
- ^ "Content not found". 3 March 2022.
- ^ Samarasekera U (July 2022). "Salmonella Typhimurium outbreak linked to chocolate". The Lancet. Infectious Diseases. 22 (7): 947. doi:10.1016/S1473-3099(22)00351-6. PMID 35636448. S2CID 249136373.
- ^ 감염증 예방에 관한 독일법 제6조 및 제7조, 감염증 슈추츠게제츠
- ^ "Anzahl der jährlich registrierten Salmonellose-Erkrankungen in Deutschland bis 2016". Statista.
- ^ 살모넬라균.질병통제예방센터
- ^ Crump JA, Luby SP, Mintz ED (May 2004). "The global burden of typhoid fever". Bulletin of the World Health Organization. 82 (5): 346–353. PMC 2622843. PMID 15298225.
- ^ Rychlik I, Barrow PA (November 2005). "Salmonella stress management and its relevance to behaviour during intestinal colonisation and infection". FEMS Microbiology Reviews. 29 (5): 1021–1040. doi:10.1016/j.femsre.2005.03.005. PMID 16023758.
- ^ a b c d Haraga A, Ohlson MB, Miller SI (January 2008). "Salmonellae interplay with host cells". Nature Reviews. Microbiology. 6 (1): 53–66. doi:10.1038/nrmicro1788. PMID 18026123. S2CID 2365666.
- ^ Kerr MC, Wang JT, Castro NA, Hamilton NA, Town L, Brown DL, et al. (April 2010). "Inhibition of the PtdIns(5) kinase PIKfyve disrupts intracellular replication of Salmonella". The EMBO Journal. 29 (8): 1331–1347. doi:10.1038/emboj.2010.28. PMC 2868569. PMID 20300065.
- ^ Mittal R, Peak-Chew SY, Sade RS, Vallis Y, McMahon HT (June 2010). "The acetyltransferase activity of the bacterial toxin YopJ of Yersinia is activated by eukaryotic host cell inositol hexakisphosphate". The Journal of Biological Chemistry. 285 (26): 19927–19934. doi:10.1074/jbc.M110.126581. PMC 2888404. PMID 20430892.
- ^ a b Choi YS, Cho WJ, Yun SH, Lee SY, Park SH, Park JC, et al. (December 2010). "A case of back pain caused by Salmonella spondylitis -A case report-". Korean Journal of Anesthesiology. 59 (Suppl): S233–S237. doi:10.4097/kjae.2010.59.S.S233. PMC 3030045. PMID 21286449.
- ^ "Bacterial protein mimics DNA to sabotage cells' defenses: Study reveals details of Salmonella infections". Sciencedaily.
- ^ Sahli M, Hemmaoui B, Errami N, Benariba F (January 2022). "Salmonella thyroid abscess". European Annals of Otorhinolaryngology, Head and Neck Diseases. 139 (1): 51–52. doi:10.1016/j.anorl.2021.05.004. PMID 34053890. S2CID 235256840.
- ^ a b Buchmeier NA, Lipps CJ, So MY, Heffron F (March 1993). "Recombination-deficient mutants of Salmonella typhimurium are avirulent and sensitive to the oxidative burst of macrophages". Molecular Microbiology. 7 (6): 933–936. doi:10.1111/j.1365-2958.1993.tb01184.x. PMID 8387147. S2CID 25127127.
- ^ Cano DA, Pucciarelli MG, García-del Portillo F, Casadesús J (January 2002). "Role of the RecBCD recombination pathway in Salmonella virulence". Journal of Bacteriology. 184 (2): 592–595. doi:10.1128/jb.184.2.592-595.2002. PMC 139588. PMID 11751841.
- ^ Thomson NR, Clayton DJ, Windhorst D, Vernikos G, Davidson S, Churcher C, et al. (October 2008). "Comparative genome analysis of Salmonella Enteritidis PT4 and Salmonella Gallinarum 287/91 provides insights into evolutionary and host adaptation pathways". Genome Research. 18 (10): 1624–1637. doi:10.1101/gr.077404.108. PMC 2556274. PMID 18583645.
- ^ den Bakker HC, Moreno Switt AI, Govoni G, Cummings CA, Ranieri ML, Degoricija L, et al. (August 2011). "Genome sequencing reveals diversification of virulence factor content and possible host adaptation in distinct subpopulations of Salmonella enterica". BMC Genomics. 12: 425. doi:10.1186/1471-2164-12-425. PMC 3176500. PMID 21859443.
- ^ Choi E, Han Y, Cho YJ, Nam D, Lee EJ (September 2017). "A trans-acting leader RNA from a Salmonella virulence gene". Proceedings of the National Academy of Sciences of the United States of America. 114 (38): 10232–10237. Bibcode:2017PNAS..11410232C. doi:10.1073/pnas.1705437114. PMC 5617274. PMID 28874555.
- ^ Kisiela DI, Chattopadhyay S, Libby SJ, Karlinsey JE, Fang FC, Tchesnokova V, et al. (2012). "Evolution of Salmonella enterica virulence via point mutations in the fimbrial adhesin". PLOS Pathogens. 8 (6): e1002733. doi:10.1371/journal.ppat.1002733. PMC 3369946. PMID 22685400.
- ^ a b Bäumler AJ, Tsolis RM, Ficht TA, Adams LG (October 1998). "Evolution of host adaptation in Salmonella enterica". Infection and Immunity. 66 (10): 4579–4587. doi:10.1128/IAI.66.10.4579-4587.1998. PMC 108564. PMID 9746553.
- ^ a b de Moraes MH, Soto EB, Salas González I, Desai P, Chu W, Porwollik S, et al. (2018). "Genome-Wide Comparative Functional Analyses Reveal Adaptations of Salmonella sv. Newport to a Plant Colonization Lifestyle". Frontiers in Microbiology. 9: 877. doi:10.3389/fmicb.2018.00877. PMC 5968271. PMID 29867794.
- ^ Johnson R, Mylona E, Frankel G (September 2018). "Typhoidal Salmonella: Distinctive virulence factors and pathogenesis". Cellular Microbiology. 20 (9): e12939. doi:10.1111/cmi.12939. PMID 30030897. S2CID 51705854.
- ^ Hapfelmeier S, Hardt WD (October 2005). "A mouse model for S. typhimurium-induced enterocolitis". Trends in Microbiology. 13 (10): 497–503. doi:10.1016/j.tim.2005.08.008. PMID 16140013.
- ^ Zinder ND, Lederberg J (November 1952). "Genetic exchange in Salmonella". Journal of Bacteriology. 64 (5): 679–699. doi:10.1128/JB.64.5.679-699.1952. PMC 169409. PMID 12999698.
- ^ Demerec M, Adelberg EA, Clark AJ, Hartman PE (July 1966). "A proposal for a uniform nomenclature in bacterial genetics". Genetics. 54 (1): 61–76. doi:10.1093/genetics/54.1.61. PMC 1211113. PMID 5961488.
- ^ Ames BN, Mccann J, Yamasaki E (December 1975). "Methods for detecting carcinogens and mutagens with the Salmonella/mammalian-microsome mutagenicity test". Mutation Research. 31 (6): 347–364. doi:10.1016/0165-1161(75)90046-1. PMID 768755.
- ^ Ge H, Lin C, Xu Y, Hu M, Xu Z, Geng S, et al. (June 2022). "A phage for the controlling of Salmonella in poultry and reducing biofilms". Veterinary Microbiology. 269: 109432. doi:10.1016/j.vetmic.2022.109432. PMID 35489296. S2CID 248195843.
- ^ Key FM, Posth C, Esquivel-Gomez LR, Hübler R, Spyrou MA, Neumann GU, et al. (March 2020). "Emergence of human-adapted Salmonella enterica is linked to the Neolithization process". Nature Ecology & Evolution. 4 (3): 324–333. doi:10.1038/s41559-020-1106-9. PMC 7186082. PMID 32094538.
- ^ Zhou Z, Lundstrøm I, Tran-Dien A, Duchêne S, Alikhan NF, Sergeant MJ, et al. (August 2018). "Pan-genome Analysis of Ancient and Modern Salmonella enterica Demonstrates Genomic Stability of the Invasive Para C Lineage for Millennia". Current Biology. 28 (15): 2420–2428.e10. doi:10.1016/j.cub.2018.05.058. PMC 6089836. PMID 30033331.
- ^ Vågene ÅJ, Herbig A, Campana MG, Robles García NM, Warinner C, Sabin S, et al. (March 2018). "Salmonella enterica genomes from victims of a major sixteenth-century epidemic in Mexico". Nature Ecology & Evolution. 2 (3): 520–528. doi:10.1038/s41559-017-0446-6. PMID 29335577. S2CID 3358440.
외부 링크
- 미국 농무부 식품안전검사국의 살모넬라균 감염 배경
- NIAID가 자금을 지원하는 생물정보자원센터인 PATRI의 살모넬라 게놈 및 관련 정보
- 상업적 및 제도적 살균 방법에 대한 질의응답 2017-06-29 웨이백 머신에서 보관.
- IFAS의 신흥 병원체로서의 살모넬라
- 살모넬라 명명법에 대한 참고 사항
- 살모넬라 운동성 비디오
- 웨이백 머신에 보관된 2011-12-09 조류 살모넬라
- 살모넬라증 개요 - 머크 수의학 매뉴얼


